一种三角帆蚌肌浆网Ca2+-ATP酶基因cDNA的全长克隆与表达分析
张爱菊,刘士力,刘金殿,张根芳,周志明,*
(1.浙江省淡水水产研究所,中国水产科学研究院东海水产研究所浙江研究中心,农业农村部淡水渔业健康养殖重点实验室,浙江省淡水水产遗传育种重点实验室,浙江 湖州 313001;2. 金华职业技术学院,浙江 金华 321000)
钙是细胞内重要的信号分子,参与许多基本细胞过程的调控[1-2]。在软体动物中,Ca2+还是参与其生物矿化过程的重要元素,对珍珠和贝壳的形成及生长非常关键。胞内钙稳态的维持是这些调控或作用正常实现的基础。在静息细胞中,细胞内、外的Ca2+含量均是相对稳定的[3]。环境Ca2+浓度是影响育珠蚌钙代谢的重要原因之一。而软体动物钙代谢,对于阐明贝壳以及珍珠形成机理和提高优质珠的产量有着重要意义。直接从环境中吸收钙是Ca2+进入淡水软体动物体内的一种重要途径[4]。因此,溶质钙外排机制对胞质钙返回静止状态、维持胞内钙稳态很重要。同时,质膜上和内质网(或肌浆网)上的Ca2+泵将细胞溶质Ca2+排出胞外或进入胞内钙库也是维持细胞调控的重要基础[5-6]。
细胞内Ca2+浓度变化主要由肌浆网(SR)对Ca2+的释放和摄入来实现[6]。SR是细胞内重要的钙贮存器,通过调节细胞内Ca2+浓度而发挥重要的生理功能。Sarco/endoplasmic reticulum calcium ATPase(SERCA)是肌浆网膜上的Ca2+泵,靠水解ATP将细胞溶质Ca2+逆浓度梯度泵入肌浆网而将细胞基质中的Ca2+泵入肌质网中储存起来[7]。迄今为止,有关软体动物SERCA基因的研究主要集中在合浦珠母贝(Pinctadafucata)、美洲牡蛎(Crassostreavirginica)等海水贝类[8],而在淡水贝类中鲜有报道。本研究通过RACE技术,成功克隆得到一种淡水贝类——三角帆蚌(Hyriopsiscumingii)SERCA基因的全长cDNA序列,并分析其核苷酸和氨基酸序列,同时采用实时荧光定量PCR(qRT-PCR)技术检测其在不同组织、不同外界Ca2+浓度刺激和同种Ca2+浓度刺激下不同时间的表达情况,以期为进一步深入研究SERCA基因的功能及其调控机理奠定基础。
1 材料与方法
1.1 实验动物
实验用1龄三角帆蚌采自浙江金华威旺养殖新技术有限公司试验基地,平均壳长65 mm,运回实验室在PVC塑料箱中暂养1周后随机选取3只,取其鳃、外套膜、性腺、肝胰腺、斧足等组织,等量相同组织混合在一起经液氮研磨后分装保存于-80 ℃超低温冰箱备用。暂养期间,采用池塘水(绿藻为主)连续充气养殖,保持水温(25±1) ℃,pH (7.6±0.1),每2 d换水1/3。
1.2 总RNA的提取及cDNA首链的合成
取三角帆蚌各组织样品按照TRIzol Reagent(Invitrogen公司)一步法提取总RNA,用核酸蛋白测定仪检测总RNA的浓度和质量,同时用琼脂糖凝胶电泳检测其完整性。之后,取各组织总RNA 500 ng按照RevertAid First Strand cDNA Synthesis Kit(Thermo公司)说明书,进行反转录合成cDNA,保存于-80 ℃备用。
1.3 全长cDNA的克隆
根据三角帆蚌转录组高通量测序得到的SERCA部分cDNA序列,设计引物RC126-1F和RC126-1R(表1)。25 μL PCR反应体系:2×GC BufferⅠ12.5 μL,dNTP Mixture(2.5 mmol·L-1)4 μL,模板cDNA 1 μL,上、下游引物(10 μmol·L-1)各0.5 μL,Taq酶(5 U·μL-1)0.2 μL,ddH2O补足至25 μL。PCR反应程序:95 ℃预变性3 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 1 min,33个循环;72 ℃延伸7 min;4 ℃保存。PCR扩增产物经1.5%的琼脂糖凝胶电泳分离检测后,用PCR产物纯化回收试剂盒(生工SK1131)回收、纯化。按照克隆试剂盒说明(TaKaRa公司)连接入pMD18-T载体,然后转化到感受态细胞(SK2301),经LB平板培养,进行蓝白斑筛选,挑取阳性克隆送至生工生物工程(上海)股份有限公司测序。测序得到600 bp左右的扩增产物,符合预期大小,并经BLAST分析初步确定为SERCA基因的部分序列。
根据已获得的中间片段,分别设计5’和3’端RACE所需的引物,所有引物均由生工生物工程(上海)股份有限公司合成(表1)。按照Clontech Smart cDNA Amplification kit操作说明书,分别合成5’-RACE Ready-cDNA和3’-RACE-Ready-cDNA作为模板,各进行二轮PCR扩增。第一轮PCR反应体系:2×GC BufferⅠ12.5 μL,dNTP Mixture(2.5 mmol·L-1)4 μL,5’-RACE Ready-cDNA或3’-RACE-Ready-cDNA 1 μL,上、下游引物(10 μmol·L-1)各0.5 μL,Taq酶(5 U·μL-1)0.2 μL,ddH2O补足至25 μL。第二轮PCR反应体系:2×GC BufferⅠ25 μL,dNTP Mixture(2.5 mmol·L-1)8 μL,第一轮或第二轮PCR稀释产物1 μL,上、下游引物各1 μL,Taq酶(5 U·μL-1)0.5 μL,ddH2O补足至50 μL。PCR反应程序、PCR扩增产物的纯化、克隆、测序与中间片段所述步骤相同。
1.4 序列分析
测序结果通过DNAStar软件中的SeqMan程序先进行载体序列的去除和拼接,获得SERCA基因全长cDNA序列,与GenBank核酸数据库及蛋白数据库作BLAST(http: //www.ncbi.nlm.nih.gov/BLAST)分析。同时将cDNA序列翻译成氨基酸序列,应用ORF Finder程序(http: //www.ncbi.nlm.nih.gov/projects/gorf/)确定正确的开放阅读框(open reading frame,ORF)。用ExPASy在线Protparam 程序(http: //www.expasy.org /tools/protparam.html)预测氨基酸序列的物理参数,HMMTOP(Tusndy and Simon 2001)和TMHMM (http://www.cbs.dtu.dk/services/TMHMM)分析跨膜结构,SignalP 3.0 server程序(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽。SMART(http://smart.embl-heidelberg.de/)预测结构域。用DNAMAN 8软件对相应的SERCA氨基酸序列进行多重序列比对分析,采用邻位相接法(neighbor-joining,NJ)构建三角帆蚌SERCA基因氨基酸序列与其他物种的NJ系统进化树,并用Bootstrap重复1 000次计算各分支的置信度[9]。
1.5 组织差异表达检测
根据已获得的全长cDNA序列,设计定量PCR引物SERCA-RTF和SERCA-RTR(表1)。采用qRT-PCR技术检测三角帆蚌SERCA基因的组织差异,以5种组织(性腺、肝胰脏、斧足、外套膜和鳃)合成的首链cDNA为模板,按照FastStart Universal SYBR Green Master (Rox)说明(Roche公司),进行qRT-PCR检测[10]。PCR反应体系:2×qPCR Mix 12.5 μL,上、下游引物(7.5 μmol·L-1)各1.0 μL,cDNA模板2.5 μL,补足ddH2O至25 μL。程序设置:95 ℃10 min进入循环;95 ℃15 s,60 ℃60 s,72 ℃30 s,共40个循环;72 ℃延伸8 min。
表1三角帆蚌SERCA基因克隆和表达分析的引物
Table1Primers designed for cloning and expression analysis of aSERCAgene forH.cumingii
1.6 环境Ca2+刺激试验
取15只暂养一周后的三角帆蚌随机分成5组,分别放入不同Ca2+浓度的20 L PVC塑料箱中,每组3个重复,试验组在曝气后的自来水中加入无水氯化钙,使水体中的Ca2+浓度分别为40、60、80和100 mg·L-1,对照组不添加氯化钙。在饲养7 d后,分别取三角帆蚌外套膜保存于-80 ℃超低温冰箱备用。
依据上述试验结果,另取暂养的三角帆蚌15只,放入Ca2+浓度为60 mg·L-1的塑料箱中,并分别于0、24、48、96和168 h取外套膜保存于-80 ℃超低温冰箱备用,每次3个重复。采用qRT-PCR技术检测所取外套膜组织中的SERCA基因表达情况。
1.7 数据分析
进行qRT-PCR检测时,以NCBI上公布的β-actin(NCBI序列号:HM045420)为内参,用于校正,内参引物见表1。数据绘图采用Excel 2013软件。数据基本统计分析、多因素方差分析、相关分析采用SPSS 16.0软件。qRT-PCR结果用2-ΔΔCT法进行数据处理[11]。
2 结果与分析
2.1 三角帆蚌SERCA基因cDNA全长序列
获得一种三角帆蚌SERCA基因全长cDNA序列,该基因全长3 326 bp(GenBank 登录号:KP313822),包括201-bp 5’-UTR区域、3 060-bp编码框(ORF)和65-bp 3’-UTR。有1个终止密码子TGA和一个多聚A尾巴(polyA),但没有多聚腺苷酸加尾信号位点(AATAAA)。
2.2 三角帆蚌SERCA氨基酸序列特征
ORF共编码1 019个氨基酸。通过预测,蛋白质相对分子质量为112.81 ku,分子式:C5052H8029N1325O1484S54,理论等电点(isoelectric point,pI)为5.42。其中,亮氨酸(Leu)含量最高,为8.4%,其次为缬氨酸(Val),含量为8.1%,色氨酸(Trp)含量最少,为1.4%。带负电荷氨基酸残基(Asp+Glu)122个,带正电荷氨基酸残基(Arg + Lys)101个。脂肪族氨基酸指数为96.54。氨基酸预测无信号肽。与其他典型SERCAs不同的是,该三角帆蚌SERCA有9个跨膜区域(图1)。
SMART分析显示,三角帆蚌SERCA氨基酸序列呈现出典型的Ca2+-ATP酶特征,由Cation-transporting P-type ATPase, N-terminal (Cation_ATPase_N)、E1-E2 ATPase-associated region(E1-E2_ATPase)、Haloacid dehalogenase-like hydrolase (Hydrolase)、Cation-transporting P-type ATPase, C-terminal (Cation_ATPase_C)四个结构域组成,分别包含77个(残基1-77)、249个(残基93-341)、380个(残基345-724)、173个(残基819-991)氨基酸残基。这些结构域中含有SERCAs的常见结构,包括磷酸化区域(phosphorylation region),异硫氰酸荧光素位点(fluorescein isothiocyanate site),FSBA结合位点(5′-(p-fluorosulfonyl) benzoyladenosinebinding site),受磷蛋白结合区(phospholamban-binding motif)以及毒胡萝卜素位点(thapsigargin sites)(图1)。
2.3 SERCA氨基酸序列同源性分析和进化树构建
经NCBI中Protein Blast 比对,与合浦珠母贝(P.fucata)SERCA异构体C和A的相似性最高,均为88%,与鳞砗磲(Tridacnasquamosa)、美洲牡蛎(C.virginica)、大瓶螺(Pomaceacanaliculata)、加洲海兔(Aplysiacalifornica)、虾夷扇贝(Patinopectenyessoensis)、海扇贝(Placopectenmagellanicus)等海洋软体动物的相似性也很高,依次为87%、87%、86%、85%和85%。此外,同其他一些物种SERCA氨基酸序列也存在不同程度的相似性(表2),说明SERCA序列高度保守。
用DNAMAN 8软件将三角帆蚌与其他17种生物的SERCA基因氨基酸序列进行多重比对,并以邻位相接法(NJ法)构建了氨基酸的进化树(图2)。结果表明,系统进化树可分为明显的两大支,其中,红螯螯虾(Cheraxquadricarinatus)占据一个分支,其它SERCA构成另一大分支,三角帆蚌首先与鳞砗磲聚在一起,然后与其他海洋软体动物,如合浦珠母贝、美洲牡蛎等紧密聚在一起,且与粉正蚓(Lumbricusrubellus)等环节动物亲缘关系较近,聚为一支,其系统发育关系基本符合传统的分类地位。


相同残基用“*”表示,半保守位置用“﹒”表示;三角帆蚌的跨膜螺旋M1-M9用阴影显示;磷酸化位点、FITC和FSBA结合位点用下划线标出;2个典型的胰蛋白酶位点R188和R505用粗体和下划线标出;典型的受磷蛋白结合区用下划线加斜体标出;预测的毒胡萝卜素位点加粗显示。缩写及登录号见表2。Identical residues were shown with an asterisk (*), and semi-conservative substitutions were labeled with a period (﹒). Transmembrane segments M1-M9 from H.cumingii were shaded in grey. The phosphorylation site, fluorescein isothiocyanate site (FITC) and the 5'-(p-fluorosulfonyl) benzoyladenosine binding site (FSBA) were underlined and labeled. Two typical tryptic sites R188 and R505 existing in sarco/endoplasmic reticulum Ca2+-ATPase (SERCA)1 and SERCA2 were marked in bold and underlined. Typical phospholamban-binding motif of SERCA2 was underlined italicized. Putative thapsigargin sites were indicated in bold. The abbreviation name and accession No. were shown as that in Table 2.图1 预测三角帆蚌SERCA氨基酸序列与合浦珠母贝的序列比对Fig.1 Amino acid sequence alignment of SERCAs from H. cumingii and P. fucata
表2三角帆蚌和其他物种SERCA氨基酸序列的相似性
Table2 Similarity of SERCA amino acid sequence amongH.cumingiiand other species
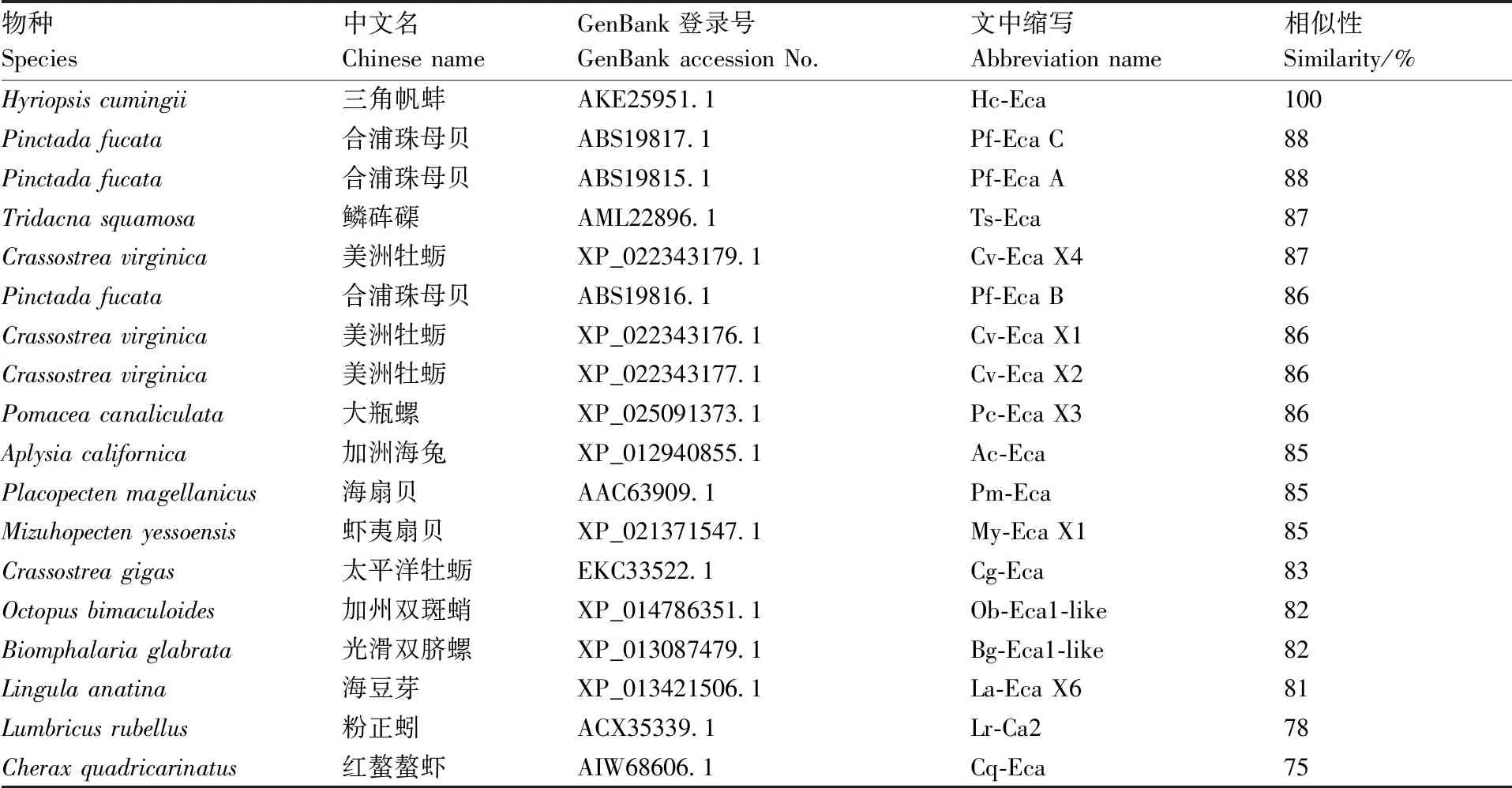
物种Species中文名Chinese nameGenBank登录号GenBank accession No.文中缩写Abbreviation name 相似性Similarity/%Hyriopsis cumingii三角帆蚌AKE25951.1Hc-Eca100Pinctada fucata合浦珠母贝ABS19817.1 Pf-Eca C88Pinctada fucata合浦珠母贝ABS19815.1 Pf-Eca A88Tridacna squamosa鳞砗磲AML22896.1 Ts-Eca87Crassostrea virginica美洲牡蛎XP_022343179.1 Cv-Eca X487Pinctada fucata合浦珠母贝ABS19816.1 Pf-Eca B86Crassostrea virginica 美洲牡蛎XP_022343176.1 Cv-Eca X186Crassostrea virginica美洲牡蛎XP_022343177.1 Cv-Eca X286Pomacea canaliculata大瓶螺XP_025091373.1 Pc-Eca X386Aplysia californica加洲海兔XP_012940855.1 Ac-Eca85Placopecten magellanicus 海扇贝AAC63909.1 Pm-Eca85Mizuhopecten yessoensis 虾夷扇贝XP_021371547.1 My-Eca X185Crassostrea gigas太平洋牡蛎EKC33522.1 Cg-Eca83Octopus bimaculoides加州双斑蛸XP_014786351.1 Ob-Eca1-like82Biomphalaria glabrata光滑双脐螺XP_013087479.1 Bg-Eca1-like82Lingula anatina 海豆芽XP_013421506.1 La-Eca X681Lumbricus rubellus 粉正蚓ACX35339.1 Lr-Ca278Cherax quadricarinatus 红螯螯虾AIW68606.1Cq-Eca75
2.4 三角帆蚌SERCA基因的表达特征
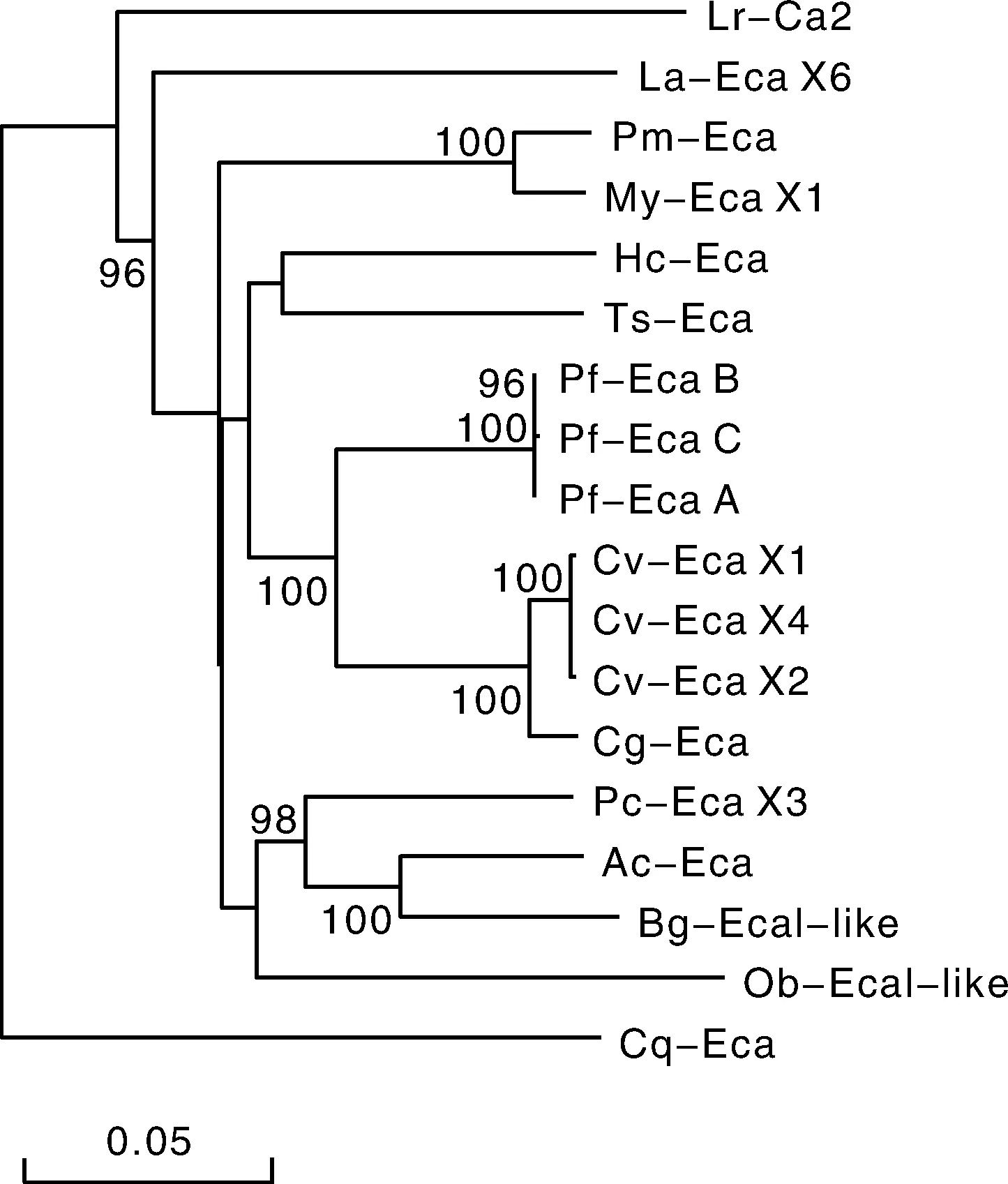
图2 根据SERCA氨基酸序列构建的NJ系统进化树Fig.2 NJ phylogenetic tree of SERCA amino acid sequence from H. cumingii and other species
2.4.1 组织表达分析
利用qRT-PCR技术检测了三角帆蚌SERCA基因在不同组织的表达差异性,结果表明三角帆蚌SERCA表达水平存在明显的组织差异,在鳃表达最高,其次为外套膜、肝胰腺,在性腺和斧足中的表达量较低(图3)。
2.4.2 环境Ca2+刺激下的SERCAmRNA表达情况
不同Ca2+浓度处理试验结果表明,随水体中Ca2+浓度逐渐升高,三角帆蚌SERCA基因在外套膜中的表达水平呈先下降后上升趋势,并在Ca2+浓度为60 mg·L-1时达到最低值,Ca2+浓度为80 mg·L-1时达到最高值,且显著高于其他试验组(图4-A)。同时在60 mg·L-1Ca2+浓度条件下,对三角帆蚌外套膜进行不同时间的表达试验,结果表明,SERCA基因的表达量随时间推移先上升,于48 h时达到最高,而后逐渐下降,但在168 h时略有上升(图4-B)。
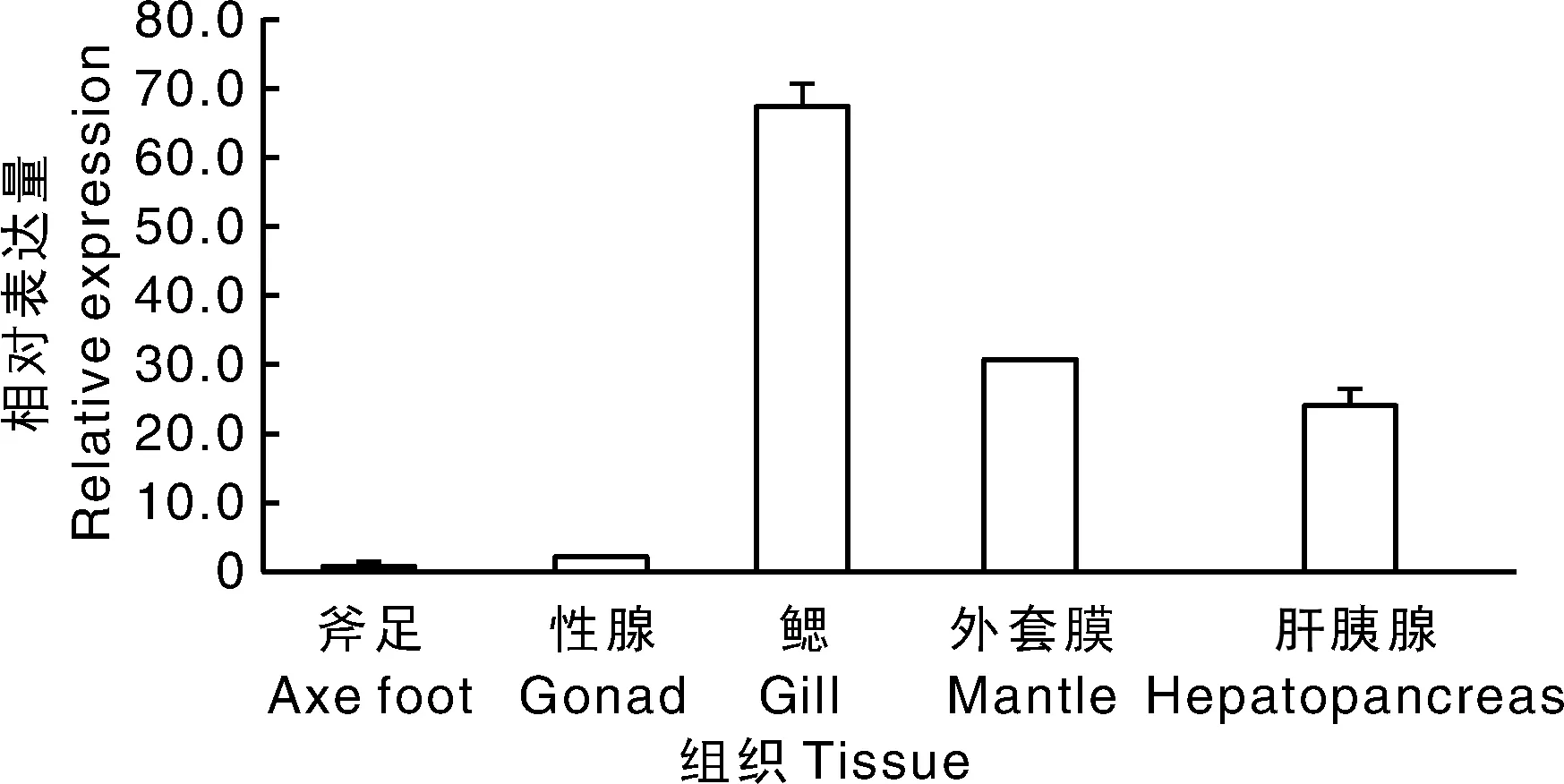
图3 三角帆蚌SERCA基因在不同组织中的相对表达量Fig.3 Relative expressions of SERCA gene in different tissues from H. cumingii
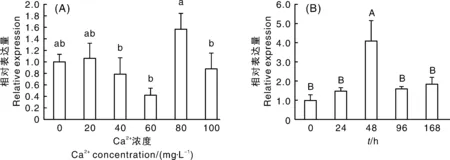
A,不同Ca2+浓度下外套膜中SERCA的相对表达量;B,60 mg·L-1 Ca2+浓度条件下外套膜中SERCA在不同时间的相对表达量。柱状图上同个系列无相同小写字母的表示各Ca2+浓度组间差异显著(P<0.05);无相同大写字母的表示时间差异极显著(P<0.01)。A, The relative expression of SERCA in the mantle under different Ca2+ concentration treatments; B, The temporal expression profile of SERCA under Ca2+ concentration of 60 mg·L-1. The different lowercase letters and capital letters meant significant difference at P<0.05 and P<0.01 respectively.图4 三角帆蚌SERCA基因的相对表达水平Fig.4 Relative expression level of SERCA detected by real-time PCR from H. cumingii
3 讨论
3.1 结构特征
SERCAs属于P型ATP酶家族,靠水解ATP将细胞溶质Ca2+逆浓度梯度泵入肌浆网而将细胞基质中的Ca2+泵入肌质网中储存起来[12]。因此,Ca2+转运功能的变化不仅会直接影响肌肉收缩功能,而且会造成细胞浆中游离Ca2+浓度增加[13]。一般来说,SERCAs由一条单一的多肽链组成,形成3个球形的胞浆区域,由螺旋杆部连接到10个跨膜螺旋(M1-M10)。然而,Fan等[8]研究表明,合浦珠母贝的一个SERCA异构体,即PSERB可能拥有11个跨膜区域。与这些SERCAs不同的是,本研究克隆的三角帆蚌SERCA基因氨基酸序列预测有9个跨膜螺旋。
研究认为,哺乳动物和鸟类的SERCA由3个同源基因(SERCA1、SERCA2和SERCA3)编码,每个基因有多种异构体[14-18],而一些无脊椎动物中SERCAs的异构体仅由一个基因编码[19-21]。同时,有学者认为,脊椎动物中的3个SERCA编码基因和无脊椎动物中的唯一一个SERCA编码基因由一个共同的SERCA祖基因衍生而来[21]。Fan等[8]的研究表明,合浦珠母贝的SERCA有3个异构体,它们因拥有不同的C末端而被区分[22]。本研究仅克隆了一种三角帆蚌SERCA基因,在氨基酸结构水平上的拼接机制仍需进一步研究。
3.2 组织特异性表达
研究表明,淡水和海水软体动物都积极地从环境中吸收钙[4],但钙代谢机制存在明显差异。海水软体动物外套膜和消化腺中几乎没有钙的存在,不在组织中贮留钙[23]。而淡水软体动物吸收的Ca2+有一部分以钙球体的形式贮藏起来备用[24]。在淡水软体动物中,斧足是主要运动器官;性腺是生殖系统的重要组成部分;肝胰腺与软体动物消化、免疫等功能密切相关;外套膜为分泌钙到矿化位点的重要组织[25];鳃是从环境中吸收钙的主要组织[26]。其中,外套膜作为珍珠形成的部位,对Ca2+具有高度通透性[27-28],其内、外表皮具有主动吸收Ca2+和储存Ca2+的功能,且Ca2+利用率高[29];鳃作为三角帆蚌的呼吸和滤食器官,对Ca2+具有较强的亲和力和较高的代谢率,是开展钙代谢过程的重要组织之一。研究表明,相比外套膜,鳃能吸收更多的Ca2+,但其利用率没有前者高[30]。由此可见,外套膜和鳃的钙代谢活动旺盛,钙稳态的维持对它们非常重要。研究表明,PSERB可在多种组织中表达,是一个潜在的“看家基因”,其分布模式与哺乳动物的SERCA2b相似;PSERA可在闭壳肌和斧足中表达,且在闭壳肌中有很高的表达量,可能为肌肉特异性异构体,有助于放松收缩的肌肉[25,31-32];PSERC可在除闭壳肌和斧足以外的组织中表达,且在鳃和外套膜中有最高的表达水平,这与非肌肉特异性异构体非常相似。在软体动物中,PSERC在鳃和外套膜中的高度表达可能体现了它在软体动物生物矿化过程中对钙稳态的重要性[8]。本研究克隆的三角帆蚌SERCA基因在鳃中表达最高,其次为外套膜、肝胰腺,在性腺和斧足中的表达量较低,说明在外套膜、鳃等钙代谢活动旺盛的组织中表达量很高,推测该基因在Ca2+吸收、转运与储存方面有着重要作用,参与珍珠形成等生物矿化过程。然而,该基因在三角帆蚌外套膜和鳃中的定位仍需进一步研究。此外,该基因在肝胰腺中的表达量也较高,推测肝胰腺也具备储存Ca2+的功能。性腺和斧足是三角帆蚌的繁殖、运动器官,其中较低的mRNA表达量,可能说明这两个组织储存Ca2+的能力较弱。今后将继续研究该三角帆蚌SERCA基因的异构体种类,进一步了解SERCA基因在维持钙稳态中的具体功能,掌握钙吸收、储藏、转运机制。
3.3 环境Ca2+刺激
环境钙浓度是影响育珠蚌钙代谢的重要原因之一。郝莹莹[24]研究结果表明,添加Ca2+时,细胞外Ca2+流动方向是从外排逐步变为内流,并随浓度增大,Ca2+流动速度增大,细胞内部的荧光信号增强。在调控途径上,有关钙代谢的调控因子可能通过钙的吸收、转运、贮藏及沉积从而参与和调控贝壳与珍珠的形成。在河蚌育珠过程中,Ca2+的吸收和转运对于珍珠的产量提高起着重要作用,加快Ca2+在珍珠囊中的沉积,是促进珍珠快速生长的重要手段。在天然水体中,河蚌生存的环境中钙含量很低,主要是靠外套膜、鳃和斧足等与周围水环境相接触的组织直接从环境中吸收钙,周围水环境中Ca2+推测是以复合体形式进入细胞内的,因为以游离的离子形态难以直接穿过外套膜表皮细胞的质膜,且要耗费一定的能量,所以部分Ca2+在膜的界面上与离子搬运体结合形成复合体进入质膜[4]。
珍珠的形成是个复杂精细的生物矿化过程,水体中保持适宜的Ca2+浓度,是珍珠生长的必备条件。李文娟等[33]研究认为,适宜的Ca2+浓度能促进三角帆蚌外套膜对Ca2+的吸收、转运与储存,而过高的Ca2+浓度则会抑制三角帆蚌外套膜的钙代谢。舒妙安等[34]研究认为,水体中适宜的Ca2+浓度(40~60 mg·L-1)能促进三角帆蚌的钙代谢过程,加快珍珠形成。本研究克隆的三角帆蚌SERCA基因在外套膜中的表达水平呈先下降后上升趋势,并在Ca2+浓度为60 mg·L-1时达到最低值,Ca2+浓度为80 mg·L-1时达到最高值,推测60 mg·L-1Ca2+浓度时外套膜积极地分泌钙到生物矿化位点参与珍珠生长,导致钙储存功能被弱化。当环境Ca2+浓度为80 mg·L-1时,外套膜的分泌功能相对弱化,而储存功能增强。至于在三角帆蚌不同生长发育阶段或外套膜不同位置的表达情况是否一致,还有待进一步研究。此外,本研究还发现三角帆蚌在适宜Ca2+浓度(60 mg·L-1)条件下,其外套膜SERCA基因的表达量随时间推移先上升,于48 h时达到最高,而后逐渐下降,由此推测48 h转运、储存Ca2+能力很强,随后降低。

