西南地区PRRSV分子流行病学调查及其类NADC30株基因组特征分析
周 群,周可磊,胡承哲,李 玉,任玉鹏,2,岳 华,2,张 斌,2*
(1.西南民族大学 生命科学与技术学院,四川 成都610041;2.国家民族事务委员会青藏高原动物疫病防控创新团队,四川成都610041)
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是由PRRS病毒(PRRSV)引起的一种猪的高度接触性传染病[1]。PRRSV基因组大小约15 kb,至少包含10个开放阅读框(ORF),ORF1a和ORF1b基因编码14个非结构蛋白(Nonstructural protein,NSP)包括NSP1α、NSP1β、NSP2~NSP6、NSP7α、NSP7β、NSP8~ORF12,参与病毒基因组的转录和复制[2];ORF2~ORF7编码病毒的结构蛋白,依次编码囊膜蛋白GP2~GP5、膜基质蛋白M和核衣壳蛋白N[3]。其中,NSP2和GP5被认为是最易变异的两个蛋白。同时,GP5蛋白也是PRRSV诱导机体产生中和抗体的主要抗原[4]。根据基因组序列及抗原特性的差异,PRRSV被分为两大基因型,即以LV株为代表的欧洲型(基因1型)和以VR2332株为代表的北美洲型(基因2型)[5]。
2013年,我国出现了在NSP2蛋白区域存在不连续的131个氨基酸缺失的类NADC30病毒株[1]。目前类NADC30病毒株已在我国大部分省份流行[6]。研究表明,2016年~2017年我国西南地区PRRSV类NADC30病毒株的流行呈上升趋势[1,7],但具体的流行情况和分子特征仍不明确。本研究主要通过采集2018年中国西南地区疑似PRRS患病保育猪的肺脏或血清等临床样本,利用RT-PCR方法对检测到的PRRSV进行分子流行病学调查,并对中国西南地区类NADC30病毒株的基因组特征和遗传进化规律进行研究,为更好地防控PRRS提供参考依据。
1 材料与方法
1.1 病料样品与主要试剂 2018年采集自我国西南地区包括四川(23/41)、重庆(6/41)、贵州(9/41)、云南(3/41)4省41个猪场共292份疑似PRRS患病保育猪的肺脏(48份)和血清(244份)样本,置于-80℃冰箱保存备用。TRIzol试剂(RNAiso Plus)、Prime-ScriptTMRT试剂盒、pMD19-T载体、DL2000 DNA Marker购自TaKaRa公司;QuickTaqHS DyeMix购自东洋纺(上海)生物科技有限公司;PCR产物纯化试剂盒和胶回收试剂盒购自OMEGA公司;E.coli DH5α感受态细胞购自天根生化科技(北京)有限公司。
1.2 引物设计与合成 参照文献[3,8]中的引物序列分别合成扩增PRRSV ORF7基因的检测引物和扩增NSP2基因的PRRSV分型引物,应用PrimerPremier5.0软件根据类NADC30病毒株SD53-1603(MH651744.1)全基因组序列分段设计引物(表1),用于该病毒全基因的扩增,引物均由上海生工生物工程技术服务有限公司合成,并用ddH2O稀释至10 pmol/L备用。
1.3 病料样本、阳性样本PRRSV分型的检测及类NADC30病毒株基因组的扩增 取适量肺脏组织剪碎研磨、离心后,取400 μL上清用于提取核酸,血清样本则直接取400 μL用于提取核酸。用TRIzol试剂提取上述样本中的PRRSV总RNA,反转录为cDNA后于-20℃保存。以292份样本的cDNA为模板,以ORF7 F/R为引物进行PRRSV检测,并利用分型引物NSP2 F/R对检测出的PRRSV进行分型。利用特异性引物(表1)对分别来自成都、宜宾、眉山、绵阳的经NSP2引物鉴定为类NADC30病毒株的样本cDNA进行全基因组PCR的分段扩增。PCR反应条件为94℃2 min;94℃30 s、54℃30 s、68℃1 min,35个循环;72℃8 min,16℃保存。PCR产物由上海生工生物工程技术服务有限公司测序,对测序结果进行序列拼接,并在NCBI中对全基因组序列进行比对分析。
1.4 类NADC30病毒株全基因组特征和遗传进化树分析 应用MegAlign和CLC sequence Viever 8对检测到的7株类NADC30病毒株全基因组和ORF5基因的核苷酸序列和氨基酸序列进行同源性分析,并通过MEGA 7.0软件用邻近法构建类NADC30病毒株的全基因组及ORF5基因系统进化树;应用SimPlot和RDP4软件对7株类NADC30病毒全基因组序列进行重组分析。
2 结果与讨论
2.1 病料样品的检测及PRRSV阳性样本分型的结果 通过对2018年从我国西南地区采集的292份疑似PRRS的临床样品经PRRSV ORF7基因的PCR检测,结果显示共检出PRRSV阳性样品129份(44.18%,95%confidence interval(CI)=38.4%~50.1%),肺脏样品PRRSV检出率为44.68%(95%CI=30.2%~59.9%,21/47),血清样品PRRSV检出率为44.08%(95%CI=37.8%~50.5%,108/245)。猪场PRRSV检出率为70.73%(95%CI=54.5%~83.9%,29/41)。与近年来我国西南地区PRRSV的检出率(22.15%~34.1%)相比明显上升[1,7],表明我国西南地区PRRSV的感染率呈逐年上升趋势。利用NSP2引物对129份阳性样本进行PCR扩增,共有64份阳性样品扩增出了PRRSV NSP2基因(49.6%),通过测序后分析该基因编码蛋白的氨基酸序列可知,有45株在NSP2蛋白中存在不连续的131个氨基酸缺失的类NADC30病毒株(70.3%,95%CI=57.6%~81.1%),8株在NSP2蛋白中存在不连续的30个氨基酸缺失的HP-PRRSV病毒株(12.5%,95%CI=5.6%~23.2%),11株为经典株(17.2%,95%CI=8.9%~28.7%),65份未分型成功。有19个猪场感染的PRRSV被分型,其中14个猪场存在类NADC30病毒株感染(73.7%,95%CI=48.8%~90.9%)。与我国山东和河北等省份一致,类NADC30病毒株已经成为我国西南地区PRRSV的优势流行株[9]。提示应加强对我国西南地区PRRSV类NADC30病毒株分子流行病学的监测和对PRRS的综合防控。
2.2 7株类NADC30病毒株基因组序列分析 对7份类NADC30病毒株样品分段扩增,并在NCBI中对拼接后的基因组序列进行比对分析,结果显示扩增的类NADC30病毒株基因组序列全长为14 915 bp~14 961 bp,GC含量为52.63%~52.79%,均在NSP2蛋白存在131(111aa+1aa+19aa)个氨基酸缺失。根据病毒株的采集时间和地理位置分别命名为SWU/MS2/2018、SWU/MS3/2018、SWU/MY5/2018、SWU/MY6/2018、SWU/YB1/2018、SWU/YB2/2018、SWU/CD1/2018,GenBank登录号依次为MK429980~MK429986。病毒基因的突变和重组,对PRRS的防控造成了阻碍。在中国西南地区关于PRRSV的报道中,类NADC30病毒株与强毒株的重组事件频繁发生[7],但本研究中的7株PRRSV基因组经分析均未与任何病毒株发生重组。7株PRRSV病毒基因组与30株国内外PRRSV病毒同源性为58.7%~96.4%,与参考株中的类NADC30病毒株同源性为85.3%~96.4%,与参考株中的NADC30病毒株(JN654459.1)同源性仅93.4%~93.8%,与山东类NADC30病毒株SD53-1606(MH651745.1)的同源性为95.6%~96.4%。7株类NADC30病毒NSP2基因进化树(图略)、ORF5基因进化树(图略)和全基因组进化树(图1)均与类NADC30病毒株聚为一大支,并且与中国近两年报道的山东病毒株SD53-1606和河北病毒株QHD1(MG687491.1)单独聚为一个分支(图1),表明7株病毒均是由类NADC30病毒株演化而来的新病毒。其基因组与NADC30病毒株(JN654459.1)的低同源性表明,我国西南地区流行的类NADC30病毒株可能出现了新的变异。这提示规模化养殖场应更加注重生物安全,完善引种程序,避免引入新的类NADC30病毒株。
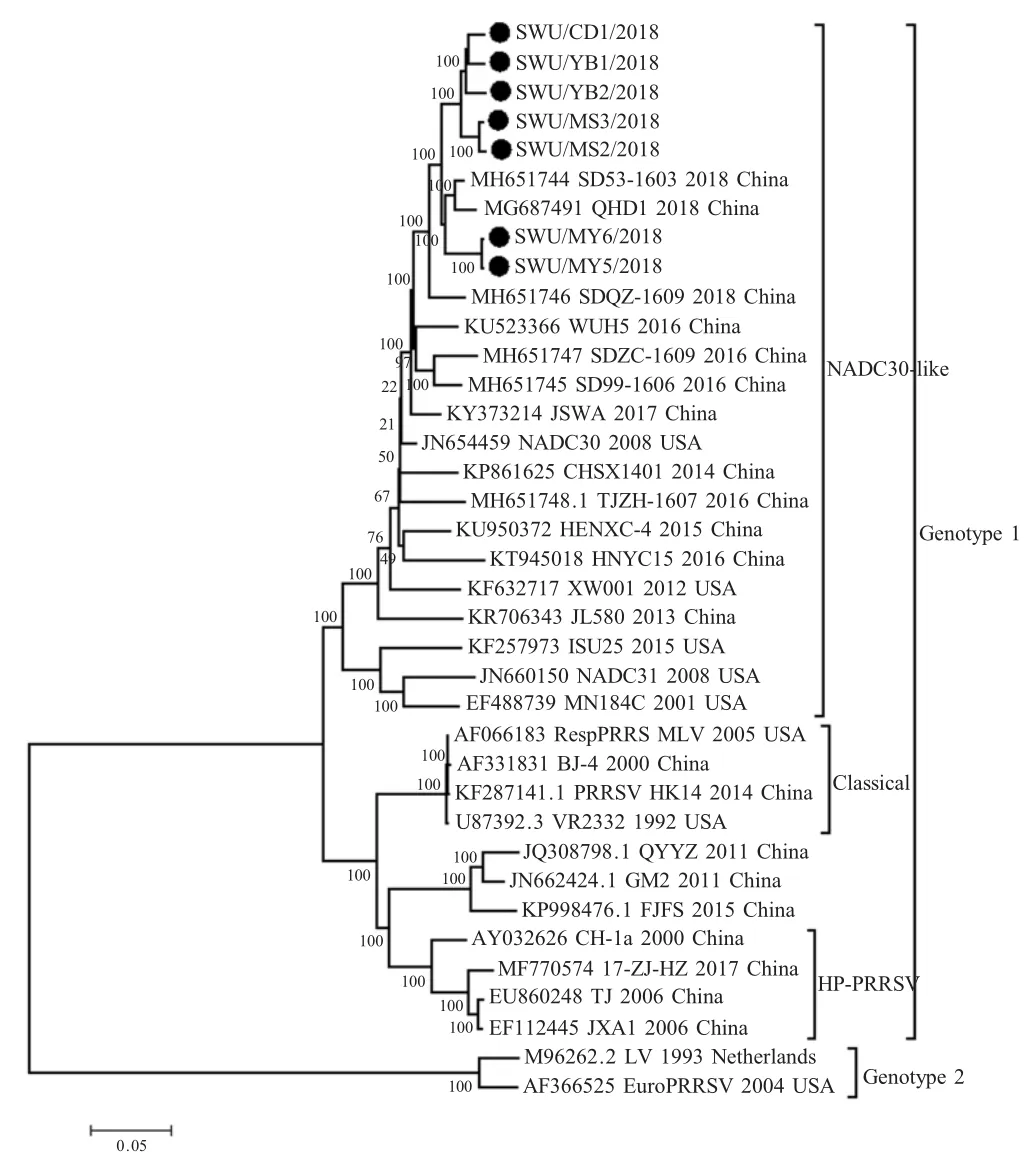
图1 7株PRRSV类NADC30病毒株全基因组遗传演化分析Fig.1 Phylogenetic tree analysis of 7 PRRSV NADC30-like strains
2.3 7株类NADC30病毒株ORF5基因序列分析对7株类NADC30病毒ORF5基因的同源性分析显示:7株病毒ORF5基因与参考株该基因核苷酸序列同源性为61.4%~97.3%,其编码蛋白GP5的氨基酸序列与参考株该蛋白氨基酸序列同源性为53.5%~95.5%。对GP5氨基酸序列分析显示,7株病毒中的部分病毒株GP5氨基酸序列出现了D54G、Y106R、K166R、V169I等突变,但7株病毒株GP5氨基酸序列中均发生了一个氨基酸(S/N33)的缺失。为进一步调查该缺失模式在PRRSV病毒株中的流行情况,将本研究中的7株病毒的ORF5基因序列与已知的395条PRRSV的ORF5基因序列比对分析,结果显示仅有16株类NADC30病毒存在同样的缺失模式,包括山东病毒株(10/16)、黑龙江病毒株(3/16)、广西病毒株(1/16)、河北病毒株(1/16)、四川病毒株(1/16)。ORF5基因的遗传进化树显示22株存在该缺失模式的病毒单独聚为一个分支(图2)。以上结果表明存在该缺失模式的病毒可能是类NADC30病毒株传入我国后遗传演化而来的新型病毒株,最早流行于山东地区,随后传入我国西南地区,目前已经在四川地区广泛流行[9]。关于存在该缺失模式病毒的动物试验中,感染存在该缺失模式病毒的仔猪具有呼吸症状明显,发热程度更高、持续时间更长等临床特征,表明存在该缺失模式的类NADC30病毒株具有较高的致病性[10],但其具体的致病机理还有待进一步研究。
本研究调查了2018年PRRS在中国西南地区的流行情况,数据表明类NADC30病毒株为我国西南地区PRRSV的主要流行株。本研究获得了7株PRRSV类NADC30病毒株全基因组序列,为分析该类病毒株的遗传进化规律和变异趋势、研发新型有效的疫苗奠定了基础。

图2 PRRSV ORF5基因遗传进化树Fig.2 Genetic phylogenetic tree of PRRSV ORF5 gene

