Krüppel-like factor 3(KLF3)基因对牛脂肪沉积的作用
郭红芳,宁越,成功,2,昝林森,2
Krüppel-like factor 3(KLF3)基因对牛脂肪沉积的作用
郭红芳1,宁越1,成功1,2,昝林森1,2
(1西北农林科技大学动物科技学院,陕西杨凌 712100;2国家肉牛改良中心,陕西杨凌 712100)
【目的】通过研究牛KLF3基因对牛脂肪分化和脂肪酸代谢关键基因表达量的作用,进而探讨KLF3 基因对脂质沉积的影响。【方法】合成干扰siRNA,筛选得到KLF3基因干扰效率最高SiRNA,分离培养秦川牛肉用新品系新生牛前脂肪细胞生长至70%-90%汇合度时转染筛选得到的KLF3基因SiRNA和阴性对照SiRNA(NC),并进行诱导分化培养至第0天和第4 天时,利用荧光定量PCR方法研究分化标志基因过氧化物酶体增殖物激活受体γ(peroxisome proliferator activated receptor γ,)、CCAAT增强子结合蛋白(CCAAT/enhancer-binding proteins α,),脂肪酸代谢关键基因脂肪酸合成酶(fatty acid synthase,)、乙酰辅酶A羧化酶(acetyl-CoA carboxylase α,)和脂肪酸结合蛋白4(fatty acid binding protein 4, FABP4)基因在牛脂肪细胞分化不同时期干扰KLF3基因处理组和对照组之间表达量的变化,同时在干扰KLF3基因处理并进行诱导脂肪细胞分化的第 4天采用油红O染色法观察干扰KLF3基因处理组和对照组之间脂滴差异及采用酶联免疫吸附测定(enzyme-linked immunosorbent assay,ELISA)方法测定甘油三酯含量进一步确定KLF3基因对牛脂肪细胞分化和脂肪酸代谢的作用。【结果】 KLF3-Si2 具有最高的干扰效率达到73%。转染KLF3-Si2后的表达量在牛脂肪细胞诱导分化的第0天 和第4 天与对照组相比分别下调了58% 和37%,达到了差异极显著(<0.01),的表达量也分别下调了64% 和41%,达到了差异极显著(<0.01)。脂质代谢的关键基因FABP4的表达量与对照组相比在牛脂肪细胞诱导分化的第0天和第4 天分别下调了89% 和60%,而的表达量干扰KLF3处理组与对照组相比在脂肪细胞诱导分化第0天和第4天分别下调了50% 和37%, 而的表达量则分别下调了73% 和19%,都分别达到了差异极显著(<0.01)。在牛脂肪细胞诱导分化第4天油红O染色和甘油三酯含量测定也发现干扰KLF3基因处理组与对照组相比脂滴减少,甘油三酯含量显著下降(<0.05)。【结论】干扰牛KLF3基因可以抑制牛脂肪细胞分化和脂肪酸代谢关键基因表达量下调,进而影响脂肪的沉积。
秦川牛;前脂肪细胞;;脂肪沉积
0 引言
【研究意义】动物体内肌内脂肪沉积,影响肉的断面大理石评分,可显著提高牛肉的嫩度、多汁性和风味等感官品质,最终影响肉质等级与经济价值[1]。因此,如何提高肌内脂肪含量来满足消费者对牛肉嫩度和风味的需要,一直是牛肉脂肪沉积生物分子调控研究的一个热点[2]。脂肪沉积量主要取决于脂肪细胞增殖和分化程度,这一过程受诸多分化转录因子的协同调控,转录因子的表达量及其活性大小决定了分化过程[3]。因此,深入研究牛脂肪沉积的分子调控机制对于改善牛肉肉质性状具有重要意义。【前人研究进展】KLF家族是一类C端含有3个锌指结构的重要的转录因子,能特异性结合于靶基因启动子区发挥其调控功能,在细胞分化、细胞增殖和凋亡方面起着重要的作用[4-7]。目前哺乳动物中研究发现KLF家族成员有17个成员,大多数成员参与脂肪细胞形成。有研究表明、和在脂肪细胞分化中主要起抑制作用[7-9],、、、、、和则主要起促进作用[10-16]。KLF3基因是KLF家族中一类重要的转录因子,最早从红细胞系中作为的同源系克隆得到[17],最初体外研究发现其在3T3-L1脂肪细胞系中募集转录辅助阻遏物CtBP结合于的启动子区发挥抑制作用[18-19]。但在体内有大量研究表明KLF3基因敲除小鼠与同窝小鼠相比明显瘦小、脂肪垫减少,进一步研究发现主要是由于白色脂肪减少而造成的[19]。另外有研究发现干扰可增加动物肠道脂肪沉积[20],而且在鸡脂肪细胞分化过程中发现虽然KLF3基因抑制脂肪细胞形成的一些关键基因的启动子活性,但促进脂肪细胞分化标志基因的启动子转录活性[21]。另外有研究发现KLF3基因在调节脂质聚集和分泌中起着重要作用。它可以通过促进脂肪酸β-氧化调节脂质代谢,另外KLF3 基因突变可导致线虫中类似哺乳类的线粒体甘油三酯转移蛋白和载脂蛋白B的基因分别减少[22]。【本研究切入点】这些研究结果表明KLF3基因在脂肪沉积过程中发挥重要的作用。目前还没有关于KLF3基因对牛脂肪细胞分化和脂质代谢的作用研究。【拟解决的关键问题】因此,本研究以牛前脂肪细胞为研究对象,通过干扰KLF3基因的表达,确定KLF3基因在牛脂肪细胞分化和脂肪酸代谢中的作用,为进一步明确KLF3基因在调控牛脂肪沉积中的作用,改善牛肉品质提供一定的理论依据。
1 材料与方法
本研究于2016年9月至2017年9月在西北农林科技大学国家肉牛改良中心实验室完成。
1.1 试验动物和所需组织
秦川牛肉用新品系新生犊牛(3日龄)3头的9个组织(心、肝、脾、肺、肾、大肠、瘤胃、肾周脂、肌肉)。
试验动物和所需组织均采自西北农林科技大学国家肉牛改良中心良繁场,分别采集3头牛的9个组织共计27个样本后立即置于液氮罐中迅速带回实验室,-80℃保存备用。
1.2 牛前脂肪细胞获取
选取临床检查无异常的秦川牛肉用新品系新生牛(3日龄),参照文献[23]试验方法从新生牛肾周脂肪组织分离得到牛前脂肪细胞。将得到的牛前脂肪细胞沉淀加入适量的含10% FBS的完全培养基重悬、吹匀,置37℃、5% CO2培养箱中培养,每2 d 换液1次,一般4—5 d长满,传代于6孔板中培养。当传代培养的牛前脂肪细胞生长至充分汇合时,用诱导分化培养基(含10% FBS培养基+5 μg·mL-1胰岛素+1 μg·mL-1地塞米松+0.5 mmol·L-13-异丁基-1甲基黄嘌呤)进行诱导分化培养2 d后,再换成脂肪细胞分化维持培养基(10% FBS+1 μg·mL-1胰岛素)进行培养2 d。
1.3 SiRNA转染
牛KLF3基因的干扰RNA(SiKLF3)和对照SiRNA(NC)由上海吉玛公司合成,序列如表1所示。当牛前脂肪细胞生长至70%—90%汇合时,先用无血清培养基饥饿细胞2 h,然后分别将5 μL干扰SiRNA 和对照组SiRNA溶解于125 μL OPTI培养基中,另外将3.75 μL lipofectin3000加入125 μL OPTI后和稀释的SiRNA混匀静置5 min后逐滴加入6孔板中。转染6 h后换成含有10% FBS的完全培养基。转染2 d后细胞生长至充分汇合时,进行诱导分化培养至第4天。
1.4 RNA提取和qRT-PCR
按照TRIZOL试剂说明书分别提取新生牛9种组织RNA和诱导分化第0天和第4天的秦川牛脂肪细胞的总RNA并分别反转录为cDNA。根据NCBI数据库提供牛、、、、和基因 mRNA 序列,利用Primer premier 5.0设计基因表达分析引物,以牛GAPDH基因为内参(NM_001034034)采用qRT-PCR方法检测各组织和基因的表达,引物见表1。20μL反应体系:2×SYBR Premix EX TaqⅡ 10 μL, 上、下游引物各0.8 μL,ROX Reference DyeⅡ 0.4 μL, 稀释至50 ng·μL-1的cDNA 2 μL,RNase-freedd-H2O 补至20 μL。反应程序为:94℃ 30 s 预变性后 95℃ 5 s, 60℃ 34 s,40个循环。每个样本3个生物学重复。用2-ΔΔCt法计算各组织和细胞中基因的相对表达量。
1.5 油红O染色和甘油三酯含量测定
将诱导分化培养至第4天的脂肪细胞用PBS洗2—3次后用4%多聚甲醛固定培养板贴壁细胞30 min后, PBS漂洗。吸取油红O 染液1 mL 浸染培养板, 30 min 后倒掉油红O 染液, PBS漂洗培养板2—3次, 至完全漂洗干净。置于倒置相差显微镜下观察, 并照相, 采集油红O染色图片。

表1 试验中所用引物
甘油三酯含量利用牛甘油三酯测定ELISA试剂盒按说明书步骤进行,经过样品和标准品准备,分别加样于96孔板,37℃反应30 min,洗板5次后加入酶标试剂,37℃ 30 min,显色10 min后加入终止液,在490 nm波长测OD值并计算。分别测定诱导分化第4 天,干扰和对照组两组之间甘油三酯的含量。每个样本设3个重复。
1.6 Western blot
将转染Si-KLF3后2 d的牛前脂肪细胞弃去培养基,室温下用PBS洗细胞3次,然后向6孔板中加入细胞裂解液,冰上裂解30 min,将细胞裂解液转至1.5 mL 离心管中12 000 r/min 4℃离心10 min后,将上清(细胞总裂解物)转至新的离心管,与蛋白上样buffer混合,在100℃煮沸10 min使蛋白质变性。每个样品取10—20 μL进行常规SDS-PAGE电泳。电泳结束后,采用BIO-RAD的MiNi Trans-Blot将样品转移至PVDF膜,将膜置于封闭液(5%脱脂奶粉的PBST),室温封闭2 h,或4℃过夜。洗去膜上的封闭液,将膜孵育在含羊抗兔的KLF3多克隆抗体(ab154531,Abcam,Cambridge,UK)或GAPDH兔单克隆抗体(ab181603,Abcam,Cambridge,UK)的PBST溶液,置于摇床上,室温反应2 h或4℃过夜;洗膜后将膜孵育在含羊抗兔IGg二抗(ab6721,Abcam,Cambridge,UK)的PBST溶液;再次洗膜后常规ECL显色。
1.7 KLF3基因生物学特征分析
应用NCBI数据库中的Blastp(http://blast. ncbi. nlm.nih.gov/Blast.cgi)分析基因的同源性;应用MEAG7.0软件构建系统进化发育树。
1.8 数据分析
数据采用Student-T和ANVOA 方法进行分析,数据表示为“平均数±标准差(Mean±SD)”以<0.05表示差异显著,<0.01表示差异极显著。
2 结果
2.1 牛KLF3基因的生物学表达特征
将牛KLF3 基因蛋白序列和GenBank公布的其他物种的序列进行Blastp比对,结果发现KLF3 的蛋白序列不但与反刍动物具有较高的同源性高达98%,而且与人和家鼠也具有很高的同源性,比对见表2。另外,构建系统进化树后发现KLF3基因与反刍动物亲缘关系较近,结果见图1。
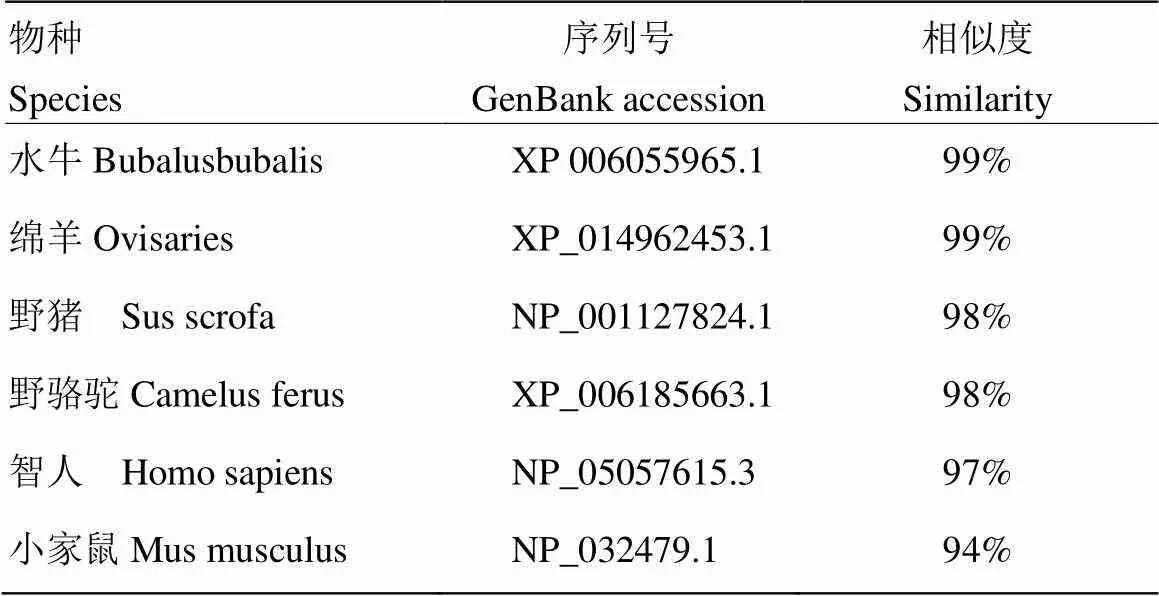
表2 牛KLF3基因蛋白Blastp比对
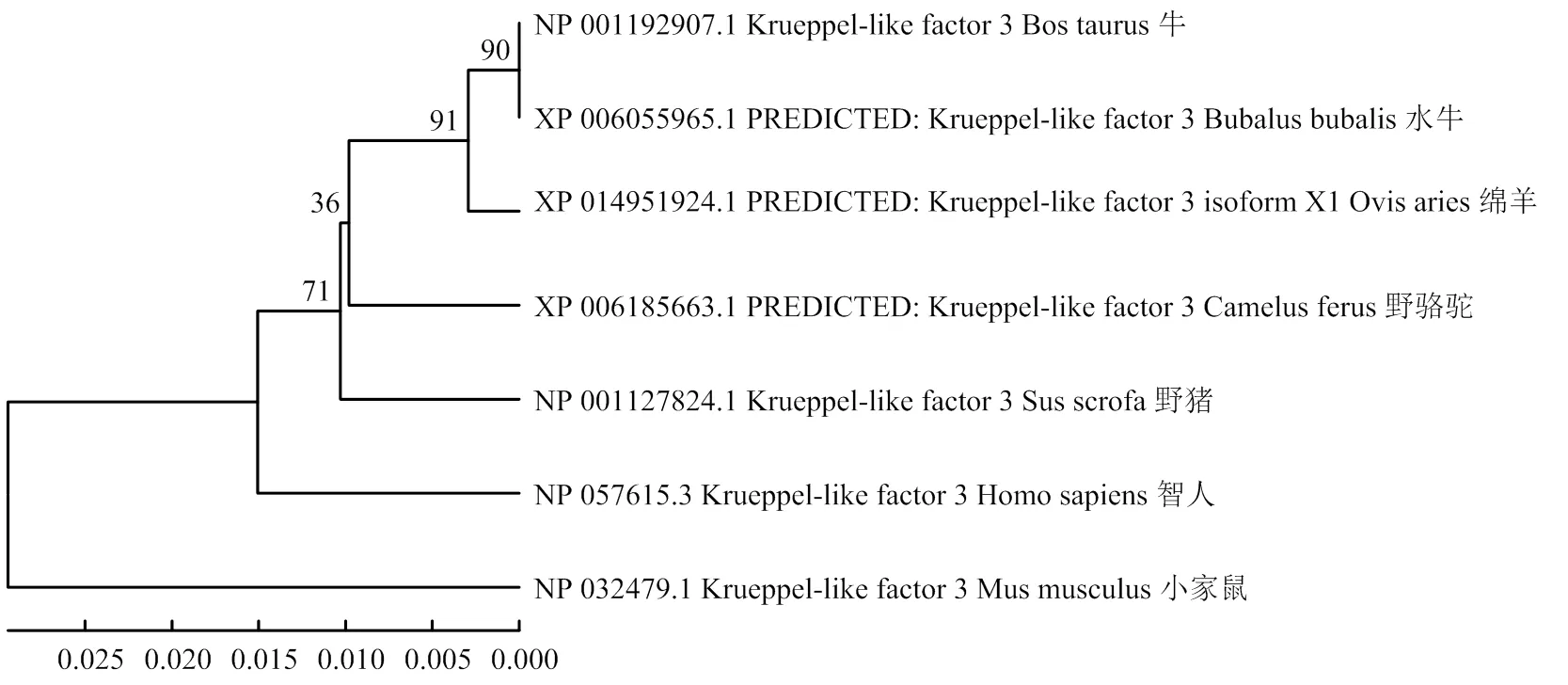
图1 牛KLF3的系统进化树
2.2 牛KLF3基因组织和脂肪细胞中的表达规律分析
qRT-PCR 结果表明KLF3 基因在新生牛的肝脏、瘤胃和脂肪中有比较高的表达量与其他组织表达量相比达到了差异显著(<0.05)(图2-A),而在肌肉中表达量最低。而KLF3 基因在牛前脂肪细胞分化中的规律发现,KLF3 基因随着脂肪细胞分化进程推进,其表达量逐渐增加(图2-B)。这表明KLF3 基因在脂肪组织形成和脂肪细胞分化中也许有比较重要的作用。
2.3 干扰KLF3基因对牛前脂肪细胞分化和脂肪酸代谢的影响
为进一步研究KLF3 基因在牛脂肪细胞分化中的作用,我们合成SiRNA干扰KLF3 基因表达,转染牛前脂肪细胞。在干扰KLF3 基因2 d 后分别提取牛 KLF3 基因干扰处理组和对照组(NC)RNA检测其干扰效率。我们发现Si2与对照组相比具有最高的干扰效率达到了73%。另外蛋白水平也发现干扰KLF3 基因处理组其蛋白水平也明显下调(图3)。并且在牛前脂肪细胞分化过程中用Si2干扰KLF3 基因后发现,在脂肪细胞分化的第0 和第4 天 KLF3 基因的表达量分别下调了73% 和 54%与对照组相比达到了极显著差异(<0.01,图4)。因此,后续试验中转染Si2用于干扰KLF3 基因的表达,并检测脂肪细胞分化标志基因和以及脂质代谢基因、和的表达量变化。
研究发现,转染Si2干扰 KLF3 基因表达后的表达量在牛脂肪细胞诱导分化的第0和第4 天与对照组相比分别下调了58%和37%,达到了差异极显著(<0.01),的表达量也分别下调了64% 和41%,达到了差异极显著(<0.01,图5)。同样干扰KLF3 基因检测对脂质代谢的关键基因的表达量变化发现脂肪细胞基因的表达量与对照组相比在脂肪细胞诱导分化的第0天和第4天分别下调了89%和60%,而的表达量干扰处理组和对照组相比在脂肪细胞诱导分化第0和第4天分别下调了50%和37%, 而的表达量则分别下调了73% 和19%,都达到了差异极显著(<0.01,图6)。

图2 KLF3基因在秦川牛新生牛中组织表达谱分析(A)和牛脂肪细胞中时序表达谱分析(B)
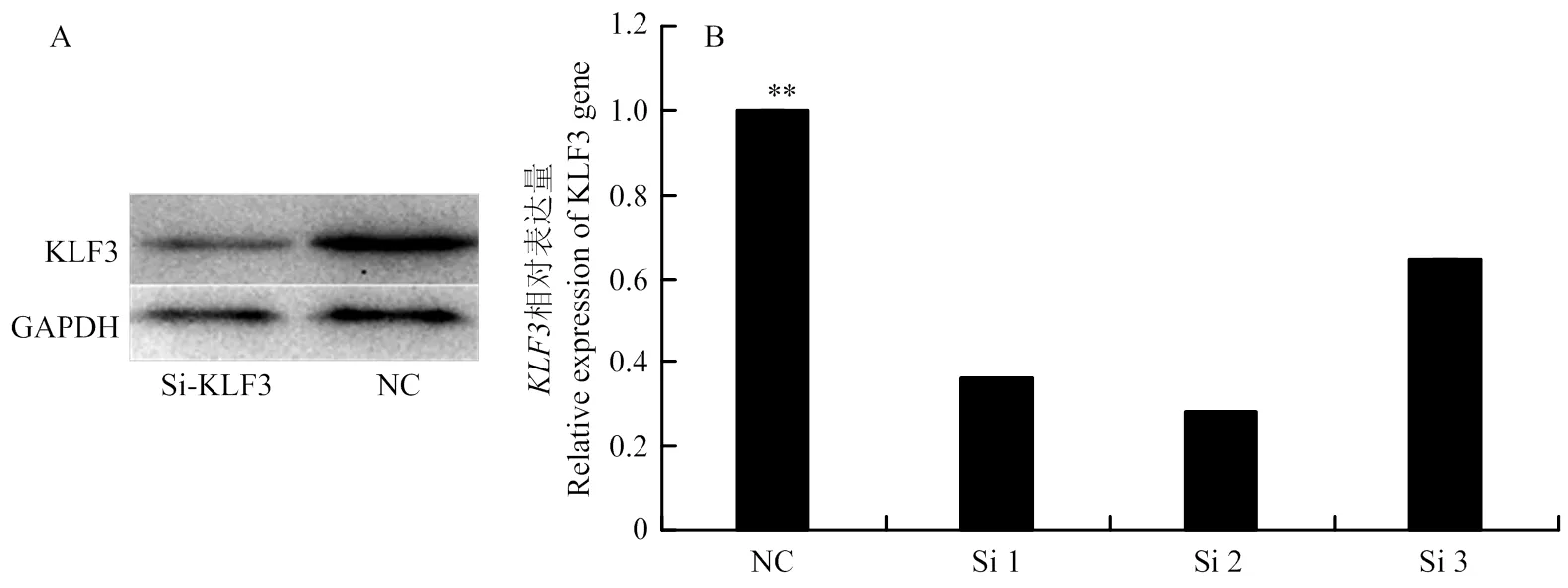
A:干扰si2- KLF3 后蛋白表达量;B:不同Si处理后KLF3的相对表达量

** 表示差异极显著,P<0.01。下同
2.4 油红O染色和细胞脂肪含量的测定
为进一步从表型上确定KLF3基因对牛脂肪细胞分化的影响,干扰牛KLF3基因处理后并诱导分化至第4天,从形态学上用油红O染色法测定干扰KLF3 基因处理组和对照组脂滴的差异。另外用牛甘油三酯测定试剂盒测定干扰处理组和对照组中甘油三酯的含量。结果表明干扰KLF3 基因抑制牛脂肪细胞分化,与对照组相比干扰处理组的脂滴较少,干扰和对照组相比甘油三酯含量明显减少并达到显著差异(<0.05,图7,图8)。

图5 牛脂肪细胞分化过程中干扰牛 KLF3 基因PPARγ和C/EBP α的相对表达量

图6 牛脂肪细胞分化过程中干扰牛KLF3 基因ACCα、FAS和FABP4 的相对表达量

图7 干扰KLF3基因并诱导牛脂肪细胞分化至第4天油红O染色图

* P<0.05
3 讨论
牛肉品质主要受脂肪沉积的影响,而脂肪沉积主要与脂肪细胞分化和脂肪酸代谢密切相关[1,24]。大量研究表明KLF家族在脂肪细胞分化中具有重要作用,并且其家族成员大多数位于上游网络调控脂肪细胞分化。KLF3 基因是KLF家族重要一员,大量研究表明其在调控脂肪细胞分化及脂质代谢中具有重要作用[17,22]。本研究通过不同物种蛋白比对和基因进化树构建发现 KLF3 基因蛋白序列不但与反刍动物具有较高同源性,高达98% 以上,而且其与人和家鼠蛋白序列同源性也高达94% 以上,这说明 KLF3 基因在不同物种中都具有重要作用,而且系统进化树比对结果发现,KLF3 基因与水牛和绵羊这类反刍动物具有更近的亲缘关系,这也侧面反映了KLF3 基因在进化过程中比较保守。另外研究发现KLF3 基因在新生牛中具有广泛表达特性并且在脂肪组织中相对表达水平较高这与在其他物种上的研究一致[19,21]。KFL3 基因的广泛表达性也表明其在机体生命活动中有着不可或缺的作用,而且其在脂肪组织中有较高表达量,并且随着脂肪细胞分化进程中其表达量逐渐增加也说明KLF3 基因也许在脂肪细胞分化和脂质沉积中起着重要作用,这也与前人关于KLF3基因在脂肪细胞分化和脂质代谢中发挥重要作用研究相一致[9,21-22]。因此,深入研究 KLF3 基因在牛脂肪细胞分化和脂质沉积作用对改善肉牛肉质性状具有重要的意义。
和作为脂肪细胞分化的中后期标志基因,在脂肪细胞分化中具有关键的作用,其表达量的变化影响着脂肪细胞的成熟[25-26]。目前关于KLF3 基因在脂肪细胞分化中的作用还不明确,ZHANG等[21]研究发现在鸡脂肪细胞中过表达 KLF3 基因可促进PPARγ 基因的启动子转录活性,而在3T3-L1细胞中有研究发现KLF3基因通过抑制基因的启动子转录活性从而抑制脂肪细胞分化[19]。本研究通过筛选最佳干扰牛KLF3 基因的SiRNA,转染至牛前体脂肪细胞并进行诱导分化,结果发现,牛脂肪细胞分化标志基因和表达量都随着KLF3 基因表达量下调明显下降(<0.01)。这表明KLF3基因在牛脂肪细胞分化过程中抑制脂肪细胞分化关键基因和表达从而抑制牛前脂肪细胞分化。不同物种中 KLF3 基因对脂肪细胞分化关键基因调控的差异也许是由于物种的差异性,因此,后续还需进一步在转录调控机制方面进行更深入的研究。
脂肪细胞的功能主要是合成并储存甘油三酯,而甘油三酯的合成在基因水平主要涉及脂肪酸的转运和合成。是脂肪酸结合蛋白,主要负责长链脂肪酸的摄取,转运及代谢调节,在脂肪酸合成过程中起关键作用[27-28]。即脂肪酸合成酶,是脂肪酸从头合成过程中的催化乙酰辅酶A和丙二酸单酰辅酶A聚合成长链脂肪酸的主要限速酶,该酶的活性及数量对动物体脂的沉积有重要影响,其表达量升高能显著增加甘油三酯的沉积[29-30]。而与一样也是动物体内脂肪酸合成的限速酶,它催化合成的丙二酰COA是长链脂肪酸合成的前体物质[31]。有研究发现KLF3 基因通过调控β-氧化调节脂肪酸代谢,另外,突变KLF3基因可使脂肪酸代谢关键基因下调[20,22],这说明KLF3基因在脂肪酸代谢中也具有重要作用。本研究发现干扰牛KLF3基因转染牛脂肪细胞后发现脂肪酸代谢基因、及的表达量也明显下调(<0.01),干扰KLF3 基因抑制脂肪酸代谢关键基因的下调,说明KLF3 基因在牛脂肪细胞中通过调节脂肪酸代谢关键基因的表达参与脂肪酸代谢从而调控甘油三酯的积累。
随后通过干扰牛KLF3 基因后,进行油红O染色并测定甘油三酯含量,发现与对照组相比,干扰牛基因组脂滴减少,甘油三酯含量下调,并且达到了差异显著(<0.05)。这进一步说明干扰KLF3 基因能够抑制牛脂肪形成并且综合脂肪细胞分化关键基因和脂肪酸代谢关键基因表达量的下调,说明干扰牛KLF3 基因抑制牛脂肪细胞分化,抑制脂滴积累。这与敲除小鼠脂肪减少结果一致[19]。总之,在牛脂肪细胞分化过程中,干扰 KLF3 基因抑制脂滴形成和甘油三酯积累。
4 结论
牛KLF3 基因通过抑制牛脂肪细胞分化标志基因和及脂肪酸代谢关键基因、及的表达抑制牛脂肪细胞分化和脂肪酸代谢,从而影响甘油三酯的积累和脂质沉积。为进一步研究牛KLF3基因调控脂质沉积、脂肪酸代谢和改善牛肉品质的转录调控提供了依据。
[1] WELLS J E, KIM M, BONO J L, KUEHN L A, BENSON A K. Meat science and muscle biology symposium:O157:H7, diet, and fecal microbiome in beef cattle.2014, 92(4): 1345-1355.
[2] ROBINSON D L, CAFE L M, GREENWOOD P L. Meat Science and Muscle Biology Symposium: developmental programming in cattle: consequences for growth, efficiency, carcass, muscle, and beef quality characteristic, 2013, 91(3): 1428-1442.
[3] LONG N M, TOUSLEY C B, UNDERWOOD K R, PAISLEY S I, MEANS W J, HESS B W, DU M, Ford S P. Effects of early-to mid- gestational undernutrition with or without protein supplementation on offspring growth, carcass characteristics, and adipocyte size in beef cattle., 2012, 90(1): 197-206.
[4] BIEKER J J. Kruppel-like factors: three fingers in many pies2001, 276(37): 34355-34358.
[5] FERNANDEZ-ZAPICO M E, LOMBERK G A, DEMARS C J, BARDSLEY M R, LIN Y H, ALMADA L L, HAN J J, MUKHOPADHYAY D, ORDOG T, BUTTAR N S, URRTTIA R. A functional family-wide screening of SP/KLF proteins identifies a subset of suppressors of KRAS-mediated cell growth2011, 435(2): 529-537.
[6] HASHMI S, ZHANG J, SIDDIQUI S S, PARHAR R S, BAKHEET R, AI-MOHANNA F. Partner in fat metabolism: role of KLFs in fat burning and reproductive behavior., 2011, 1(2): 59-72.
[7] KAWAMURA Y, TASUSHI Y, KAWAMORI R, MAEDA S. Overexpression of Kruppel-Like factor 7 regulates adipocytokine gene expressions in human adipocytes and inhibits glucose-induced insulin secretion in pancreatic beta-cell line, 2006, 20(4): 844-856.
[8] BANERJEE S S, FEINBERG M W, WATANABE M, GARY S, HASPEL R L, DENKINGER D J, KAWAHARA R, HAUNER H, JAIN M K. The Kruppel-like factor KLF2 inhibits peroxisome proliferator-activated receptor-gamma expression and adipogenesis.2003, 278(4): 2581-2584.
[9] BELL-ANDERSON K S, FUNNELL A P, WILLIAMS H, JUSOH M, SCULLY T, LIM W F, BURDACH J G, MAK K S, KNIGHT A J, HOY A J, NICHOLAS H R, SAINSBURY A, TURNER N, PEARSON R C, CROSSLEY M. Loss of Kruppel-like factor 3 (KLF3/BKLF) leads to upregulation of the insulin-sensitizing factor adipolin (FAM132A/CTRP12/C1qdc2).2013, 62(8): 2728-2737.
[10] BIRSOY K, CHEN Z, FRIEDMAN J. Transcriptional regulation of adipogenesis by KLF4, 2008, 7(4): 339-347.
[11] MORI T, SAKAUE H, IGUCHI H, GOMI H, OKADA Y, TAKASHIMA Y, NAKAMURA K, NAKAMURA T, YAMAUCHI T, KUBOTA N, KADOWAKI T, MATSUKI Y, OGAWA W, HIRAMATSU R, KASUGA M. Role of Kruppel-like factor 15 (KLF15) in transcriptional regulation of adipogenesis., 2005, 280(13): 12867-12875.
[12] PEI H, YAO Y, YANG Y, LIAO K, WU J R. Kruppel-like factor KLF9 regulates PPAR gamma transactivation at the middle stage of adipogenesis2011, 18(2):. 315-327.
[13] SMALL K S, HEDMAN A K, GRUNDBERG E, NICA A C, THORLEIFSSON G, KONG A, THORSTEINDOTTIR U, SHIN S Y, RICHARDS H B, CONSORTIUM G, INVESTIGATORS M, Consortium D, Soranzo N, Ahmadi K R, Lindgren C M, Stefansson K, Dermitzakis E T, Deloukas P, Spector T D, McCarthy M I, Mu T C. Identification of an imprinted master trans regulator at the KLF14 locus related to multiple metabolic phenotypes (vol 43, pg 561, 2011)., 2011. 43(10): 1040.
[14] LI D, YEA S, LI S, CHEN Z, NARLA G, BANCK M, LABORDA J, TAN S, FRIEDMAN J M, FRIEDMAN S L, WALSH M J. Kruppel-like factor-6 promotes preadipocyte differentiation through histone deacetylase 3-dependent repression of DLK1, 2005, 280(29): 26941-26952.
[15] YAMAMOTO K, SAKAGUCHI M, MEDINA R J, NIIDA A, SAKAGUCHI Y, MIYAZAKI M, KATAOKA K, HUH N H. Transcriptional regulation of a brown adipocyte-specific gene, UCP1, by KLF11 and KLF152010, 400(1): 175-180.
[16] JIANG S, WEI H K, SONG T X, YANG Y, ZHANG F, ZHOU Y F, PENG J, JIANG S W. KLF13 promotes porcine adipocyte differentiation through PPARgamma activation2015, 5: 28.
[17] PEARSON R C, FUNNELL A P, CROSSLEY M. The mammalian zinc finger transcription factor Kruppel-like factor 3 (KLF3/BKLF)2011, 63(2): 86-93.
[18] DEWI V, KWOK A, LEE S, LEE M M, TAN Y M, NICHOLAS H R, ISONO K, WIENERT B, MAK K S, KNIGHTS A J, QUINLAN K G, CORDWELL S J, FUNNELL A P, PEARSON R C, CROSSLEY M. Phosphorylation of Kruppel-like factor 3 (KLF3/BKLF) and C-terminal binding protein 2 (CtBP2) by homeodomain-interacting protein kinase 2 (HIPK2) modulates KLF3 DNA binding and activity.2015, 290(13): 8591-8605.
[19] SUE N, JACK B H, EATON S A, PEARSON R C, FUNNELL A P, TURNER J, CZOLIJ R, DENYER G, BAO S, MOLERO-NAVAJAS J C, PERKINS A, FUJIWARA Y, ORKIN S H, BELL-ANDERSON K, CROSSLEY M. Targeted disruption of the basic Kruppel-like factor gene (Klf3) reveals a role in adipogenesis.2008. 28(12): 3967-3978.
[20] ZHANG J, HASHMI S, CHEEMA F, AL-NASSER N, BAKHEET R, PARHAR R S, AL-MOHANNA F, GAUGLER R, HUSSAIN M M, HASHMI S. Regulation of lipoprotein assembly, secretion and fatty acid beta-oxidation by Kruppel-like transcription factor, klf-3.2013, 425(15): 2641-2655.
[21] ZHANG Z W, WU C Y, LI H, WANG N. Expression and functional analyses of Kruppel-like factor 3 in chicken adipose tissue, 2014. 78(4): 614-623.
[22] Zhang J, Bakheet R, Parhar R S, Huang C H, Hussain M M, Pan X, Siddiqui S S, Hashmi S. Regulation of fat storage and reproduction by Kruppel-like transcription factor KLF3 and fat-associated genes in Caenorhabditis elegans2011, 411(3): 537-553.
[23] 郭红芳, 昝林森, 孙永刚. 牛前体脂肪细胞的分离培养及诱导分化西北农林科技大学学报(自然科学版), 2014, 42(2): 1-6.
GUO H F, ZAN L S, SUN Y G. Primary culture and differentiation of bovine preadipocytes, 2014, 42(2): 1-6. (in Chinese )
[24] Callaway T R, Edrington T S, Nisbet D J. Meat science and muscle biology symposium: ecological and dietary impactors of foodborne pathogens and methods to reduce fecal shedding in cattle,2014, 92(4): 1356-1365.
[25] KUDO M, SUGAWARA A, URUNO A, TAKEUCHI K, ITO S. Transcription suppression of peroxisome proliferator-activated receptor gamma2 gene expression by tumor necrosis factor alpha via an inhibition of CCAAT/ enhancer-binding protein delta during the early stage of adipocyte differentiation2004, 145(11): 4948-4956.
[26] GRIMALDI P A. The roles of PPARs in adipocyte differentiation, 2001. 40(4): 269-281.
[27] GARIN-SHKOLNIK T, RUDICH A, HOTAMISLIGIL G S, RUBINSTEIN M. FABP4 attenuates PPARgamma and adipogenesis and is inversely correlated with PPARgamma in adipose tissues.2014, 63(3): 900-911.
[28] MITA T, FURUHASHI M, HIRAMITSU S, ISHII J, HOSHINA K, ISHIMURA S, FUSEYA T, WATANABE Y, TANAKA M, OHNO K, AKASAKA H, OHNISHI H, YOSHIDA H, SAITOH S, SHIMAMOTO K, MIURA T. FABP4 is secreted from adipocytes by adenyl cyclase- PKA- and guanylyl cyclase-PKG-dependent lipolytic mechanisms.2015, 23(2): 359-367.
[29] SEMENKOVICH C F. Regulation of fatty acid synthase (FAS), 1997, 36(1): 43-53.
[30] 张力莉, 徐晓峰, 动物脂肪酸合成酶基因表达调控的研究进展畜牧与兽医, 2012, 44(4): 101-103.
ZHANG L L, XU X F. Advances in research on regulation of animal fatty acid synthase gene expression.2012, 44(4): 101-103. (in Chinese)
[31] MAO J, DEMAYO F J, LI H, ABU-ELHEIGA L, GU Z, SHAIKENOV T E, KORDARI P, CHIRALA S S, HEIRD W C, WAKIL S J. Liver-specific deletion of acetyl-CoA carboxylase 1 reduces hepatic triglyceride accumulation without affecting glucose homeostasis., 2006, 103(22): 8552-8557.
The Effect of Krüppel-like Factor 3 (KLF3) Gene on Bovine Fat Deposition
GUO HongFang1, NING Yue1, CHENG Gong1,2, ZAN LinSen1,2
(1College of Animal Science and Technology, Northwest Agriculture and Forestry University, Yangling 712100, Shaanxi;2National Beef Cattle Improvement Center, Yangling, 712100, Shaanxi)
【Objective】This study aimed to investigate the effects of bovine Krüppel-like factor 3 (KLF3) gene on bovine adipocytes differentiation and fatty acid metabolism, and to explore the effect of KLF3 gene on lipid deposition. 【Method】This research synthesized interference RNA (siRNA) of, and then transfected KLF3 gene SiRNA and negative to the Qinchuan cattle preadipocytes when the cells grew to a confluence of 70%-90%. The QPCR method was used to determine the expression of adipocyte differentiation marker genesandas well the key genes,andgene interfered. At the same time, on the 4th day of inducing adipocyte differentiation after interfering with KLF3 gene treatment, oil red O staining was used to observe the difference of lipid droplets between the KLF3 gene treatment group and the control group, and the enzyme-linked immunosorbent assay (ELISA) method was used to determine the triglyceride content to further determine the effect of the KLF3 gene on bovine adipocyte differentiation and lipid metabolism. 【Result】The results indicated that KLF3-Si2 had the highest interference efficiency, which was 73%. After transfected with KLF3-Si2, on the 0th and 4th day of bovine adipocytes induced differentiation, the expression ofwas extremely significant (<0.01) down-regulated by 58% and 37%, respectively; the expression ofwas also extremely significant (<0.01) down-regulated by 64% and 41%, respectively; the expression level of, a key gene for lipid metabolism, was down-regulated by 89% and 60%, respectively; while the expression ofinterfered with thetreatment group and the control group was down-regulated by 50% and 37%, respectively; the expression ofwas down-regulated by 73% and 19%, respectively, and all the difference was extremely significant (<0.01). On the 4th day of induced differentiation, oil red O staining and triglyceride content determination also showed that the lipid droplets were decreased and the triglyceride content was significantly decreased in the silence KLF3 gene treated group compared with the control group (<0.05). 【Conclusion】Interfering with bovine KLF3 gene could inhibit bovine adipocytes differentiation and fatty acid synthesis key genes expression, thus affecting fat deposition.
Qinchuan cattle; preadipocytes;; fat deposition
10.3864/j.issn.0578-1752.2019.07.014
2018-10-10;
2019-01-22
国家重点研发计划(No.2018YFD0501700)、国家863计划(No.2013AA102505)、国家肉牛牦牛产业技术体系(CARS-37)
郭红芳,E-mail:guohongfangkl@126.com。通信作者昝林森,E-mail:zanlinsen@163.com
(责任编辑 林鉴非)

