F17大肠杆菌在湖羊羔羊个体脾脏中LncRNA表达谱变化
黄赛男,金澄艳,鲍建军,王悦,陈炜昊,吴天弋,王利宏,吕晓阳, 高雯,王步忠,朱国强,戴国俊,孙伟,5
F17大肠杆菌在湖羊羔羊个体脾脏中LncRNA表达谱变化
黄赛男1,金澄艳1,鲍建军2,王悦1,陈炜昊1,吴天弋1,王利宏1,吕晓阳1, 高雯1,王步忠3,朱国强4,戴国俊1,孙伟1,5
(1扬州大学动物科学与技术学院,江苏扬州 225009;2南京新九州农牧科技有限公司,南京 210000;3江苏西来原生态农业有限公司,江苏泰州 225300;4扬州大学兽医学院,江苏扬州 225009;5扬州大学教育部农业与农产品安全国际合作联合实验室,江苏扬州 225009)
【目的】通过筛选对大肠杆菌()F17菌毛非腹泻型与腹泻型的绵羊脾脏中差异表达的lncRNA,来探究lncRNA对绵羊抗腹泻的作用。【方法】本研究通过对湖羊羔羊口服F17菌株获得非腹泻和腹泻型个体,利用羔羊肠道细菌计数、病理组织切片验证攻毒成功性;构建非腹泻组和腹泻组羔羊脾脏的cDNA文库,使用Illumina HiSeq 2500平台进行配对测序;通过Gene Ontology(GO)和KEGG Pathway富集分析对差异表达转录本功能描述和细胞通路分析,利用FPKM法估计lncRNA和mRNA转录物的表达水平,并用高通量测序技术RNA-seq筛选出非腹泻和腹泻个体脾脏中的差异表达lncRNA;然后利用荧光定量PCR技术检测了非腹泻组和腹泻组羔羊脾脏组织中DE lncRNA和DE mRNA的表达水平,来验证筛选的DE lncRNA在非腹泻组过程中发挥作用。【结果】羔羊口服F17菌株后,出现非腹泻和腹泻两种表型,腹泻组羔羊肠道中的细菌数量显著高于非腹泻组(<0.05),同时腹泻组羔羊空肠黏膜组织出现不同程度的损伤,色泽暗沉,小肠绒毛部分脱落。笔者利用RNA-seq在非腹泻和腹泻羔羊脾脏中筛选出34个差异表达的(DE)lncRNA,703个的DE mRNA,随机选择一共12个DE lncRNA和DE mRNA,用q-PCR验证它们在非腹泻型和腹泻型羔羊体内的相对表达水平,发现与RNA-seq结果一致。通过Gene Ontology(GO)和KEGG Pathway富集分析,将DE lncRNA与GO 数据库进行比对的结果表明一共有34条lncRNA被注释和分类到302个功能亚类中,绵羊蛋白质结合(GO:0005515),细胞核(GO:0005634),poly(A)RNA结合(GO:0044822),细胞质(GO:0005737),组织重塑(GO:0048771),内肽酶活性的调节(GO:0052548)),6-磷酸果糖-2-激酶/果糖-2,6-双磷酸酶复合物(GO:0043540),磷脂酰肌醇磷酸化(GO:0046854),果糖-2,6-二磷酸2-磷酸酶活性(GO:0004331),钙依赖性磷脂酶C活性(GO:0050429)等10 个功能亚类的lncRNA较多,而其余的功能亚类的lncRNA分布较少。将DE lncRNA与KEGG 通路数据库进行比对的结果表明,一共有34条lncRNA被注释和归类到149个KEGG 通路中,绵羊甲状腺激素信号通路(路径:ko04919),Spliceosome(路径:ko03040),白细胞跨内皮迁移(路径:ko04670),神经营养因子信号通路(路径:ko04722),溶酶体(路径:ko04142),MAPK信号通路 - 途径(路径: ko04011),鞘脂信号通路(路径:ko04071),吞噬体(路径:ko04145),氧化磷酸化(路径:ko00190)等9 个KEGG 通路的lncRNA较多,而其余的KEGG 通路的lncRNA分布较少。 通过lncRNA-mRNA相互作用网络分析,发现6个共表达基因:MYO1G、TIMM29、CARM1、ADGRB1、SEPT4、DESI2。【结论】探究了对于腹泻产生非腹泻和腹泻型羔羊脾脏中lncRNA的表达谱,发现了非腹泻和腹泻羔羊脾脏中差异表达的lncRNA,有助于找出羔羊如何抵抗腹泻的发生机制,为羔羊抵抗腹泻提供科学的依据。
大肠杆菌F17;lncRNA;湖羊羔羊
0 引言
【研究意义】羊大肠杆菌病是规模化羊场最为常见高发的细菌性疾病之一,传统沿用的抗生素治疗方案存在诸多缺陷。检测影响绵羊大肠杆菌非腹泻型相关基因的表达情况是分析绵羊抗病分子机制的基础,从而可以发现与抗大肠杆菌性状相关的候选基因。【前人研究进展】1966年,Orskov等[1]首先报道了猪源大肠杆菌黏附性抗原K88,后证实黏附性抗原K88是一种依赖于质粒表达的蛋白质抗原[2],电子显微镜下观察到其形态为暴露在菌体表面的丝状物。目前已经鉴定出多种动物源产肠毒素大肠杆菌(ETEC)菌毛,包括K88、K99、987P、F17和F41等都是动物源ETEC重要的黏附性毒力因子。lncRNA(Long non-coding RNA)是长度大于200个核苷酸的非编码RNA。大量的研究表明,lncRNA 与人类各种肿瘤、心血管疾病和代谢疾病的发生具有密切的联系和调控作用。值得注意的是,近年来越来越多的研究表明,lncRNA在抗病毒等天然免疫反应中也具有重要的调控作用[3-7],相比于人的lncRNA,羊的lncRNA功能研究较滞后,而且大多集中在分析lncRNA对羊肌肉生长发育、睾丸发育、毛囊发育等性状的调控作用[8-10]。【本研究切入点】目前,关于绵羊抗病方面研究还不多,少量的研究主要集中在疾病防治方面[11-12],而绵羊抗病的分子遗传学基础研究鲜有报道。【拟解决的关键问题】利用RNA-seq,对大肠杆菌F17菌毛非腹泻型与腹泻型绵羊个体脾脏中差异表达lncRNA进行筛选,并基于顺式(cis)机制进行靶基因预测以及功能注释分析,筛选出关键lncRNA。在此基础上利用qPCR进行验证。本研究从lncRNA层面上,加深了对绵羊非腹泻组大肠杆菌F17菌毛的认识,同时有望确定绵羊非腹泻型大肠杆菌F17菌毛的功能基因,解决中国地方羊品种对大肠杆菌病的抗病育种关键问题,为今后制订抗大肠杆菌病遗传选育策略奠定基础和提供理论依据。
1 材料与方法
1.1 试验设计和样本采集
试验用绵羊于2016年12月购自泰州市西来原生态农业有限公司。随机选择生长发育良好、日龄体重相近的18只3日龄羔羊,并将羔羊全部隔离饲养。先用10%羔羊奶粉(表1)饲喂,以确保在试验前适应饮食需要。5日龄时饲喂12.5%羔羊奶粉和大肠杆菌F17菌液(4.6×108CFU/mL),同时保证自由饮水。每天记录羔羊粪便形态(表2),在有一些羔羊持续腹泻2 d后,将羔羊分为非腹泻组和腹泻组,并对羊进行安乐死。用4%多聚甲醛收集肠道组织。用RNAlater收集每只羔羊的肝脏、脾脏、十二指肠、空肠和回肠,并立即在液氮中冷冻以进行RNA提取。
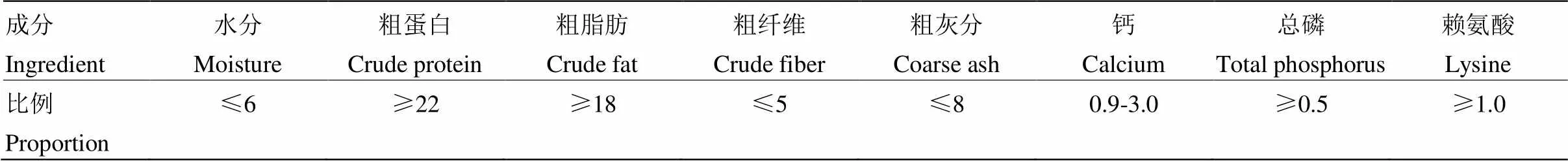
表1 奶粉成分(每100克奶粉)
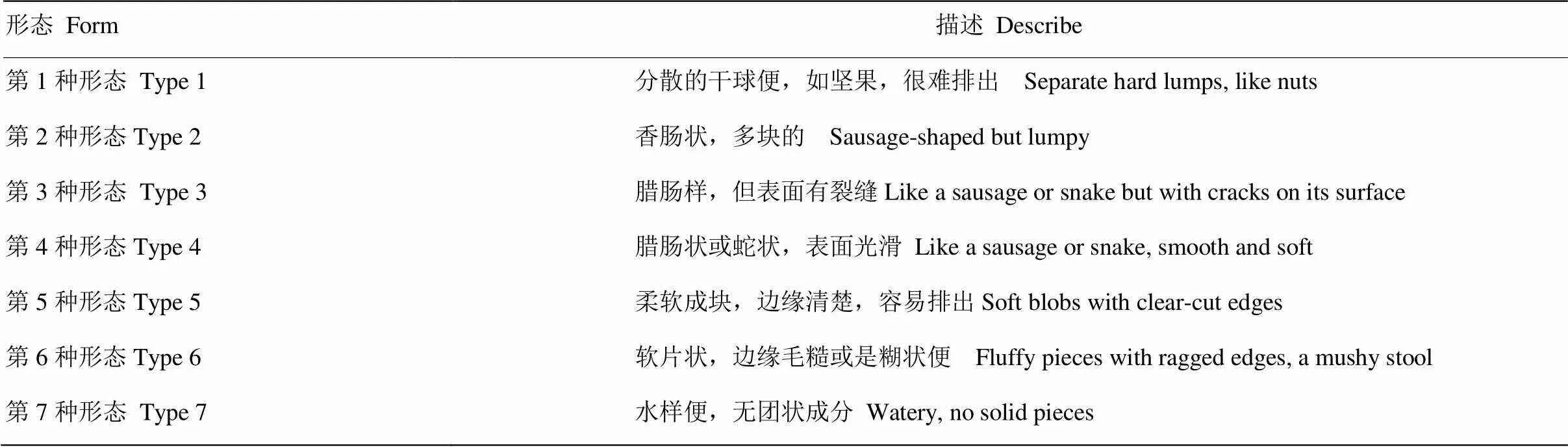
表2 粪便形式量[20]
1.2 H.E.染色
用0.9%盐水洗涤空肠组织,并在室温下用4%多聚甲醛固定48 h,然后进行组织学分析。接下来,用苏木精-伊红染色7 μm切片,并在显微镜下观察空肠上皮形态。
1.3 文库建设和测序
从每组中的3只绵羊脾脏提取总RNA,并使用NanoDrop 2000超微量分光光度计和Agilent 2100生物分析仪进行质控。用Ribo-Zero TM试剂盒(Epicenter, Madison, WI, USA)除去核糖体RNA。将RNA片段化(平均长度约为200 bp),然后通过逆转录合成和纯化cDNA。使用Qubit®dsDNA HS测定试剂盒进行PCR扩增和纯化后,选择使用NEBNext®Ultra™RNA文库制备试剂盒进行文库构建。在上海欧易生物医学科技有限公司使用Illumina HiSeq 2500平台,对文库进行末端配对测序(测序读长为150 bp)。
1.4 鉴定lncRNA和mRNA
过滤原始数据以消除低质量读长,使用映射到参照基因组(Ovis aries v4.0)的干净读长进行组装。用CPC[13]、CNCI[14]、Pfam[15]、PLEK[16]四种编码潜能分析方法,将来自未知转录物中推定的编码RNA和非编码RNA候选物进行分类。将最小长度和外显子数设置为阈值,滤出推定的编码RNA,长度超过200 nt的两个外显子的转录物被选为lncRNA候选物。使用cuffcompare归类不同类型的lncRNA,主要包括intergenic lncRNA(字符为u),intronic lncRNA(字符为i), anti-sense lncRNA(字符为x),sense- overlapping lncRNA(字符为o)。
1.5 差异表达分析
由于FPKM法[17]同时考虑了测序深度和转录本长度对fragments计数的影响,因此使用FPKM值(fragments Per kb per Million reads,是每百万fragments中来自某一基因每千碱基长度的fragments数目)估计lncRNA和mRNA转录物的表达水平。使用DESeq[18]软件检测两组之间DE基因的数量和FPKM值。在利用RNA-seq数据比较分析两个样品中同一个转录本是否存在差异表达的时候,选取两个标准:一是FoldChange,就是两样品中同一个转录本表达水平的变化倍数;二是p-value或FDR(padjust),FDR值的计算方法先要对每个转录本计算p-value,再用FDR错误控制法对p-value作多重假设检验校正。默认筛选差异的条件为<0.05且| log2(倍数变化)|>1。
1.6 GO和KEGG通路分析
得到差异表达转录本之后,对差异表达转录本进行GO富集分析,对其功能进行描述(结合GO注释结果)。统计每个GO条目中所包括的差异转录本个数,并用Fisher's exact test计算每个GO条目中差异转录本富集的显著性(默认筛选差异的条件为<0.05)。KEGG[19]是有关通路的主要公共数据库,利用KEGG数据库对差异转录本进行通路分析(结合KEGG注释结果),并用Fisher's exact test计算每个通路条目中差异转录本富集的显著性(默认筛选差异的条件为<0.05),通路分析对试验结果有提示作用,通过差异转录本的通路分析,可以找到富集差异转录本的通路条目,寻找不同样品的差异转录本可能和哪些细胞通路的改变有关。
1.7 预测DE lncRNA的靶基因
通过计算多个基因之间的Pearson相关系数和值预测DE lncRNA的靶基因。使用|corelation|≥0.7并且≤0.05来过滤转录物,选择与黏附功能相关通路的DE lncRNA,并通过顺式和反式作用预测所有DE lncRNA的靶基因。
1.8 验证DE lncRNA的表达水平
为验证筛选的DE lncRNA在非腹泻组过程中发挥作用,用q-PCR检测了非腹泻组和腹泻组羔羊脾脏组织中一共12种DE lncRNA和DE mRNA的表达水平,使用2-ΔΔCt方法将每个RNA的相对定量归一化为GAPDH,lncRNA的引物见表3。
1.9 统计分析
使用SPSS软件(版本20.0)分析所有数据,使用单因素方差分析(ANOVA)分析差异转录本的相对表达量,并使用Tukey检验进行多重比较。<0.05被认为具有统计学意义。每组包含3个样品,每个试验重复3次。
2 结果
2.1 非腹泻组和腹泻组肠道细菌数量比较及组织学形态观察
根据粪便形态[20],将试验个体分为非腹泻组(12、13、14p)与腹泻组(15、16、17p)。在腹泻组中,细菌计数在4.7×108至1.9×109之间,但在非腹泻组中细菌计数下降到了5.1×106至9.0×107。与非腹泻组相比,腹泻组羔羊肠道细菌数量明显较高(<0.05,表4,图1),腹泻组羔羊空肠黏膜组织出现不同程度的损伤,色泽暗沉,细胞死亡、裂解,黏膜下层留下较大间隙,肠绒毛部分脱落,肠道黏膜中丰富的毛细血管都被极大的破坏,难以在切片中找到相应结构(图2)。
表3 GAPDH,DE lncRNA和mRNA的引物
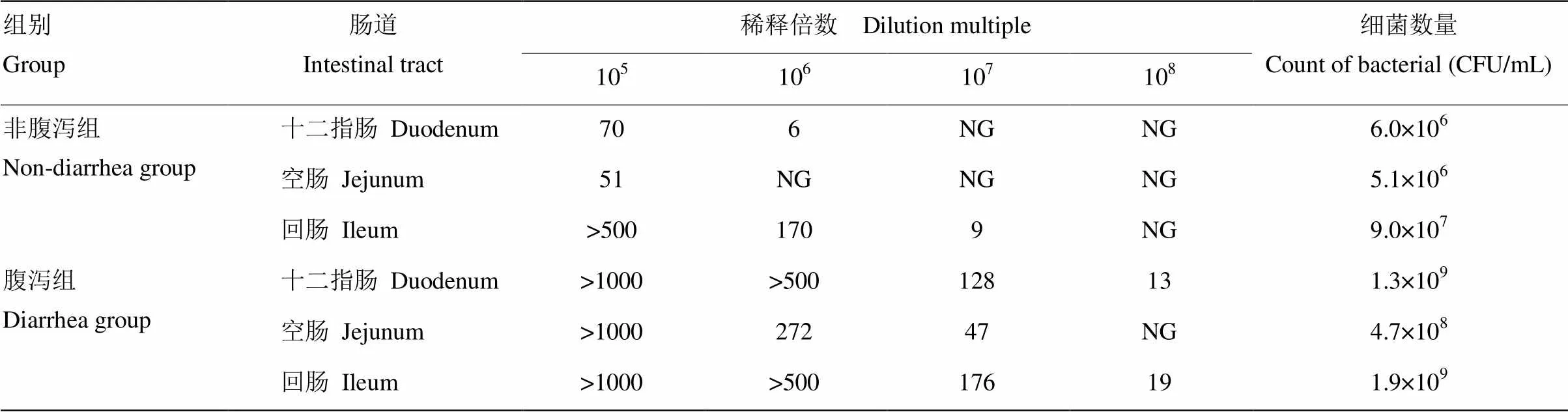
表4 非腹泻组与腹泻羔羊肠道细菌数量对比

图1 非腹泻组与腹泻羔羊肠道细菌数量对比

图2 非腹泻组和腹泻组羔羊空肠组织学形态观察
2.2 绵羊脾脏RNA测序概述
构建非腹泻组和腹泻组羔羊脾脏的cDNA文库,使用Illumina HiSeq 2500平台进行测序,分别产生了354 943 820和370 616 990个原始读长。文库的GC含量分别为48.33%和49.67%。将清洁读长中的有效读长映射到ovis aries v4.0参考基因组,超过73.5%的读长被映射到基因组。映射到参考序列多个位置的序列数目低于4.5%,超过70%的读长被唯一映射到参考序列。大约35%的读长映射到基因组的正、负链。此外,通过注释分析映射到外显子区域(约60%)的读长数量高于基因间和内含子区域。这些结果表明,匹配效率很高,大多数读长映射到外显子区域。
2.3 绵羊脾脏中转录本的鉴定
在绘制参考序列后,我们从42 460个汇编的转录物中鉴定出已知的1 988个lncRNA和38 843个mRNA。lncRNA长度主要分布于200—5 000 bp,平均长度为2 124 bp。此外,lncRNA主要为intergenic lncRNA(字符为u),intronic lncRNA(字符为i),包含2—3个外显子(图3)。
2.4 分析和验证差异转录本
利用FPKM值估计lncRNA和mRNA转录物的表达水平,其中lncRNA转录本表达水平相对较低(图4)。
在<0.05和| log2(倍数变化)|>1的条件下,筛选出14个上调和20个下调的DE lncRNA,370个上调和333个下调的DE mRNA(图5)。
为了进一步验证RNA-seq的可靠性,随机选择一共12个DE lncRNA和DE mRNA,用q-PCR验证它们在非腹泻组和腹泻组羔羊体内的相对表达水平,发现与RNA-seq结果一致(图6),表明RNA-seq数据是可靠的。这些分析还表明,高通量测序具有检测低表达水平(0<FPKM<1)基因的优点。

图3 lncRNA长度、类型、预测lncRNA的外显子数目统计

图4 lncRNA(左)和mRNA(右)转录本表达水平箱线图

图5 非腹泻组和腹泻组型羔羊之间差异表达的lncRNAs(左)和mRNAs(右)

图6 DE lncRNA和mRNA在非腹泻组和腹泻组羔羊体内的相对表达水平
2.5 DE lncRNA的GO功能注释和KEGG 通路富集分析
在“lncRNA名称--功能预测Term”之间的对应关系中(附件),分别选取预测可信度(按p-value排序)最高的Top 500个预测关系,对其中各个功能预测Term进行频次计数,统计功能注释较多的GO(或通路)term,反映该试验中得到的差异lncRNAs功能分布的整体情况(图7)。
DE lncRNA与GO 数据库进行比对的结果表明,一共有34条lncRNA被注释和分类到302个功能亚类中。图7显示了DE lncRNA数量排名前30 位的功能亚类。结果显示,绵羊蛋白质结合(GO:0005515)、细胞核(GO:0005634)、poly(A)RNA结合(GO:0044822)、细胞质(GO:0005737)、组织重塑(GO:0048771)、内肽酶活性的调节(GO:0052548))、6-磷酸果糖-2-激酶/果糖-2,6-双磷酸酶复合物(GO:0043540)、磷脂酰肌醇磷酸化(GO:0046854)、果糖-2,6-二磷酸2-磷酸酶活性(GO:0004331)、钙依赖性磷脂酶C活性(GO:0050429)等10 个功能亚类的lncRNA较多,而其余的功能亚类的lncRNA分布较少。

DE lncRNA与KEGG 通路数据库进行比对的结果表明,一共有34条lncRNA被注释和归类到149个KEGG 通路中。图7显示了DE lncRNA数量排名前30 位的KEGG 通路。结果显示,绵羊甲状腺激素信号通路(路径:ko04919)、Spliceosome(路径:ko03040)、白细胞跨内皮迁移(路径:ko04670)、神经营养因子信号通路(路径:ko04722)、溶酶体(路径:ko04142)、MAPK信号通路-途径(路径:ko04011)、鞘脂信号通路(路径:ko04071)、吞噬体(路径:ko04145)、氧化磷酸化(路径:ko00190)等9 个KEGG 通路的lncRNA较多,而其余的KEGG 通路的lncRNA分布较少。
2.6 LncRNAs与其临近编码基因的基因组共表达结果
对于感兴趣的DE lncRNAs,笔者搜索其上下游100 kb范围内的所有编码基因,并与该lncRNA有显著共表达(皮尔森相关性计算)的基因取交集。这些在基因组上临近且表达模式上存在共表达的基因很可能被该lncRNA所调控。因此,发现6个基因可能被该相关lncRNA所调控(表5)。

表5 差异lncRNAs的cis调控
3 讨论
由于转录组分析的快速发展,lncRNAs在过去几年作为细胞发育中的新型调节剂受到广泛关注[21]。目前已经鉴定的lncRNAs主要与癌症相关,如前列腺癌[22]、胃癌[23]、肺癌[24]以及乳腺癌[25]等,以及与生殖功能相关的lncRNA[26-29]。然而,迄今为止,关于羔羊腹泻的lncRNA,特别是绵羊的研究报道很少。湖羊是一种具有高繁殖力和对湿热气候适应性较强的中国特有品种,可以全年在室内饲养。在这项研究中,不仅提供了绵羊腹泻过程中lncRNA的第一个概况,还研究了lncRNA在抗病过程中的可能作用。
长期以来,羔羊腹泻对牧场造成了严重的经济损失。在研究中,发现lncRNA的表达水平低于mRNA,与绵羊睾丸组织一致[30],并且lncRNAs和mRNAs的平均长度比猪更长(分别为1 713和1 983 bp[27]。搜索其上下游100 kb范围内的所有编码基因,并与该lncRNAs有显著共表达(皮尔森相关性计算)的基因取交集,发现6个与lncRNA共表达的基因:MYO1G(肌球蛋白IG)、TIMM29(线粒体内膜的转位酶29)、CARM1(共激活因子相关的精氨酸甲基转移酶1)、ADGRB1(黏附G蛋白偶联受体B1)、SEPT4(septin 4)、DESI2(脱嘌呤的肽酶2)。
MYO1G在维持B细胞淋巴细胞的细胞刚度(cell stiffness)方面扮演了很重要的角色,MYO1G的缺失降低细胞刚度,影响细胞黏附,扩散,吞噬作用和B细胞淋巴细胞的內吞过程[31]。关于TIMM29的研究报道比较少,最近研究报道TIMM29被鉴定为哺乳动物TIMM22蛋白复合物第一个特异性组分,同时其在TIMM23蛋白的组装过程中扮演重要角色[32-33]。CARM1,是蛋白质精氨酸甲基转移酶家族(protein arginine methyltransferase,PRMTs)的成员之一,含 有高度保守结构域的具有甲基转移酶活性的酶。CARM1 敲除的小鼠出生时就死亡[34],这表明CARM1为产后生存所必须。后来研究还发现,CARM1抑制能够促进HIV-1活化[35]。ADGRB1属于跨膜蛋白家族——黏附G蛋白偶联受体(aGPCRs)的成员之一,aGPCR家族的定义特征是保守的GAIN结构域,具有自体蛋白水解活性并且可以切割第一跨膜结构域附近的受体,研究发现通过GAIN结构域切割显示的新的N末端茎可以直接激活作为束缚激动剂的ADGRB1[36]。Septins是一个具有GTP 酶活性的高度保守的细胞骨架蛋白家族,肿瘤抑制因子SEPT4是Septins 家族成员之一,其可诱导癌细胞凋亡[37]。研究发现,SEPT4 基因突变的小鼠精子环及其邻近的皮质结构紊乱,引起精子活力低下甚至不运动,最终导致不育症的发生[38-39]。DESI2 基因是一种促凋亡基因,体外试验表明DESI2基因过表达可诱导胰腺癌等肿瘤细胞凋亡,能有效抑制部分癌细胞增殖,其与IP10的组合基因治疗能显著抑制肿瘤生长,并有效延长了患肿瘤小鼠的存活时间[40-41]。
总共703个mRNA和34个已知的lncRNA在非腹泻组和腹泻组之间显着差异表达,其中14个上调和20个下调DE lncRNA。另外,确定了两组中共有1 942个新的lncRNA。搜索lncRNA上下游100 kb范围内的所有编码基因,并与该lncRNA有显著共表达(皮尔森相关性计算)的基因取交集,发现6个基因可能被该相关lncRNA所调控。为了进一步验证RNA-Seq结果,采用q-PCR验证12种已知的lncRNA和mRNA的表达水平,结果一致。
GO是一种广泛用于研究基因功能关系的生物信息学工具。对34个DE lncRNA进行GO分析,在Top 500的预测关系对中,有16个DE lncRNA富集到蛋白结合(GO:0005515)条目。KEGG 通路分析表明,信号通路如Sphingolipid signaling 通路(path:ko04071)、Axon guidance(path:ko04360)、Glycosylphosphatidylinositol (GPI)- anchor biosynthesis(path:ko00563)可能是DE lncRNA共表达基因的重要KEGG途径,相关的lncRNA可能潜在地参与菌毛黏附肠道黏膜的过程。然而,这些通路在抗病过程中的作用很大程度上仍然是未知的。
4 结论
研究了对于腹泻产生非腹泻型和腹泻型羔羊脾脏中lncRNA的表达谱,以进一步了解其在绵羊抗病发生过程中的调控作用。发现了非腹泻组和腹泻组羔羊脾脏组织中差异表达的lncRNA。有助于找出羔羊如何抵抗腹泻的发生机制。此外,进一步研究这些lncRNA可以为羔羊抵抗腹泻提供科学的依据。
[1] Orskov I, Orskov F. Episome-carried surface antigen K88 ofI. Transmission of the determinant of the K88 antigen and influence on the transfer of chromosomal markers.1966, 91(1): 69-75.
[2] Stirm S, Orskov I, Orskov F. K88, an episome-determined protein antigen of.,1966,209(5022): 507-508.
[3] Kim T, Jeon Y J, Cui R, Lee J H, Peng Y, Kim S H, Tili E, Alder H, Croce C M. Role of MYC-regulated long noncoding RNAs in cell cycle regulation and tumorigenesis., 2015, 107(4). doi: 10.1093/jnci/dju505.
[4] Ouyang J, Zhu X M, Chen Y H, Wei H T, Chen Q H, Chi X J, Qi B M, Zhang L F, Zhao Y, Gao F, Wang G S, Chen J L. NRAV, a long noncoding RNA, modulates antiviral responses through suppression of interferon-stimulated gene transcription., 2014, 16(5): 616-626.
[5] Li Z, Rana T M. Decoding the noncoding: prospective of lncRNA-mediated innate immune regulation., 2014, 11(8): 979-985.
[6] Turner M, Galloway A, Vigorito E. Noncoding RNA and its associated proteins as regulatory elements of the immune system., 2014, 15(6): 484-491.
[7] Heward J A, Lindsay M A. Long non-coding RNAs in the regulation of the immune response., 2014, 35(9): 408-419.
[8] Ren C, Deng M, Fan Y, Yang H, Zhang G, Feng X, Li F, Wang D, Wang F, Zhang Y. Genome-wide analysis reveals extensive changes in LncRNAs during skeletal muscle development in Hu Sheep., 2017, 8(8):283-313.
[9] Zhang Y, Yang H, Han L, Li F, Zhang T, Pang J, Feng X, Ren C, Mao S, Wang F. Long noncoding RNA expression profile changes associated with dietary energy in the sheep testis during sexual maturation., 2017, 7(1): 5180.
[10] Yue Y, Guo T, Yuan C, Liu J, Guo J, Feng R, Niu C, Sun X, Yang B. Integrated analysis of the roles of long noncoding RNA and coding RNA expression in sheep () skin during initiation of secondary hair follicle.. 2016, 11(6): e: 0156890.
[11] 徐兴文. 羊大肠杆菌病防治. 中国畜禽种业, 2017, 13(4):112-113.
XU X W. Prevention and treatment of sheep.2017, 13(4):112-113. (in Chinese)
[12] 张文静. 羊大肠杆菌病的防控措施. 畜牧兽医科技信息, 2017(6):76.
ZHANG W J. Prevention and control measures for sheep colibacillosis.2017(6): 76. (in Chinese)
[13] Kong L, Zhang Y, Ye ZQ, Liu XQ, Zhao SQ, Wei L, Gao G.ess the protein-coding potential of transcripts using sequence features and support vector machine.2007, 36: 345-349.
[14] Sun L, Luo H T , Bu D C , Zhao G G , Yu K t, Zhang C h , Liu Y n , Chen R s, Zhao Y. Utilizing sequence intrinsic composition to classify protein-coding and long non-coding transcripts., 2013 , 41 (17) :166.
[15] Finn R D, Bateman A, Clements J, Coggill P, Eberhardt R Y, Eddy S R, Heger A, Hetherington K, Holm L, Mistry J, Sonnhammer E L, Tate J, Punta M. The Pfam protein families database., 2014, 42(Database issue):222-230.
[16] Li A, Zhang J, Zhou Z. PLEK: a tool for predicting long non-coding RNAs and messenger RNAs based on an improved k-mer scheme., 2014, 15(1):311.
[17] TRAPNELL C, Williams B A, Pertea G, Mortazavi A, Kwan G, van Baren M J, Salzberg S L, Wold B J, Pachter L. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation., 2010, 28(5): 511-515.
[18] Anders S, Huber W. Differential expression analysis for sequence count data., 2010, 11: R106.
[19] Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M, Itoh M, Katayama T, Kawashima S, Okuda S, Tokimatsu T, Yamanishi Y. KEGG for linking genomes to life and the environment.. 2008, 36: 480-484.
[20] Lewis S J, Heaton K W. Stool form scale as a useful guide to intestinal transit time., 1997, 32(9): 920-924.
[21] Veneziano D, Nigita G, Ferro A. Computational approaches for the analysis of ncRNA through deep sequencing Te1chniques., 2015, 3(77):77.
[22] Martignano F, Rossi L, Maugeri A, Gallà V, Conteduca V, De Giorgi U, Casadio V, Schepisi G. Urinary RNA-based biomarkers for prostate cancer detection., 2017, 473:96-105.
[23] Hao Y P, Qiu J H, Zhang D B, Yu C G. Long non-coding RNA DANCR, a prognostic indicator, promotes cell growth and tumorigenicity in gastric cancer., 2017, 39(6): 1010428317699798.
[24] Huang Q, Chi Y, Deng J, Liu Y, Lu Y, Chen J, Dong S. Fine particulate matter 2.5 exerted its toxicological effect by regulating a new layer, long non-coding RNA., 2017, 7(1): 9392.
[25] Li M, Li X, Zhuang Y, Flemington E K, Zhen L, Shan B. Induction of a novel isoform of the lncRNA HOTAIR in Claudin-low breast cancer cells attached to extracellular matrix., 2017, 11(12): 1698-1710.
[26] Yoneda R, Satoh Y, Yoshida I, Kawamura S, Kotani T, Atsushi. A genomic region transcribed into a long noncoding RNA interacts with the Prss42/Tessp-2 promoter in spermatocytes during mouse spermatogenesis, and its flanking sequences can function as enhancers., 2016, 83(6): 541-557.
[27] Ran M, Chen B, Li Z, Wu M, Liu X, He C, Zhang S, Li Z. Systematic identification of long non-coding RNAs in immature and mature porcine testes., 2016, 94(4):77.
[28] Cabili M N, Trapnell C, Goff L, Koziol M, Tazon-Vega B, Regev A, Rinn J L. Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses., 2011, 25(18):1915.
[29] Derrien T, Johnson R, Bussotti G, Tanzer A, Djebali S, Tilgner H, Guernec G, Martin D, Merkel A, Knowles DG, Lagarde J, Veeravalli L, Ruan X, Ruan Y, Lassmann T, Carninci P, Brown J B, Lipovich L, Gonzalez J M, Thomas M, Davis C A, Shiekhattar R, Gingeras T R, Hubbard T J, Notredame C, Harrow J, Guigó R. The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression., 2012,22:1775-1789.
[30] GLSS B S, DINGER M E. The specificity of long moncoding RNA expression, 2015, 1859(1): 16-22.
[31] López-Ortega O, Ovalle-García E, Ortega-Blake I, Antillón A, Chávez-Munguía B, Patiño-López G, Fragoso- Soriano R, Santos-Argumedo L. Myo1g is an active player in maintaining cell stiffness in B-lymphocytes., 2016, 73(5):258-268.
[32] Callegari S, Richter F, Chojnacka K, Jans DC, Lorenzi , Pacheu-Grau D, Jakobs S, Lenz C, Urlaub H, Dudek J, Chacinska A, Rehling P. TIM29 is a subunit of the human carrier translocase required for protein transport., 2016, 590(23):4147-4158.
[33] Kang Y, Baker M J, Liem M, Louber J, McKenzie M, Atukorala I, Ang C S, Keerthikumar S, Mathivanan S, Stojanovski D.Tim29 is a novel subunit of the human TIM22 translocase and is involved in complex assembly and stability.,5, (2016-08-14), 2016, 5: e17463.
[34] Yadav N, Lee J, Kim J, Shen J, Hu MC, Aldaz CM, Bedford M T. Specific protein methylation defects and gene expression perturbations in coactivator-associated arginine methyltransferase 1-deficient mice., 2003, 100(11): 6464-6468.
[35] Zhang Z, Nikolai B C, Gates L A, Jung S Y, Siwak E B, He B, Rice A P, O'Malley B W, Feng Q. Crosstalk between histone modifications indicates that inhibition of arginine methyltransferase CARM1 activity reverses HIV latency., 2017, 45(16): 9348-9360.
[36] Kishore A, Purcell R H, Nassiri-Toosi Z, Hall R A. Stalk-dependent and stalk-independent signaling by the adhesion G protein-coupled receptors GPR56 (ADGRG1) and BAI1 (ADGRB1)., 2015, 291(7):3385.
[37] Jeon T W, Yang H, Lee CG,Oh ST , Seo D , Baik I H , Lee E H , Yun I , Park KR , Lee Y H. Electro-hyperthermia up-regulates tumour suppressor Septin 4 to induce apoptotic cell death in hepatocellular carcinoma., 2016, 32(6):1-9.
[38] Ihara M, Kinoshita A, Yamada S, Tanaka H, Tanigaki A, Kitano A, Goto M, Okubo K, Nishiyama H, Ogawa O, Takahashi C, Itohara S, Nishimune Y, Noda M, Kinoshita M. Cortical organization by the septin cytoskeleton is essential for structural and mechanical integrity of mammalian spermatozoa., 2005, 8(3): 343-352.
[39] Kissel H, Georgescu M M, Larisch S, Manova K, Hunnicutt G R, Steller H. The Sept4 septin locus is required for sperm terminal differentiation in mice., 2005, 8(3) : 353-364.
[40] Shen C C, Cui X Y, He Y, Kang Y H, Yi C, Yang J L, Gou L T. High phosphorylation status of AKT/mTOR signal in DESI2- reduced pancreatic ductal adenocarcinoma., 2015, 21(2):267-272.
[41] Lin C, Yan H, Yang J, Li L, Tang M, Zhao X, Nie C, Luo N, Wei Y, Yuan Z. Combination of DESI2 and IP10 gene therapy significantly improves therapeutic efficacy against murine carcinoma., 2017, 8(34):56281-56295.
Changes of LncRNA Expression Profile in Spleen of Diarrhea and Non-diarrhea Individuals in F17 of Hu Sheep Lamb
HUANG SaiNan1, JIN ChengYan1, BAO JianJun2,WANG Yue1, CHEN WeiHao1, WU TianYi1, WANG LiHong1, LÜ XiaoYang1, GAO Wen1, WANG BuZhong3, ZHU GuoQiang4, DAI GuoJun1, SUN Wei1,5
(1College of Animal Science and Technology, Yangzhou University, Yangzhou 225009, Jiangsu;2Nanjing New Kyushu Agriculture and Animal Husbandry Technology Co., Ltd., Nanjing 210000;3Jiangsu Source Ecological Agriculture Co., Ltd., Taizhou 225300, Jiangsu;4College of Veterinary Medicine, Yangzhou University, Yangzhou 225009, Jiangsu;5Joint Laboratory of International Cooperation in Agriculture and Agricultural Products Safety of the Ministry of Education, Yangzhou University, Yangzhou 225009, Jiangsu)
【Objective】The objective of this study was to investigate the effect of lncRNA on anti-diarrhea in sheep by screening lncRNA differentially expressed inF17 fimbriae non-diarrhea and diarrhea sheep spleen. 【Method】 In this study, individuals with non-diarrhea and diarrhea were obtained by oral administration ofF17 strain to Lake Lamb. The success of the challenge was verified by using intestinal counts and pathological sections of the lambs. A cDNA library of spleen from lambs in non-diarrhea group and diarrhea group was constructed and sequenced by using Illumina HiSeq 2500 platform. Functional description and cell pathway analysis of differentially expressed transcripts were performed by Gene Ontology (GO) and KEGG Pathway enrichment analysis by using FPKM method. The expression levels of lncRNA and mRNA transcripts were screened by high-throughput sequencing technology RNA-seq for differential expression of lncRNA in spleens of non-diarrhea and diarrhea individuals; then, Quantitative PCR was used to detect spleen tissues in non-diarrhea and diarrhea lambs. The expression levels of differentially expressed (DE) lncRNA and DE mRNA were used to verify the role of screened DE lncRNA in the non-diarrhea group. 【Result】 After oral administration ofF17 strain, there were two phenotypes of non-diarrhea and diarrhea. The number of bacteria in the intestine of the diarrhea group was significantly higher than that in the non-diarrhea group (<0.05), and the jejunal mucosa of the diarrhea group appeared different degrees of damage, dull color, part of the small intestine villi off. We used RNA-seq to screen 34 DE lncRNAs and 703 DE mRNAs in non-diarrhea and diarrhea lamb spleens. A total of 12 DE lncRNA and DE mRNA were randomly selected and verified by q-PCR. Relative expression levels in the diarrhea and non-diarrhea lambs were found to be consistent with RNA-seq results. The comparison between DE lncRNA and GO database by GO and KEGG pathway enrichment analysis indicated that a total of 34 lncRNAs were annotated and classified into 302 functional subclasses. There were more than one functional subclass of lncRNA, such as sheep protein binding (GO: 0005515), nuclear (GO: 0005634), poly (A) RNA binding (GO: 0044822), cytoplasm (GO: 0005737), tissue remodeling (GO: 0048771), regulation of endopeptidase activity (GO: 0052548) ), 6-phosphate fructose-2-kinase/fructose-2,6-bisphosphatase complex (GO: 0043540), phosphatidylinositol phosphorylation (GO: 0848654), fructose-2, 6.2-phosphite 2-phosphatase activity (GO: 0004331) and calcium-dependent phospholipase C activity (GO: 0050429), while the remaining functional subclasses had less lncRNA distribution. The alignment of DE lncRNA with the KEGG pathway database indicated that a total of 34 lncRNAs were annotated and classified into 149 KEGG pathways, the sheep thyroid hormone signaling pathway (path: ko04919), Spliceosome (path: ko03040), white blood cell cross Endothelial migration (path: ko04670), neurotrophin signaling pathway (path: ko04722), lysosome (path: ko04142), MAPK signaling pathway-pathway (path: ko04011), sphingolipid signaling pathway (path: ko04071), phagocytosis the body (path: ko04145), oxidative phosphorylation (path: ko00190) and other 9 KEGG pathways had more lncRNAs, while the remaining KEGG pathways had less lncRNA distribution. Through lncRNA-mRNA interaction network analysis, we found six co-expressed genes: MYO1G, TIMM29, CARM1, ADGRB1, SEPT4, and DESI2. 【Conclusion】 This study explored the expression profile of lncRNA in the spleen of non-diarrhea and diarrhea lambs for diarrhea. It was found that lncRNA differentially expressed in the spleen of non-diarrhea and diarrhea lambs, which helped to find out how lambs resist diarrhea and provided a scientific basis for lambs to resist diarrhea.
F17; lncRNA; Lake lamb
10.3864/j.issn.0578-1752.2019.07.015
2018-09-18;
2018-12-03
国家自然科学基金(31872333)、科技部家养动物平台项目、江苏省重点研发计划(现代农业)项目(BE2018354)、江苏省农业重大新品种创制项目(PZCZ201739)、江苏省农业科技自主创新项目(CX(18)2003)、江苏高校优势学科建设工程资助项目、江苏省高校自然科学研究重大项目(17KJA230001)、江苏省六大高峰人才项目和扬州大学研究生创新工程项目(XKYCX17_060、SJCX18_0804)
黄赛男,E-mail:1481258911@qq.com。金澄艳,E-mail:1342315339@qq.com。黄赛男和金澄艳为同等贡献作者。通信作者孙伟,E-mail:dkxmsunwei@163.com
(责任编辑 林鉴非)

