花生膜联蛋白基因家族成员的结构和表达分析
王慧敏 李新国 万书波 张智猛 丁 红 李国卫 高文伟,*彭振英,,*
花生膜联蛋白基因家族成员的结构和表达分析
王慧敏1李新国2万书波3张智猛4丁 红4李国卫2高文伟1,*彭振英1,2,*
1新疆农业大学, 新疆乌鲁木齐 830052;2山东省农业科学院生物技术研究中心 /山东省作物遗传改良与生态生理重点实验室, 山东济南 250100;3山东省农业科学院, 山东济南 250100;4山东省花生研究所, 山东青岛 266100
植物膜联蛋白(annexin)是一类钙依赖性磷脂结合蛋白, 参与调控植物代谢和生长发育并协同调节抗旱、耐盐等多种抗逆反应, 其结构在不同植物中具有物种特异性。为系统分析花生膜蛋白基因家族, 对30个花生annexin (annexin of,)基因进行了生物信息学分析。结果表明 30个不均匀分布在13条染色体上, 其中A、B基因组中各有13个和17个。含有2~8个内含子, 但大多数含有5~6个内含子。聚类分析表明, 植物annexin聚类关系比较复杂, 低等植物、单子叶、双子叶植物annexin间隔分布, AnnAhs穿插其中, 分布于各个分支中; 但在各个小分支中, AnnAhs 基本上都与双子叶植物annexin聚在一起, 其中与大豆、苜蓿、向日葵亲缘关系较近, 其次是拟南芥; 但个别AnnAhs与单子叶植物和低等植物annexin聚在一起。30个AnnAhs均无跨膜结构域, 其中有16个AnnAhs定位于细胞质, 其余定位不明确。对可变剪切分析显示, 仅有11个发生可变剪切事件, 占38%; 根中发生的最多, 其次是叶中, 种子中最少。根据表达谱数据分析发现,在seed2和根中表达量较高, 其次是seed1, 在叶中表达量较低。本文可为花生抗性育种提供一定的理论支撑。
花生; 膜联蛋白; 基因结构分析; 可变剪切分析; 表达模式分析
膜联蛋白(annexin)是一类钙依赖性磷脂结合蛋白, 广泛存在于除酵母外的真核生物细胞中, 在进化上属于保守的多基因家族[1]。来源于脊椎动物、无脊椎动物、真菌和单细胞真核生物、植物和原生生物的 annexin被分为 A、B、C、D、E五大类[2], 动物annexin属于A类和B类, 参与细胞中的胞饮、胞吐、细胞骨架、离子通道、细胞增殖、细胞凋亡等多种重要的功能调节及多种信号调节[3-4]。植物annexin属于D类, 约占植物总蛋白含量的0.1%[5], 与动物annexin在分子量、氨基酸序列及钙离子与磷脂结合的能力上, 具有较高的同源性[2]。
目前, 动物annexin的研究主要集中在肺癌[6]、胃癌[7]、卵巢癌[8]、结直肠癌(CRC)[5]等相关疾病上, 如研究annexin在肿瘤细胞中的表达水平及其与临床特征的相关性[6]。ANXA3与肿瘤细胞的化疗药物有关, 对新药开发与新疗法探究有重要意义[9]; Annexin A2能够激活纤溶系统, 调控细胞骨架和重肌动蛋白, 在肿瘤侵袭和转移中具有非常重要的作用[10]。
植物annexin研究起步晚于动物, 但是近几年取得了初步进展。研究表明, 植物annexin参与了细胞多个生命过程, 对许多物质的合成途径具有重要的调节作用。如小麦在小麦花药中特异表达, 但是在温敏核不育系中不能被低温诱导表达, 表明特异性下调与小麦雄性不育具有相关性[11]; 棉花影响棉纤维的伸长, 并参与次生细胞壁的生物合成[12]。植物基因家族庞大, 其家族成员各自具有不同的功能分工和时空表达模式。植物的表达具有组织特异性, 在植物生长的各个阶段、不同器官、组织中的表达模式各有不同, 并且参与植物代谢和生长发育的调节过程[13]。拟南芥中8个基因在各组织部位的表达水平各不相同, 其中只在幼苗下胚轴表达, 而主要在幼苗胚轴和花丝中表达[14]。
Annexin亚细胞定位的多样性也反应出其功能的多样性, 其定位与胞质中的Ca2+浓度、细胞所处pH值以及外界环境有关[15-16]。Annexin通常为胞质蛋白, 具有可溶型和结合型两种形式。Annexin常常和液泡、细胞核、质膜以及高尔基体等结合在一起[17-19]。Annexin也能够可逆地与细胞骨架组成成分或介导细胞和胞外基质间互作的蛋白结合。褚翠萍等[16]研究表明, 拟南芥中annexin2可能通过微丝骨架调节与介导的囊泡运输参与细胞分泌活动。植物annexin的表达受各种生物及非生物胁迫的影响, 且逆境胁迫会影响annexin的亚细胞定位。欧洲甜瓜受到机械损伤时, 薄壁细胞中的annexin 会从胞质中重新定位于质膜上[20]。
花生是世界范围内广泛种植的油料和经济作物之一, 在国民经济占有重要地位。目前关于花生的研究多集中在产量和品质上, 而关于抗逆性研究进展缓慢。早期研究主要集中在抗逆性综合评价及抗逆种质筛选上, 而关于抗性机制研究相对较少。目前花生全基因组测序工作已经完成[21], 为系统研究花生的功能基因奠定了基础。本课题组王靖从花生“鲁花14”中克隆了2个花生基因,和, 并用烟草进行了耐盐性检验, 发现在盐胁迫下表达下调[22]。何美敬等[23]从花生中克隆获得6个基因, 发现它们在盐、旱、重金属、低温和激素处理下表达量均发生了改变。然而到目前为止, 有关花生基因在非生物胁迫中的作用机制还没有得以系统研究。本文通过对进行全基因组生物信息学分析, 并利用转录组数据分析其表达模式和可变剪接, 以期为深入研究其功能和调控机制奠定基础。
1 材料与方法
1.1 AnnAh全基因组检索以及其他物种annexin获得
运用“annexin”关键词在花生基因组数据库(https://www.peanutbase.org/)中搜索, 获得30个家族基因(表1), 命名为(of)。运用“annexin”关键词在NCBI (https://www.ncbi.nlm.nih.gov/)上搜索其他物种基因并下载其蛋白序列。搜索物种为拟南芥(, Ath)、大豆(, Gma)、苜蓿(, Mtr)、向日葵(, Han)、谷子(Sit)、水稻(, Osa)、玉米(, Zma)、卷柏(, Smo)、伪矮海链藻(, Tps)、三角褐指藻(Ptr)、绿色鞭毛藻(, Olu)、水蕨(, Cri)和小立碗藓(, Ppa), 对以上物种名缩写为大写属的首字母加小写种的前2个字母。命名方法为其蛋白序列号加物种名缩写(图1)。
1.2 AnnAh生物信息学分析
利用Mega5.0软件构建annexin蛋白序列进化树, 构建方法为邻近法。用GSDS (http://gsds.cbi.pku.edu.cn/)在线软件分析基因结构。利用TMHMM (http://www.cbs.dtu.dk/services/TMHMM/)在线软件分析AnnAhs跨膜结构域。在NCBI的Protein BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)上进行AnnAhs蛋白序列完整性分析。利用MEME (http://meme-suite.org/)和NCBI的Conserved domains (https://www.ncbi.nlm.nih. gov/Structure/cdd/wrp)在线软件分析AnnAhs保守结构域。利用Softberry (http://linux1.softberry.com/berry)在线软件进行AnnAhs亚细胞定位预测。
1.3 AnnAhs的表达模式和可变剪切分析
分别取花生品种“丰花1号”根、叶、果针入土30 d种子(seed1)、果针入土50 d种子(seed2)材料提取总RNA构建cDNA文库, 用链特异性测序方法测序。目前已将原始测序数据上传至NCBI数据库(编号为PRJNA354652)。用Cufflinks软件(http://cufflinks. cbcb.umd.edu/)计算各基因或转录本的表达丰度,用FPKM 来表示[24]。根据各的FPKM值, 用HemI软件进行表达模式分析。用ASprofile[25]软件对的可变剪切分别进行分类和数量统计。预测的可变剪切形式为TSS, 即转录起始位点可变剪切; TTS为转录终止位点可变剪切; SKIP为外显子跳跃; IR为内含子滞留; AE为可变外显子5'或3'端可变剪切。
2 结果与分析
2.1 AnnAh在染色体上的分布
从花生基因组中共检索到30个基因, 其中有12个序列不完整(表1)。它们不均匀地分布在13条染色体上, 其中A基因组中有13个, B基因组中17个。在Araip.B08、Aradu.A08、Araip.B10最多, 各有4个; 在Aradu.A10、Araip.B03各有3个; 在其他8条染色体上分布较少, 分别有1~2个。
2.2 植物annexin聚类分析
将AnnAhs与其他植物annexin构建进化树(图10)显示, 植物annexin聚类关系比较复杂, 低等植物、单子叶、双子叶植物annexin间隔分布, AnnAhs穿插其中, 分布于各个分支中, 显示出annexin进化关系的复杂性。在一些次级分支中, 单、双子叶植物annexin聚在一起, 低等植物annexin聚为另一分支; 而在有些小分支中, 高等植物annexin直接和低等植物annexin聚在一起, 如玉米NP_001266716.1和绿色鞭毛藻XP_001420377.1聚在一起, 显示出二者的高度相似性。

表1 Anneixn家族基因
(续表1)

基因名称Gene name染色体位置Chr. position氨基酸个数Amino acid number亚细胞定位Subcellular localization蛋白完整性Protein integrityAnnexin结构域Annexin domain重复序列Repetitive sequence其他结构域Other domains Aradu.MBZ2MAradu.A10333-完整Complete220 Araip.1LR8IAraip.B0289C不完整Incomplete220 Araip.KR6F4Araip.B03323-完整Complete222 Araip.X0F2SAraip.B03317-完整Complete440 Araip.CCM9GAraip.B03286-不完整Incomplete430 Araip.RGY04Araip.B04142C不完整Incomplete110 Araip.R8WRMAraip.B04207-不完整Incomplete320 Araip.HD6QLAraip.B05316C完整Complete440 Araip.0MR1XAraip.B05315C完整Complete440 Araip.FX5SIAraip.B07316C完整Complete440 Araip.J58EQAraip.B08135C不完整Incomplete220 Araip.YGP4JAraip.B08315-完整Complete330 Araip.6KP6UAraip.B08295-完整Complete330 Araip.Y8EDRAraip.B08321-完整Complete440 Araip.X9BIGAraip.B10317C完整Complete440 Araip.Z0Q6QAraip.B10311C完整Complete430 Araip.WA456Araip.B10315C完整Complete430 Araip.RPP1MAraip.B10364-完整Complete220 Aradu.V26BDAradu.A03279C完整Complete440 Aradu.86DERAradu.A03286-不完整Incomplete430
C: 细胞质; -: 其他定位。
C: cytoplasm; -: other localization.
在各个小分支中, AnnAhs基本上都与双子叶植物annexin聚在一起, 其中与大豆、苜蓿、向日葵亲缘关系较近, 其次是拟南芥。个别AnnAhs与单子叶植物玉米和谷子聚在一起, 如Araip.WA456与玉米XP_008656201.2聚在一起, Araip.R8WRM与谷子XP_004986983.2聚在一起。这些分析结果显示出AnnAhs家族成员的复杂性。
2.3 AnnAhs基因结构分析
由于、.和.序列太短, 故只分析其余27个的基因结构(图2)。分析结果表明,基因序列中至少有2个内含子, 最多有8个内含子, 多数含有5~6个内含子。.、.、.和.内含子特别长, 而其外显子序列与其他基因相差并不大, 保守性较强。
27个中有9对同源基因, 同源基因间具有类似的基因结构(图2)。如和.为一对同源基因, 它们都具有6个外显子, 但是第3个内含子长度差异较大。.和.分别具有4个和3个外显子, 如果.第2个内含子发生了滞留, 则会产生一个大外显子, 该大外显子与.第2个外显子大小相同。
2.4 AnnAhs跨膜结构域与保守结构域分析
对AnnAhs进行跨膜结构域分析, 发现这30 个AnnAhs均无跨膜结构域, 表明它们均为胞质可溶性蛋白。
图3显示, 在motif2中有一个保守“isoleucine- arginine-isoleucine” motif, 即IRI基序[26], 27个AnnAhs均有IRI基序; 在motif4中有一个GXGT基序[27], 15个AnnAhs含有GXGT基序。
利用NCBI中Conserved domains 进行分析(表1), 发现大多数AnnAhs有4个annexin结构域, 即4个功能结构域[22]。Aradu.SCC75、Aradu.23XWK、Aradu.N8MUP、Araip.YGP4J、Aradu.4J11T、Araip. 6KP6U、Aradu.KJ1YM和Araip.R8WRM各有3个, Aradu.MBZ2M、Araip.RPP1M、Araip.KR6F4各有2个。另外, Araip.KR6F4还含有2个不同的结构域, 即ALDH_F10_BADH结构域和Ribosomal_L18结构域。前者属于ALDH-SF超家族, 即醛脱氢酶家族, 具有氧化还原酶活性。后者是一个核糖体蛋白, 属于L18家族, 在细胞中与RNA分子的核心结构域结合, 以发挥功能。Araip.KR6F4有此2个结构域说明该基因可能具有这方面的功能。

图1 植物annexin蛋白聚类分析
红色: 双子叶植物; 绿色: 单子叶植物; 黑色: 低等植物; 黑色圆点: AnnAhs。
Red: dicotyledonous; green: monocotyledonous; black: lower plants; black dot: AnnAhs.
2.5 AnnAhs亚细胞定位预测分析
对30个AnnAhs进行亚细胞定位预测分析显示, 有16个AnnAhs定位于细胞质(表1), 分别是Aradu.3L5NK、Araip.HD6QL、Aradu.WYZ5E、Araip.0MR1X、Aradu.SCC75、Araip.X9BIG、Aradu.9BC7H、Araip.FX5SI、Aradu.23XWK、Aradu.IZQ3Z、Araip.Z0Q6Q、Araip.WA456、Araip.J58EQ、Araip.RGY04、Araip.1LR8I和Aradu.V26BD, 其余14个定位不明确。
2.6 AnnAhs可变剪切分析
根据“丰花1号”转录组数据分析的可变剪接情况(表2)表明, 有11个具有可变剪接现象, 占38%。共检测到TSS、TTS、AE和ES四种可变剪接类型, 分别占15.4%、30.8%、30.8%和23.0%。没有发现IR类型。在根中发生的可变剪切数目最多, 其次是叶中, 种子中最少, 显示出的可变剪接在根系发育以及在应对环境胁迫中的重要作用。在4个组织中均发生了可变剪接,.在3个组织中均发生了可变剪接, 其余9个只在1~2个组织中发生了可变剪接。

图2 AnnAhs基因结构分析
因部分无UTR, 故均用ORF进行基因结构分析。
Some ofhave no UTRs, so theORF is used to analyze the gene structures.
2.7 AnnAhs表达模式分析
利用“丰花1号”转录组数据中各基因的FPKM值对其表达模式进行分析。其中.、.、.、.、.、.、.在4个组织中FPKM值均为0, 故不予分析。其余23个基因按照FPKM值作图(图4)表明, 各在不同组织中的表达量存在显著差异。总体而言, 各在seed2和根中表达量较高, 其次是seed1, 在叶中表达量较低。其中.和在种子中特异性高表达, 说明二者对种子发育的重要性。各基因在根中的表达量显著高于叶中, 显示出在根系发育中的重要作用。
3 讨论
植物anneixn是一个庞大而保守的基因家族[28], 不仅在植物的生长发育中具有重要功能, 还参与抗寒、抗旱及耐盐等多种反应[29]。目前关于植物annexin应对非生物胁迫的研究取得一定进展, 但是其作用机制研究还比较少。本文主要利用花生基因组和转录组数据对进行全基因组系统分析, 可为研究的生理机制与功能提供一定的理论基础。
在聚类分析中发现, 植物annexin聚类关系比较复杂, 各类annexin间隔分布, AnnAhs穿插其中。AnnAhs基本上都与双子叶植物annexin聚在一起, 个别AnnAhs与单子叶植物聚在一起, 说明AnnAhs家族基因的复杂性。Jami等[27]对149个植物annexin构建进化树, 并将其分成9个组。并发现, annexin多基因家族似乎随着基因组的复杂而扩大, 可能是与基因复制相关, 而这种扩增意味着该家族可能在植物应对环境胁迫中具有重要作用[27,30]。花生是异源四倍体, 遗传背景复杂, 在整个基因组中含有30个annexin基因, 比其他植物中annexin个数都要多, 可能是在进化过程中通过复制形成的, 类似情况在苔藓、大豆中也有报道[30-31]。
在保守结构域分析中发现AnnAhs都含有 IRI基序, 大部分AnnAhs含有GXGT基序。有研究认为 IRI 可以促成植物annexin与F-actin的结合, F-actin在植物生长发育过程中参与许多重要生命活动, 如细胞分泌、细胞分裂、细胞极性生长、细胞形态维持及物质运输等[32]。含有IRI基序说明这部分annexin可能是通过F-actin而起作用的。在拟南芥annexin家族的8个成员中有3个(AtANN3、AtANN4和AtANN4)不含有IRI基序[33], 说明并不是所有成员都具有IRI 基序, 这部分annexin可能具有其他的调控方式。Araip.KR6F4比较特殊, 还含有ALDH _F10_BADH和Ribosomal_L18结构域。这种现象在annexin蛋白家族中少见, Araip.KR6F4的功能也需要进一步验证与探究。

图3 AnnAhs的保守结构域分析
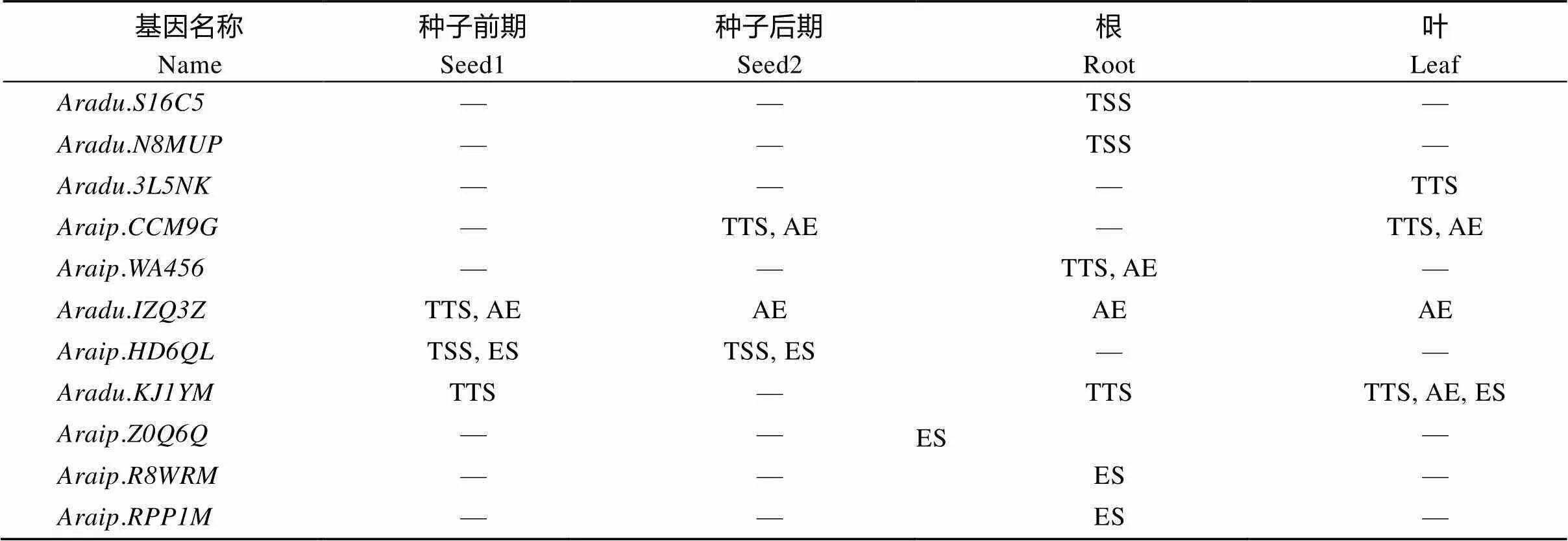
表2 AnnAhs可变剪切分析
TSS: 转录起始位点替换; TTS: 转录终止位点替换; AE: 外显子替换 5'或3'端剪接; ES: 外显子跳跃。
TSS: transcription start site; TTS: transcription terminal site; AE: alternative exon ends (5', 3', or both); ES: exon skipping.

图4 AnnAhs表达模式分析
亚细胞定位对研究蛋白质功能具有重要作用[28,34], 植物annexin在细胞质、质膜、内膜系统以及核膜均有定位, 但是定位于细胞质中的较多些, 可能与其家族蛋白基因在膜功能中起重要作用相关[15]。而且植物annexin的亚细胞定位与细胞质中的Ca2+浓度、细胞所处pH值以及外界环境均有关系[15-16]。拟南芥Ann At2、Ann At3和Ann At6定位于细胞质和细胞核; Ann At5 定位于过氧化物酶体; Ann At7定位于细胞质[33-34]。棉花中AnnGh3和AnnGh6定位于细胞质[35]; MeAnn1定位于木薯体胚细胞的细胞质和细胞核[36]。小麦中的annexin在低温胁迫下, 以p39和p22.5的形式定位于质膜[37]。蒺藜苜蓿MtANN1定位于核膜[38]。芹菜VCaB42在 Ca2+存在时定位于液泡膜[19]。本研究中预测的AnnAhs大部分定位在细胞质中, 少数部分定位不明确, 因此需要进一步用实验来验证。这些结果暗示Annexin成员在不同的细胞生理过程和不同的环境条件下发挥不同作用。
可变剪接是调控真核生物转录组和蛋白质组多样性的重要分子机制, 也是一种在转录后 RNA 水平调控基因表达的重要机制, 在生物发育中具有重要作用[39]。本研究发现有11个在不同组织中发生了可变剪切, 这种现象在苔藓、拟南芥和水稻的annexin中也有报道[27]。据统计, 单子叶植物annexin发生AS的情况较双子叶植物多。水稻中2个annexin的转录本(Os09g23160, Os02g51750)发生了AS, 而玉米种至少有5个annexin基因发生AS。在双子叶植物中, 拟南芥annexin (At5g65020, AnnAt2)发生AS。当然, 关于AS调控annexin的作用机制仍需进一步研究。
表达模式对基因的功能研究具有重要作用。植物annexin在不同组织中的表达模式具有显著差异。褚翠萍[14]对拟南芥中8个annexin基因的表达模式分析表明, Ann At1、Ann At6、Ann At7在幼苗、花和种子中都有表达; Ann At2主要在子叶、胚轴和花萼中表达; Ann At3主要在叶片、幼苗下胚轴和花萼中表达; Ann At4 只在幼苗下胚轴表达; Ann At5主要在幼苗胚轴和花丝中表达; Ann At8 在叶片、花萼和种子中表达; Ann At3、Ann At6、Ann At7、Ann At8 在保卫细胞中表达[34]。这种表达模式的差异反应了不同annexin成员功能分工的差异。又有研究表明, 许多annexin是在根中发挥功能的。例如玉米annexin能够加强玉米根冠原生质体的胞吐作用[40]; Annexin P35在豌豆胚芽和根部的幼嫩区液泡细胞和根冠外围分泌粘液细胞中含量最高[41]; 烟草Ntann12能够调节根的极性运输[42];在根毛发育中可被细胞分裂素诱导表达[43]。本研究表明, 除了7个没有表达外, 剩余23个的表达模式各不相同, 但是总体而言它们在seed2和根中的表达水平较高, seed1次之, 而在叶片中的表达量最低。这说明不同成员的功能分工是不同的, 如.和.在seed1和seed2特异性高表达, 显示出二者对种子发育的重要性;.在根中的表达量最高, 表明其在根发育中具有重要作用; 不同在种子发育不同阶段所起的作用也是不同的, 随着种子发育的日趋成熟, 种子内部各个器官逐渐分化出来, 越来越多的参与其中, 表达量也随之增加。
4 结论
本文利用生物信息学方法探讨了花生膜联蛋白基因家族成员的基因结构、跨膜结构域、保守结构域与亚细胞定位的特点, 并利用转录组数据对其进行了可变剪接以及表达模式分析, 为以后进行花生膜联蛋白基因的克隆与功能验证提供了一定的理论基础。
[1] Ahmed I, Yadav D, Shukla P, Kirti P B. Heterologous expression ofannexin, AnnBj2 confers salt tolerance and ABA insensitivity in transgenic tobacco seedlings.,2018, 18: 569–529.
[2] 李瑞梅, 王雨晴, 张帆, 袁帅, 耿梦婷, 姚远, 刘姣, 段瑞军, 符少萍, 胡新文, 郭建春. 植物膜联蛋白(Annexin)的研究进展. 基因组学与应用生物学, 2017, 36: 2128–2136. Li R M, Wang Y Q, Zhang F, Yuan S, Geng M T, Yao Y, Liu J, Duan R G, Fu S P, Hu X W, Guo J C. Research progress on plant annexin., 2017, 36: 2128–2136 (in Chinese with English abstract).
[3] 叶卫华. 膜联蛋白A7(annexin A7)在胃癌凋亡中作用的研究. 河北医科大学博士学位论文, 河北石家庄, 2017. Ye W H. The Role of A7(Annexin A7) in Apoptosis of Gastric Carcinoma. PhD Dissertation ofHebei Medical University, Shijiazhuang, Hebei, China, 2017 (in Chinese with English abstract).
[4] Galvão I, Vago J P, Barroso L C, Tavares L P, Queiroz-Junior C M, Costa V V, Carneiro F S, Ferreira T P, Silva P M, Amaral F A, Sousa L P, Teixeira M M. Annexin A1 promotes timely resolution of inflammation in murine gout., 2017, 47: 585–596.
[5] Konopkapostupolska D, Clark G. Annexins as overlooked regulators of membrane trafficking in plant cells., 2017, 18: 863.
[6] Hua K L, Li Y, Zhao Q, Fan L Q, Tan B B, Gu J B. Downregulation of Annexin A11 (ANXA11) inhibits cell proliferation, invasion, and migration via the AKT/GSK-3β pathway in gastric cancer.,2018, 24: 149–160.
[7] Pi F, Zhang H, Li H, Thiviyanathan V, Gorenstein D G, Sood A K, Guo P. RNA nanoparticles harboring annexin A2 aptamer can target ovarian cancer for tumor-specific doxorubicin delivery.(UK), 2017, 13: 1183–1193.
[8] Yu S, Bian H, Gao X, Gui L. Annexin A9 promotes invasion and metastasis of colorectal cancer and predicts poor prognosis., 2018, 41: 2185–2192.
[9] 杜锐. Annexin A3 (ANXA3)对乳腺癌的调控研究与乳腺癌患者来源移植瘤模型的建立. 中国科学技术大学博士学位论文, 安徽合肥, 2017. Du R. The Role of Annexin A3 in Regulating Breast Cancer and the Establishment of Breast Cancer PDX Model. PhD Dissertation of University of Science and Technology of China, Hefei, Anhui, China, 2017 (in Chinese with English abstract).
[10] 刘琰. Annexin A2对卵巢癌生物学行为的影响及机制研究. 郑州大学博士学位论文, 河南郑州, 2017. Liu Y. The Effects and Mechanism of Annexin A2 on Biological Behavior of Ovarian Cancer. PhD Dissertation of Zhengzhou University, Zhengzhou, Henan, China, 2017 (in Chinese with English abstract).
[11] Xu L, Tang Y M, Gao S Q, Su S C, Hong L, Wang W W, Fang Z F, Li X Y, Ma J X, Quan W, Sun H, Li X, Wang Y B, Liao X Z, Gao J G, Zhang F G, Li L, Zhao C P. Comprehensive analyses of the annexin gene family in wheat., 2016, 17: 415.
[12] Zhang F, Jin X X, Wang L K, Li S F, Wu S, Cheng C Z, Zhang T Z, Guo W Z. A cotton annexin affects fiber elongation and secondary cell wall biosynthesis associated with Ca2+influx, ROS homeostasis, and actin filament reorganization., 2016, 171: 1750–1770.
[13] 楚璞. 莲膜联蛋白的鉴定及其在种子耐热性和活力中的功能研究. 中山大学博士学位论文, 广东广州, 2011. Chu P. Identification and Functional Analysis of aAnnexin Involved in Seed Thermotolerance and Vigor. PhD Dissertation of Sun Yat-Sen University. Guangzhou, Guangdong, China, 2011 (in Chinese with English abstract).
[14] 褚翠萍. 拟南芥膜联蛋白的基因表达、亚细胞定位和AnnAt3响应非生物胁迫的初步研究. 山东农业大学硕士学位论文, 山东泰安, 2012. Chu C P. Gene Expression, Subcellular Localization of Arabidopsis Annexins and Abiotic Stress Response of Ann At3. MS Thesis of Shandong Agricultural University, Tai’an, Shandong, China, 2005 (in Chinese with English abstract).
[15] 何美敬, 穆国俊, 侯名语, 陈焕英, 崔顺立, 杨鑫雷, 刘立峰. 植物膜联蛋白的结构及功能研究进展. 西北植物学报, 2013, 33: 2567–2574. He M J, Mu G J, Hou M Y, Chen H Y, Cui S L, Yang X L, Liu L F. Advances in structure and function of plant annexins.2013, 33: 2567–2574 (in Chinese with English abstract).
[16] 褚翠萍, 任秋萍, 孔兰静, 王秀玲. 拟南芥膜联蛋白2的亚细胞定位研究. 中国细胞生物学学报, 2017, 39: 873–880. Zhu C P, Ren Q P, Kong L G, Wang X L. The subcellular localization of annexin 2 in Arabidopsis., 2017, 39: 873–880 (in Chinese with English abstract).
[17] Clark G B, Dauwalder M, Roux S J. Purification and immunolocalization of an annexin-like protein in pea seedlings., 1992, 187: 1–9.
[18] Clark G B, Dauwalder M, Roux S J. Immunological and biochemical evidence for nuclear localization of annexin in peas., 1998, 36: 621–627.
[19] Seals D F, Parrish M L, Randall S K. A 42-kilodalton annexin-like protein is associated with plant vacuoles., 1994, 106: 1403–1412.
[20] Thonat C, Mathieu C, Crevecoeur M, Penel C, Gaspar T, Boyer N. Effects of a mechanical stimulation of localization of annexin-like proteins ininternodes., 1997, 114: 981–988.
[21] 郑昌林. 花生基因组中重复基因间基因置换的比较分析. 华北理工大学硕士学位论文, 河北唐山, 2017. Zheng C L. Comparative Analysis of Repetitive Gene Replacement in Peanut Genome. MS Thesis of North China University of Science and Technology, Tangshan, Hebei, China, 2017 (in Chinese with English abstract).
[22] 王靖. 花生膜联蛋白基因和的功能验证. 山东大学硕士学位论文, 山东济南, 2014.Wang J. Functional Research of Annexin GeneandfromL. MS Thesis of Shandong University, Jinan, Shandong, China, 2017 (in Chinese with English abstract).
[23] 何美敬, 杨鑫雷, 崔顺立, 穆国俊, 侯名语, 陈焕英, 刘立峰. 花生膜联蛋白基因(Annexin)家族的分子克隆和鉴定.中国作物学会学术年会论文集, 2014. He M J, Yang X L, Cui S L, Mu G J, Hou M Y, Chen H Y, Liu L F. Molecular cloning and identification of annexin gene family from peanut. In: Proceedings of the Crop Science Society of China2014 (abstract in Chinese).
[24] Trapnell C, Williams B A, Pertea G, Mortazavi A, Kwan G, van Baren M J, Salzberg S L, Wold B J. Transcript assembly and quantification by RNA Seq reveals unannotated transcripts and isoform switching during cell differentiation., 2010, 28: 511–515.
[25] Florea L, Li S, Salzberg S L.Thousands of exon skipping events differentiate among splicing patterns in sixteen human tissues., 2013, 2: 188.
[26] Clark G B, Dauwalder M, Roux S J. Immunolocalization of an annexin-like protein in corn., 1994, 14: 341–346.
[27] Jami S K, Clark G B, Ayele B T, Ashe P, Kirti P B. Genome-wide comparative analysis of annexin superfamily in plants., 2012, 7: 47801.
[28] 王立科, 张边江, 唐宁, 杨平, 周峰, 陈全战. 植物膜联蛋白基因研究进展. 分子植物育种, 2016, 14: 2060–2065. Wang L K, Zhang B J, Tang N, Yang P, Zhou F, Chen Q Z. Research advances of annexin genes in plants., 2016, 14: 2060–2065 (in Chinese with English abstract).
[29] Vogel C, Chothia C. Protein family expansions and biological complexity., 2006, 2: 48.
[30] Schmutz J,Cannon S B, Schlueter J, Ma J X, Mitros T, Nelson W, Hyten D L, Song Q J, Thelen J J, Cheng J L, Xu D, Hellsten U, May G D, Yu Y, Sakurai T, Umezawa T, Bhattacharyya M K, Sandhu D, Valliyodan B, Lindquist E, Peto M, Grant D, Shu S Q, Goodstein D, Barry K, Futrell-Griggs M, Abernathy B, Du J C, Tian Z X, Zhu L C, Gill N, Joshi T, Libault M, Sethuraman A, Zhang X C, Shinozaki K, Nguyen H T, Wing R A, Cregan P, Specht J, Grimwood J, Rokhsar D, Stacey G, Shoemaker R C, Jackson S A. Genome sequence of the palaeopolyploid soybean., 2010, 463: 178–183.
[31] Kandasamy M K, Burgos-Rivera B, McKinney E C,Ruzicka D R, Meagher R B. Class-specific interaction of profilin and ADF isovariants with actin in the regulation of plant development., 2007, 19: 3111–3126.
[32] Koehler P E, Odell G V. Factors affecting the formation of pyrazine compounds in sugara-mine reactions., 1970, 18: 895–898.
[33] Clark G B, Sessions A, Eastburn D J, Roux S J. Differential expression of members of the annexin multigene family in Arabidopsis., 2001, 126: 1072–1084.
[34] 李立奇, 万瑛. 蛋白质的亚细胞定位预测研究进展. 免疫学杂志, 2009, 25: 602–604. Li L Q, Wan Y. Advancement of predicting proteins in subcellular localization sites., 2009, 25:602-604 (in Chinese with English abstract).
[35] 李兵. 棉花()膜联蛋白基因克隆鉴定及功能研究. 华中师范大学博士学位论文, 湖北武汉, 2012. Li B. Molecular Characterization and Function of the Cotton Annexin Genes. PhD Dissertation of Central China normal University, Wuhan, Hubei, China, 2012 (in Chinese with English abstract).
[36] 王雨晴, 张帆, 李瑞梅, 胡新文, 郭建春. 木薯基因的克隆及表达分析. 分子植物育种, 2015, 13: 2477–2483. Wang Y Q, Zhang F, Li R M, Hu X W, Guo J C. Cloning and expression ofgene from cassava (Crantz)., 2015, 13: 2477–2483 (in Chinese with English abstract).
[37] Breton G, Vazqueztello A, Danyluk J, Sarhan F. Two novel intrinsic annexins accumulate in wheat membranes in response to low temperature., 2000, 41: 177–184.
[38] Carvalho-Niebel F D, Timmers A C J, Chabaud M, Defaux-Petras A, Barker D G. The Nod factor-elicited annexin MtAnn1 is preferentially localised at the nuclear periphery in symbiotically activated root tissues of., 2002, 32: 343–352.
[39] 章天骄. 可变剪接的生物信息数据分析综述. 生物信息学, 2012, 10: 61–64. Zhang T J. Review of bioinformation data analysis with alternative splicing., 2012, 10: 61–64 (in Chinese with English abstract).
[40] Wilkinson J Q, Lanahan M B, Conner T W, Klee H J. Identification of mRNAs with enhanced expression in ripening strawberry fruit using polymerase chain reaction differential display., 1995, 27: 1097–1108.
[41] 那杰, 杨怀义. 植物Annexins研究进展. 辽宁师范大学学报(自然科学版), 2004, 27: 464–467. Na J, Yang H Y. Aadvances in the study of plant annexins.(Nat Sci Edn), 2004, 27: 464–467 (in Chinese with English abstract)
[42] Baucher M, Oukouomi L Y, Vandeputte O M, Bopopi J M, Moussawi J, Vermeersch M, Mol A, Jaziri M E, Homble F, Perez-Morga D.annexin expression is induced by auxin in tobacco roots., 2011, 62: 4055–4065.
[43] 巩子英, 宋雪, 陈功友, 朱家璧, 俞冠翘, 邹华松. 苜蓿膜联蛋白MtAnn3基因的鉴定及其在根毛发育中的功能. 科学通报, 2012, 57: 431–437. Gong Z Y, Song X, Chen G Y, Zhu J B, Yu G Q, Zou H S. Identification ofgene of alfalfa and its function in root hair development., 2012, 57: 431–437 (in Chinese).
Structure and expression analysis of the members of peanut annexin gene family
WANG Hui-Min1, LI Xin-Guo2, WAN Shu-Bo3, ZHANG Zhi-Meng4, DING Hong4, LI Guo-Wei2,GAO Wen-Wei1,*, and PENG Zhen-Ying1,2,*
1Xinjiang Agricultural University, Wulumuqi 830052, Xinjang, China;2Research Center of Biotechnology, Shandong Academy of Agricultural Sciences / Shandong Key Laboratory of Crop Genetic Improvement and Ecology and Physiology, Jinan 250100, Shandong, China;3Shandong Academy of Agricultural Sciences, Jinan 250100, Shandong, China;4Shandong Peanut Research Institute, Qingdao 266100, Shandong, China
Annexin is a kind of calcium-dependent phospholipid binding proteins involved in the regulation of plant metabolism, growth and development, drought resistance and salt tolerance, and its structure is species-specific in different plants. In order to have a systematic analysis of thegene family of peanut, we identified 30genes from the peanut genome database, and analyzed their characteristics using bioinformatics method. Peanut annexin (annexin of,) genes were unevenly distributed on 13 chromosomes, with 13 in A genome and 17 in B genome. There were 2 to 8 introns in the, with 5 to 6 introns in most. Phylogenetic analysis showed that the clustering relationship was complex. The annexins of lower plant, monocotyledonous and dicotyledonous plants were distributed at interval, with AnnAhs inserting in each branch. However, in each small branch, AnnAhswere basically clustered with the dicotyledonous plant annexins, and close to soybean, alfalfa and sunflower, followed byArabidopsis; but severalAnnAhs were associated with monocotyledonous and lower plant annexins. All 30 AnnAhs had no transmembrane domain, and 16 of them were located in cytoplasm, and the others’ localization was uncertain. Results of alternative splicing (AS) analysis ofshowed that only 11experienced AS, which accounted for about 38% of all; the AS events occurred most in roots, followed by leaves, and the least in seeds. The expression level ofwas high in seed2 and root, followed by seed1, and lower in leaf. The comprehensive analysis ofcan provide some theoretical support for peanut resistance breeding.
peanut; annexin; gene structure analysis; alternative splicing analysis; expression pattern analysis
2018-04-19;
2018-10-08;
2018-11-05.
10.3724/SP.J.1006.2019.84056
彭振英, E-mail: pengzhenying2005@126.com; 高文伟, E-mail: gww0911@163.com
E-mail: whuiminyspa@163.com
本研究由山东省自然科学基金重大基础研究项目(植物根系耐盐、抗旱、耐瘠薄的调控机理), 山东省农业科学院农业科技创新工程项目(CXGC2018B05)和山东省自然科学基金项目(ZR2014YL043)资助。
This study was supported by the Major Basic Research Project of Shandong Natural Science Foundation, Shandong Academy of Agricultural Sciences Agricultural Science and Technology Innovation Project (CXGC2018B05), and Shandong Natural Science Foundation (ZR2014YL043).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20181101.1045.014.html

