甘蓝型油菜PEBP基因家族的鉴定与表达分析
荐红举 杨 博 李阳阳 杨 鸿 刘列钊 徐新福 李加纳
甘蓝型油菜基因家族的鉴定与表达分析
荐红举**杨 博**李阳阳 杨 鸿 刘列钊 徐新福 李加纳*
西南大学农学与生物科技学院 / 油菜工程研究中心 / 西南大学现代农业科学研究院, 重庆 400716
植物磷脂酰乙醇胺结合蛋白(phosphatidylethanolamine-binding protein, PEBP)基因对于控制开花时间具有重要作用。油菜作为世界上最重要的油料作物之一, 与模式植物拟南芥具有相似的开花习性。但有关油菜开花基因的功能研究相对较少。本研究利用拟南芥PEBP基因家族蛋白序列在油菜基因组内进行BlastP分析, 获得油菜PEBP家族成员, 并对其进行基因结构分析、motif预测、复制事件、进化树构建、选择压力分析和组织表达分析。结果表明, 共有26个油菜基因成员得到鉴定, 大部分成员含有4个外显子和3个内含子, motif-1和motif -2是PEBP成员的特征基序, 超过76.9%的成员属于片段复制扩增事件。进化树分析显示, PEBP分为3个亚家族, 基于转录组测序的组织表达数据表明油菜26个成员具有非常明显的组织表达特性。以上研究结果, 极大丰富了我们对甘蓝型油菜开花基因及调控模式的认识, 为进一步的分子育种提供了理论基础。
油菜; 开花基因; PEBP; 进化; 表达
开花是植物通过有性生殖传递后代的关键前提, 开花时间是影响作物产量和品质最重要的农艺性状之一。相比于其他性状, 开花时间对外界环境更加敏感。因此, 控制开花时间对于提高不同地域的作物产量具有重要意义。截至目前, 有关开花候选基因的解析主要集中在拟南芥等模式植物中。在拟南芥中, 超过300个开花基因得到鉴定[1], 这些基因根据其参与的过程主要分为六大途径, 即春化过程、光周期途径、外界温度、赤霉素途径、自发途径和内在因素[2-4]。虽然不同的基因参与不同的开花调控通路, 但各通路被几个关键基因协同调控, 如()、()、()和()[5]。
FT基因编码一个称为“促花素”的可以长距离移动的信号蛋白, 在开花调控中起着核心调控作用[6]。该基因属于植物磷脂酰乙醇胺结合蛋白PEBP (phosphatidylethanolamine-binding protein)基因, PEBP基因编码蛋白包含一个保守结构域(InterPro: IPR00891)。进化研究表明, 该蛋白家族分为3个亚家族, 即FT、TFL1 (TERMINAL FLOWER 1)和MFT (MOTHER OF FT AND TFL1)。其中, FT主要是诱导开花, TFL1抑制开花, 而MFT主要参与种子发育和萌发过程[7-9]。在拟南芥中,家族包含6个基因成员, 即、、()、()、()和[10]。其中,、和促进开花,、和抑制开花[11-13]。在拟南芥中, 2个基因,和是开花激活因子, 突变该基因将延迟开花[11,14]。在长日照环境中,和在叶子韧皮部的伴胞细胞中上调表达[15]。作为的旁系同源基因,在营养生长期的成熟顶端分生组织的内表皮中少量积累, 而开花之后, 其表达量显著下调[16-17]。TFL1蛋白是一个移动信号, 可从顶端分生组织的内表皮移动到外表皮[18]。虽然和在序列上非常相似, 都可以与FD (FLOWERING LOCUS D)互作来调控FD依赖的靶基因, 但在调控开花过程中的作用是相反的[19]。在高盐胁迫中, BFT可以通过调节光周期来适应外界胁迫[20]。MFT特异诱导下胚轴和根的转变, 其突变体表现为在种子萌发过程中对ABA的超敏反应, 而且在萌发的种子中,的表达直接受到ABA-INSENSITIVE3 (ABI3)和ABI5的调控, 同时, MFT通过反馈抑制ABI5的表达来促进胚的发育[21]。
相比于单个基因成员的研究, 在植物基因组范围内对的鉴定愈发重要且普遍家族相对较少。比如, 在大豆中有23个成员得到鉴定, 几乎是拟南芥基因数目的4倍[22], 其中GmFT2a和GmFT5a通过调控光周期途径来控制开花[23]。在单子叶模式植物水稻中, 有19个家族得到鉴定, 其中()和()被研究的最为透彻[24]。在短日照环境中, Hd3a促进水稻开花, 而的同源基因主要在被抑制时起到控制开花的功能[25]。在玉米的24个家族中, 只有跟拟南芥的功能最为相似[26]。
油菜, 作为世界上最重要的油料作物之一, 与模式植物拟南芥具有相似的开花习性。虽然有很多通过基因定位和高通量测序技术研究油菜开花基因或通路的报道, 但是稳定的QTL或者关键调控基因或因子尚未得到解析[27-28]。相比于QTL定位方法筛选候选基因, 以拟南芥基因功能作为参考, 并结合RNA-Seq和分子实验来筛选并鉴定开花相关基因, 然后通过全基因组比较分析作物开花候选基因是非常方便和准确的。这种方式已经在萝卜中得到运用[29]。通过这种方式还可以为进一步的功能鉴定以及开花通路调控因子提供候选基因。家族成员的进化分析在大豆、棉花、郁金香和小麦等作物中已有报道[10,29-32], 但油菜中尚无有关该基因家族的鉴定、进化和表达分析。本研究利用拟南芥PEBP家族成员的蛋白序列为参考, 通过BlastP方式, 获得甘蓝型油菜以及亲本种白菜和甘蓝的PEBP家族成员, 并分析其进化、选择压力以及组织表达, 可为油菜开花性状候选基因的研究提供理论参考。
1 材料与方法
1.1 序列的获得
从拟南芥数据库(https://www.arabidopsis.org/)中获取拟南芥基因的蛋白序列, 并以此序列在BRAD数据库(http://brassicadb.org/brad/index. php)中进行BlastP, 阈值E<10–5, 筛选甘蓝型油菜、白菜和甘蓝的同源基因, 并下载相应蛋白和CDS序列。
1.2 油菜PEBP基因结构、motif预测以及重复基因检测
根据油菜基因家族的基因组DNA及CDS序列, 并利用在线工具GSDS (http://gsds.cbi.pku.edu. cn/)分析其基因结构。利用在线工具MEME (http:// meme-suite.org/)预测油菜基因家族的保守结构元件, 并设置motifs数量为5个, 宽度为6~50。基因结构和motif结果均利用TBtools工具展示。用软件MCScanX 分析26个油菜基因家族的复制情况, 默认其参数。
1.3 序列比对及进化分析
利用Clustal W软件对获取的拟南芥、甘蓝型油菜、白菜和甘蓝基因组内的PEBP的序列进行完全比对。用MEGA 6.0软件, 通过邻接法(Neighbor Joining, NJ)构建基因分子进化树, 设BootStrap抽样次数为1000, 默认其余参数, 并用软件Figtree展示。
1.4 选择压力分析
用MEGA 6.0内置的Muscle对拟南芥和甘蓝型油菜之间的共线性基因对做codon比对[33], 然后用软件KaKs_Calculator 2.0计算共线性基因之间的选择压力[34]。
1.5 甘蓝型油菜PEBP家族成员的组织表达分析
利用课题组前期完成的不同发育时期的‘中双11’共111个组织器官的转录组数据(PRJNA358784)分析甘蓝型油菜的表达情况, 样品详细信息见附表1。对各成员的表达量取log2FPKM后利用MeV 4.9.0 (http://en.bio-soft.net/chip/MeV.html)绘制热图展示。
2 结果与分析
2.1 甘蓝型油菜、白菜和甘蓝PEBP基因家族成员筛选
为筛选并鉴定甘蓝型油菜及亲本种白菜和甘蓝基因组内基因成员, 利用拟南芥的6个PEBP蛋白序列在BRAD网站上进行BlastP分析(E<10–5), 分别获得26、12和11个甘蓝型油菜、白菜和甘蓝基因成员(表1)。值得一提的是, 在甘蓝基因组内没有发现亚家族同源基因, 可能在进化过程中被丢失。
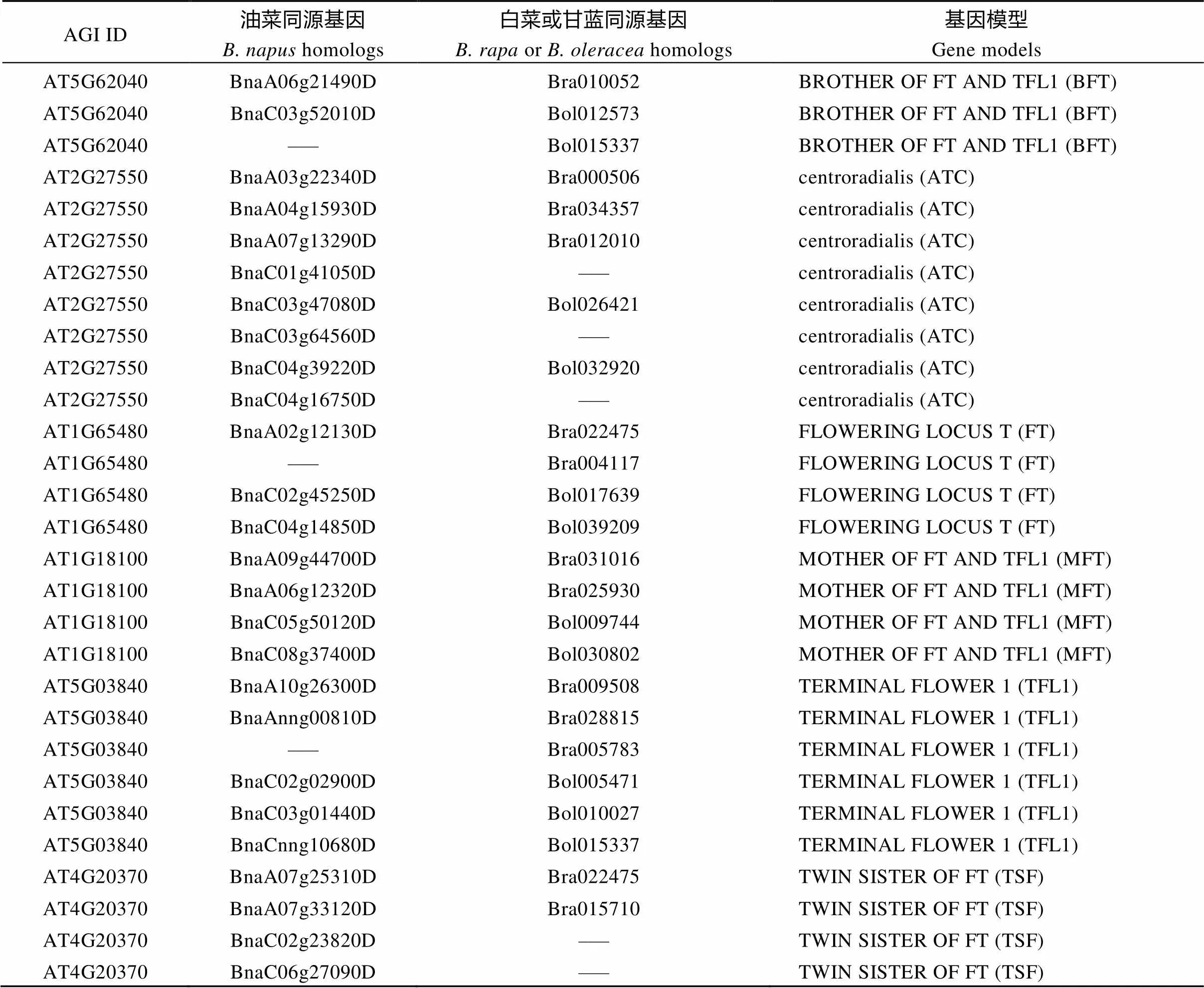
表1 利用BlastP方法获得油菜、白菜、甘蓝和拟南芥PEBP同源基因
2.2 基因结构与motif预测
基因结构决定其功能, 为进一步分析油菜基因家族的功能, 利用其CDS和基因组DNA序列分析其外显子-内含子结构, 并按照其进化树顺序展示(图1)。除了()和()含有3个外显子和2个内含子之外, 其余基因家族成员均含有4个外显子和3个内含子。Motif预测结果表明, Motif-1和Motif-2是基因家族的特征性motif, 除()和()只含有Motif-1和Motif-2外, 其余成员还含有Motif-3、Motif-4和Motif-5, 说明Motif-3、Motif-4和Motif-5的功能也相当保守(图2)。
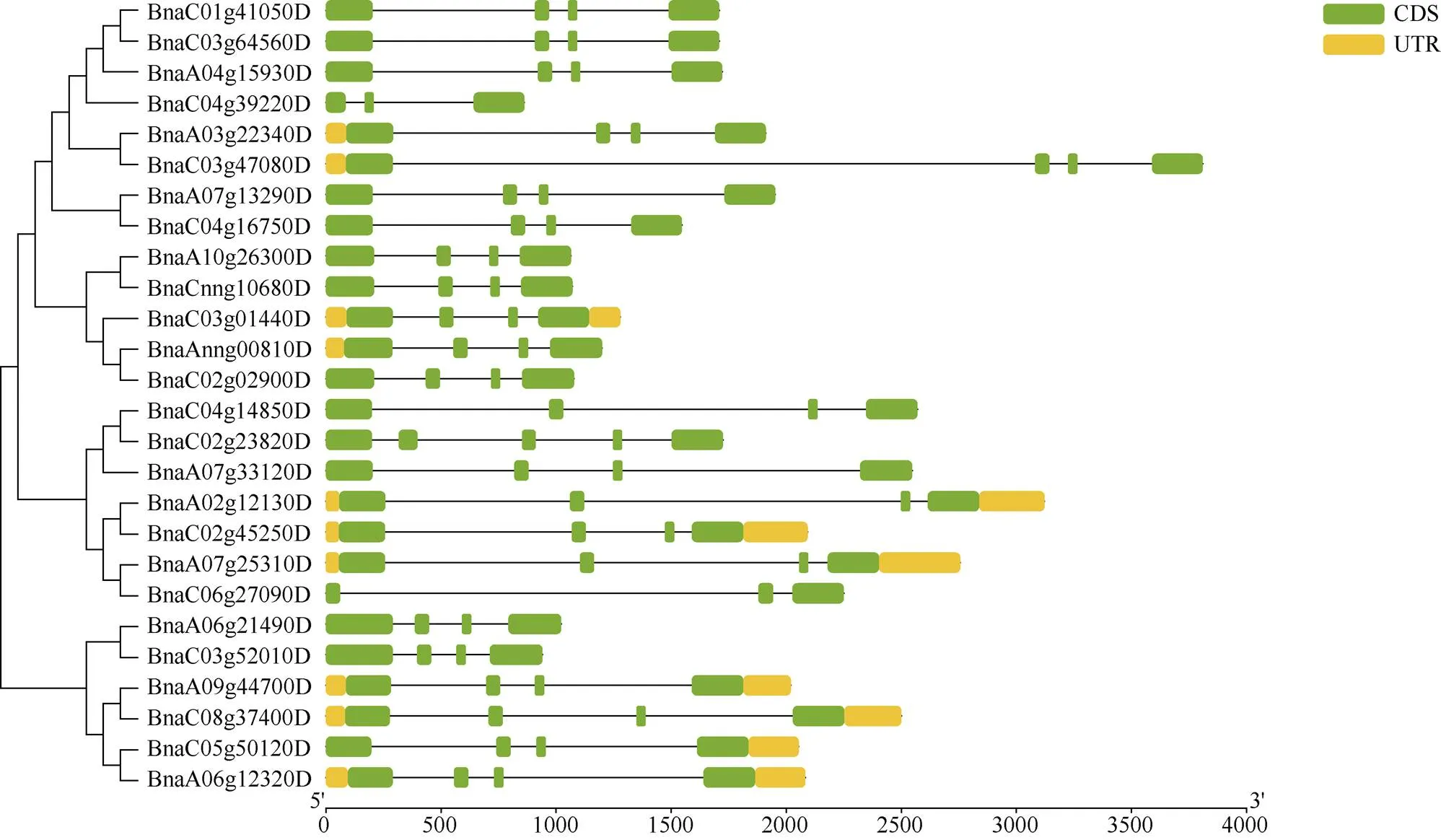
图1 油菜PEBP进化树与基因结构
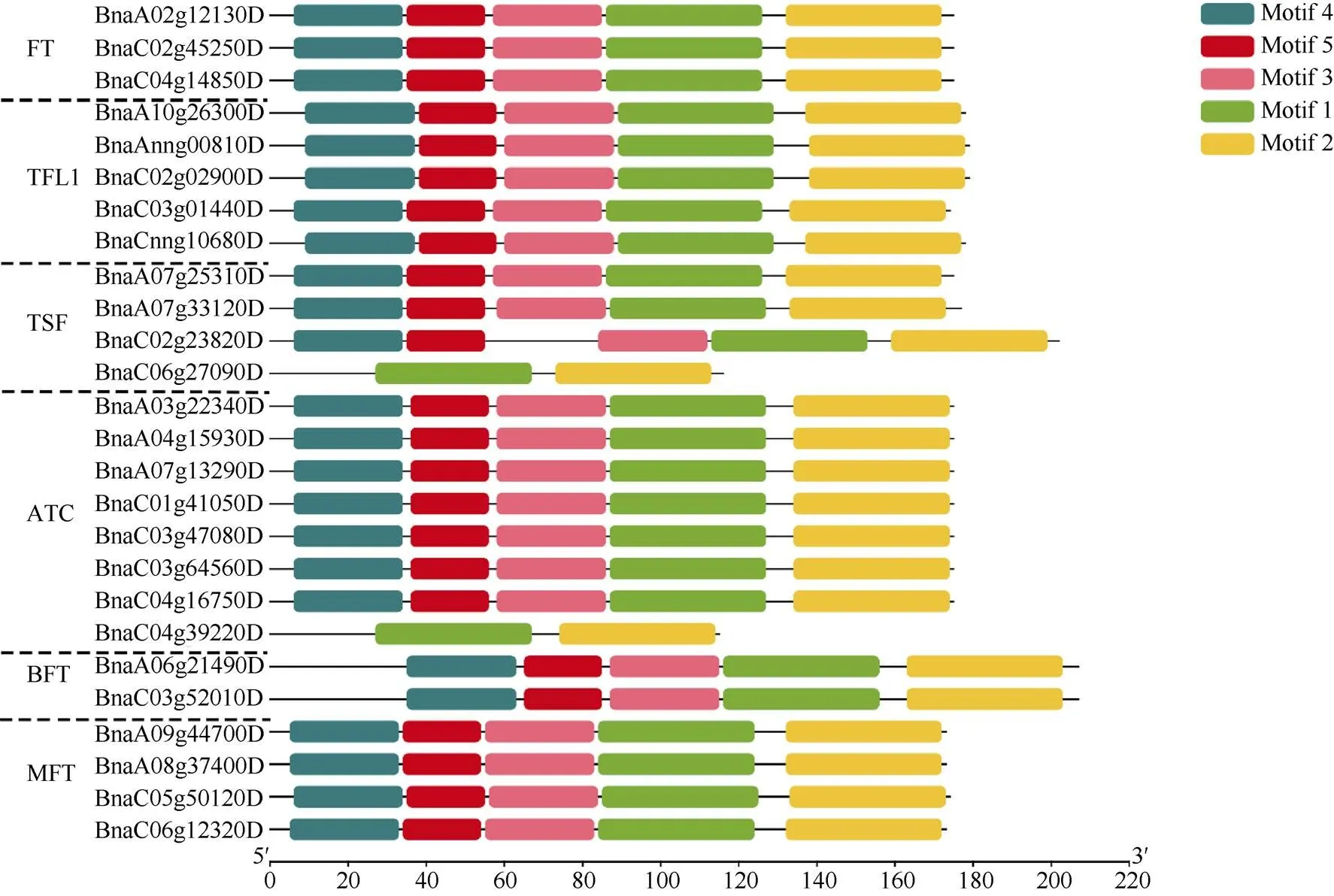
图2 油菜PEBP基因家族motif预测
为检测油菜基因家族的扩增情况, 检测了26个成员复制情况, 发现超过76.9%的成员都属于片段重复, 说明片段复制在油菜家族扩增中起着重要作用。
2.3 序列比对与进化树构建
利用Clustal W软件将4个物种PEBP蛋白序列进行多序列比对, 并导入DNAMAN中展示(图3)。用MEGA6.0软件, 通过邻接法(Neighbor Joining, NJ) 构建基因分子进化树(图4), 表明该基因可分为3个亚家族, 第一亚家族包括ATC(13)和TFL1(11), 第二亚家族包括FT(6)和TSF(6), 第三亚家族包括BFT(5)和MFT(8)。

图3 油菜与拟南芥PEBP蛋白序列多重比对
2.4 选择压力分析
在进化分析中, 常用Ka/Ks或dN/dS来计算某基因是否受到自然选择作用。如果Ka/Ks>1, 认为有正选择效应; 如果Ka/Ks=1, 认为存在中性选择; 如果Ka/Ks<1, 则认为有纯化选择作用。以拟南芥家族基因为参照估算进化过程中甘蓝型油菜基因家族的选择压力(表2)。表明, 油菜基因家族的Ka/Ks均小于1, 即受到纯化选择, 其中和亚家族的Ka/Ks较大,和亚家族的Ka/Ks较小, 表明和亚家族进化更保守。
2.5 甘蓝型油菜PEBP基因家族成员的组织表达分析
为解析甘蓝型油菜基因家族的功能, 利用本课题组前期完成的不同发育时期的中双11材料的共111个组织器官的转录组数据分析甘蓝型油菜基因家族的表达情况(图5)。3个基因成员中,在各组织器官中表达量均较低或者不表达, 其余两个成员在开花后茎秆、叶片和发育的角果皮中具有较高的表达量, 其他组织器官表达量均较低; 4个成员中, 除在花器官中具有一定表达之外, 其他成员在所有检测样品中几乎均不表达; 4个成员中,和在茎秆、叶片、蕾、雄蕊、花药、花丝以及不同发育时期的种子中有一定表达, 其余样品表达量均较低, 剩余2个成员在所有检测样品中几乎均不表达; 8个油菜成员中, 除了和在胚根和下胚轴中有表达之外, 其余成员均不表达; 2个BFT中,在不同发育时期的叶子中表达, 其余组织器官中不表达,在所有检测的样品中均不表达或者表达量很低; 4个成员均在发育的种子、种胚、胚芽和种皮中特异表达, 其他组织器官中不表达。以上结果表明, 油菜家族具有较高的组织表达特性, 部分同源基因功能可能发生分化。
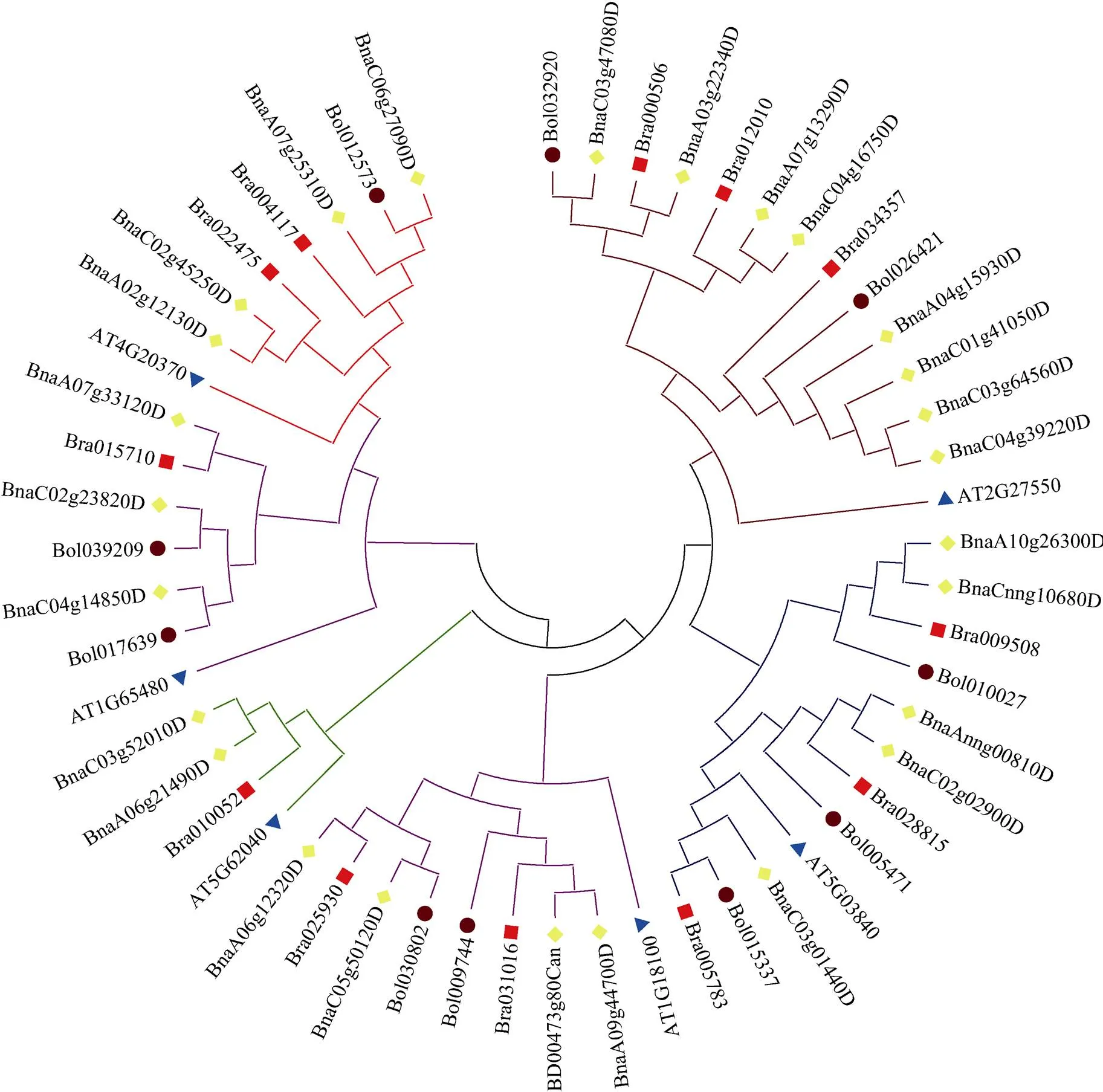
图4 油菜、白菜、甘蓝和拟南芥PEBP基因家族进化分析
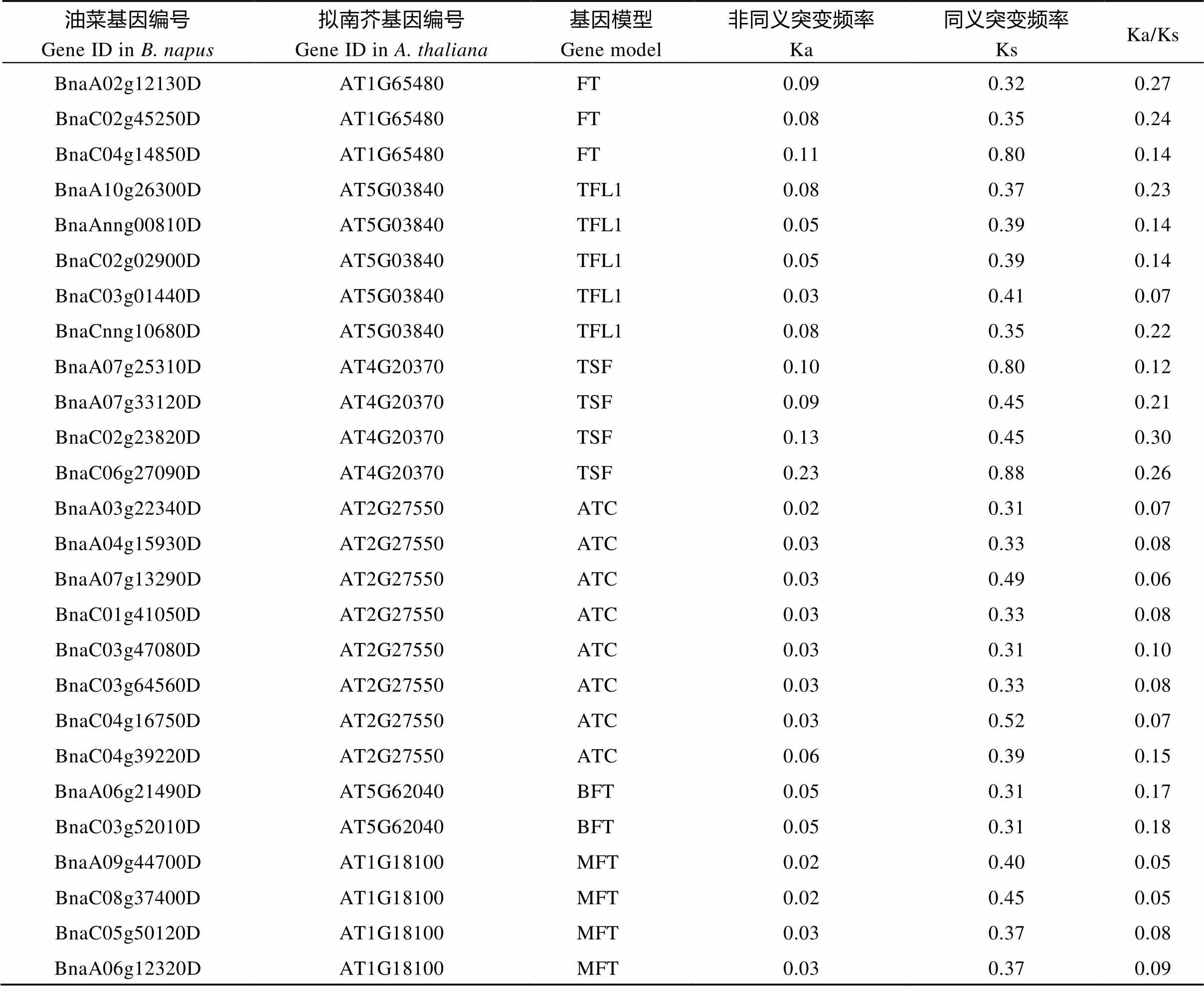
表2 油菜和拟南芥PEBP同源基因的Ka/Ks

图5 油菜PEBP家族成员的组织表达分析
横坐标样品名称详见附表1, 蓝色表示低表达, 黄色表示高表达。
Samples were listed in Supplementary table 1. Blue shows low expression levels and yellow shows high expression levels.
3 讨论
随着白菜、甘蓝和甘蓝型油菜基因组数据的释放, 芸薹属植物的遗传和分子功能研究进入一个新的时代。有关油菜产量性状如千粒重、农艺性状如株高以及抗性性状如核盘菌抗性的研究已经有大量报道, 而且利用分子标记技术对开花时间等数量性状QTL的定位同样有很多研究。PEBP家族功能非常保守而且在动植物中参与多种生物学过程[35]。其家族成员的功能在拟南芥、水稻、葡萄等作物中有一定研究, 但油菜中除一例报道外[36], 对该基因家族的功能特点研究相对较少。
基于同源比对的方法, 我们在油菜基因组内筛选出26个基因家族成员, 分别包括2个、8个、3个、4个、5个和4个。进化树表明, 共包含3个亚家族, 其中ATC和TFL1亚家族, 为开花抑制因子; FT和TSF亚家族, 为开花促进因子; BFT和MFT属于第三亚家族。结构分析表明, 大分部成员含有4个外显子和3个内含子, 表明油菜基因家族进化非常保守。Motif预测表明Motif-1和-2是PEBP基因家族特征性基序。和亚家族的Ka/Ks较大,和亚家族的Ka/Ks较小, 表明和亚家族进化更保守。
家族成员的组织器官时空表达特性在拟南芥、葡萄和水稻等作物中已有报道[37-39]。但油菜中尚无有该基因的组织表达特性的研究。在本研究中, 3个基因成员除在各组织器官中表达量均较低或者不表达, 其余两个成员在开花后器官如茎秆、叶片和发育的角果皮中具有较高的表达量, 其他组织器官中表达量均较低, 这与拟南芥中的表达模式一致[40]。4个成员中, 只有在花器官中具有一定表达, 其他成员均不表达, 而棉花和主要在根中特异表达[30]。作为开花抑制因子, 油菜基因仅在子叶和萌发的根中表达, 这与拟南芥基因的表达模式一致。5个基因成员中, 只有在花器官中有表达, 而其他成员均不表达, 表明为的主要关键成员, 该基因与功能类似, 都抑制植物开花[41]。与TFL1有功能冗余的2个油菜基因, 只有在不同发育时期的叶子中表达[42]。基因主要在种子中表达, 且对ABA有响应[21], 油菜的4个基因成员在不同发育时期的种子中均有较高水平表达。
[1] Bouché F, Lobet G, Tocquin P, Périlleux C. FLOR-ID: an interactive database of flowering-time gene networks in, 2016, 44(D1): D1167–D1171.
[2] Srikanth A, Schmid M. Regulation of flowering time: all roads lead to Rome, 2011, 68: 2013–2037.
[3] Putterill J, Laurie R, Macknight R. It’s time to flower: the genetic control of flowering time, 2004, 26: 363–373.
[4] Roux F, Touzet P, Cuguen J, Le Corre V. How to be early flowering: an evolutionary perspective, 2006, 11: 375–381.
[5] Fornara F, de Montaigu A, Coupland G. SnapShot: control of flowering in Arabidopsis, 2010, 141(3), doi: 10.1016/j.cell. 2010.04.024.
[6] Turck F, Fornara F, Coupland G. Regulation and identity of florigen: FLOWERING LOCUS T moves center stage, 2008, 59: 573–594.
[7] Kikuchi R, Kawahigashi H, Ando T, Tonooka T, Handa H. Molecular and functional characterization ofgenes in barley reveal the diversification of their roles in flowering, 2009, 149: 1341–1353.
[8] Karlgren A, Gyllenstrand N, Källman T, Sundström J F, Moore D, Lascoux M, Lagercrantz U. Evolution of thegene family in plants: functional diversification in seed plant evolution, 2011, 156: 1967–1977.
[9] Tao Y B, Luo L, He L L, Ni J, Xu Z F. A promoter analysis of(), a seed-preferential gene from the biofuel plant, 2014, 127: 513–524.
[10] Peng F Y, Hu Z, Yang R C. Genome-wide comparative analysis of flowering-related genes in Arabidopsis, wheat, and barley., 2015, 2015: 874361.
[11] Yamaguchi A, Kobayashi Y, Goto K, Abe M, Araki T.() acts as a floral pathway integrator redundantly with FT, 2005, 46: 1175–1189.
[12] Huang N C, Jane W N, Chen J, Yu T S.homologue () acts systemically to inhibit floral initiation in Arabidopsis, 2012, 72: 175–184.
[13] Yoo S Y, Kardailsky I, Lee J S, Weigel D, Ahn J H. Acceleration of flowering by overexpression of(), 2004, 17: 95–101.
[14] Kardailsky I, Shukla V K, Ahn J H, Dagenais N, Christensen S K, Nguyen J T, Chory J, Harrison M J, Weigel D. Activation tagging of the floral inducer, 1999, 286: 1962–1965.
[15] Jang S, Torti S, Coupland G. Genetic and spatial interactions between,andduring the early stages of floral induction in Arabidopsis, 2009, 60: 614–625.
[16] Bradley D, Ratcliffe O, Vincent C, Carpenter R, Coen E. Inflorescence commitment and architecture in Arabidopsis, 1997, 275: 80–83.
[17] Ratcliffe O J, Bradley D J, Coen E S. Separation of shoot and floral identity in Arabidopsis, 1999, 126: 1109–1120.
[18] Conti L, Bradley D.is a mobile signal controlling Arabidopsis architecture, 2007, 19: 767–778.
[19] Hanano S, Goto K. Arabidopsisis involved in the regulation of flowering time and inflorescence development through transcriptional repression, 2011, 23: 3172–3184.
[20] Ryu J Y, Lee H J, Seo P J, Jung J H, Ahn J H, Park C M. The Arabidopsis floral repressor BFT delays flowering by competing with FT for FD binding under high salinity, 2014, 7: 377–387.
[21] Xi W, Liu C, Hou X, Yu H.regulates seed germination through a negative feedback loop modulating ABA signaling in Arabidopsis, 2010, 22: 1733–1748.
[22] Wang Z, Zhou Z, Liu Y, Liu T, Li Q, Ji Y, Li C, Fang C, Wang M, Wu M, Shen Y, Tang T, Ma J, Tian Z. Functional evolution of phosphatidylethanolamine binding proteins in soybean and Arabidopsis, 2015, 27: 323–336.
[23] Nan H, Cao D, Zhang D, Li Y, Lu S, Tang L, Yuan X, Liu B, Kong F.andredundantly and differentially regulate flowering through interaction with and upregulation of the bZIP transcription factor GmFDL19 in soybean, 2014, 9: 0097669.
[24] Chardon F, Damerval C. Phylogenomic analysis of thegene family in cereals, 2005, 61: 579–590.
[25] Komiya R, Ikegami A, Tamaki S, Yokoi S, Shimamoto K.andare essential for flowering in rice, 2008, 135: 767–774.
[26] Meng X, Muszynski M G, Danilevskaya O N. Thegene functions as a floral activator and is involved in photoperiod sensitivity in maize, 2011, 23: 942–960.
[27] Raman H, Raman R, Coombes N, Song J, Prangnell R, Bandaranayake C, Tahira R, Sundaramoorthi V, Killian A, Meng J, Dennis E S, Balasubramanian S. Genome-wide association analyses reveal complex genetic architecture underlying natural variation for flowering time in canola, 2016, 39: 1228–1239.
[28] Xu L, Hu K, Zhang Z, Guan C, Chen S, Hua W, Li J, Wen J, Yi B, Shen J, Ma C, Tu J, Fu T. Genome-wide association study reveals the genetic architecture of flowering time in rapeseed (L.)., 2016, 23: 43–52.
[29] Wang J, Qiu Y, Cheng F, Chen X, Zhang X, Wang H, Song J, Duan M, Yang H, Li X. Genome-wide identification, characterization, and evolutionary analysis of flowering genes in radish (L.), 2017, 18, doi: 10.1186/s12864-017-4377-z.
[30] Zhang X, Wang C, Pang C, Wei H, Wang H, Song M, Fan S, Yu S. Characterization and functional analysis of PEBP family genes in upland cotton (L.), 2016, 11: 0161080.
[31] Książkiewicz M, Rychel S, Nelson M N, Wyrwa K, Naganowska B, Wolko B. Expansion of the phosphatidylethanolamine binding protein family in legumes: a case study ofL.homologs,and, 2016, 17, doi: 10.1186/s12864-016-3150-z.
[32] Leeggangers H A C F, Rosilio-Brami T, Bigas-Nadal J, Rubin N, van Dijk A D J, Nunez de Caceres Gonzalez F F, Saadon-Shitrit S, Nijveen H, Hilhorst H W M, Immink R G H, Zaccai M.andgenes and their putative roles in flowering time control, 2018, 59: 90–106.
[33] Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6: molecular evolutionary genetics analysis version 6.0, 2013, 30: 2725–2729.
[34] Wang D P, Wan H L, Zhang S, Yu J. Gamma-MYN: a new algorithm for estimating Ka and Ks with consideration of variable substitution rates, 2009, 4, doi: 10.1186/1745-6150-4-20.
[35] Danilevskaya O N, Meng X, Hou Z, Ananiev E V, Simmons C R. A genomic and expression compendium of the expandedgene family from maize, 2008, 146: 250–264.
[36] Guo Y, Hans H, Christian J, Molina C. Mutations in singleandparalogs of rapeseed (L.) and their impact on flowering time and yield components, 2014, 5: 282.
[37] Carmona M J, Calonje M, Martínez-Zapater J M. The/gene family in grapevine, 2007, 63: 637–650.
[38] Carmel-Goren L, Liu Y S, Lifschitz E, Zamir D. Thegene family in tomato, 2003, 52: 1215–1222.
[39] Hedman H, Källman T, Lagercrantz U. Early evolution of thegene family in plants, 2009, 70: 359–369.
[40] Kobayashi Y, Kaya H, Goto K, Iwabuchi M, Araki T. A pair of related genes with antagonistic roles in mediating flowering signals, 1999, 286: 1960–1962.
[41] Baumann K, Venail J, Berbel A, Domenech M J, Money T, Conti L, Hanzawa Y, Madueno F, Bradley D. Changing the spatial pattern ofexpression reveals its key role in the shoot meristem in controlling Arabidopsis flowering architecture, 2015, 66: 4769–4780.
[42] Yoo S J, Chung K S, Jung S H, Yoo S Y, Lee J S, Ahn J H.() has-like activity and functions redundantly within inflorescence meristem development in Arabidopsis, 2010, 63: 241–253.
附表1 ‘中双11’的111个样品信息
Supplementarey table 1 Information of 111 materiels from ‘Zhongshuang 11’
编号Code生育期Growth stage器官Organ取样时间Treatment time编号Code生育期Growth stage器官Organ取样时间Treatment time Ro_24h种子萌发胚根萌发后24 hSe_7d青荚期种子花后7 d Ro_48h种子萌发胚根萌发后48 hSe_10d青荚期种子花后10 d Ro_72h种子萌发胚根萌发后72 hSe_13d青荚期种子花后13 d Hy_24h种子萌发下胚轴萌发后24 hSe_19d青荚期种子花后19 d Hy_48h种子萌发下胚轴萌发后48 hSe_21d青荚期种子花后21 d Hy_72h种子萌发下胚轴萌发后72 hSe_24d青荚期种子花后24 d Co_24h种子萌发子叶萌发后24 hSe_27d青荚期种子花后27 d Co_48h种子萌发子叶萌发后48 hSe_30d青荚期种子花后30 d Co_72h种子萌发子叶萌发后72 hSe_35d青荚期种子花后35 d GS_12h种子萌发整个种子萌发后12 hSe_40d青荚期种子花后40 d GS_24h种子萌发整个种子萌发后24 hSe_43d青荚期种子花后43 d Ro_s苗期-温室根Se_46d青荚期种子花后46 d Ro_s_f苗期-大田根Se_49d青荚期种子花后49 d Ro_b蕾期根Em_19d青荚期种胚花后19 d Ro_i初花期根Em_21d青荚期种胚花后21 d Ro_f盛花期根Em_24d青荚期种胚花后24 d St_b蕾期茎秆Em_27d青荚期种胚花后27 d St_i初花期茎秆Em_30d青荚期种胚花后30 d St_f盛花期茎秆Em_35d青荚期种胚花后35 d St_24d青荚期茎秆花后24 dEm_40d成熟期种胚花后40 d St_50d成熟期茎秆花后50 dEm_43d成熟期种胚花后43 d Le_s苗期-温室叶片Em_46d成熟期种胚花后46 d Le_s_f苗期-大田叶片Em_49d成熟期种胚花后49 d LeY_b蕾期幼叶Ra_40d成熟期胚芽花后40 d LeY_i初花期幼叶Ra_43d成熟期胚芽花后43 d LeY_f盛花期幼叶Ra_46d成熟期胚芽花后46 d LeY_10d青荚期幼叶花后10 dRa_49d成熟期胚芽花后49 d LeY_24d青荚期幼叶花后24 dSC_19d青荚期种皮花后19 d LeY_30d青荚期幼叶花后30 dSC_21d青荚期种皮花后21 d LeO_b蕾期成熟叶片SC_27d青荚期种皮花后27 d LeO_i初花期成熟叶片SC_30d青荚期种皮花后30 d LeO_f盛花期成熟叶片SC_35d青荚期种皮花后35 d LeO_10d青荚期成熟叶片花后10 dSC_40d青荚期种皮花后40 d LeO_24d青荚期成熟叶片花后24 dSC_43d成熟期种皮花后43 d LeO_30d青荚期成熟叶片花后30 dEn_21d青荚期内种皮花后21 d Bu_b蕾期蕾En_24d青荚期内种皮花后24 d Ao_i初花期花柄Ep_24d青荚期外种皮花后24 d Ao_f盛花期花柄Ep_30d青荚期外种皮花后30 d Cal_i初花期萼片Fu_27d青荚期珠柄花后27 d Cal_f盛花期萼片Fu_35d青荚期珠柄花后35 d Pe_i初花期花瓣SP_3d青荚期角果皮花后3 d Pe_f盛花期花瓣SP_5d青荚期角果皮花后3 d Pi_f_un盛花期雌蕊-未授粉SP_7d青荚期角果皮花后7 d Pi_i初花期雌蕊SP_10d青荚期角果皮花后10 d
(续附表1)
编号Code生育期Growth stage器官Organ取样时间Treatment time编号Code生育期Growth stage器官Organ取样时间Treatment time Pi_f盛花期雌蕊SP_13d青荚期角果皮花后13 d Sta_i初花期雄蕊SP_16d青荚期角果皮 Sta_f盛花期雄蕊SP_19d青荚期角果皮花后19 d At_i初花期花药SP_21d青荚期角果皮花后21 d At_f盛花期花药SP_24d青荚期角果皮花后24 d Cap_i初花期花丝SP_27d青荚期角果皮花后27 d Cap_f盛花期花丝SP_30d青荚期角果皮花后30 d IT_b蕾期主序顶端SP_35d青荚期角果皮花后35 d IT_i初花期主序顶端SP_40d成熟期角果皮花后40 d IT_f盛花期主序顶端SP_43d成熟期角果皮花后43 d Se_3d青荚期种子花后3 dSP_46d成熟期角果皮花后46 d Se_5d青荚期种子花后3 d
Identification and expression analysis ofgene family in oilseed rape
JIAN Hong-Ju**, YANG Bo**, LI Yang-Yang, YANG Hong, LIU Lie-Zhao, XU Xin-Fu, and LI Jia-Na*
College of Agronomy and Biotechnology / Chongqing Engineering Research Center for Rapeseed / Academy of Agricultural Sciences, Southwest University, Chongqing 400715, China
The plant phosphatidylethanolamine-binding protein (PEBP) genes play an important role in controlling flowering time. Rapeseed, as one of the most important oil crops in the world, has similar flowering habits with. In this study, the PEBP family protein sequences ofwere used to perform BlastP analysis in the rapeseed genome. The members of the rape PEBP family were obtained, and the gene structure analysis, motif prediction, duplication analysis, phylogenetic tree construction, selective pressure analysis and tissue expression analysis were carried out on the family members. A total of 26 rapeseed PEBP gene members were identified, and most of them contained four exons and three introns, and motif-1 and motif-2 were the characteristics of PEBP members. Over 76.9% members were segmental duplicated. Phylogenetic tree analysis showed that PEBP was divided into three subfamilies. The tissue-specifics analysis based on RNA-Seq data showed that 26 PEBP members of rapeseed had very obvious tissue expression characteristics. All these results greatly enrich our understandings of flowering genes and regulation patterns in, and provide a theoretical basis for further molecular breeding in.
; flowering genes; PEBP; evolution analysis; expression analysis
2018-07-09;
2018-10-08;
2018-11-01.
10.3724/SP.J.1006.2019.84095
李加纳, E-mail: ljn1950@swu.edu.cn, Tel: 023-68250642
**同等贡献(Contributed equally to this work)
荐红举, E-mail: jianhongju1989@126.com, 杨博, E-mail: sheepneck@hotmail.com
本研究由高等学校学科创新引智基地项目“作物种质资源利用创新引智基地”(B12006), 重庆市民生工程主题专项项目(cstc2016shms- ztzx80020)和重庆市研究生创新项目(CYS17078)资助。
This study was supported by the Project of Intellectual Base for Discipline Innovation in Colleges and Universities (B12006), the Special Project of Chongqing People’s Livelihood, and Chongqing Graduate Student Research Innovation Project (CYS17078).
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20181030.1734.008.html

