原发性肝细胞癌GOLM1基因异常表达和相关miRNA和lncRNA预测
王 蕾,马 军,刘宗文,王 健,张中冕,张 玲,张 芳,周士霞,张萌迪,邢仪通,孙甜甜,马佳康,段芳龄
郑州大学第二附属医院 1.放射科; 2.检验科; 3.放疗科; 4.肿瘤科;河南 郑州 450014; 5.郑州大学附属肿瘤医院肝胆外科; 6.郑州大学第二附属医院ICU; 7.郑州大学第二附属医院消化内科
原发性肝细胞癌(hepatocellular carcinoma,HCC)是一种常见的恶性肿瘤,肿瘤相关死亡率位列前三位[1],其致癌作用主要与肝炎病毒、饮酒、脂肪肝等引起的肝炎或肝硬化有关。HCC的诊断主要依靠病史、血清AFP水平及超声、CT或磁共振等影像学检查,由于缺乏高灵敏度和特异度的标志物,早期诊断难以实现,治疗效果不能令人满意,因此,有必要进一步寻找更有效的诊断标志物和治疗靶点。
GOLM1(GP73)是高尔基体跨膜蛋白,在HCC组织中表达。一些数据显示,血清GOLM1是诊断HCC的可靠生物标志物,其灵敏度和特异度可能优于目前使用的生物标志物,如AFP[2-3]。然而,最近的一些研究发现,GOLM1在良性肝脏疾病中也有表达,甚至肝硬化患者血清GOLM1水平也显著高于HCC患者[4-5],因此对于GOLM1在HCC诊断中的作用还需要进一步探讨。
非编码RNA,包括miRNA(微小RNA)、lncRNA(长链非编码RNA)等,对基因表达的调控越来越受到重视,并有可能对肿瘤的发生、发展起重要作用,在HCC中,也发现多个非编码RNA与其发生、发展及预后相关,这些基因既可作为肿瘤诊断的潜在标志物,也可成为治疗靶点[6-7]。
本研究分析了来自Oncomine数据库中HCC相关病变中GOLM1 mRNA的表达,用组织芯片和免疫组化(IHC)技术验证HCC组织和癌旁组织中GOLM1蛋白的表达,并用生物信息学技术预测其相关的非编码RNA。
1 材料与方法
1.1在线数据分析GOLM1mRNA在HCC中的表达使用Oncomine(https://www.oncomine.org/)和UALCAN(http://ualcan. path.uab.edu/)在线工具,分析公开数据库中GOLM1 mRNA在HCC患者中的表达,并从TCGA数据库中下载HCC患者mRNA和非编码RNA测序数据和临床病理信息,进行相关性分析。
1.2GOLM1蛋白的组织芯片免疫组化染色和病理临床特征的相关性分析包含90例HCC患者成对癌组织和癌旁组织标本的组织阵列来自上海Outdo生物技术有限公司(HLivH180Su06,SOBC,中国上海),详细信息可以在线下载(http://www.superchip.com.cn/biology/tissue.html)。手术时间为2006年2月至2007年5月,随访时间截止至2012年2月。免疫组化具体方法为常规脱蜡、水化和抗原热修复的组织标本,商业阻断剂(迈新生物技术)用于阻断非特异性染色。将组织芯片加抗人GOLM1抗体(Santa Cruez,sc-365817,1∶100)在4 ℃下孵育过夜;用PBS冲洗,加二抗(SP试剂盒)孵育1 h;PBS洗净后,进行DAB显色、苏木精复染。PBS代替一抗作为阴性对照。根据染色强度和染色细胞百分比,由2名未知临床病理特征的研究人员评估IHC染色结果。颜色强度评分:无颜色,0分;浅黄色,1分;棕黄色,2分;深棕色,3分。阳性细胞百分率:<10%,0分;10%~30%,1分;>30%,3分。利用两者的积分,评价标本结果,综合评分>3为阳性病例。
1.3TCGA转录组数据下载和分析下载TCGA数据库中的HCC转录组测序数据,利用R和Perl软件进行mRNA、miRNA和lncRNA差异谱分析,矫正后P值设定为0.05,差异倍数logFC设定为2,并进行共表达分析和靶基因预测。
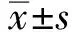
2 结果
2.1HCC、肝纤维化和不典型增生组织GOLM1mRNA表达升高首先,我们使用Oncomine在线数据,进行HCC中GOLM1 mRNA表达的Meta分析,在4个HCC与正常对照研究中,GOLM1 mRNA均显示为高表达基因(见图1A)。接着用UALCAN在线工具对TCGA数据中的HCC和正常对照组的GOLM1 mRNA表达水平进行比较,在HCC中GOLM1的表达明显高于癌旁正常组织;另外,我们下载了TCGA转录组表达和部分病理临床数据,经分析发现,GOLM1 mRNA表达与HCC的分级、患者临床分期及总生存时间相关(见图1B、表1)。
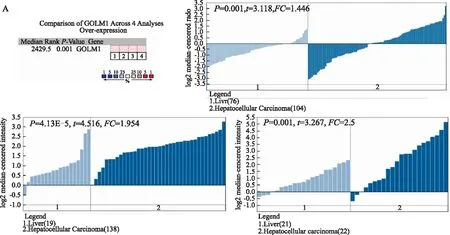
图1在线数据分析GOLM1mRNA在HCC中的异常表达A:GOLM1 mRNA表达分析(HCC和正常肝组织对比,Oncomine Meta分析),综合分析4个HCC研究的GOLM1 mRNA差异表达情况(癌和正常),结果显示GOLM1是HCC中高表达基因;B:TCGA转录
组测序数据分析结果,GOLM1 mRNA在HCC中明显高于正常组织,并和总生存期相关(K-M分析)
Fig1GOLM1mRNAexpressioninHCCbyonlineanalysis
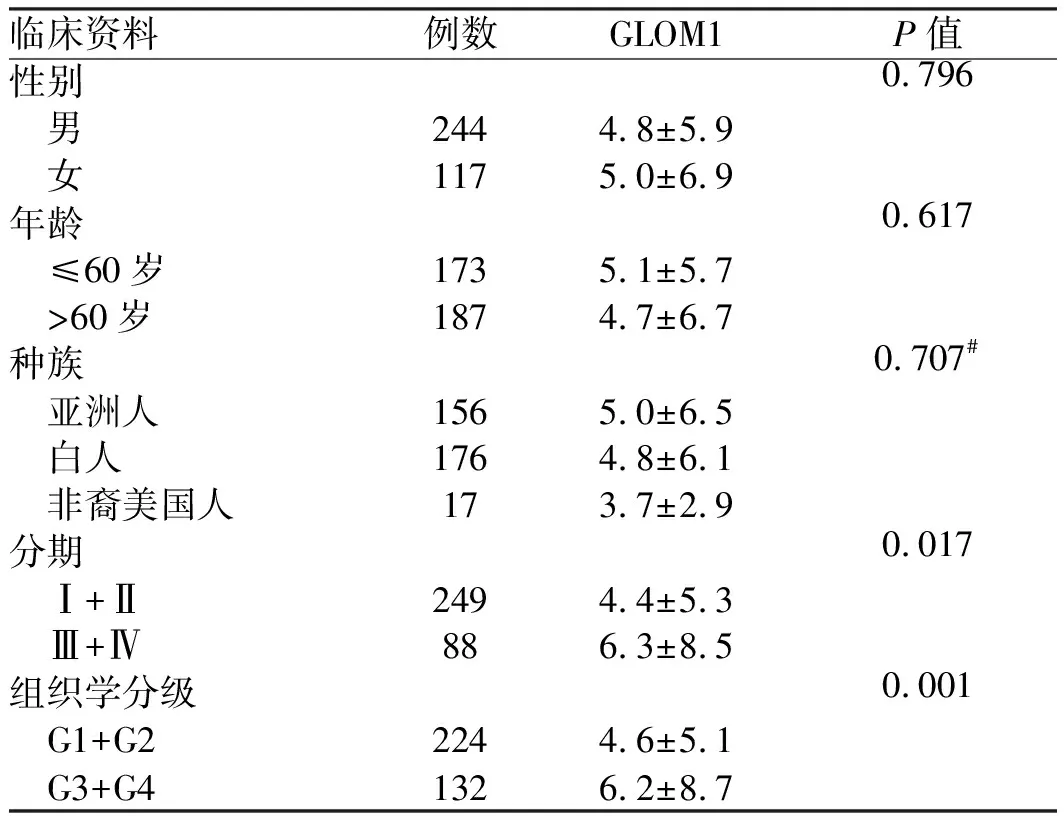
表1 GOLM1 mRNA表达水平与HCC患者病理临床特征相关性分析(TCGA数据) Tab 1 Correlation analysis between GOLM1 mRNA expression and clinicopathological characteristics of HCC (TCGA data)
注:#代表三个种族组间ANOVA分析,其他为两组间t检验。
Oncomine数据分析显示,GOLM1 mRNA表达在肝硬化(上)和不典型增生病变(下)中的表达均高于正常肝组织(见图2)。
2.2基因芯片检测GOLM1蛋白在HCC组织中的表达GOLM1阳性信号定位于HCC细胞的核周区或细胞质中,癌旁组织中的非肿瘤细胞在细胞质中呈弥散细颗粒状(见图3),经统计学分析,GOLM1蛋白阳性率在癌组织和癌旁正常组织中差异无统计学意义(71.1%vs72.2%),其表达与病理临床特征之间无显著相关性(见表2),与总生存时间、无病生存时间也无明显关系。
2.3GOLM1相关miRNA和lncRNA生物信息学预测TCGA转录组数据下载后进行差异基因分析,通过TargetScan在线预测GOLM1的相关miRNA后,和HCC中差异的miRNA取交集,共发现48个GOLM1基因相关的miRNA。通过mRNA和lncRNA共表达分析并和HCC中差异lncRNA取交集,发现6个差异lncRNA和GOLM1呈正相关(见图4、表3)。
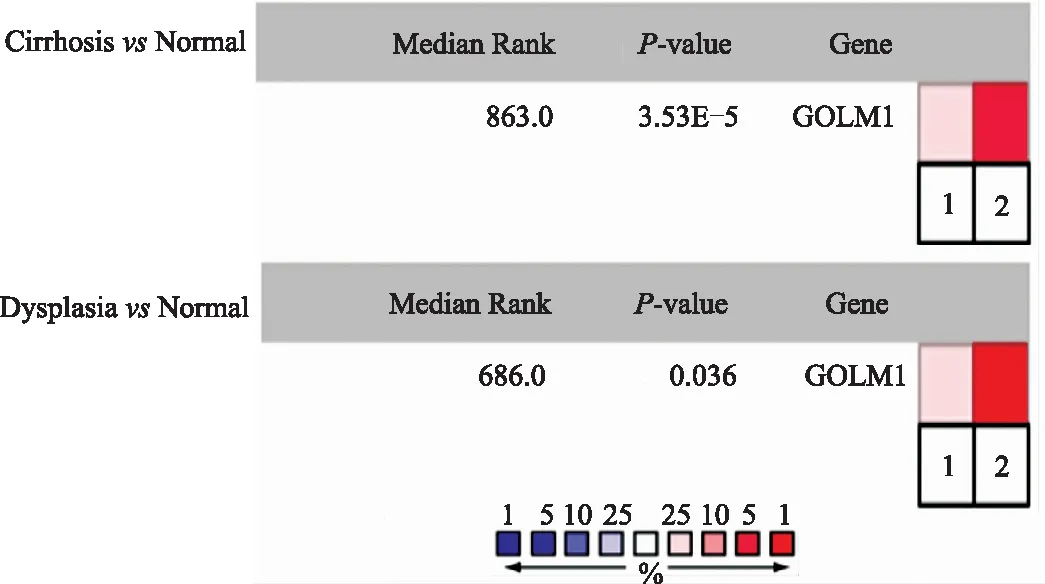
图2 GOLM1 mRNA在肝硬化或不典型增生结节中的异常表达Fig 2 Abnormal expressions of GOLM1 mRNA in cirrhosis and dysplasia tissues of liver
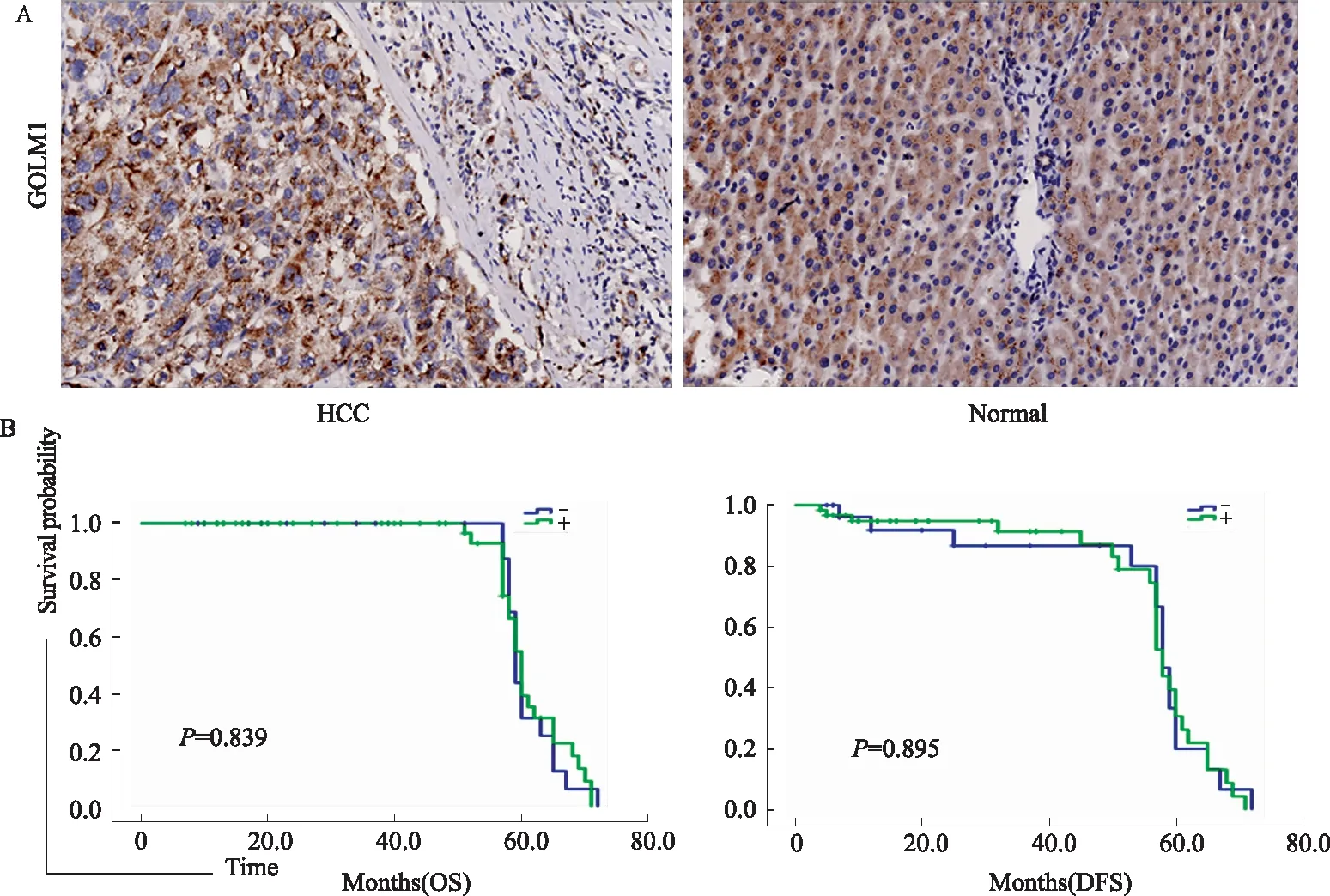
图3GOLM1蛋白在HCC组织中的表达和生存分析A:阳性染色的HCC和癌旁组织免疫组化结果(SP法,200×),癌旁组织染色为弥漫性,HCC组织着色多为粗颗粒状;B:GOLM1蛋白表达和HCC患者总生存时间(OS)、无复发生存时间(DFS)相关性分析
Fig3GOLM1proteinexpressioninHCCandsurvivalanalysis
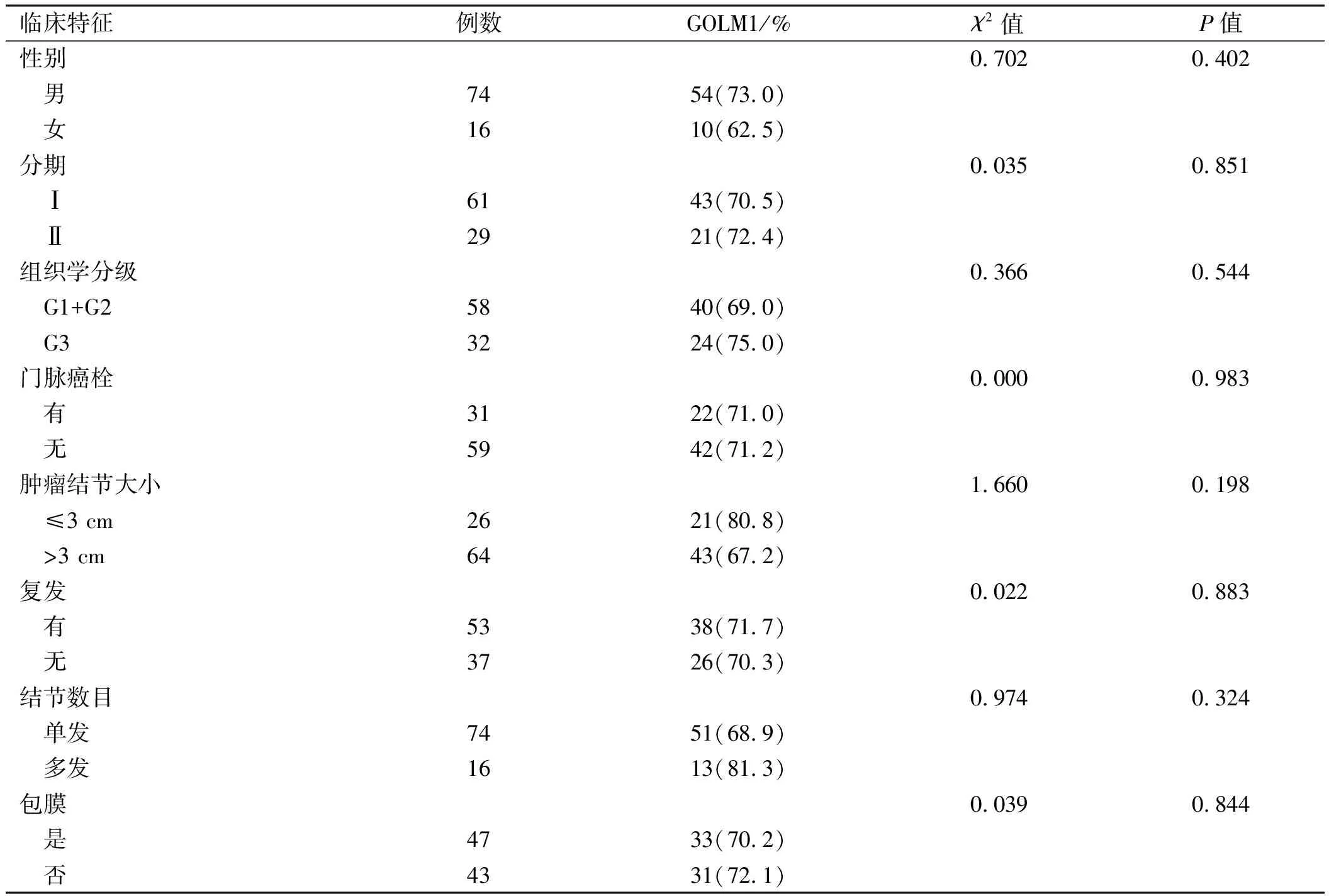
表2 GOLM1蛋白表达和HCC患者病理临床特征之间的相关性分析(组织芯片)Tab 2 Correlation analysis between expression of GOLM1 protein and clinicopathological features of HCC (tissue microarray)
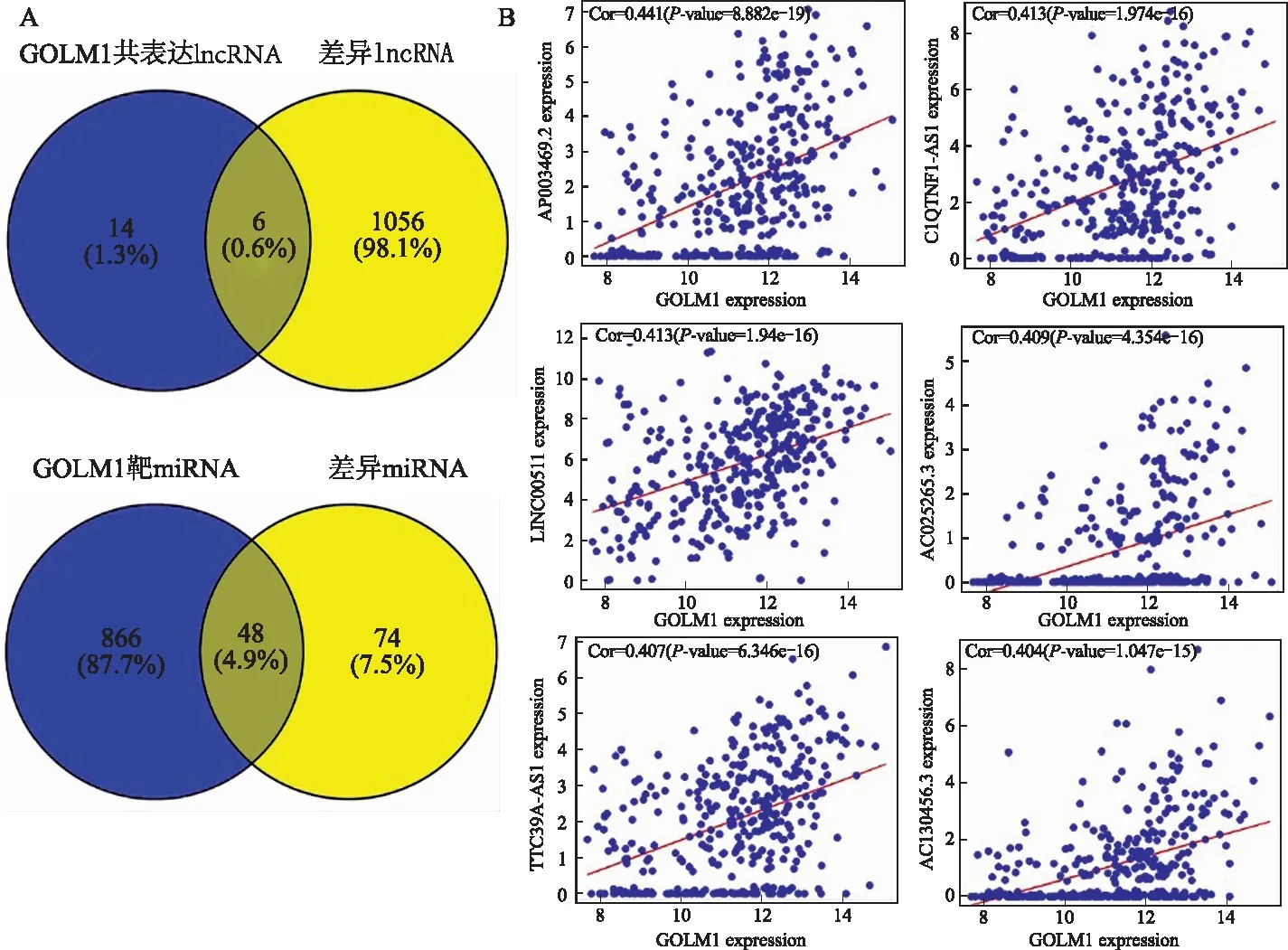
注:cor:相关系数。
图4TCGA转录组数据分析在HCC中GOLM1mRNA相关的miRNA和lncRNAA:通过TargetScan预测GOLM1相关的miRNA(蓝),和HCC中差异的miRNA(黄)用Venn图取交集,共得到48个miRNA;B:利用R软件函数cor.test分析和GOLM1基因
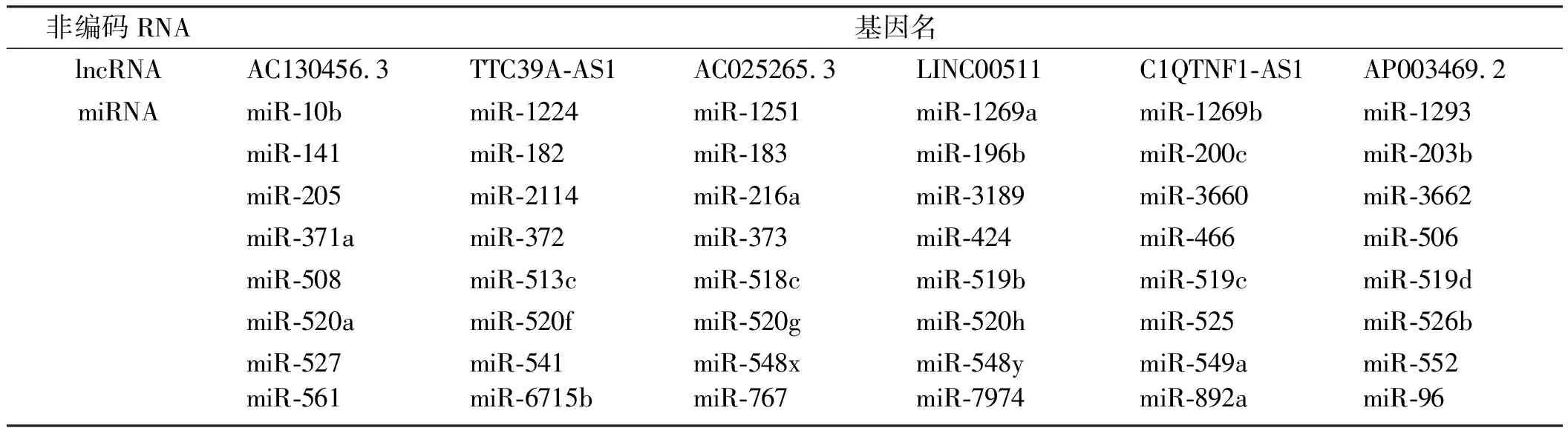
有共表达关系的lncRNA(蓝),和HCC中差异lncRNA(黄)取交集,得到6个lncRNA
3 讨论
虽然在生理情况下GOLM1主要在胆管上皮细胞中表达,肝细胞中很少表达,但多种病因可导致GOLM1在肝脏疾病中表达异常。早期报道表明,血清GOLM1检测可作为诊断HCC的潜在标志物,甚至优于AFP[2]。然而,另外一些研究表明,HCC患者和肝硬化患者血清GOLM1均可呈高水平表达,因此GOLM1在HCC诊断中的可靠性受到质疑[3-5]。根据SHAN等[8]研究,GOLM1蛋白在HCC组织中表达最高,从高度不典型增生、慢性乙型肝炎到正常肝细胞逐渐降低(IHC和Western blotting),但高级别不典型增生与HCC差异无统计学意义;此外,该研究在血清的检测结果中显示,肝硬化患者血清GOLM1最高。提示GOLM1蛋白作为肝良恶性疾病鉴别诊断的指标尚缺乏足够的证据支持。因此,进一步评价GOLM1对HCC的诊断价值具有重要意义。
为了进一步研究GOLM1是否可以作为HCC的特异性诊断标志物,并分析其预后价值,Oncomine在线工具可进行多个研究的Meta分析功能,本研究利用该数据库的mRNA表达数据进行在线分析,发现HCC组织中GOLM1 mRNA表达高于正常组织,肝硬化、不典型增生组织中的表达量也高于正常组织,由于在线数据分析功能所限,所以这些结果不能同时显示HCC、肝硬化、不典型增生、正常之间的比较结果。TCGA数据库mRNA测序结果也显示,GOLM1 mRNA在HCC中高表达,并与HCC患者的总生存时间相关。然而,我们的组织芯片结果显示,其蛋白水平变化在HCC与癌旁正常组织中差异无统计学意义;这些结果提示,癌组织GOLM1 mRNA水平变化可能比蛋白检测更适合于作为HCC的诊断标志物。
GOLM1 mRNA可能与HCC的发生、发展密切相关,为了更深入地了解GOLM1基因的调控,本研究利用生物信息学技术分析,分析了TCGA数据库中mRNA、miRNA和lncRNA之间的关系,发现了48个可能调控GOLM1表达的miRNA,及6个与GOLM1 mRNA表达呈正相关的lncRNA。lncRNA是近几年发现的具有重要调控功能的非编码RNA,其作用机制复杂,哺乳动物已发现的lncRNA数量多达10万条以上,很多lncRNA目前也仅仅知道其核苷酸序列,本研究中这6个lncRNA,多数功能还不明确,也无与GOLM1相互作用文献报道。其中仅LINC00511有癌基因的作用,分别在肺癌中通过EZH2/P57信号通路、在膀胱癌中通过Wnt/β-cantenin信号通路促进肿瘤的发生、发展[9-11]。
miRNA的功能研究相对较多,大多数miRNA和mRNA结合,诱导后者的降解,从而导致其功能抑制,但最近研究发现,胞核中的miRNA也具有调控基因转录功能。本文预测的GOLM1相关的miRNA中,一些已在肝癌中报道,其中以miR-200家族研究最多[12-14],多数具有抑癌基因作用,如miR-200c-5p可通过MAD2L1基因抑制HCC的增殖和转移[15]。另外,在肺腺癌中,miR-200a可抑制GOLM1,抑制肿瘤细胞的增殖[16]。本研究预测的这些miRNA对GOLM1的调控机制还需要更多的实验证实。

