TCGA数据库中肝癌相关差异长链非编码RNA筛选和功能预测
孙金旗, 马佳康, 任凯凯, 李 南, 李雨濛, 马 军,3
郑州大学第二附属医院 1.检验科; 2.肿瘤科,河南 郑州 450014; 3.郑州大学消化疾病研究所
肝癌是全世界高发的恶性肿瘤,其发病率也呈逐年增高的趋势[1-2]。近年来,虽然针对肝癌的研究取得不断进展,但仍面临着严峻的挑战。随着全基因组和转录组测序技术的发展,肝癌相关基因日益受到关注,其中的长链非编码RNA(long non-coding RNA,lncRNA)成为肿瘤研究的热点[3-4]。lncRNA是一种非编码RNA,其转录本的长度超过200个核苷酸,lncRNA可以和microRNA(miRNA)相互作用,作为一种相互竞争的内源性RNA(competing endogenous RNA,ceRNA)来调节靶基因的表达,这在肿瘤的起源和发展中扮演着重要的角色[5]。由美国政府发起的癌症和肿瘤基因图谱(TCGA)计划[6],通过应用基因测序技术,绘制人类全部癌症的基因组变异。本文下载了TCGA数据库中的肝癌转录组数据,利用perl语言和R语言进行系统分析,旨在找出肝癌相关基因,为今后的实验研究提供依据。
1 资料与方法
1.1一般资料使用网站专用的工具下载肝癌的mRNA基因表达数据和miRNA基因表达数据,收集患者的临床信息。肝癌的基因测序数据424例,男281例,女143例,其中包括癌组织374例,癌旁正常组织50例。基因表达量用于差异基因分析和ceRNA网络构建,患者的临床信息用于生存分析。
1.2差异基因分析链接http://bioconductor.org/biocLite.R网站下载R语言数据分析包“edgeR”和图形创建包“gplots”,设置文件路径读入基因表达数据,将癌组织的基因数据与正常组织的基因数据进行比对分析,在R语言的分析代码中设置差异倍数(foldChange)≥2,差异的显著性(padj:调整P值)<0.01。
1.3生存分析TCGA数据库中下载的患者临床信息包含了患者的生存状态及生存时间,提取生存状态和生存时间后,用R语言中生存分析包“survival”寻找生存相关的差异表达mRNA、lncRNA和miRNA。
1.4构建ceRNA网络利用TCGA数据库中差异表达的mRNA、lncRNA、miRNA,通过miRcode(www.mircode.org)来预测与差异lncRNA相互作用的miRNA,并与差异miRNA取交集。利用这些交集中的差异表达的miRNA在3个数据库(miRDB、miRTarBase、TargetScan)中做比对分析后找出差异表达的miRNA和差异表达的mRNA之间的对应关系。Cytoscape可以把三者之间的对应关系进行可视化处理,构建一个ceRNA网络图。
2 结果
2.1差异表达的mRNA、lncRNA和miRNA对肝癌的测序数据进行分析后,共识别出差异表达的mRNA 1 992个,其中高表达的基因1 787个,低表达的基因205个;差异表达的lncRNA1 082个,其中高表达的基因1 024个,低表达的基因58个;差异表达的miRNA 122个,其中高表达的基因119个,低表达的基因3个(见图1~2)。

注:横坐标:底数是10,矫正P值取负对数;纵坐标:底数是2,差异倍数取对数。黑色:无差异基因,红色:高表达差异基因,
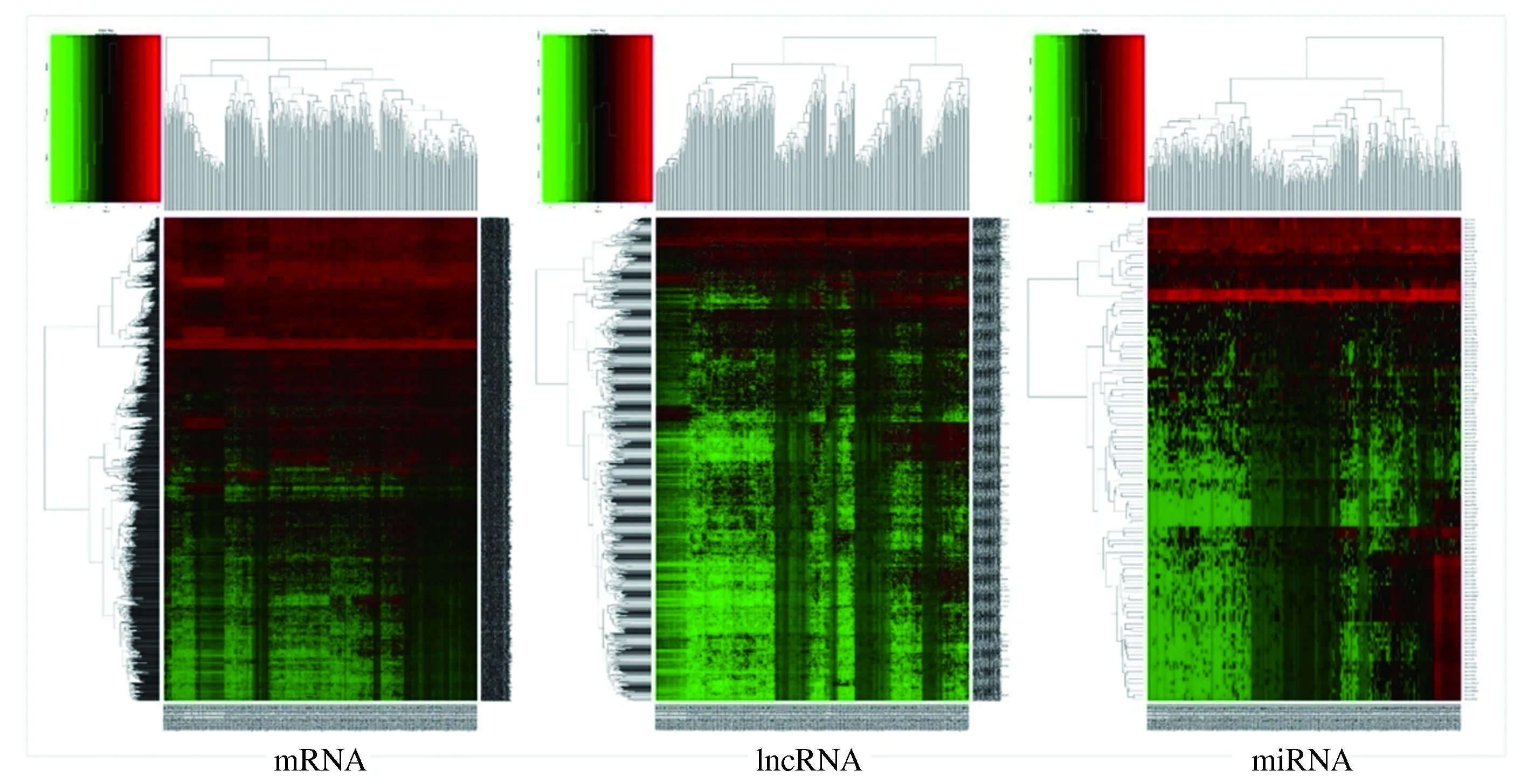
注:红色:高表达差异基因,绿色:低表达差异基因。
2.2生存分析为了寻找在肝癌中重要的lncRNA,我们通过miRcode数据库筛选和差异表达的miRNA相关的lncRNA 77个,对77个lncRNA进行Cox多因素生存分析,设置P<0.01为筛选标准,最终发现与肝癌预后相关的6个lncRNA组合(见表1、图3),其中高表达的有5个,与肝癌的生存期呈正相关;低表达的有1个,与肝癌的生存期呈负相关。与77个差异lncRNA相关的差异miRNA有16个,通过三个数据库(miRDB、miRTarBase、TargetScan)预测得到的靶mRNA和差异表达的mRNA取交集,最终获得与肝癌有关的mRNA有35个;同样设置P<0.01为筛选标准,对16个miRNA和35个mRNA做Cox多因素生存分析,发现与肝癌生存相关的miRNA有1个(见表2、图4),mRNA有20个(见表3、图5)。

表1 Cox回归分析中筛选的差异lncRNATab 1 Differentially expressed lncRNAs screened from Cox regression analysis

表2 Cox回归分析中筛选的差异miRNATab 2 Differentially expressed miRNAs screened from Cox regression analysis

表3 Cox回归分析中筛选的差异mRNATab 3 Differentially expressed mRNAs screened from Cox regression analysis
2.3构建ceRNA网络为了更好地阐明肝癌基因潜在的作用途径和网络,我们依据差异表达的mRNA、lncRNA和miRNA调控关系,构建了ceRNA网络关系图。使用Cytoscape-3.5.1对肝癌的相关基因进行了可视化,为了使图形更加清晰易读,我们只保留了有5个或5个以上连接点的lncRNA(见图6)。
在ceRNA网络中,包含了26个lncRNA,通过连接点计算后,我们发现有3个lncRNA TCL6、HOTTIP、PVT1与miRNA连接点≥10,推测这3个lncRNA可能在肝癌的发生、发展中起关键作用。有6个连接点以上的lncRNA还有16个,包含了肝癌生存期相关的3个基因AP002478.1、MYLK-AS1、C2orf48,这3个基因也可以作为后续肝癌研究中重点关注的对象。
我们还发现,低表达的hsa-mir-424和高表达的hsa-mir-519d也在肝癌的基因调控中发挥着重要作用。肝癌基因中与has-mir-424有关联的mRNA有13个,lncRNA有29个;与hsa-mir-519d有关联的mRNA有9个,lncRNA有23个(见图7)。hsa-mir-424和hsa-mir-519调控的mRNA中有10个与肝癌的生存后预后呈负相关,有1个mRNA(CPEB3)与肝癌生存后预后呈正相关。

图3 肝癌相关的6个lncRNA的生存曲线 Fig 3 Survival curves of 6 lncRNAs associated with liver cancer

图4 肝癌相关的1个miRNA的生存曲线 Fig 4 Survival curve of one miRNA associated
3 讨论
在消化系统常见的恶性肿瘤中,肝癌的死亡率排在第三,仅次于胃癌和食管癌,给人民的健康安全带来严重的危害[7]。随着分子生物学的进步和高通量基因组测序技术的发展,发现了越来越多的与癌症相关的基因,其中就包括lncRNA,在癌症发生和发展中起重要作用,lncRNA可通过表观遗传调控、RNA转录和功能调控、蛋白定位和活性调节、组蛋白修饰等机制调控癌症的发生、发展[8-9],在肝癌的发病、进展和癌细胞浸润和转移中发挥重要的调控作用[10]。
在我们的研究中,下载了TCGA数据库中的全部肝癌的转录组数据,确定了差异表达的lncRNA、miRNA、mRNA。然后又通过可视化的软件Cytoscape建立了ceRNA网络,揭示了三者之间相互作用的关系。生存分析的使用让我们了解到哪些基因与癌症的生存和预后有关,为下一步的实验提供依据。已经有很多相关的研究揭示了lncRNA和肝癌之间的关系,TSANG等[11]报道,HOTTIP的低表达会减少HOXA基因的表达,从而消弱肝癌的生长和转移。而HOTTIP的高表达会增加肝癌生长和转移的风险,在我们的研究中也证实肝癌患者中的HOTTIP是高表达的lncRNA。我们对包括HOTTIP在内的77个差异表达的lncRNA进行了生存分析,结果显示,低表达的HTR2A-AS1与肝癌的生存呈负相关,高表达的AC073352.1、AL359878.1、AP002478.1、C2orf48、MYLK-AS1与肝癌的生存呈正相关。通过miRcode的注释,和miRNA相互作用关系较强的lncRNA有TCL6、HOTTIP、PVT1,SU等[12]报道低表达的TCL6与肾细胞癌的预后不良有关,本研究中,TCL6是高表达的基因,ceRNA网络中显示TCL6作用的miRNA达到12个。TSENG等[13]发现在原发性人类肿瘤中,PVT1与髓细胞瘤癌基因表达相关,而在超过98%的髓细胞瘤癌基因复制增加的癌症中,PVT1的表达量也相应增加。lncRNA AP002478.1低表达提示肝癌预后良好,目前对这个lncRNA的功能还未见报道。

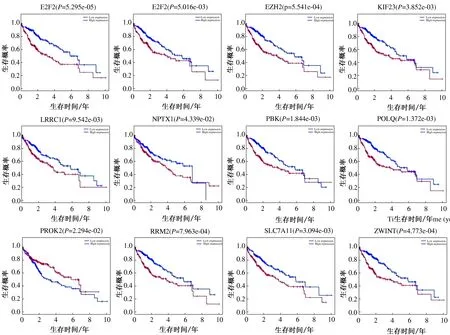
图5 肝癌相关的20个mRNA的生存曲线 Fig 5 Survival curves of twenty mRNAs associated with liver cancer
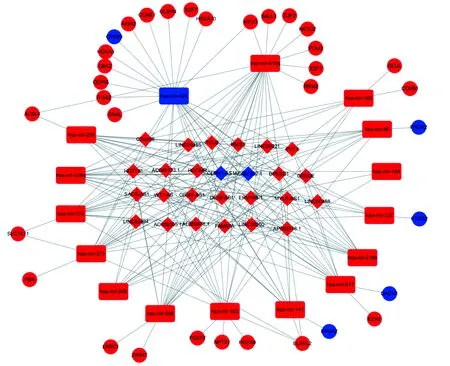
注:○表示mRNA,▭表示miRNA,◇表示lncRNA,红色是高表达基因,蓝色是低表达基因。

注:○表示mRNA,▭表示miRNA,◇表示lncRNA,红色是高表达基因,蓝色是低表达基因。
在ceRNA网络中,lncRNA可以作为miRNA“海绵”,对miRNA的作用有抑制作用。WANG等[14]研究发现,HOTAIR通过对hsa-mir-217的下调来促进肝癌的扩散和发展;MAHMOUDI等[15]观察到了hsa-mir-137在抑制癌细胞分化和增殖方面的作用。但我们通过374例TCGA数据库的肝癌与癌旁组织对比发现,低表达的hsa-mir-137肝癌患者存活时间更长,这些结果的差异还需要更多的证据来验证。其他两个关键的miRNA是hsa-mir-424和hsa-mir-519d,在ceRNA的网络中,has-mir-424和13个mRNA、29个lncRNA有相互调控的关系,has-mir-519d和9个mRNA、23个lncRNA有相互调控的关系调控,更值得关注的是,hsa-mir-424和hsa-mir-519调控的靶基因中有11个mRNA(E2F2、KIF23、POLQ、RRM2、E2F1、CCNE1、CLSPN、E2F7、CEP55、CBX2、CPEB3)与肝癌的生存期相关。据此推断hsa-mir-424和hsa-mir-519d在肝癌的调控机制中发挥着重要作用,针对hsa-mir-424和hsa-mir-519d的研究可能会对肝癌的发生、发展调控带来新的机遇。

