辽宁地区野生和栽培羊肚菌ITS序列及ISSR多态性分析*
曹 君,王 红,张 敏,刘俊杰,吕立涛
(辽宁省农业科学院 食用菌研究所,辽宁 沈阳 110161)
羊肚菌(MorchellaDill. Ex Pers.∶ Fr.)是可形成大型子实体的食药用真菌,属子囊菌门(Ascomycata)盘菌亚门(Pezizomycotina)盘菌纲(Pezizomycetes)盘菌目(Pezizales)羊肚菌科(Morchellaceae)[1],因其子囊果表面呈现不规则的凹陷,形似羊肚而得名,羊肚菌味道鲜美,营养丰富,是一种珍稀的食用菌品种,同时在中医记载中也可以作为药用菌。李时珍《本草纲目》(菜部二十八卷,菜之五,蘑菰蕈)记载“……一种状如羊肚,有蜂窠眼者,名羊肚菜”,主要功能为“益肠胃,化痰理气”。刘波《中国药用真菌》记载羊肚菌“益肠胃,化痰理气。治消化不良,痰多气短”[2]。
羊肚菌在北美、亚洲、欧洲均有分布,在中国大部分区域也有广泛的分布。辽宁地区羊肚菌研究起步较晚,以野生资源分布研究为主,主要记录地点集中于朝阳、沈阳、辽阳以及东部山区,成为当地重要的珍稀食用菌资源[3-6]。近年来,部分经选育后的南方羊肚菌品种进入辽宁,在温室大棚中实现了羊肚菌人工栽培。但是由于栽培面积、场地和技术所限,现有可人工栽培的羊肚菌产量有限,且栽培产品的风味、口感以及价格等与辽宁野生羊肚菌差距较大,因而尚未见辽宁本地野生菌种实现人工栽培成功的报道。
为解决传统形态学分类造成的同种异名和同名异种问题,DNA分子标记被引入应用于食用菌遗传多样性分析。其中基于PCR技术的ITS序列分析和ISSR多态性分析等技术由于其特异性优良、准确性较高以及操作较为简便等优点得到广泛的应用。ITS序列是rDNA基因中,5.8S rDNA和28S rDNA基因间隔序列,其相对进化速度较快,作为分子标记被广泛地应用于真菌的亲缘关系分析系统发育关系分析。Wipf等[7-8]对羊肚菌属部分种的ITS序列的多态性进行分析,并以此为依据将区分出黑色和黄色2个羊肚菌复合群。杜习慧[9-11]等利用包括ITS序列在内的多基因谱系一致性系统发育学物种识别法(genealogical concordance phylogenetic species recognition,GCPSR),首次系统地进行了中国范围内羊肚菌资源的系统发育研究,并确定了中国分布的30个羊肚菌物种,包括11种未命名物种。此外,许多研究均证明ITS序列对于不同区域范围内羊肚菌野生或栽培资源的分类鉴定具有重要作用[12-16]。ISSR技术是由SSR技术发展而来,具有模板质量要求低、多态性丰富等优点,可以进行种内个体遗传差异性分析。刘文丛等[17]通过ISSR聚类分析将云南西北地区11个羊肚菌种群56个样品分为三大组,证明不同的分组与地理距离有明显的相关性。魏巍等[18]对四川地区16个野生羊肚菌样品进行ISSR聚类分析,并发现不同遗传背景个体的代谢产物或活性物质存在明显的差异。
本研究综合运用ITS序列分析和ISSR多态性分析技术,在分子水平上研究辽宁地区引进的羊肚菌人工栽培菌种和本地野生菌种之间的遗传多样性,为扩大羊肚菌人工栽培菌种范围,合理开发和保护辽宁野生羊肚菌资源,实现辽宁野生品种人工栽培提供理论依据。
1 材料与方法
1.1 试验材料
人工栽培羊肚菌样品为2015年~2016年于北镇、苏家屯、岫岩、宽甸等地日光温室中采集,共收集样品15株。野生羊肚菌样品为2016年~2017年从辽宁省野生羊肚菌主要产区朝阳、凌源、康平以及沈阳东陵地区等地采集,共收集野生样品14株,见表1,样品采集后置于密封袋中低温保存。
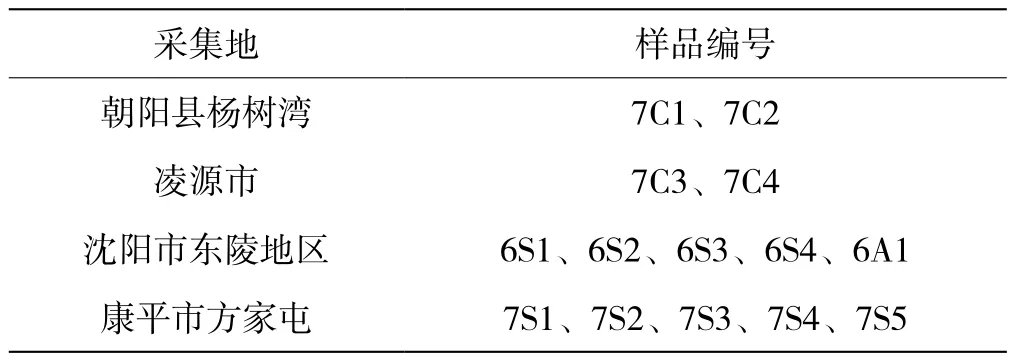
表1 野生羊肚菌样品采集信息Tab.1 Information of Morchella samples collcted
1.2 总DNA提取
取子囊菌菌盖内部组织0.3 g左右,低温充分研磨后,采用E.Z.N.A. Fungal DNA Kit试剂盒提取总DNA,提取物经1.0%琼脂糖凝胶电泳检测后分装,于-20℃条件下保存。
1.3 ITS序列扩增与数据分析
ITS序列PCR扩增建立20 μL的PCR反应体系,如表2所示。其中2条PCR引物分别为ITS1(5’-TCCGTAGGTGAACCTGCGG-3’)和 ITS4(5’-TCCTCCGCTTATTGATATGC-3’)。PCR 扩增反应程序为95℃预变性4 min,然后95℃变性30 s,57℃退火30 s,72℃延伸60 s,30个循环,最后72℃延伸7 min。PCR反应产物经1.0%琼脂糖凝胶电泳检测后,送检目的条带进行测序。根据ITS测序结果利用Mega 6.0软件进行分析,并构建系统发育树。

表2 PCR扩增反应体系Tab.2 PCR reaction system
1.4 ISSR-PCR扩增与数据分析
ISSR多态性分析PCR扩增建立20 mL的PCR反应体系,如表2所示。根据武冬梅[19]所用引物进行进一步的多态性筛选,PCR扩增反应程序为94℃预变性5 min,94℃变性45 s,53℃退火45 s,72℃延伸60 s,30个循环,72℃延伸7 min。PCR反应产物经1.0%琼脂糖凝胶电泳检测。
电泳结果通过人工分辨统计条带,其中清晰度高、重复性好、分辨率高的条带可以用于ISSR多态性分析。在Excel表格中将图形资料转换成数据资料,即通过“0/1”表型数据矩阵的二元数据模型来表现ISSR扩增结果,每条可阅读到的条带可视为一个等位基因赋值为1,不同样品中同一位置无条带的赋值为0,由此建立原始数据矩阵,从中筛选具有足够多态性的ISSR引物。获得的高多态性ISSR数据,应用NTSYS-pc 2.1软件计算样品之间的相似系数,并进行UPGMA聚类分析。
2 结果与分析
2.1 ITS分子鉴定
试验样品提取总DNA经ITS序列扩增测序后,所得数据与羊肚菌多基因序列模标数据库MorchellaMLST(http∶//www.cbs.knaw.nl/morchella/)进行比对,15株栽培菌株属于黑色系羊肚菌类群的Morchella sextelata(Mel-6)、Morchella septimelata(Mel-7)和Morchella importuna(Mel-10),而 14株野生菌株也均为羊肚菌属物种,属于黄色系的Morchellasp.(Mes-6)和Morchellasp.(Mes-9)。应用 mega 6.0软件,以肋状皱盘菌Disciotis venosa(Pers.)Boud为外群,bootstrap次数1 000,采用最大似然法(maximum likelihood methods,ML)进行构建系统发育树,见图1。
从图1可见,15株栽培菌株与14株野生菌株完全分离。其中栽培菌株中Y15Z001、Y16Z003、Y16Z007、Y16Z009和 Y16Z016与Mel-10聚 为 一支(bootstrap值100%),证明这5株栽培羊肚菌均在系统发育学分类上为梯棱羊肚菌(Morchella importuna,Mel-10);菌株Y16Z017与Mel-7聚为一支(bootstrap值98%),证明为七妹羊肚菌(Morchella septimelata,Mel-7);其余9株栽培菌株均与Mel-6聚为一支(bootstrap值94%),证明为六妹羊肚菌(Morchella sextelata,Mel-6)。野生羊肚菌样品中,6S2、7C1、7C2、7C3、7C4、7S1、7S2、7S3、7S4、7S5与Mes-6聚为一支(bootstrap值95%),而6A1、6S1、6S3和6S4与Mes-9聚为一支(bootstrap值87%)。

图1 基于ITS序列构建的羊肚菌系统发育树Fig.1 Phylogentic tree based on rDNA ITS sequence
2.2 ISSR-PCR扩增结果
ISSR引物通过进行稳定性、特异性和重复性筛选,其中12条引物的扩增位点多态性较好且条带较为清晰。同过对已收集到的29个羊肚菌菌株进行扩增,12条引物共获得114个位点,片段大小分布在300 bp~2 000 bp,其中具多态性的位点106个,占总位点数的93.0%,每条引物多态性位点数为5个~12个,平均8.83个,见表3。
2.3 ISSR聚类分析
应用NTSYS-2.10e软件对采集的14株羊肚菌野生菌株和15株栽培菌株的ISSR扩增结果合并进行聚类分析,得到聚类图如图2所示,相关性系数r=0.93136,聚类结果较好。

表3 用于ISSR分析的12条引物及其扩增位点Tab.3 12 primers a and their amplification sites for ISSR
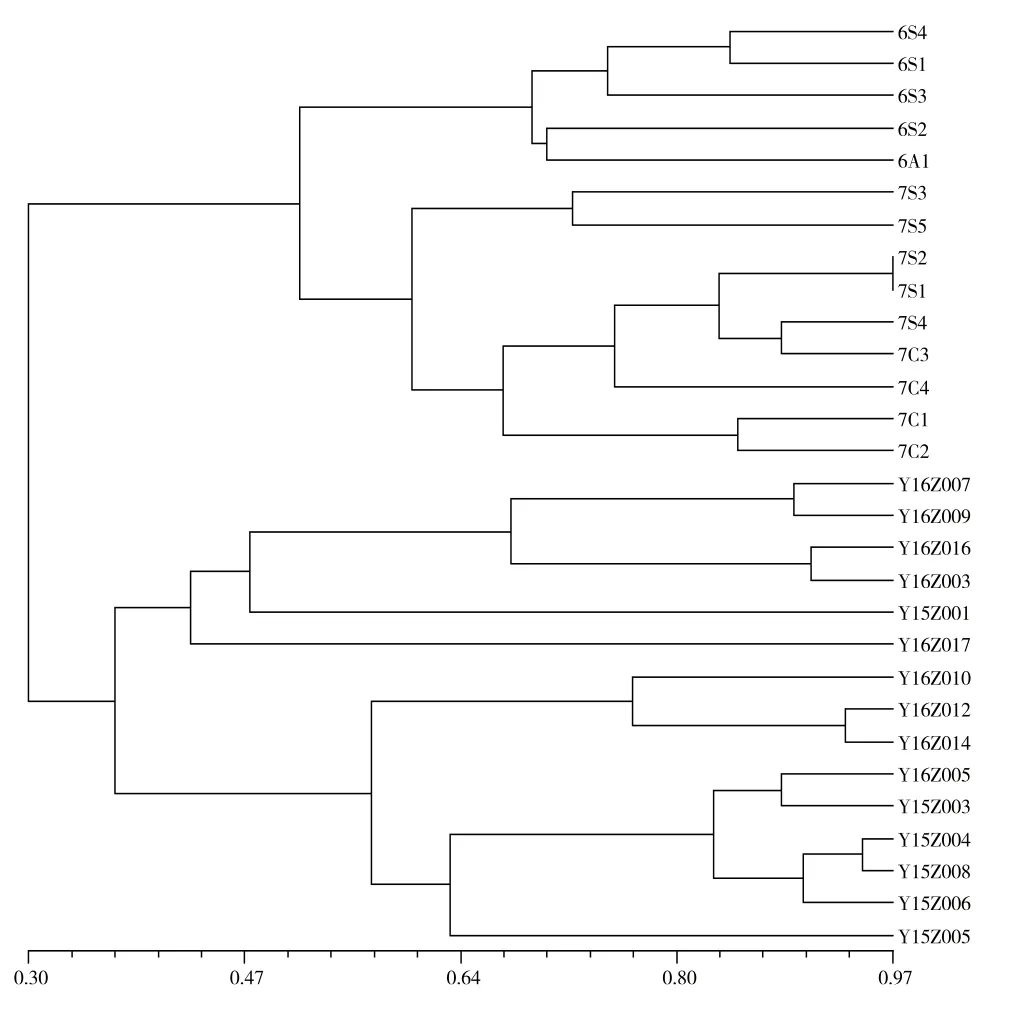
图2 羊肚菌ISSR分析遗传聚类图Fig.2 UPGMA dendrogram based on result of the ISSR
由图中2可见,在相似水平在37%左右时,供试样本分为野生和栽培2个大类群。在栽培类群中,相似水平43%左右时,收集的栽培菌株分为2个亚类群,Y15Z001、Y16Z003、Y16Z007、Y16Z009、Y16Z016和Y16Z017为聚为一支,其中Y16Z017在相似水平42%左右时又与该亚类群中其他5株样品分开,而其他9株栽培菌株样本在相似水平57%左右时仍然聚为一支。这一结果与ITS序列分析中Mel-10、Mel-7和Mel-6三种人工栽培羊肚菌系统发育种分类结果相同。
野生菌株中,相似水平50%左右时,分为2个亚类群,7S3、7S5、7S2、7S1、7S4、7C3、7C4、7C1和7C2聚为一个亚类群,其中7S1和7S2在相似水平达到97%时仍然聚为一支,遗传相似性最大,说明这2个菌株的亲缘关系最近。而6S4、6S1、6S3、6S2和6A1在相似水平68%左右时仍然聚为一支。
综合基于ITS序列构建的ML树和基于ISSR多态性位点构建的UPGMA聚类分析结果,二者均表明辽宁地区的野生羊肚菌资源与现有栽培品种具有明显的差异,遗传相似性较低。栽培菌株中,两种遗传标记均可以对Mel-6、Mel-7和Mel-10进行区分,但是ML系统发育树中三者间分枝的bootstrap值较低(≤80%),且ISSR聚类分析中Mel-7和Mel-6相似性较高,与ITS数据分析存在差异,表明在数据上仍存在一定的争议性。而野生菌株样品划分出的两个亚类群中,6S2在ML系统发育树中归于Mes-6,而在ISSR数据分析中与6A1、6S1、6S3和6S4(Mes-9)相似性更高,表现出较大的差异性,这可能是与两种遗传标记进化速度的差异有关。
3 结论
根据杜习慧[20]等观点,中国羊肚菌分为黑色和黄色2个复合群,现有的可实现人工稳定栽培的Mel-6(六妹)、Mel-7(七妹)和Mel-10(梯棱)等均属于黑色羊肚菌复合群,而辽宁地区野生羊肚菌则属于黄色羊肚菌类群。近几年来,由于羊肚菌市场价格远高于香菇、木耳等传统食用菌品种,羊肚菌的人工栽培成为农业产业发展的新热点。辽宁地区的种植面积也在不断地扩大,但所用的黑色野生羊肚菌组织分离菌种均原产于四川等地,由于生境变化剧烈,这些未经过人工改良或选育的菌种,在日光温室条件下也容易因外界气候变化等因素造成生产过程中病害严重、子实体品质下降等问题。因此亟需充分利用本地野生羊肚菌资源,经驯化和改良后进行人工栽培,从而满足辽宁地区羊肚菌的生产需求。本研究为实现本地野生羊肚菌资源的合理利用,选育适合辽宁地区环境特征的羊肚菌品种做好基础准备和技术支撑。
本研究利用DNA分子标记对辽宁省内15株羊肚菌样本及14株野生资源样本进行了分类鉴定和遗传多样性分析。综合基于ITS序列构建的ML树和基于ISSR多态性位点构建的UPGMA聚类分析结果,证明了与引种自四川等地的栽培菌株相比,辽宁地区野生羊肚菌具有非常大的遗传差异性。表明辽宁地区的野生羊肚菌资源与现有栽培品种具有明显的差异,遗传相似性较低。栽培菌株中,两种遗传标记均可以对Mel-6、Mel-7和Mel-10进行区分,结果与杜习慧等人研究结果相似。但是ML系统发育树中三者间分枝的bootstrap值较低(≤80%),且ISSR聚类分析中Mel-7和Mel-6相似性较高,与ITS数据分析存在差异,表明在数据上仍存在一定的争议性。需要在后续研究中引入ef1-α、rpb和LSU rDNA等遗传标记进行多基因联合分析,提高遗传信息的分析精度。而野生菌株样品划分出的2个亚类群中,6S2在ML系统发育树中归于Mes-6,而在ISSR数据分析中与6A1、6S1、6S3和6S4(Mes-9)相似性更高,表现出较大的差异性,这可能是与两种遗传标记进化速度的差异有关,还可能是ISSR多态性位点对于所选样本基因覆盖程度不足造成的,在后续试验中需要进一步筛选更多、更为合适的ISSR多态性位点。

