基于重组酶和终止子的状态调控开关设计
张嵩元,邱建辉,王宣,董一名,李昱龙,张益豪,欧阳颀
基于重组酶和终止子的状态调控开关设计
张嵩元1*,邱建辉1*,王宣2,董一名1,李昱龙1,张益豪1,欧阳颀1
1 北京大学 定量生物学中心与生命科学联合中心,北京 100871 2 清华大学 生命科学学院与生命科学联合中心,北京 100084

张嵩元, 邱建辉, 王宣, 等. 基于重组酶和终止子的状态调控开关设计. 生物工程学报, 2018, 34(12): 1874–1885.Zhang SY, Qiu JH, Wang X, et al. Design of recombinase and terminator-based genetic switches for cell state control. Chin J Biotech, 2018, 34(12): 1874–1885.
合成生物学研究常用基因开关来调控细胞的状态以实现相应功能。已有的基因开关往往需要持续的输入信号来维持特定的开关状态,开关功能的维持需要持续消耗能量,并且对扰动较为敏感。文中利用了位点特异性重组酶的倒位效应反转终止子,构建了一种转录层次的状态调控开关,使得脉冲信号即可触发开关状态改变,并在下一次信号来临前稳定维持当前状态。应用自下而上的工程化思想,文中先后对重组酶和终止子进行了单独表征和组合表征,探究了二者之间的相互影响,筛选出了相互兼容的组合,成功实现了细胞的单次、二次状态切换。最后,此开关成功地被用于构建生物七段译码器,显示出了其较好的应用潜力。
位点特异性重组酶,终止子,调控开关,状态转换
生命体中发生的许多生物过程都可以抽象为“开关” (Switch)[1]。如细胞分化时,某些外界信号触发“分化开关”,使得干细胞基因选择性表达以执行特定功能。正常情况下,这种状态切换是非常稳定的。一旦发生,即使分化信号不再存在,细胞的状态也不容易被逆转,除非再次接受一组特定脱分化信号的刺激。一些构象可改变的蛋白也具有“开关”的性质,如光受体蛋白[2]、压力[3]/电压[4]/配体[5]门控离子的通道蛋白等。当外界信号(光、小分子物质、电位差) 存在时,蛋白被活化以执行功能,当外界信号不复存在时蛋白立即变为失活状态。
目前在合成生物学领域,多种自然或人工的“开关”已经投入生物学应用中,如诱导型启动 子[6]、光敏离子通道[7-8]、Toehold Switch[9]等。这些开关大多需要输入信号的维持才可保持在开启状态,这个过程会不断地消耗能量,状态维持往往依赖于细胞内蛋白、核酸分子等的浓度维持在某一平衡浓度,外界干扰诸如细胞分裂,细胞缺乏营养等很容易打破平衡,虽然这些开关已经用于多种场景,但它们难以像细胞分化那样进行稳定的、自身可持续的状态切换。
上述依赖外界信号维持状态的“开关”具有一个共同特点——状态切换对应着生物大分子结构变化。我们可以想象,当生物大分子的序列而不是结构发生变化时,尤其是DNA序列发生变化时,“开关”造成的状态切换将是稳定的且是可自我维持的。
位点特异性重组酶 (Site-specific recombinases,SSRs) 介导的重组反应被广泛用于遗传工程操作中来改变DNA序列,如著名的Cre/LoxP和Flp/FRT基因敲除系统。SSRs具有一对特异的识别位点 (Recognition site)——attB位点和attP位点。根据重组位点的序列和排列的方向性,位点特异性重组有3种可能的结果,即整合(Integration)、切离(Excision) 和倒位(Inversion),如图1所示,当两位点反向位于同一DNA上时,将产生倒位生成attL位点和attR位点[11]。位点特异性重组系统可分为两类:酪氨酸重组酶(Tyrosine recombinase) 和丝氨酸重组酶(Serine recombinase),后者中的大丝氨酸重组酶亚家族的识别位点简单且高度特异,且反应方向控制严 格——只有当重组定向性因子(Recombination directionality factor,RDF) 存在时才能识别attL与attR序列,逆转上述反应重新生成attB、attP位点[12]。
除了传统的基因敲除,Timothy K. Lu研究团队利用重组酶整合单链DNA进入基因组[13]或直接翻转DNA序列[14]成功开发了多种DNA存储器,使细胞以DNA序列为载体“记录”外界事件。在医疗领域中,研究者通过在肠道益生菌中构建这种存储器,可以实现对肠道环境的检测[15]。研究者可利用重组酶翻转启动子、终止子、编码序列等有功能基因元件构建新型逻辑线路,使细胞能够对外界信息产生更复杂的响应模式[14,16-17]。在此过程中一系列具有良好正交性的重组酶被筛选出来,如大丝氨酸重组酶亚家族的phiC31、Bxb1、TP901-1和酪氨酸家族的Int系列等[18]。
终止子是合成生物学中重要的元件,可用于终止转录、基因线路间的绝缘等。以其是否依赖于ρ因子可以分为两类:依赖ρ因子的终止子需要额外的ρ因子终止转录[19],不依赖ρ因子的终止子可利用自身形成茎环结构及polyU序列终止转录过程[20]。此外,根据是否在两个转录方向上均具有转录终止能力可分为双向(Bidirectional)终止子[21]和单向(Unidirectional) 终止子。2013年Chen等报道了其表征的来自大肠杆菌的和人工设计的共计582个终止子的元件库[22],我们可以从这个库中挑选适合的终止子用于转录调控开关的设计。

图1 当两个识别位点排列方式不同时重组酶表现插入/删除/翻转效应
在本研究中,我们利用位点特异性重组酶的倒位效应翻转终止子,构建了一种稳定的转录状态调控开关。首先我们单独表征了终止子和重组酶的性能:通过测试正向反向接入时的转录终止能力从终止子库中筛选出单向强终止子;通过测试重组酶的翻转效率得到最优的重组酶-RBS-诱导物浓度组合。然后我们分别将一对、两对重组酶识别序列引入终止子两侧。考虑识别序列与终止子的互相影响,我们再次测试了转录终止能力,筛选并表征了多组兼容的组合。接着,使用相应的重组酶作用于这些组合成功地实现了“开关”功能,并表征了此系统的性能。最后,通过组合上述开关制成了“生物七段译码器”,实现了数字显示图像的切换。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
TOP10,购自北京博迈德基因技术有限公司。
pTAC-pSB4C5 (pSC101 ori,repA,CmR,lacⅠ,pTAC,RiboJ,sfGFP),由pSB4C5质粒(parts.igem. org/Part:pSB4C5)改造而成,IPTG诱导后可表达增强型绿色荧光蛋白。RiboJ作为绝缘子防止RBS上游非翻译区对翻译的影响[23]。启动子和RBS间可连入终止子及重组酶识别位点,用于表征终止子性能,或作为重组酶的作用靶标,以实现状态切换的开关功能,作为“报告质粒”,保存于本实验室。
pBAD诱导表达质粒(ColE1 ori,KanR,AraC,pBAD,RBS,Recombinase),作为“表达系统”在阿拉伯糖诱导下表达重组酶,为状态切换提供驱动力。RBS序列插入位点两端含一对BⅠ位点,用于更换RBS以探寻最适重组酶表达量。保存于本实验室。
pTAC诱导表达质粒(p15A ori,AmpR,LacI,pTAC,RBS,Recombinase),在IPTG诱导下表达重组酶,功能与pBAD诱导表达载体相同。保存于本实验室。
重组酶测试质粒(pSC101 ori,CmR,RFP,attB,pTAC,GFP),用来表征重组酶效率。包含一个可以被翻转的pTAC启动子,翻转前后分别表达绿色、红色荧光蛋白。保存于本实验室。
1.1.2 主要试剂
LB培养基与M9培养基。质粒小提试剂盒(康宁生命科学(吴江) 有限公司)。胶回收试剂盒(天根生化科技(北京) 有限公司)。2×EasyPCR SuperMix (北京全式金生物技术有限公司)。T4 DNA连接酶和限制性内切酶RⅠ、Ⅰ、Ⅰ、Ⅰ,BⅠ(NEB)。诱导物:IPTG、阿拉伯糖。
1.2 方法
1.2.1 终止子性能的表征
初步选择:我们根据已有的对自然及人工终止子的表征数据[22]初步选择一些具有较强转录终止能力的终止子进行进一步测试。
质粒构建:终止子性能表征的基因线路如图2A所示。利用先合成单链寡核苷酸再退火的方法得到一双链DNA片段,其两端含有Ⅰ位点,中间是一段随机序列的双链DNA片段。该序列与普通终止子长度相似(50 bp),含50%GC,已证明对转录无影响[14]。利用RⅠ、Ⅰ双酶切pTAC-pSB4C5质粒并胶回收,连入上述短片段。得到的质粒可以利用Golden Gate assembly 法一步快速替换随机序列为各种终止子。同时,由于此处随机序列可认为不影响转录,故此质粒可作为终止子表征时的阳性对照。对于每一个候选终止子,利用先合成单链寡核苷酸再退火的方法,分别得到其正向(Forward,F) 与反向(Reverse,R) 双链DNA片段(规定正向为其在生物体中自然存在方向或能发挥转录终止功能的方向)。该片段中,终止子序列两端设计有Ⅰ酶切位点。利用Golden Gate assembly法将片段插入前述质粒中。分别在诱导型启动子后与sfGFP上设计引物,菌落PCR后测序检测是否成功连入。
诱导培养:挑平板菌落于含抗生素的LB培养基中,37 ℃、250 r/min振荡培养12 h。取培养物1∶100稀释于深孔板中,37 ℃、1 500 r/min培养2 h。取少量培养物用含抗生素的 M9培养基1∶100稀释于另一深孔板中,加入IPTG设置终浓度为0.01 mmol/L、0.1 mmol/L和1 mmol/L,同上条件培养6 h。取少量培养物用含2 mg/mL卡那霉素或35 μg/mL氯霉素的PBS缓冲液1∶5稀释于96孔板中,以抑制蛋白质生成。空白对照组为不转上述质粒并经过相同培养步骤的.TOP10。
流式细胞仪检测:用流式细胞仪(BD LSRFortessa™) 的488 nm激发光检测细胞的绿色荧光强度,设置进样体积(Injection volumn) 为15 μL,流速(Flow rate) 为1 μL/s,每孔样品最小样本量为10 000个细胞。流式数据用FlowJo®软件分析得到荧光强度平均值。
表征:各组样品平均值减去通过空白对照组测得的本底荧光,得到净荧光强度。定义终止子强度(Terminator strength,Ts) 为阳性对照(含不阻止转录的随机序列)的荧光强度([GFP]random) 与实验组(含终止子) 荧光强度([GFP]ter) 的比值。
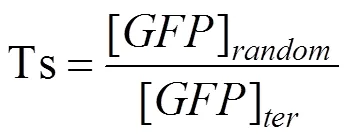
该值越大表明终止子的转录终止能力越强。若 反向接入时该值小于1说明终止子是个潜在的启动子[24] (Cryptic promoter)。通过计算每一终止子的正 向与反向Ts的比值来验证终止子是否为单向的(Unidirectional),该值越大说明终止子正反向接入的作用差异越大,更具有“开关”的性质。基于以上两个计算值,我们制定了终止子的筛选标准:正反向的Ts值均大于0.9 (没有潜在启动子);正反向Ts比值大 于10 (ON、OFF状态差异足够明显);正反向中较 大的Ts大于10(OFF状态时基因表达受严格抑制);正反向中较小的Ts不大于1.2 (ON状态时基因表达 基本不被抑制)。满足标准的终止子进入下一步筛选。
1.2.2 重组酶翻转效率的表征及表达系统的改进
我们从RBS Calculator[25]和iGEM元件库中得到了一系列RBS序列,先合成单链寡核苷酸再退火得到双链DNA。利用Golden Gate assembly法更换RBS序列,构建不同表达强度的pBAD诱导的Bxb1、phiC31重组酶表达质粒和pTAC诱导的TP901-1、Int2重组酶表达质粒。将其与重组酶测试质粒共转入.TOP10中以构建重组酶效率测试体系(图3A)。在双抗平板上挑取单菌落于LB培养基中37 ℃、1 000 r/min振荡培养12 h。取2 μL菌液加入含抗生素和不同浓度诱导物(0、10–5、10–4、10–3、10–2、10–1、100、101、102mmol/L)的M9培养基中,深孔板37 ℃、1 000 r/min诱导培养15 h。取2 μL培养物加入198 μL PBS中,用流式细胞仪测量诱导结果,通过统计细菌表型可以看到是否发生翻转,利用FlowJo®软件分析可得重组酶效率。
1.2.3 终止子两侧引入一对重组酶识别位点
终止子的转录终止能力依赖RNA形成的茎环结构。考虑到重组酶识别序列会同样具有特殊构象,这不但会影响终止子功能,其本身也会对转录造成一定影响,我们进行了第二轮筛选,以得到互相兼容的识别位点-终止子组合,基因线路如图2B所示。

图3 重组酶翻转效率与表达系统的优化
质粒构建:以1.2.1中pTAC-pSB4C5的酶切胶回收产物为载体,酶切连接法连入一短片段,得到的质粒可以作为插入重组酶识别位点以及终止子的载体。为了方便高通量构建,我们在两个重组酶识别序列插入位点两端各设计了一对BⅠ切割位点,终止子两端设计了一对Ⅰ切割位点,这样就可以用Golden Gate assembly法一步高效替换重组酶识别序列和终止子,形成不同组合。重组酶识别位点信息通过单链退火的方法得到(序列请见2017.igem.org/Team:Peking)。其余构建、检验步骤同1.2.1。阳性对照组含有某重组酶识别序列但终止子以随机序列替代。诱导培养步骤与1.2.1相同。
酶标仪检测:我们采用酶标仪(Thermo Scientific Varioskan©Flash) 粗测样品的荧光强度(激发波长485 nm、510 nm发射波长) 与值,取二者比值则为单位数量细菌的相对荧光强度。与1.2.1中的单细胞平均荧光强度具有相同统计意义。公示表征步骤同1.2.1。筛选得到表现良好的组合做进一步筛选。
1.2.4 终止子两侧引入两对重组酶识别位点
产生多种状态需要重组酶的多次作用,故需要多对重组酶识别位点。两对识别位点将对终止子的开关性能产生更大影响,故需要进一步表征筛选。含两对识别位点的报告质粒如图2C所示。
质粒构建:将pTAC-pSB4C5的诱导型启动子删除,更换为组成型表达的J23119启动子。合成一段双链DNA片段连入该质粒。该片段含两对嵌套的重组酶位点与一个终止子。外侧的重组酶位点固定为一表征较好的高效重组酶,内侧位点及终止子按1.2.2设计并可以更换。诱导培养、酶标仪检测、公式表征步骤同1.2.2。
1.2.5 重组酶翻转终止子性能表征

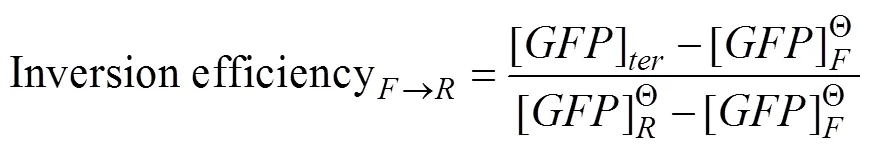
两次翻转:将报告质粒与对应的两个重组酶表达质粒共转入.TOP10中,三抗平板上培养。其他步骤同一次翻转。
1.2.6 演示:七段译码器三状态转换
由1.2.3和1.2.5的实验,我们得到了基于重组酶翻转作用和终止子的基因“开关”。当终止子两侧引入两对识别位点时,意味着可以通过控制两种重组酶表达,对终止子进行两次翻转。算上初始状态,该开关可以产生3个状态(即“开-关-开”或“关-开-关”)。
电子器件中常见的七段译码器示数变化“1-2-3”可作为上述过程的形象演示。如图5A所示,利用1.1.1–1.2.5的材料与方法我们可以在96孔板上实现“生物基的七段译码器”。考虑每个显示管的明暗变化顺序,7个显示管有5种状态转换模式(000、111、011、010、101,0、1分别代表暗、亮),因此要使用5种报告质粒与重组酶表达质粒的组合。每次手动状态切换时,将上一状态的菌液1∶100稀释于含相应抗生素和诱导物(依次10 mmol/L阿拉伯糖、0.1 mmol/L IPTG) 的M9培养基中振荡培养12 h得到后一状态。诱导结束后依据1.2.2中方法得到单位数量细菌的荧光强度,并在蓝光板上对其拍照。
2 结果与分析
2.1 终止子性能的表征
共选取24个已报道的强力终止子纳入我们的系统中进行表征(序列请见2017.igen.org/Team:Peking)。
比较4个IPTG浓度梯度下的Ts,我们发现随诱导物浓度加大,对于具有较强转录终止能力的终止子方向,其Ts总体呈现先迅速上升后略微下降的趋势。若某一方向转录终止能力弱,变化趋势则不明显(图2D嵌入图以强力终止子435的Ts随诱导物浓度变化的趋势为例)。同时我们发现高浓度诱导物会对细胞产生一定代谢负担和毒性,故选择0.1 mmol/L IPTG为最终诱导浓度。24个终止子依据其正反向Ts可分为单向终止子与双向终止子(图2D)。我们最终得到了6个符合前述4条标准的终止子(435、322、309、221、836、855),其中435表现最佳,正向Ts高于1 000,反向Ts接近1,是典型的单向强终止子。
2.2 重组酶翻转效率的表征及表达系统改进
我们所应用的RBS来源于RBS calculator[25]和iGEM网站(序列请见2017.igem.org/Team:Peking,纯数字代号的来自RBS calculator,“B00XX”来自iGEM Parts,“字母+数字”由B0034突变获得)。图3B显示测试质粒被重组酶翻转前后流式细胞仪的测试结果,横轴为红色荧光强度,纵轴为绿色荧光强度。翻转前细胞位于对角线以上,主要表达GFP,翻转后细胞位于对角线下,主要表达RFP。我们把诱导表达重组酶后对角线下的细胞比例定义为重组酶效率。重组酶和phiC31利用各个RBS在不同阿拉伯糖诱导浓度下的效率如图3C所示。我们发现在不加诱导物时均有一定程度泄露表 达,低浓度诱导物时重组酶效率变化不大,但在10 mmol/L时效率有一个阶跃式上升,最高可达90%左右。IPTG诱导也有类似的结果。因此选择10 mmol/L阿拉伯糖和0.1 mmol/L IPTG作为最 适诱导浓度。进而测试了各重组酶在最适诱导浓度下应用不同RBS时的效率(图3D),最终选择878、1 668、A6分别作为Bxb1、TP901-1、phiC31的RBS,因为其泄露较低,最适诱导浓度时效率较高。
2.3 终止子两侧引入一对重组酶识别位点
单独的重组酶位点也会对转录造成一定影响,可被视为“终止子”。测试的重组酶位点中,Int2有最强的“终止子”效应(Ts=23.0),甚至强于221F (Ts=16.6)。其他较为微弱:phiC31 (Ts=2.00)、TP901-1 (Ts=1.42)。
共测试了14个重组酶位点+终止子组合的Ts值(图2E)。编号phiC31 435代表终止子ECK120034435两侧有重组酶phiC31的识别序列,其他以此类推。在所有组合中,int2 221拥有最强的转录终止效应(Ts=35.3)。满足标准的其他组合依次为phiC31 435 (28.3)、Bxb1 435 (21.1)、TP901-1 221 (17.7)、TP901-1 435 (17.3)、int2 855 (17.2)。
值得注意的是,重组酶识别序列与终止子的Ts并非为线性叠加关系。例如,int2与435F在识别序列与终止子中各自具有最大的Ts值,但是二者组合的Ts值在所有组合中最弱(Ts=5.37)。TP901-1与221F在识别序列与终止子中各自具有最小的Ts值,但是二者组合在一起效果则很强。此外,二者的组合可能在序列内部形成启动子,比如TP901-1 221R。
考虑到int2与TP901-1的重组酶表达质粒的启动子为pTAC,较pBAD泄露较大。我们选择Bxb1 435与phiC31 435进行1.2.5中的一次翻转测试。
2.4 终止子两侧引入两对重组酶识别位点
我们选取了翻转效果较稳定的重组酶Bxb1、 phiC31、TP901-1的识别位点,与终止转录效果最好的单向强终止子435组合在一起进行性能表征,结果如图2F所示。最终,我们选取了性能最好的组合结构“Bxb1 attB-TP901-1 attB-435- TP901-1 attP-Bxb1 attP”和“Bxb1 attB-phiC31 attB-435-phiC31 attP-Bxb1 attP”进行1.2.5中的两次翻转实验。之后将其分别简称为Bxb1 TP901-1 435和Bxb1 phiC31 435。
2.5 重组酶翻转终止子性能表征
将报告质粒Bxb1 435和phiC31 435与相应的pBAD诱导表达质粒共转进行了重组酶一次翻转终止子的实验(图4A)。计算结果(图4C)显示有些翻转效率 (Inversion efficiency)超过100%,有些则不足50%,与2.2.3中理想值偏差较大。经过分析,可能存在如下原因:第一,attB-F-attP经过翻转得到attL-R-attR,而不是我们计算inversion efficiency所使用的attB-R-attP的理想状态;第二,重组酶作用于pTAC-pSB4C5质粒翻转终止子的效率与2.2.2中重组酶作用于重组酶测试质粒翻转启动子的效率存在不同,可能是终止子影响识别位点的空间结构,造成翻转效率的变化。
两次翻转使用报告质粒Bxb1 TP901-1 435和Bxb1 phiC31 435及其对应的重组酶表达质粒(图4B),得到的结果如图4D所示。
2.6 演示:七段译码器三状态转换
依照1.2.6我们得到了图5B所示的结果。每个加样孔中的数字代表单位的荧光强度。单凭肉眼可以观察到较为明显的状态变化,数字图案清晰可辨,视觉上成功实现了生物七段译码器。数据上若以50作为定义开关状态转换的阈值,亦可判定实验的成功。
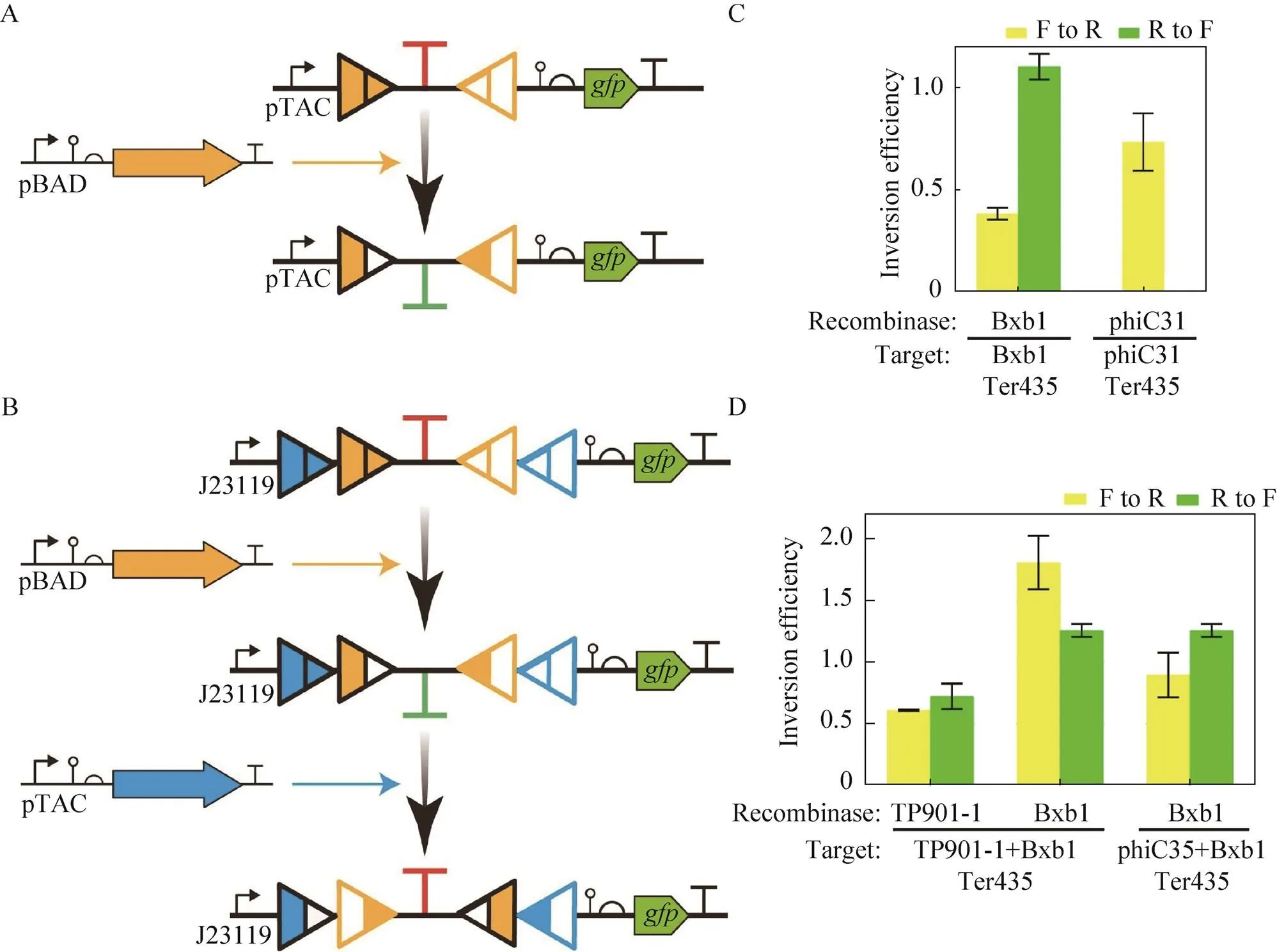
图4 重组酶翻转终止子的效率

图5 生物七段译码器
3 结论
本研究通过组合重组酶识别位点和终止子构建了一种稳定可维持的转录调控开关。该开关可以作为一个组件,模块化地应用于基因线路构建中,使细胞可以感受短暂的脉冲信号从而改变自身状态。通过细致的表征,我们发现了重组酶识别位点和终止子之间的相互影响是开关设计的一个瓶颈,从而揭示了此类开关的重要设计原则——寻找相互影响较小的终止子和识别位点,为大规模设计此类开关提供了一种思路。
我们通过依次表达两个重组酶作用于终止子两侧的两对识别位点,实现了两次状态切换。已有研究表明当重组定向性因子(Recombination directionality factor,RDF)存在时,重组酶可识别attL与attR序列发生重组反应,重新生成attB、attP位点。因此,本开关的一种优化方式是引入RDF,使得只需要一种重组酶、一对识别位点便可实现两次状态转换。这将避免多对识别位点对终止子的影响,提高本开关的性能。
如果要使这种状态转换自动化,创造更复杂的基因线路,可以在此系统中引入“计时器”。例如利用基于三因子负反馈的生物振荡器[26]节律表达重组酶及其RDF,作用于经过设计的含此类开关的基因线路,理论上可以实现循环的状态转换过程。
此外,本研究具有较大的应用价值。工业上,可以利用此类状态开关可设计的次序性,让一种工程菌在发酵罐中依次合成不同产物,例如含有多种单体的高分子化合物的生物合成。医疗上,可以设计一种含此种开关的益生菌,使之在患者体内在合适的时间按一定次序给药。基础研究中,此类开关能够为通过“建物致知”的策略为研究细胞周期与生物节律提供新的思路和方法。
[1] Bittner M. A window on the dynamics of biological switches. Nat Biotechnol, 2005, 23(2): 183–184.
[2] Pathak GP, Strickland D, Vrana JD, et al. Benchmarking of optical dimerizer systems. ACS Synth Biol, 2014, 3(11): 832–838.
[3] Martinac B, Buechner M, Delcour AH, et al. Pressure-sensitive ion channel in. Proc Natl Acad Sci USA, 1987, 84(8): 2297–2301.
[4] Payandeh J, Scheuer T, Zheng N, et al. The crystal structure of a voltage-gated sodium channel. Nature, 2011, 475(7356): 353–358.
[5] Jensen AA, Spalding A. Allosteric modulation of G-protein coupled receptors. Eur J Pharm Sci, 2004, 21(4): 407–420.
[6] Xu YQ, Ma CQ, Tao F, et al. Bacterial promoter recognition and application. Chin J Biotech, 2010, 26(10): 1393–1403 (in Chinese). 徐友强, 马翠卿, 陶飞, 等. 细菌启动子识别及应用研究进展. 生物工程学报, 2010, 26(10): 1393–1403.
[7] Boyden ES, Zhang F, Bamberg E, et al. Millisecond-timescale, genetically targeted optical control of neural activity. Nature Neurosci, 2005, 8(9): 1263–1268.
[8] Levskaya A, Chevalier AA, Tabor JJ, et al. Synthetic biology: engineeringto see light. Nature, 2005, 438(7067): 441–442.
[9] Green AA, Silver PA, Collins JJ, et al. Toehold switches:--designed regulators of gene expression. Cell, 2014, 159(4): 925–939.
[10] Hasty J, Pradines J, Dolnik M, et al. Noise-based switches and amplifiers for gene expression. Proc Natl Acad Sci USA, 2000, 97(5): 2075–2080.
[11] Grindley NDF, Whiteson KL, Rice PA. Mechanisms of site-specific recombination. Annu Rev Biochem, 2006, 75: 567–605.
[12] Lewis JA, Hatfull GF. Control of directionality in integrase-mediated recombination: examination of recombination directionality factors (RDFs) including Xis and Cox proteins. Nucleic Acids Res, 2001, 29(11): 2205–2216.
[13] Farzadfard F, Lu TK. Genomically encoded analog memory with preciseDNA writing in living cell populations. Science, 2014, 346(6211): 1256272.
[14] Siuti P, Yazbek J, Lu TK. Synthetic circuits integrating logic and memory in living cells. Nat Biotechnol, 2013, 31(5): 448–452.
[15] Mimee M, Tucker AC, Voigt CA, et al. Programming a Human commensal bacterium,, to sense and respond to stimuli in the murine gut microbiota. Cell Syst, 2015, 1(1): 62–71.
[16] Bonnet J, Yin P, Ortiz ME, et al. Amplifying genetic logic gates. Science, 2013, 340(6132): 599–603.
[17] Roquet N, Soleimany AP, Ferris AC, et al. Synthetic recombinase-based state machines in living cells. Science, 2016, 353(6297): aad8559.
[18] Yang L, Nielsen AAK, Fernandezrodriguez J, et al. Permanent genetic memory with >1-byte capacity. Nat Methods, 2014, 11(12): 1261–1266.
[19] Epshtein V, Dutta D, Wade J, et al. An allosteric mechanism of Rho-dependent transcription termination. Nature, 2010, 463(7278): 245–249.
[20] Adhya S, Gottesman M. Control of transcription termination. Annu Rev Biochem, 1978, 47: 967–996.
[21] Postle K, Good RF. A bidirectional rho-independent transcription terminator between the.gene and an opposing gene. Cell, 1985, 41(2): 577–585.
[22] Chen YJ, Liu P, Nielsen AAK, et al. Characterization of 582 natural and synthetic terminators and quantification of their design constraints. Nature Methods, 2013, 10(7): 659–664.
[23] Lou CB, Stanton B, Chen YJ, et al. Ribozyme-based insulator parts buffer synthetic circuits from genetic context. Nat Biotechnol, 2012, 30(11): 1137–1142.
[24] Brophy JA, Voigt CA. Antisense transcription as a tool to tune gene expression. Mol Syst Biol, 2016, 12(1): 854.
[25] Salis HM, Mirsky EA, Voigt CA. Automated design of synthetic ribosome binding sites to control protein expression. Nat Biotechnol, 2009, 27(10): 946–950.
[26] Elowitz MB, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403(6767): 335–338.
团队简介:
2017年北京大学iGEM团队由来自生命科学学院、信息科学技术学院、医学部共13名在校本科生组成。北大iGEM团队提出了一套在细菌内搭建遗传时序逻辑线路,使其能够自动按顺序执行对应功能的设计框架,充分借鉴了数字理论中时序逻辑概念,在生物系统中构建时钟 (Clock)、分频器 (Frequency Divider)、解释器 (Interpreter) 三个基本模块。基于重组酶和终止子元件所实现的细胞状态调控便是其核心内容之一。北大iGEM团队在2017年iGEM大赛中获得了最佳信息处理项目奖提名 (Nominated Best Information Processing Project),并获得金牌。

(本文责编 郝丽芳)
Design of recombinase and terminator-based genetic switches for cell state control
Songyuan Zhang1*, Jianhui Qiu1*, Xuan Wang2, Yiming Dong1, Yulong Li1, Yihao Zhang1, and Qi Ouyang1
1 Center for Quantitative Biology and Center for Life Sciences, Peking University, Beijing 100871, China 2 Center for Life Sciences, Tsinghua-Peking Center for Life Sciences, Tsinghua University, Beijing 100084, China
Various genetic switches have been developed to let engineered cells perform designed functions. However, a sustained input is often needed to maintain the on/off state, which is energy-consuming and sensitive to perturbation. Therefore, we developed a set of transcriptional switches for cell states control that were constructed by the inversion effect of site-specific recombinases on terminators. Such a switch could respond to a pulse signal and maintain the new state by itself until the next input. With a bottom-up design principle, we first characterized the terminators and recombinases. Then the mutual interference was studied to select compatible pairs, which were used to achieve one-time and two-time state transitions. Finally, we constructed a biological seven-segment display as a demonstration to prove such switch’s immense potential for application.
site-specific recombinase, terminator, genetic switch, state transition
June 30, 2018;
October 22, 2018
Talent Training Program, Educational and Teaching Reform Project of Central College.
Yihao Zhang. Tel/Fax: +86-10-62744020; E-mail: yihaozhang@pku.edu.cn
Qi Ouyang. Tel/Fax: +86-10-62744020; E-mail: qi@pku.edu.cn
10.13345/j.cjb.180270
*These authors contributed equally to this study.
中央高校教育教学改革拔尖人才培养项目资助。

