猪源转录因子AP-2δ通过增强rep基因启动子活性促进猪圆环病毒2型的复制
王越,宋东峰,林翠,李佳容,王胜男,顾金燕,周继勇
猪源转录因子AP-2δ通过增强基因启动子活性促进猪圆环病毒2型的复制
王越1,宋东峰1,林翠2,李佳容1,王胜男1,顾金燕1,周继勇2
1 南京农业大学 免疫研究所,江苏 南京 210095 2 浙江大学 农业部动物病毒学重点实验室,浙江 杭州 310058

王越, 宋东峰, 林翠, 等. 猪源转录因子AP-2δ通过增强rep基因启动子活性促进猪圆环病毒2型的复制. 生物工程学报, 2018, 34(12): 1985–1995.Wang Y, Song DF, Lin C, et al. Porcine transcription factor AP-2δ promotes porcine circovirus type 2 replication through enhancing the activity of the rep gene promoter. Chin J Biotech, 2018, 34(12): 1985–1995.
本研究旨在探究猪圆环病毒2型 (Porcine circovirus type 2, PCV2) 能否借助基因启动子区的转录因子结合位点调控自身基因转录,并寻找参与这一调控过程的宿主因子。凝胶迁移实验证实基因启动子具有核蛋白结合活性。DNA-pull down联合液相-串联质谱分析鉴定到了可与基因启动子结合的猪源转录因子AP-2δ (Porcine transcription factor AP-2δ, poTFAP2δ)。双荧光素酶报告基因试验、实时荧光定量PCR、免疫印迹及间接免疫荧光等试验证明poTFAP2δ不仅可以特异性地增强基因启动子活性,而且可以在PCV2感染过程中促进和基因的转录、翻译及PCV2病毒滴度。文中揭示了PCV2利用宿主蛋白促进自身增殖的分子机制,为进一步从病毒与宿主互作角度阐明PCV2的致病机制提供新的研究思路,亦为PCV2高效疫苗的研制提供理论基础。
猪圆环病毒2型,基因启动子,转录因子结合位点,猪源转录因子AP-2δ
猪圆环病毒2型 (Porcine circovirus type 2,PCV2) 属于圆环病毒科,无囊膜,直径约12−23 nm,具有大约1.7 kb的单链环状DNA基因组[1],是猪圆环病毒相关疾病 (PCV2-associated disease, PCVAD) 的主要病原体,给养猪业造成了巨大的经济损失[2-5]。
PCV2的基因编码衣壳蛋白Cap,作为PCV2唯一的结构蛋白及主要的免疫原性蛋白,Cap蛋白在病毒入侵及致病性方面发挥重要作用[6-8]。PCV2的基因编码两个病毒复制相关蛋白Rep和Rep′,Rep和Rep′能够与位于基因间区复制起点 () 内的特定序列结合,启动PCV2复制[9-12]。PCV2基因启动子区存在一个类干扰素刺激反应元件 (Interferon-stimulated response element,ISRE),这个类ISRE作为一个转录因子结合位点 (Transcription factor binding sites,TFBS),可以促进基因转录及病毒增殖[13],本研究旨在探究PCV2基因启动子是否具有利用类ISRE等TFBSs调控自身基因转录的生物学特性,并鉴定参与这一调控过程的宿主蛋白。
研究表明,某些病毒的基因启动子可以与宿主转录因子结合,通过调控病毒基因的启动子活性而调节自身基因的转录及翻译。如单纯疱疹病毒1型 (Herpes simplex virus type 1,HSV-1)、人乳头瘤病毒 (Human papillomavirus,HPV) 的启动子均具有利用宿主转录因子调节启动子活性的功能。本研究通过分析发现,PCV2基因启动子上除了拥有一个已知的类ISRE,还存在其他一些潜在的TFBSs,这些TFBSs是否具有生物学功能?何种宿主蛋白可与之结合并调控基因转录?
本研究通过DNA-pull down联合液相-串联质谱分析 (Liquid chromatography-tandem mass spectrometric,LC-MS/MS) 等试验鉴定出猪源转录因子AP-2δ (Porcine transcription factor AP-2δ,poTFAP2δ) 可以在体外与基因启动子结合。据报道,TFAP2家族蛋白具有与病毒基因启动子结合并调控启动子活性的功能,如TFAP2能与EB病毒 (Epstein-Barr virus, EBV) 及人类免疫缺陷病毒 (Human immunodeficiency virus,HIV) 等病毒的启动子结合,调控病毒基因的表达[14-16]。本研究通过一系列试验证明,poTFAP2δ不仅能够显著增强PCV2基因启动子活性及/基因的转录和翻译,还能在一定程度上促进PCV2的病毒滴度。
1 材料与方法
1.1 细胞和病毒
无PCV污染的猪肾细胞系 (Porcine kidney epithelial cell line,PK15) 由本实验室保存,在含有8%胎牛血清的RPMI-1640培养基中培养。PCV2 HZ0201毒株 (GenBank登录号:AY188355) 为本实验室分离保存,病毒毒价为106.4TCID50/0.1 mL。
1.2 主要试剂及载体
双荧光素酶报告系统质粒pGL3-Basic (Basic) 和pRL-TK均购自Promega公司;真核表达载体pCMV-N-Myc (Myc-Empty Vector, Myc-EV) 由本实验室保存;RPMI-1640培养基购自Hyclone公司;胎牛血清购自Biological Industries公司;双荧光素酶报告基因检测试剂盒、细胞核蛋白与细胞浆蛋白抽提试剂盒均购自碧云天公司;限制性内切酶购自New England BioLabs公司;蛋白Marker、EMSA化学发光试剂盒、尼龙膜均购自Thermo Fisher Scientific公司;0.22 μm无菌滤器购自Millipore公司;DNA胶纯化试剂盒购自Axygen公司;质粒小提试剂盒购自Tiangen公司;荧光定量PCR试剂 (SYBR Green Master Mix)、一步克隆法试剂盒 (Clonexpress II one step cloning kit)、增强型ECL化学发光检测试剂盒均购自诺唯赞公司;转染试剂Lipofectamine 3 000及链霉亲和素磁珠均购自Invitrogen公司;鼠抗Myc单抗购自Biolegend公司;兔抗β-actin单抗购自杭州华安生物技术有限公司;鼠抗Rep单抗 (2E11) 及鼠抗Cap单抗 (5E11) 为本实验室制备并保存;HRP标记羊抗鼠IgG、HRP标记羊抗兔IgG、FITC标记羊抗鼠IgG均购自KPL公司。
1.3 rep基因启动子区潜在TFBSs预测及分析
PCV2基因启动子位于基因的上游,从–193 nt至,共193 bp (图1)。本研究用在线数据库JASPAR (http://jaspar.genereg.net/) 对PCV2基因启动子进行潜在TFBSs的预测,对预测分值较高 (score>10) 的位点进行后续分析。
1.4 凝胶迁移实验 (Electrophoretic mobility shift assay, EMSA) 验证rep基因启动子的核蛋白结合活性
根据基因启动子潜在TFBSs预测及分析结果,本研究设计了用于EMSA试验的互补生物素标记的单链核酸探针p119biotin-s和p119biotin-a (表1) 及未标记的竞争探针并委托金斯瑞生物科技有限公司进行合成。p119biotin-s和p119biotin-a为5¢端生物素标记的长度为119 nt (–133 nt至–15 nt,图1) 的互补单链核苷酸序列,包含EGR、SP、KLF、ZNF、YY、TFAP2、XBP、Crem、Atf、IRF等主要潜在TFBSs,竞争探针为没有生物素标记的同序列单链探针。单链探针进行退火操作后可获得用于EMSA试验的双链生物素标记探针p119biotin及竞争探针p119。
细胞核蛋白抽提按细胞核蛋白与细胞浆蛋白抽提试剂盒操作说明进行,将PK15细胞接种于25 cm2细胞瓶,常规培养至细胞密度约为90%,从3×106个PK15细胞中抽提细胞核蛋白用于EMSA试验。EMSA试验按照EMSA化学发光试剂盒操作说明进行,反应体系如下:p119biotin (1×104fmol/L),2 μL,10×结合缓冲液2 μL,细胞核蛋白提取物4 μL,p193 (1×103pmol/L),4 μL。

表1 本研究所用引物及探针序列
1.5 DNA-pull down联合LC-MS/MS鉴定结合蛋白
为了探索能与PCV2基因启动子结合的宿主转录因子,本研究采用DNA-pull down联合LC-MS/MS技术进行鉴定。具体步骤如下:将5 μg p119biotin与500 μg PK15细胞核蛋白提取物室温孵育30 min,加入链霉亲和素标记的磁珠充分混匀,4 ℃翻转过夜,利用磁力架反复清洗3次,最后将沉淀进行LC-MS/MS蛋白鉴定 (上海博苑生物科技有限公司)。
1.6 重组质粒的构建
poTFAP2δ基因根据NCBI数据库中的预测序列 (GenBank登录号:XM_003356639) 委托生工生物工程 (上海) 股份有限公司进行基因合成,并利用一步克隆法构建真核表达载体pCMV-N- Myc-poTFAP2δ (Myc-poTFAP2δ)。PCV2基因启动子测试载体pGL3-Basic-p193 (p193) 由本实验室保存。
1.7 双荧光素酶报告基因试验
PK15细胞接种于24孔细胞板,常规培养24 h至细胞密度约为75%,用Lipofectamine 3000将Basic/pRL-TK、p193/pRL-TK分别与Myc-EV和Myc-poTFAP2δ共转染PK15细胞,转染24 h后依据双荧光素酶检测试剂盒说明书进行相对荧光素酶活性检测,分析poTFAP2δ对基因启动子活性的影响。
1.8 实时荧光定量PCR (Quantitative real-time PCR, qPCR)
将PK15细胞接种于12孔板,常规培养24 h至细胞密度约为75%,用Lipofectamine 3000将Myc-EV和Myc-poTFAP2δ分别转染PK15细胞,转染24 h后,以感染复数 (Multiplicity of infection, MOI) =1的PCV2 HZ0201毒株感染该细胞,在感染后24、48、72 h收取细胞,trizol法抽提病毒RNA,取10 ng进行反转录得病毒cDNA,用qPCR (SYBR法) 检测、的mRNA水平。qPCR引物如表1所示,两步法qPCR反应程序按SYBR Green Master Mix使用说明书进行。
1.9 免疫印迹试验 (Western blotting, WB)
将Myc-EV和Myc-poTFAP2δ在PK15细胞上瞬时过表达后接种PCV2 HZ0201 (MOI=1),方法如1.8所述,病毒感染24、48、72 h后收集细胞,裂解蛋白样品后WB检测Rep及Cap的蛋白表达水平。WB所用一抗分别为鼠抗Myc单抗 (1∶1 000)、兔抗β-actin单抗 (1∶1 000)、鼠抗Rep单抗2E11 (1∶500) 和鼠抗Cap单抗5E11 (1∶500),二抗分别为HRP标记羊抗鼠IgG (1∶7 500) 和HRP标记羊抗兔IgG (1∶7 500)。最后加入ECL显色液用Amersham Imager 600超敏多功能成像仪显色,用ImageJ软件进行灰度值分析,计算Rep和Cap对β-actin的比率。
1.10 间接免疫荧光试验 (Indirect immunofluorescent assay, IFA) 测定PCV2病毒滴度
将Myc-EV和Myc-poTFAP2δ在PK15细胞上瞬时过表达后接种PCV2 HZ0201 (MOI=1),方法如1.8所述,在感染后24、48、72 h收取板子,反复冻融3次后收集病毒样品,4 ℃、12 000 r/min离心10 min后收取上清,利用IFA测定样品中病毒的半数组织培养感染量 (Median tissue culture infectious dose, TCID50)。IFA所用一抗为鼠抗Cap单抗5E11 (1∶500),二抗为FITC标记羊抗鼠IgG (1∶350)。最后,用Reed-Muench方法计算各样品中PCV2的TCID50。
2 结果与分析
2.1 PCV2 rep基因启动子上存在多个潜在的TFBSs
为了探究PCV2基因启动子是否具有利用宿主转录因子促进自身启动子活性的功能,本研究利用JASPAR数据库对基因启动子进行潜在TFBSs的预测分析,结果发现,该启动子上存在多处潜在的TFBSs,如图1所示,评分较高 (score>10) 的有EGR、SP、KLF、ZNF、YY、TFAP、XBP、Crem、Atf、IRF等TFBSs,这些位点集中在基因启动子的中间位置 (–133 nt至–15 nt, 119 bp),所以推测这段序列是基因启动子的转录因子结合核心区域。
2.2 PCV2 rep基因启动子具有核蛋白结合活性
根据潜在TFBSs的预测结果,将基因启动子的关键序列119 bp合成标记探针p119biotin及竞争探针p119,利用EMSA验证基因启动子的核蛋白结合活性。结果如图2所示,当标记探针中加入PK15细胞核蛋白提取物时,p119biotin出现了明显的迁移条带,说明PK15细胞核蛋白提取物中存在与p119biotin结合的宿主蛋白;但是,当加入竞争探针p119时迁移条带消失,说明这种蛋白与探针的结合具有高度特异性。

图1 PCV2 rep基因启动子区潜在TFBSs预测图
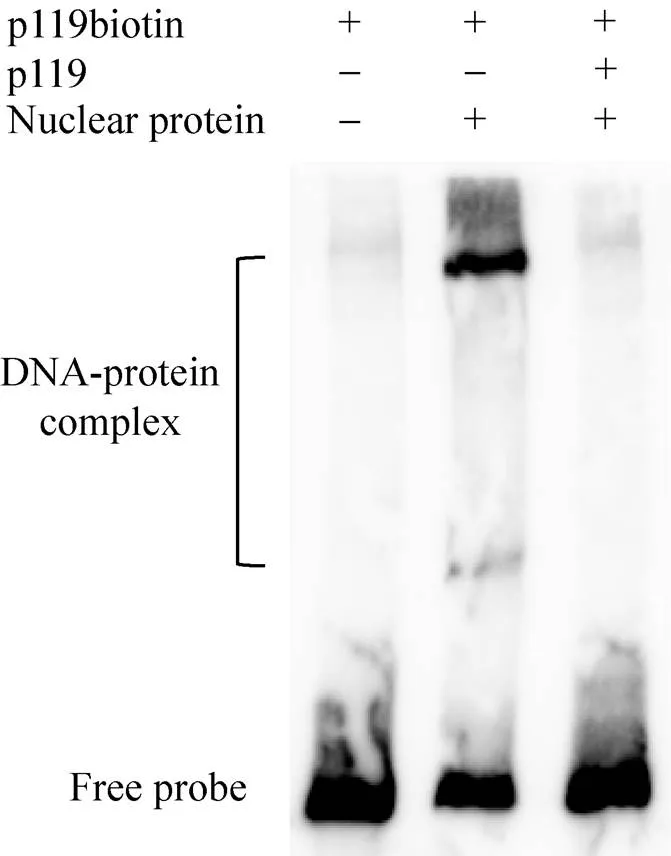
图2 EMSA验证rep基因启动子的核蛋白结合活性
2.3 poTFAP2δ可以与PCV2 rep基因启动子结合
为了探究何种核蛋白可以与PCV2基因启动子结合,本研究以p119biotin为探针进行DNA-pull down联合LC-MS/MS试验,将DNA- pull down后能与p119biotin结合的细胞核蛋白进行LC-MS/MS分析,质谱鉴定结果显示,与基因启动子序列结合的蛋白中有poTFAP2δ,这个发现与基因启动子的潜在TFBSs预测结果相吻合,所以推测poTFAP2δ可能在基因的转录启动方面发挥重要作用。
2.4 poTFAP2δ对PCV2 rep基因启动子的活性具有促进作用
为了探究poTFAP2δ是否可以特异性增强基因启动子活性,本研究比较了基因启动子在poTFAP2δ瞬时过表达及对照PK15细胞中的启动子活性差异。结果如图3所示,在poTFAP2δ瞬时过表达的PK15细胞中,p193的平均相对荧光素酶活性为16.95,而其在对照PK15细胞中的平均相对荧光素酶活性仅为7.732,差异显著,这个结果充分证明poTFAP2δ对基因启动子活性具有显著的促进作用。

图3 poTFAP2δ对PCV2 rep基因启动子活性的影响
2.5 poTFAP2δ对PCV2 rep、cap基因的转录及翻译具有促进作用
poTFAP2δ可以促进基因的启动子活性,那么是否也能促进基因的转录和翻译,从而进一步促进基因的转录及翻译?为了探究这个问题,试验将PCV2分别接种poTFAP2δ瞬时过表达及对照PK15细胞,感染24、48、72 h后检测、基因mRNA及蛋白表达水平的差异。结果如图4所示,在poTFAP2δ瞬时过表达的PK15细胞中,PCV2mRNA的平均表达水平在病毒感染后24、48、72 h分别是对照组的2.133、1.587、2.444倍,而PCV2mRNA的平均表达水平在病毒感染后24、48、72 h分别是对照组的2.196、1.993、1.811倍。不论在病毒感染后24、48 h还是72 h,poTFAP2δ过表达后病毒基因mRNA水平均显著上调,说明在PCV2感染过程中poTFAP2δ可以持续显著促进及基因的转录。
poTFAP2δ是否能进一步促进Rep及Cap蛋白的表达?结果如图5所示,将WB试验所检测的Rep、Cap蛋白与β-actin蛋白条带进行灰度值比值分析,发现在poTFAP2δ瞬时过表达的PK15细胞中,PCV2 Rep蛋白的平均相对表达水平在病毒感染后24、48、72 h分别为0.295、1.858、3.985,而对照组为0.183、1.144、2.361,说明在PCV2感染后24、48、72 h,poTFAP2δ过表达均能上调Rep蛋白的表达量,虽然感染后24、48 h上调不显著,但是72 h上调极显著;同样的,在poTFAP2δ瞬时过表达的PK15细胞中,PCV2 Cap蛋白的平均相对表达水平在病毒感染后24、48、72 h分别是0.520、1.390、2.790,而对照组为0.512、0.883、1.997,说明在PCV2感染后24、48、72 h,poTFAP2δ过表达均能上调Cap蛋白的表达量,虽然感染后24 h上调不显著,但48 h上调显著,72 h上调极显著。这两组试验结果说明poTFAP2δ过表达能在PCV2感染的整个过程中持续促进Rep及Cap蛋白的表达,且这种促进作用随着感染进程的推进而增强。依据以上实验结果推测,在PCV2感染过程中,poTFAP2δ不仅可以促进基因的转录及翻译,而且能通过促进Rep蛋白的表达进一步促进PCV2基因的转录及翻译。
2.6 poTFAP2δ对PCV2病毒增殖具有一定的促进作用
为了确认poTFAP2δ是否可以增加PCV2病毒滴度,本研究利用IFA对PCV2在poTFAP2δ瞬时过表达及对照PK15细胞上的TCID50进行测定,结果如图6所示。在对照PK15细胞上,PCV2感染后24、48、72 h的平均滴度为103.250/0.1 mL、103.667/0.1 mL、104.333/0.1 mL,而在poTFAP2δ瞬时过表达PK15细胞中则为103.417/0.1 mL、104.000/0.1 mL、104.917/0.1 mL。瞬时过表达组与对照组PCV2在感染后24、48、72 h的病毒滴度比值分别为1.469、2.153和3.837。虽然检验分析显示,poTFAP2δ瞬时过表达组与对照组的差异并不显著,但是滴度比值显示,poTFAP2δ对PCV2的滴度还是具有一定的促进作用,说明poTFAP2δ可以在一定程度上促进PCV2的增殖。

图4 poTFAP2δ对rep及cap基因mRNA表达水平的影响
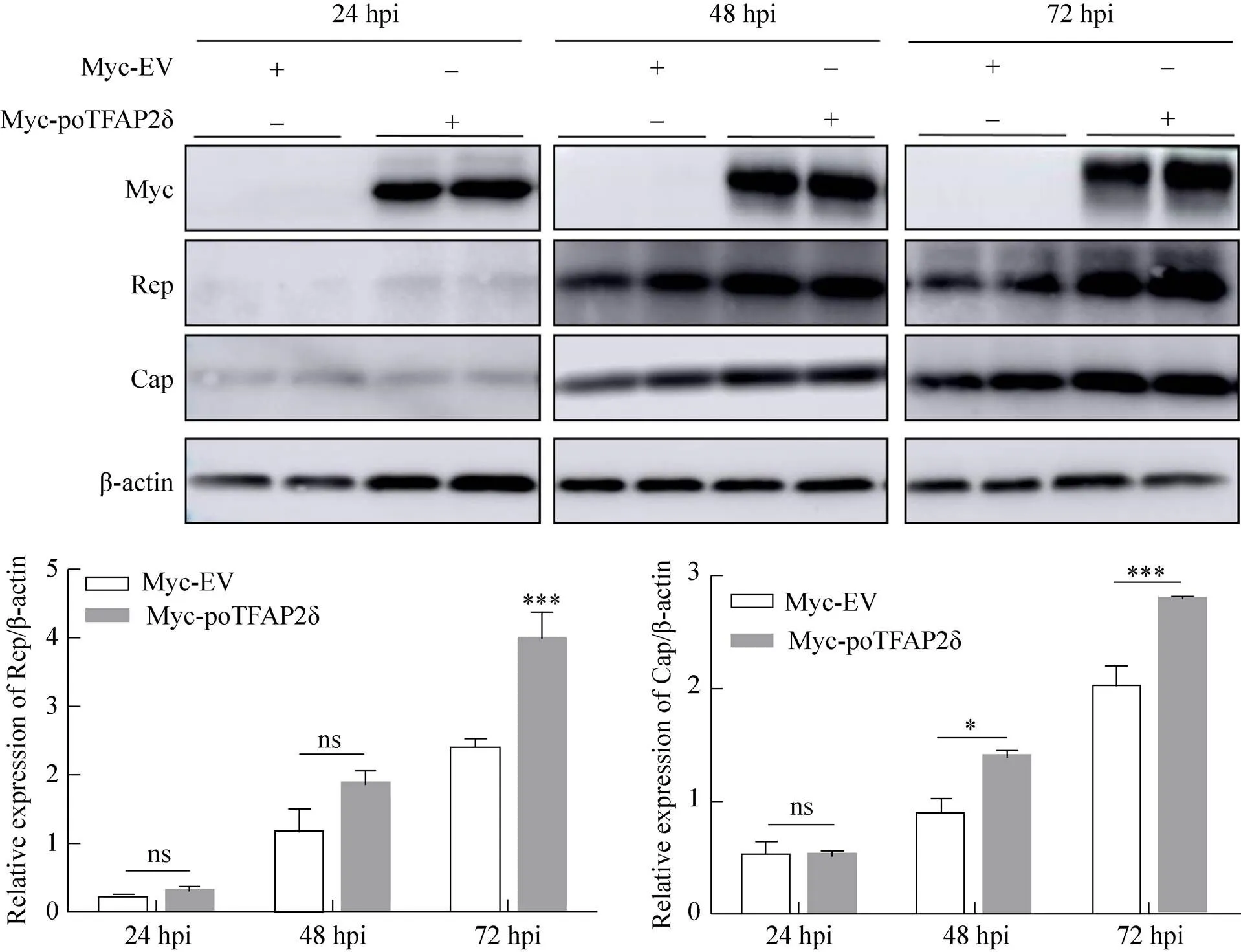
图5 poTFAP2δ对Rep及Cap蛋白表达水平的影响
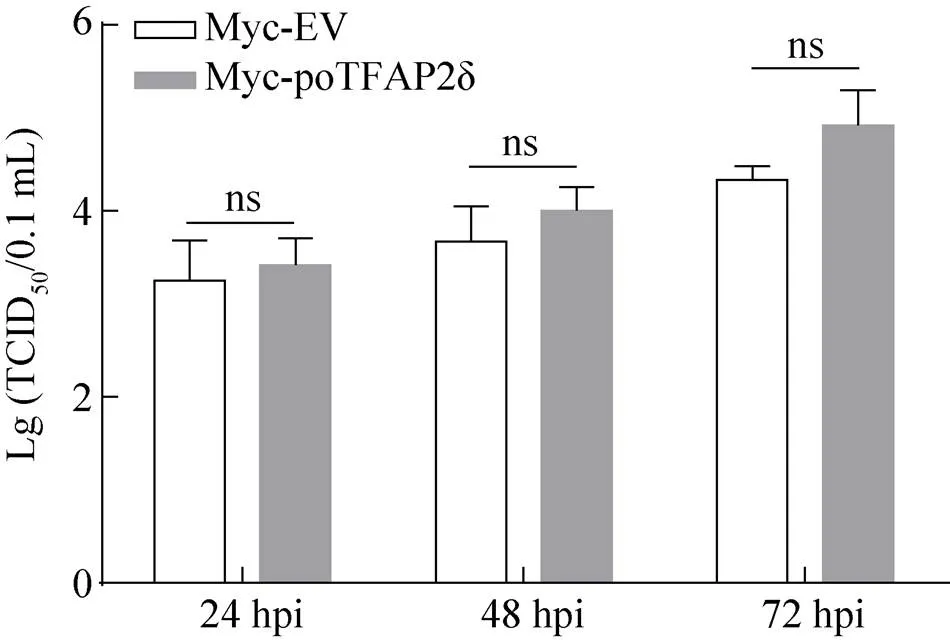
图6 poTFAP2δ对PCV2病毒滴度的影响
3 讨论
作为严格的细胞内寄生微生物,病毒利用宿主蛋白调控自身增殖是许多病毒的普遍生存法则。在自然界中,病毒利用宿主转录因子调控自身基因启动子活性,从而调节病毒基因转录及翻译的情况并不罕见,如HIV-1基因组5¢末端的长末端重复序列 (Long terminal repeat, LTR),作为启动子,LTR可以借助NF-κB、SP及TATA盒等TFBSs与宿主转录因子结合调控自身的基因转录[17-18];HSV-1的LAT启动子,可以与宿主细胞的信号转导及转录激活因子结合,调控病毒启动子活性[19];HPV的E6/E7基因启动子P97,可以与细胞因子YY1结合而下调自身基因转录活性[20];恒河猴疱疹病毒 (Rhesus rhadinovirus, RRV) 的复制和反式激活子 (Replication and transactivator, Rta) 的启动子,可以与宿主Sp蛋白结合,启动Rta的转录翻译,实现RRV的潜伏感染[21]。
PCV2作为最小的动物病毒之一,基因组仅为1.7 kb,表达的蛋白非常有限,为了更好地生存,它是否也存在利用宿主蛋白调控自身增殖的复制机制?前期研究发现,PCV2基因启动子区存在一个类ISRE,与PCV2基因启动子的活性、病毒的复制增殖及干扰素促进PCV2增殖的功能均相关,但具体机制还未得到阐释。PCV2基因启动子是否能像HIV-1 LTR等病毒启动子一样存在一系列TFBSs,通过与宿主转录因子的结合调控病毒基因转录,从而突破自身编码蛋白有限的困境?
本研究通过在线数据库对PCV2基因启动子区进行潜在TFBSs的预测,结果显示,这个启动子上除了有类ISRE,还存在诸如EGR、SP、KLF、TFAP2等一系列TFBSs,这些位点均集中在启动子中部的119 bp (–133 nt至–15 nt),特别是位于–133 nt至–120 nt的位点“GGCGGGGGTGGAGG”、位于–86 nt至–69 nt的位点“TGGCTGCGGGGGC GGTGT”和位于–46 nt至–33 nt的位点“TTGGA TACGTCATA”,这三个位点均含有一些分值较高的潜在TFBSs,推测可能是启动子与宿主转录因子结合的核心区域。
EMSA证明了基因启动子确实能与宿主核蛋白结合,DNA-pull down联合LC-MS/MS将这些结合蛋白鉴定为包含有转录因子的一系列宿主核蛋白 (数据未展示),其中包括poTFAP2δ。TFAP2家族是序列特异性的DNA结合蛋白,在动物发育、分化和肿瘤发生过程中调控基因表达,发挥重要作用[22]。哺乳动物普遍存在5个TFAP2基因 (TFAP2α/β/γ/δ/ε),分别编码TFAP2α/β/γ/δ/ε蛋白。TFAP2家族常表达于中枢神经系统,基因缺失小鼠实验表明TFAP2家族对发育极其关键,常为致死性缺失[23-30]。TFAP2δ与TFAP2家族其他成员的相似度最低,对于TFAP2家族蛋白功能极其重要的8个保守残基,TFAP2δ仅有3个[31-33]。据报道,TFAP2家族蛋白具有与病毒基因启动子结合并调控启动子活性的功能,如TFAP2α/β/γ能与EBV的LMP1基因启动子结合,促进LMP1蛋白的表达,稳定EBV的潜伏感染[14-15];TFAP2还可以与HIV-1的LTR结合,调控HIV-1基因的表达[16]。巧合的是,在预测的基因启动子潜在TFBSs结果中也存在TFAP2位点,那么poTFAP2δ是否可以与基因启动子结合而调节基因的转录?通过启动子活性试验、qPCR、WB及IFA等一系列试验,表明poTFAP2δ不仅可以促进基因启动子的活性,而且可以在PCV2感染过程中持续促进及基因的转录及翻译,最后对PCV2的病毒滴度也有一定的促进作用。
PCV2自身不编码复制酶,主要依靠细胞有丝分裂S期表达的酶及蛋白进行滚环复制,PCV2的复制起始依赖于Rep及Rep′蛋白与PCV2复制子的结合,所以基因的转录翻译在PCV2复制增殖中发挥至关重要的作用。我们基于本研究的结果推测,poTFAP2δ可以特异性地与基因启动子区某位点结合启动或促进基因的转录,翻译所得的大量Rep及Rep′蛋白启动病毒复制,产生更多的PCV2基因组及基因转录所需的模板,导致Cap蛋白的大量表达,保证了PCV2病毒包装所需衣壳蛋白及基因组的供给,于是病毒滴度增加。从试验结果可见,poTFAP2δ对PCV2 Rep、Cap蛋白及病毒滴度的促进均具有时间依赖性,随着PCV2感染进程的推进,这种促进作用越来越强,说明以上推测的poTFAP2δ对PCV2复制增殖的促进是一个不断累加的循环过程。至于poTFAP2δ是否与预测的TFAP2位点“TGGCTGCGGGGGCGGTGT” (–86 nt至–69 nt) 结合,还需要大量的后续试验验证。
本研究首次证明了PCV2基因启动子具有通过利用宿主转录因子poTFAP2δ促进病毒基因转录、翻译及病毒滴度的生物学功能,从病毒利用宿主因子的角度阐释了PCV2的复制机制。该研究结果为进一步从病毒与宿主互作角度阐明PCV2的致病机制提供新的研究思路,亦为PCV2高效疫苗的研制提供了理论基础。
[1] Finsterbusch T, Mankertz A. Porcine circoviruses— small but powerful. Virus Res, 2009, 143(2): 177–183.
[2] Madson DM, Opriessnig T. Effect of porcine circovirus type 2 (PCV2) infection on reproduction: disease, vertical transmission, diagnostics and vaccination. Anim Health Res Rev, 2011, 12(1): 47–65.
[3] Nayar GP, Hamel A, Lin L. Detection and characterization of porcine circovirus associated with postweaning multisystemic wasting syndrome in pigs. Can Vet J, 1997, 38(6): 385–386.
[4] Segalés J, Sitjar M, Domingo M, et al. First report of post-weaning multisystemic wasting syndrome in pigs in Spain. Vet Rec, 1997, 141(23): 600–601.
[5] Gu JY, Xing G, Lei J, et al. Porcine circovirus type 2 and PCV2-systemic disease - a review. Chin J Biotech, 2015, 31(6): 880–891 (in Chinese).顾金燕, 邢刚, 雷静, 等. 猪圆环病毒2型与猪圆环病毒相关性系统疾病的回顾及展望. 生物工程学报, 2015, 31(6): 880–891.
[6] Zhang LK, Li YF, Xie LB, et al. Generation and characterization of recombinant classical swine fever virus C-strain expressing the cap protein of porcine circovirus type 2. Chin J Biotech, 2018, 34(2): 216–223 (in Chinese).张玲楷, 李永锋, 谢利豹, 等. 表达猪圆环病毒2型Cap蛋白的重组猪瘟兔化弱毒疫苗株的构建与鉴定. 生物工程学报, 2018, 34(2): 216–223.
[7] Wang XW, Li YF, Jiang P. Construction and immunogencity of recombinantadenoviruses expressing Cap protein of PCV2 and GP5protein of PRRSV in mice. Chin J Biotech, 2009, 25(11): 1639–1645 (in Chinese).王先炜, 李玉峰, 姜平. 表达猪圆环病毒2型Cap和猪繁殖与呼吸综合征病毒GP5融合蛋白重组腺病毒的构建和免疫原性测定. 生物工程学报, 2009, 25(11): 1639–1645.
[8] Fan HY,Chen HC,Tong TZ, et al. The expression of porcine circovirus Type 2ORF2 genein insect cells and its character. Chin J Biotech, 2005, 21(6): 123–126 (in Chinese).樊惠英, 陈焕春, 佟铁铸, 等. 猪圆环病毒2型ORF2基因在昆虫细胞中的表达及其特性. 生物工程学报, 2005, 21(6): 123–126.
[9] Cheung AK. The essential and nonessential transcription units for viral protein synthesis and DNA replication of porcine circovirus type 2. Virology, 2003, 313(2): 452–459.
[10] Cheung AK. Identification of the essential and non-essential transcription units for protein synthesis, DNA replication and infectious virus production oftype 1. Arch Virol, 2004, 149(5): 975–988.
[11] Mankertz A, Hillenbrand B. Replication of porcine circovirus type 1 requires two proteins encoded by the viralgene. Virology, 2001, 279(2): 429–438.
[12] Steinfeldt T, Finsterbusch T, Mankertz A. Rep and Rep' protein oftype 1 bind to the origin of replication. Virology, 2001, 291(1): 152–160.
[13] Gu JY, Zhang Y, Lian X, et al. Functional analysis of the interferon-stimulated response element of porcine circovirus type 2 and its role during viral replicationand. Virol J, 2012, 9: 152.
[14] Jansson A, Johansson P, Yang W, et al. Role of a consensus AP-2 regulatory sequence within the Epstein-Barr virus LMP1 promoter in EBNA2 mediated transactivation. Virus Genes, 2007, 35(2): 203–214.
[15] Murata T, Noda C, Narita Y, et al. Induction of Epstein-Barr Virus Oncoprotein LMP1 by Transcription Factors AP-2 and Early B Cell Factor. J Virol, 2016, 90(8): 3873–3889.
[16] Perkins ND, Agranoff AB, Duckett CS, et al. Transcription factor AP-2 regulates human immunodeficiency virus type 1 gene expression. J Virol, 1994, 68(10): 6820–6823.
[17] Hiscott J, Kwon H, Génin P. Hostile takeovers: viral appropriation of the NF-κB pathway. J Clin Invest, 2001, 107(2): 143–151.
[18] Kilareski EM, Shah S, Nonnemacher MR, et al. Regulation of HIV-1 transcription in cells of the monocyte-macrophage lineage. Retrovirology, 2009, 6: 118.
[19] Kriesel JD, Jones BB, Dahms KM, et al. STAT1 binds to the herpes simplex virus type 1 latency-associated transcript promoter. J Neurovirol, 2004, 10(1): 12–20.
[20] Lace MJ, Yamakawa Y, Ushikai M, et al. Cellular factor YY1 downregulates the human papillomavirus 16 E6/E7 promoter, P97,andfrom a negative element overlapping the transcription-initiation site. J Gen Virol, 2009, 90(Pt 10): 2402–2412.
[21] Demaster LK, Rose TM. A critical Sp1 element in the rhesus rhadinovirus (RRV) Rta promoter confers high-level activity that correlates with cellular permissivity for viral replication. Virology, 2014, 448: 196–209.
[22] Eckert D, Buhl S, Weber S, et al. The AP-2 family of transcription factors. Genome Biol, 2005, 6(13): 246.
[23] Feng WG, Williams T. Cloning and characterization of the mouse AP-2ε gene: a novel family member expressed in the developing olfactory bulb. Mol Cell Neurosci, 2003, 24(2): 460–475.
[24] Moser M, Pscherer A, Roth C, et al. Enhanced apoptotic cell death of renal epithelial cells in mice lacking transcription factor AP-2β. Genes Dev, 1997, 11(15): 1938–1948.
[25] Moser M, Dahmen S, Kluge R, et al. Terminal renal failure in mice lacking transcription factor AP-2 β. Lab Invest, 2003, 83(4): 571–578.
[26] Nottoli T, Hagopian-Donaldson S, Zhang J, et al. AP-2-null cells disrupt morphogenesis of the eye, face, and limbs in chimeric mice. Proc Natl Acad Sci USA, 1998, 95(23): 13714–13719.
[27] Schorle H, Meier P, Buchert M, et al. Transcription factor AP-2 essential for cranial closure and craniofacial development. Nature, 1996, 381(6579): 235–238.
[28] Wang HV, Vaupel K, Buettner R, et al. Identification and embryonic expression of a new AP-2 transcription factor, AP-2ε. Dev Dyn, 2004, 231(1): 128–135.
[29] Wenke AK, Rothhammer T, Moser M, et al. Regulation of integrin α10 expression in chondrocytes by the transcription factors AP-2ε and Ets-1. Biochem Biophys Res Commun, 2006, 345(1): 495–501.
[30] Zhang J, Hagopian-Donaldson S, Serbedzija G, et al. Neural tube, skeletal and body wall defects in mice lacking transcription factor AP-2. Nature, 1996, 381(6579): 238–241.
[31] Li XD, Glubrecht DD, Mita R, et al. Expression of AP-2δ in the developing chick retina. Dev Dyn, 2008, 237(11): 3210–3221.
[32] Wankhade S, Yu YH, Weinberg J, et al. Characterization of the activation domains of AP-2 family transcription factors. J Biol Chem, 2000, 275(38): 29701–29708.
[33] Zhao F, Satoda M, Licht JD, et al. Cloning and characterization of a novel mouse AP-2 transcription factor, AP-2δ, with unique DNA binding and transactivation properties. J Biol Chem, 2001, 276(44): 40755–40760.
(本文责编 陈宏宇)
Porcine transcription factor AP-2δ promotes porcine circovirus type 2 replication through enhancing the activity of thegene promoter
Yue Wang1, Dongfeng Song1, Cui Lin2, Jiarong Li1, Shengnan Wang1, Jinyan Gu1, and Jiyong Zhou2
1 Institute of Immunology, Nanjing Agricultural University, Nanjing 210095, Jiangsu, China 2 Key Laboratory of Animal Virology of Ministry of Agriculture, Zhejiang University, Hangzhou 310058, Zhejiang, China
Several putative transcription factor binding sites (TFBSs) exist in the PCV2gene promoter. To explore if porcine circovirus type 2 (PCV2) could regulate the viral replication by using these TFBSs, we conducted electrophoretic mobility shift assay (EMSA), DNA-pull down and liquid chromatography-tandem mass spectrometric (LC-MS/MS) assays. EMSA confirmed the binding activity of thegene promoter with nuclear proteins of host cells. DNA-pull down and LC-MS/MS identified the porcine transcription factor AP-2δ (poTFAP2δ) could bind the PCV2gene promoter.Dual-luciferase reporter assay, quantitative real-time PCR, Western blotting and indirect immunofluorescent assay demonstrated that poTFAP2δ could not only promote the activity of thegene promoter, but also enhance the transcription/translation activity of the/gene and the virus titer of PCV2 during the entire life cycle of PCV2 infection. This study revealed the molecular mechanism of PCV2 using host proteins to enhance the viral replication, provided a new perspective for studying the pathogenic mechanism of PCV2 from virus and host interactions, and provided a theoretical basis for developing highly effective PCV2 vaccines.
porcine circovirus type 2,gene promoter, transcription factor binding sites, porcine transcription factor AP-2δ
February 22, 2018;
May 9, 2018
National Key Technology R & D Program of China (No. 2015BAD12B01), National Natural Science Foundation of China (No. 31402198), the Fundamental Research Funds for the Central Universities (Nos. KJQN201524, KYZ201730), the Priority Academic Program Development of Jiangsu Higher Education Institutions.
s:Jinyan Gu. Tel: +86-25-84396068; Fax: +86-25-84395166; E-mail: gjy@njau.edu.cn
国家科技支撑计划 (No. 2015BAD12B01),国家自然科学基金 (No. 31402198),中央高校基本科研业务费 (Nos. KJQN201524, KYZ201730),江苏高校优势学科建设资助。
2018-06-06
10.13345/j.cjb.180069
http://kns.cnki.net/kcms/detail/11.1998.Q.20180605.1654.001.html

