利用生物信息学数据库筛选药物药动学、药效学相关基因及其多态性位点
张 远,何 霞,串俊兰,钟 磊,喻冬柯,熊 萱
0 引言
药物基因组学是建立在“基因多态性是形成个体差异的重要原因”这一理论基础之上,主要研究遗传变异如何影响个人对药物反应,并根据遗传变异特点选择治疗指数高、毒副作用小的药物,保障药物治疗的安全性和有效性,是列入“十三五”国家科技创新规划“精准医疗”的重要部分之一。但药物的有效性和安全性并非单一基因和多态性位点可以决定的,针对基因-基因、基因-环境交互作用的研究是药物基因组学未来的方向。然而,如何选取药物相关应答的基因及多态性位点是临床研究中大多数研究者面临的一个难题。虽然全基因组或外显子测序可以涵盖更全面的基因和多态性位点,但花费较高,难以应用于临床;从文献中筛选位点犹如大海捞针,难以选择到证据级别较高,与临床药物应答相关性较大的潜在的基因和位点。生物信息学的诞生为解决这类问题提供了帮助。生物信息学专门从事对基因组研究相关生物信息的获取、加工、储存、分配、分析和解释。如果能从数据库中方便、快速地检索到证据级别较高、临床相关性较大的药物相关基因和位点,不但可以节约时间、经费,还能一定程度上降低数据维度,为研究者进行基因-基因、基因-环境的研究设计阶段和后期数据统计处理提供一定的便利。
重度抑郁症(MDD),也被称为抑郁障碍,保守估计我国大约有7 000万人受其影响。作为一种高致残性疾病,抑郁障碍已经成为我国一个重大的公共卫生问题[1]。抗抑郁药物虽然可以有效治疗MDD,但个体间差异也较大:约30%~40%的患者没有得到完全缓解[2]。西酞普兰及其S-对映体艾司西酞普兰属于选择性5-羟色胺再摄取抑制剂(SSRIs),是MDD一线治疗用药。然而,早有证据显示,遗传差异对(艾司)西酞普兰的反应有一定影响。美国FDA已将CYP2D6(细胞色素P450家族2D6)和CYP2C19(细胞色素P450家族2C19)的遗传效应写入了药品说明书。同时,有报道,SLC6A4(溶质载体家族6成员4) rs4795541、BDNF(脑源性神经营养因子)Val66Met、HTR2A(5-羟色胺2A受体)rs6311、TPH2(色氨酸羟化酶2)rs1800532等与西酞普兰的药效学或药动学相关[3-5]。但与其他疾病不同的是,抑郁障碍的缓解受环境因素影响也很大:如年龄、性别、婚姻状况、教育程度、是否进行了心理治疗、居住地、职业和收入等[6-7]。因此,对于抗抑郁药物个体化差异的研究仅关注遗传差异性是远远不够的。本研究以抗抑郁药物西酞普兰为例,介绍几种常用的生物信息学数据库的搜索方式,为基因及多态性位点的筛选模式提供参考。
1 材料与方法
1.1 生物信息学数据库搜索筛选与(艾司)西酞普兰相关基因 本研究所选取的生物信息学数据库均为公共免费数据库。GenCards数据库是由以色列魏茨曼科学研究所(Weizmann Institute of Science)和Crown人类基因组中心(Crown Human Genome Center)整合各方资源所建立,包含了非常全面的关于基因及其产物的生物医学信息。它界面友好,使用户能方便地找到感兴趣的研究内容。UniProt是由欧洲生物信息学研究所(European Bioinformatics Institute)、美国蛋白质信息资源(Prontein Information Resource)以及瑞士生物信息研究所(Swiss Institute of Bioinformatics)等机构共同组成的UniProt协会(UniProt Consortium)编辑、制作的一个信息资源,旨在提供一个有关蛋白质序列及其相关功能方面的广泛的、高质量的并可免费使用的共享数据库。PharmGKB是由美国国立卫生院创建,收集了史上最完整的与药物基因组相关的基因型和表型信息,该数据库的特点是提供了高证据级别的基于基因型药物剂量指南推荐。以上3个数据库可以直接使用药名搜索,本研究检索词为 “citalopram OR escitalopram”(“西酞普兰OR艾司西酞普兰”)。
1.2 ClinVar筛选所选基因多态性位点 ClinVar也是一个公共免费数据库,由NCBI(美国国立生物技术信息中心)建立。收集了大量的基因突变与疾病或药物的关联信息,还对信息的级别进行了测评和注解。将以上3个数据库所搜索得到的基因名称输入ClinVar,并通过注解筛选多态性位点。
1.3 利用文献数据库筛选基因 为对比生物信息学数据库搜索得到的基因和文献数据库搜索得到的基因结果一致性,本研究以“(citalopram OR escitalopram)AND gene polymorphism”为检索词,搜索PubMed和中国生物医学文献数据库(CBM),时间到2017年12月31日截止。研究内容为探讨(艾司)西酞普兰和基因多态性的相关性临床试验。搜集文献的名称、年限、第一作者及所研究的基因。以基因研究文献的篇数界定该基因是否为(艾司)西酞普兰的临床相关性较大的基因,即热点基因。
2 结果
2.1 基因筛选结果 将检索词输入GeneCards,共得到140条结果(相关性得分1.1~21.2),从UniProtKB得到13条结果,从PharmGKB得到26条结果(证据级别1A级~4级)(表1)。本研究选取GeneCards的前20条结果(相关性得分在5.63以上)和PharmGKB的前20条结果(证据级别均在3级以上)。3个生物信息学数据库中有4个共同基因被确定:分别是SLC6A4、CYP2C19、CYP2D6和ABCB1(多药耐药基因1)(图1)。
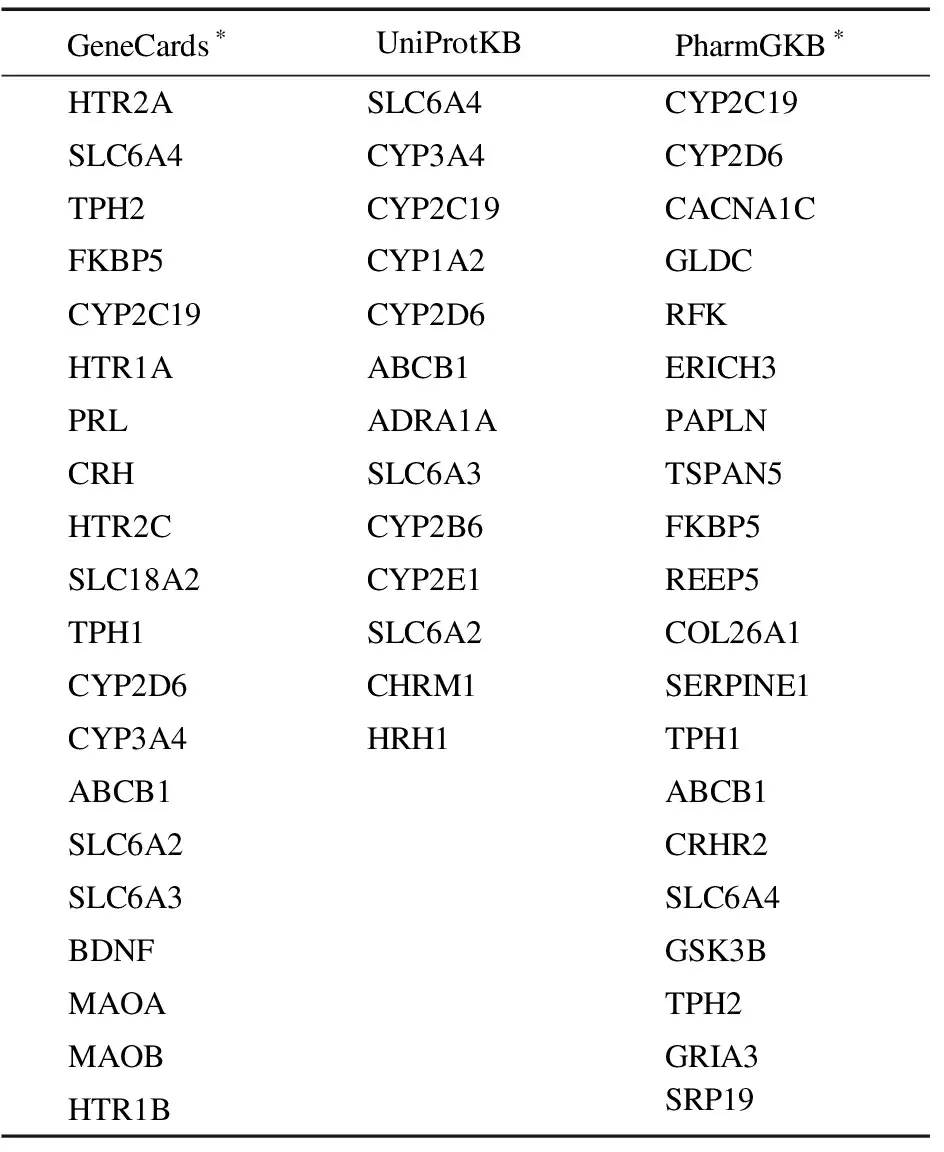
表1 GeneCards、PharmGKB、UniprotKB数据库搜索(艾司)西酞普兰相关基因
注:*前20条结果目
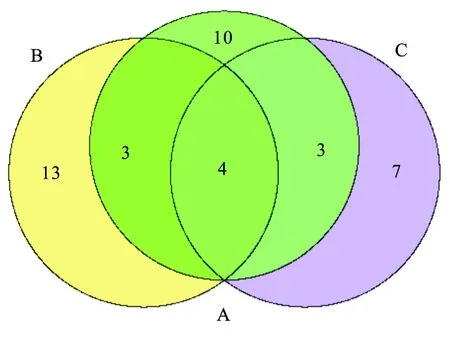
图1 GeneCards、PharmGKB、UniprotKB数据库搜索(艾司)西酞普兰相关基因重叠数文氏图
注:A:GeneCards;B:PharmGKB;C:UniprotKB
2.2 多态性位点筛选结果 将上述4个基因名称分别输入ClinVar数据库中,根据“条件”(Conditions)和“临床意义”(Clinical Significants)的描述选择与药物药动学、药效学相关的位点。其中,SLC6A4基因选取的是rs1131692236;CYP2C19是rs12248560,rs28399504、rs4986893,rs4244285,rs56337013;CYP2D6是rs730882251,rs267608319,rs730882170,rs16947,rs35742686,rs3892097,rs5030865,rs5030655,rs373000587,rs267608275,rs1065852;ABCB1是rs2032582 rs1045642(表2)。

表2 ClinVar数据库搜索基因多态性位点
2.3 文献筛选结果 将检索词输入文献数据库PubMed和CBM中,一共得到173篇文献,其中PubMed 159篇,CBM 14篇。查阅题目和摘要后剔除动物实验、综述、表观遗传学研究、全基因组关联分析及重复研究之后一共有38篇文献符合要求。其中研究基因频率从高到低排列是SLC6A4(13/38)、CYP2C19(12/38)、CYP2D6(6/38)、TPH1(5/38)、HTR2A(4/38)、BDNF(3/28)、ABCB1(2/38)、TPH2(2/38)、CYP3A4(2/38)、MAOA(2/38)、HTR6(1/38)、HTR5A(1/38)、HTR2C(1/38)、HTR1A(1/38)、CYP3A5(1/38)、CRHBP(1/38)、CNR1(1/38)、ABCC1(1/38)、5-HT1A(1/38)。见图2。
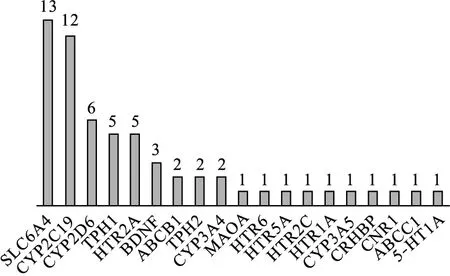
图2PubMed和CBM文献搜索(艾司)西酞普兰相关基因研究频率从高到低柱状图
注:A:GeneCards;B:PharmGKB;C:UniprotKB
3 讨论
随着人类基因组测序的完成,基因组学已进入“后基因时代”,也称“功能基因组时代”,即从揭示生命所有遗传信息到了分子水平的功能研究之上,由此诞生了一系列以研究功能为目的的“组学”科学,如“药物基因组学”、“环境基因组学”等。目前全球已有很多研究机构通过深度测序获得了有关疾病或药物反应的基因编码,但大多数全基因组关联分析(GWAS)虽耗费了大量的人力物力,却并未找到关键基因多态性位点,或者结果报道不一致[8-9]。对于临床一线的科研人员来说,重复GWAS研究或者从海量文献中筛选目标基因多态性位点做临床表型的关联分析会消耗大量的时间和经费,几乎难以完成。于是,了解和掌握生物信息学数据库的使用是一个可以让研究过程事半功倍的方法。
与文献搜索对比结果显示,GeneCards、PharmGKB和UniprotKB共有的4个基因中有三个(SLC6A4、CYP2C19、CYP2D6)是排列在文献搜索结果的前3位,但ABCB1没有在文献搜索的结果前4项中,而是在TPH1(色氨酸羟化酶1)、HTR2A(5-羟色胺受体2A)和BDNF之后。TPH1、HTR2A和BDNF虽然都在GeneCards中出现,但相关性得分分别为13.59、26.2、9.53,排名分别是11、1、17,TPH1、HTR2A和BDNF中任何一个都没在UniprotKB中出现而PharmGKB中只有TPH1(证据级别为3级)。由此看来,与搜索文献数据库相比,GeneCards数据库较为全面和准确,只是如果按相关性高低来筛选基因,可能会遗漏潜在相关性较大的基因,如果全部检测,则可能造成时间和经费的浪费。当然,仅靠文献数量来确定该基因是否与临床相关性较大是有一定片面性:虽然研究该基因的研究者多,但研究质量可能不高,如研究样本较少、未采用金标准检测方法等,而研究数量少的可能反而质量较高。或者,研究药效学的基因(如靶蛋白等,与缓解率有关[4-5,10-11])本身就比研究药动学的基因(如各种代谢酶或转运体,与不良反应有关[12-17])吸引更多的临床关注。此外,采用本研究的方式筛选到基因和位点后,还需要搜索该位点在特定人种中的突变频率,如果突变位点频率太低,则将其纳入临床研究中检测的意义并不大。以这次的结果为例,在中国汉族人群中,CYP2C19*17、*4、*3的突变频率均在10%以下,而*5没有数据报道;对于CYP2D6,只有rs16947和*10有较高的(>10%)突变频率,其余位点频率均较低或没有报道。突变频率的信息可以在NCBI的dbSNP数据库中查到。
综上所述,采用生物信息学数据库搜索基因+ClinVar搜索多态性位点的检索模式可以节约大量时间和精力,从文献中查找基因和位点,为后续试验设计和统计带来方便,值得向对基因多态性感兴趣的临床一线工作人员推荐。

