翻译后修饰与结核分枝杆菌耐药调控网络
谢龙祥,党艺方,谢建平
谢建平 西南大学生命科学学院现代生物医药研究所副所长,博导,研究员 (二级),中国微生物学理事/基础微生物/分子微生物与生物工程专委会副主任、中国微生物学会教学工作委员会委员、中国遗传学会微生物遗传专委会委员、中国医促会结核病防治分会结核病基础组副主任、中华医学会结核病分会基础专委会委员、重庆微生物学会副理事长、重庆免疫学会副理事长,中国毒理学会毒素毒理专委会理事、《遗传》副主编、《生物工程学报》、《药学学报》、《微生物学报》、《中国抗生素杂志》和编委,享受国务院政府特殊津贴专家(2010),教育部新世纪优秀人才计划入选者(2011),重庆市缙云英才入选者。主要围绕结核病持续开展研究,先后主持或者主研国家传染病科技重大专项、国家重点研发计划、国家自然科学基金等20余项,近5年以通讯作者发表SCI论文80余篇。主译/副主编《图解微生物学实验指南》、《结核病免疫学》、《分枝杆菌分子生物学》、《微生物学》、《药学细胞生物学》和《DNA科学导论》等教材10余部。《生活中的DNA科学》获教育部精品视频课程(2014) 和重庆市教学技术成果二等奖(2016)。“结核病防控新措施的基础研究”获重庆市自然科学三等奖(2015)。

翻译后修饰与结核分枝杆菌耐药调控网络
谢龙祥1,党艺方1,谢建平2
1 河南大学 基础医学院 医学生物信息研究所,河南 开封 475004 2 西南大学 生命科学学院 现代医药生物研究所,重庆 400715

谢龙祥, 党艺方, 谢建平. 翻译后修饰与结核分枝杆菌耐药调控网络.生物工程学报, 2018, 34(8): 1279–1287.Xie LX, Dang YF, Xie JP. Post-translational modification and regulatory network of Mycobacterium tuberculosis antibiotic resistance. Chin J Biotech, 2018, 34(8): 1279–1287.
目前对于结核分枝杆菌(,Mtb) 耐药产生机制研究得较多,但对其调控机制的研究较少。翻译后修饰(Post-translational modifications,PTMs) 在结核菌多种生理途径(如代谢、应激反应等) 中发挥重要调控作用,而它们和结核菌耐药之间的关系逐渐引起了研究者的关注。文中介绍了结核菌抗生素耐受机制以及存在的一些PTMs,重点讨论了PTMs在调控结核杆菌耐药机制中的潜在作用,以期为新型抗结核药物研发提供新的切入点。
翻译后修饰,结核分枝杆菌,抗生素,耐药
由结核分枝杆菌 (以下简称结核菌) 引起的结核病是人类健康的主要威胁之一。据报道,全球每年有1 040万人感染结核病,180万人死于结核病[1]。近些年,由于抗生素的滥用导致多耐药 (Multidrug resistant,MDR) 和广泛性耐药 (Extensively drug-resistant,XDR) 结核菌的出现,这使得结核病疫情愈加严重。因此,深入理解结核菌耐药机制并在此基础上开发新型抗结核药物就显得尤为迫切和重要。
结核菌对许多抗生素是天然耐药的,这主要是由于结核菌具有厚、蜡状、疏水的细胞被膜[1],体内存在可使药物降解失活或修饰的酶[3-4]。此外,结核菌存在大量的药物外排泵系统,这也可以赋予它们一定水平的临床耐药性[5]。除了天然耐药外,大多数结核菌的耐药表型可以归结于染色体突变造成的[6]。这些染色体突变可以通过修饰或过表达药物靶标,或活化前体药物来赋予菌株不同水平的耐药性。表1概括了导致结核菌耐药常见的染色体突变靶标[6]。虽然目前关于结核菌耐药产生机制研究得较为透彻,但其具体的调控机制仍不清楚。

表1 结核菌耐药机制及常见的染色体突变靶标
蛋白质的许多PTMs在高等和低等生物中均存在,它们可参与水解或将修饰基团转移到蛋白质的一个或多个氨基酸。这些PTMs可能影响蛋白质的活化状态、定位、转换和/或与其他蛋白质的相互作用[7]。随着依赖质谱 (Mass spectrometry,MS) 的蛋白质组学技术快速发展,细菌中大量的蛋白质被发现存在PTMs,例如磷酸化 (Phosphorylation)、乙酰化 (Acetylation)、类泛素化 (Pupylation) 等,并且一些蛋白质的PTMs被证明在细菌的多种生理途径中发挥重要调控作用。2015年,Nakedi等解析并比较了对数时期的快生型耻垢分枝杆菌和慢生型牛分枝杆菌BCG的蛋白质磷酸化组,发现牛分枝杆菌呈现复杂的蛋白质磷酸化网络,而该网络可以调控细胞分裂、细胞壁合成、快速应对压力反应等细胞途径[8]。2017年,Birhanu等利用定量乙酰化蛋白质组学方法分析了3种Mtb临床分离菌株 (2种谱系7分离株和谱系4 H37Rv标准株),鉴定了953个蛋白质中存在2 490个class-Ⅰ乙酰化位点、2 349个O乙酰化位点和141个Nε乙酰化位点[9]。乙酰化蛋白质被发现参与中心代谢、翻译和应激反应以及抗生素耐受[9]。鉴于PTMs在应对环境压力中的重要作用,人们推测PTMs可能也参与抗生素耐受过程[10]。文中提供了PTMs (也扩展包含了抗生素的化学修饰) 可能调控结核菌耐药的证据。
1 在结核菌中抗生素暴露和PTMs之间是否存在联系?
在过去数年中,多个研究暗示PTMs和抗生素暴露之间可能存在一定的关联。在经典的磷酸化修饰中,蛋白质激酶转移ATP上的γ-磷酸到蛋白质的特定氨基酸 (通常为丝氨酸、苏氨酸和酪氨酸) 上来启动信号转导[11]。结核菌基因组包含11个丝氨酸/苏氨酸磷酸激酶基因 (Serine/threonine protein kinases,STPK),也就是、和–[12]。分析已报道的转录组数据发现[12],结核菌在卷曲霉素 (Capreomycin,Cap)、阿米卡星 (Amikacin,Ami)、链霉素 (Streptomycin,SM) 处理条件下,基因分别下调4.76倍、2.11倍、2.14倍;在四环素 (Tetracycline,Tet) 处理条件下,基因下调3.41倍;在异烟肼 (Isoniazid,INH)处理条件下,基因上调2.71倍;在利福平(Rifampicin,Rif) 处理条件下,基因上调5.2倍;在罗红霉素 (Roxithromycin,Rox) 和链霉素 (Streptomycin,SM) 处理条件下,基因分别上调2.14倍和2倍。值得注意的是,有研究表明过表达PknA和PknB激酶 (其属于编码参与细胞形状调控和细胞壁合成基因的操纵子的一部分) 会导致结核菌生长变慢并改变细胞形态,或的部分敲除会导致结核菌形态变窄和变长[14]。此外,缺失基因会导致耻垢分枝杆菌和结核菌对多种抗生素的敏感[15]。这些数据表明负责PTMs的关键酶或PTMs可能和抗生素处理之间有一定的功能联系。
2 PTMs是如何作用导致结核菌耐药改变呢?
抗生素可以利用多种机制靶向关键途径,例如核酸和蛋白质合成、被膜完整性以及活性氧等。有趣的是,结核菌的各种PTMs也参与这些功能。因此,PTMs活动的变动可能导致细胞途径的变化,进而调控结核菌的抗生素耐受 (图1)。
2.1 可能参与细胞壁合成
研究表明Wag31的磷酸化在耻垢分枝杆菌抗生素耐受中发挥作用[16]。Wag31是细胞分裂蛋白质DivIVA的同源蛋白,它可以调控分枝杆菌的生长、形态和极性细胞壁合成。Wag31蛋白也可以与ACCase酶亚基AccA3相互作用,进而调控脂质和分枝菌酸 (Mycolic acids) 合成。AccA3对酰基链延伸是至关重要的,因为它可以将乙酰辅酶A (Acetyl-CoA) 转换成丙二酰辅酶A (Malonyl-CoA)。AccA3过表达可以导致渗透性降低进而增加耻垢分枝杆菌对利福平和新生霉素 (Novobiocin) 的抗性。Wag31-AccA3的相互作用可以促进细胞壁的稳定性[17]。Wag31可被分枝杆菌激酶PknA磷酸化,且Wag31的磷酸化可以帮助其蛋白质-蛋白质相互作用,也可调控和促进肽聚糖 (Peptidoglycan,PG) 生物合成。
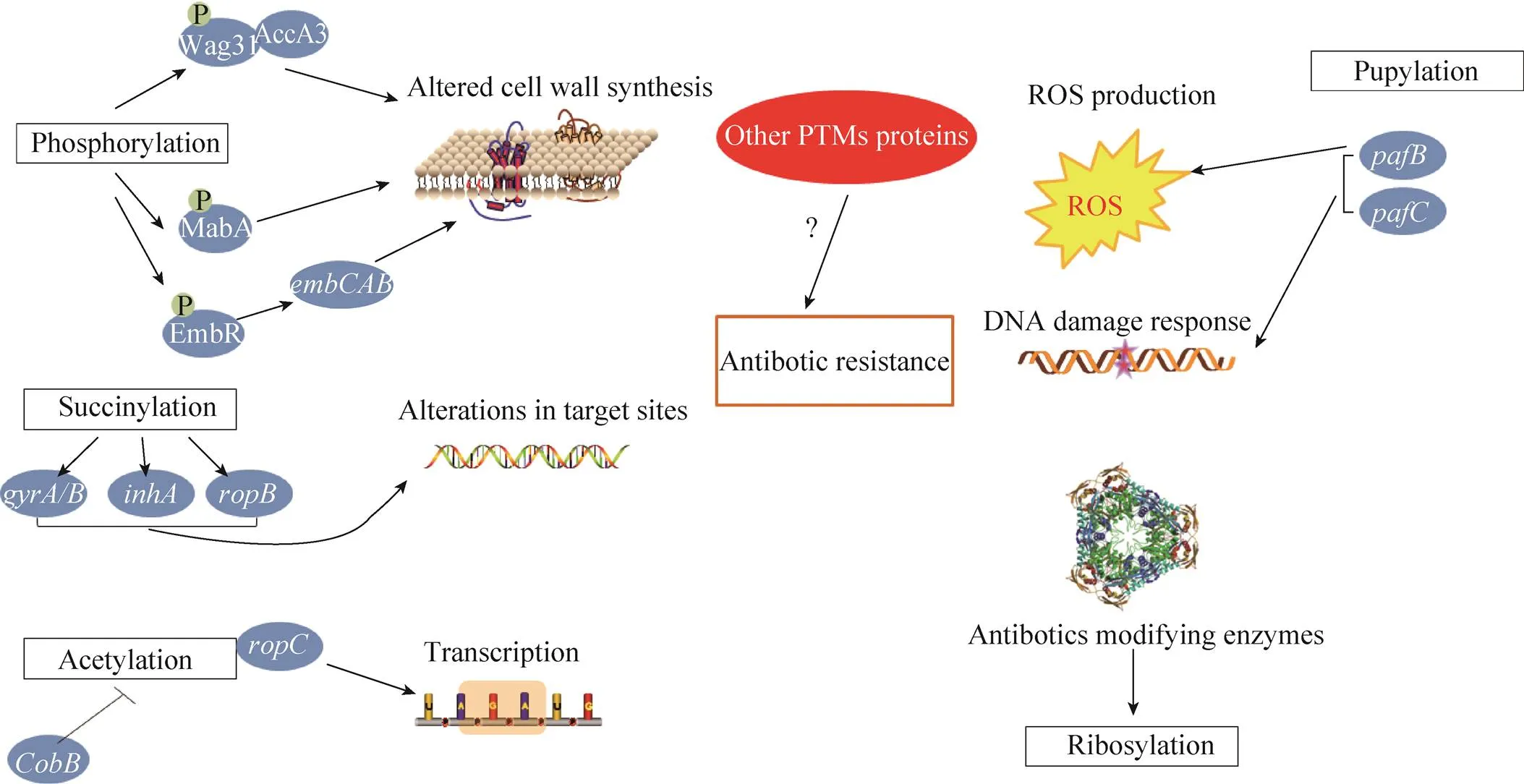
图1 结核菌中PTMs和抗生素耐受之间的联系
此外,结核菌MabA (β酮脂酰基载体蛋白还原酶) 的磷酸化可以调控分枝菌酸生物合成途径[18]。包括PknB在内的一些Ser/Thr蛋白激酶体外和体内可以对分枝杆菌的MabA进行磷酸化。质谱分析和定点突变鉴定了3个磷酸苏氨酸,其中Thr191是主要的磷酸化受体。与野生型蛋白质相比,用来模拟组成型磷酸化的MabA_T191D突变体的酮酰还原酶活性明显下降[18]。在BCG中组成型过表达MabA_T191D会严重损害分枝杆菌的生长,导致分枝菌酸从头合成途径明显受到抑制[18]。Ando等研究表明MabAg609a的沉默突变可以通过增加的转录水平来赋予结核菌对INH的耐受[19]。
参与细胞壁组分阿拉伯聚糖合成的基因簇(阿拉伯聚糖转移酶) 是乙胺丁醇的作用靶标,而EmbR是基因转录激活器。2003年,Molle等发现PknH可以对含有FHA磷酸化蛋白质识别区域的EmbR磷酸化[20]。2006年,Sharma等研究证明EmbR的磷酸化可以影响脂阿拉伯甘露糖/脂甘露糖(LAM⁄LM)比率,而该比率是分枝杆菌毒力和对抗结核药物乙胺丁醇耐受的重要决定因素[21]。同年,他们又证明PknA和PknB是导致EmbR磷酸化的新型调控器,而上文中我们提到PknA﹑PknB与结核菌的生长和细胞形态有密切关系[22]。
2.2 可能参与转录调控
一些抗生素可以改变转录机器 (Transcriptional machinery),而PTMs已被发现可以调控此过程。Nε-赖氨酸乙酰化是一种由乙酰化酶和去乙酰化酶所催化的可逆的、广泛的、动态的调控机制,它可以影响蛋白质的多种特性,包括酶活性、DNA-蛋白质相互作用、亚细胞定位、转录活性和蛋白质稳定性等[23]。2015年,Gu等研究表明依赖烟酰胺腺嘌呤二核苷 (NAD+) 的去乙酰酶MSMEG_5175可以通过两条途径来参与异烟肼抗性:1) 直接调控胞内NAD或NADH水平;2) 影响RNA聚合酶β′亚基 (RpoC) 乙酰化水平控制全局性基因转录水平[24]。与包含pMV261的耻垢分枝杆菌空载菌 (mc2155-pMV261) 相比,MSMEG_5175过表达菌株 (mc2155-MS5175) 具有较低的胞内NAD水平但较高的异烟肼抗性。定量蛋白质组学分析表明,MSMEG_5175的过表达可以导致34个蛋白质上调表达和72个蛋白质下调表达。这些差异性蛋白质参与包括代谢激活、转录、翻译、抗氧化和DNA损伤在内的多种细胞途径。在mc2155-MS5175菌株中,过氧化氢酶 (KatG) 的mRNA和蛋白质水平均发生了下调。此外,免疫共沉淀和蛋白质组学联合分析表明mc2155-MS5175中27个蛋白质的乙酰化水平下调,其中RpoC的乙酰化水平的改变可能影响它的自身功能并导致整体基因转录的变化[24]。
2.3 可能参与活性氧产生和DNA损伤反应
抗生素诱导细菌产生的ROS (Reactive oxygen species) 已被深入研究。尽管曾有不少争议,但是最新研究表明氨苄青霉素、卡那霉素、诺氟沙星、莫西沙星等抗生素都可诱导细菌产生ROS[25-26]。原核类泛素化蛋白 (Prokaryotic ubiquitin-like protein,Pup) 由64个氨基酸组成,相对分子量为6 900,是一种存在于分枝杆菌中类似真核生物泛素 (Ubiquitin,Ub) 的小分子蛋白质[27]。分枝杆菌的蛋白质酶体和Pup组成了完整的Pup-蛋白酶体系统 (PPS)。当蛋白质被Pup共价标记后,修饰的蛋白质将被蛋白酶体降解或参与修饰后的调控过程,最终影响细菌各个生命活动。位于同一个操纵子的Pup连接酶PafA、PafB和PafC是分枝杆菌和其他放线菌中Pup-蛋白酶体系统的一部分。2015年,我们课题组研究表明耻垢分枝杆菌突变菌株对氟喹诺酮类抗生素 (包括莫西沙星、诺氟沙星、氧氟沙星和环丙沙星) 是超敏感的,但是对其他抗生素例如异烟肼、利福平、氯霉素和卷曲霉素的敏感性是没有改变的。回补实验恢复了和野生型耻垢分枝杆菌表型[28]。突变菌株对H2O2是非常敏感的,并且铁螯合剂 (联吡啶) 和羟基自由基清除剂 (硫脲) 可以消除此差异[28]。最近Olivencia等证明参与分枝杆菌DNA损伤反应[29]。耻垢分枝杆菌突变菌株的蛋白质组学分析揭示参与DNA损伤反应的各种蛋白质 (包括重组酶RecA) 的胞内水平是降低的,同时,突变菌株对DNA损伤试剂敏感性增加[29]。
2.4 参与抗生素的直接修饰
分枝杆菌中存在一些酶,这些酶可以将某些化学基团转移到抗生素上对其进行修饰,而该修饰可以降低靶标的结合能力进而使药物失活。Eis是基因编码的乙酰化酶 (GNAT) 超家族蛋白,它可促进耻垢分枝杆菌在人类巨噬细胞系U-937的传代过程中的存活。此外,由RNA聚合酶σ因子SigA调控的Eis表达也可增进W-Beijing菌株在单核细胞中的胞内存活。2009年,Zaunbrechera等研究证明Eis的过量表达可赋予结核菌对卡那霉素的抗性[30]。2011年,Gikalo等研究表明Eis的突变在莫斯科区域结核菌临床菌株卡那霉素抗性发展中是至关重要的[31],随后Chen等发现Eis可以直接对多种氨基糖苷类抗生素的多个氨基乙酰化[32]。
ADP-核糖转移酶可以将ADP-ribosyl基团转移到抗生素,进而抑制抗生素接近靶标。例如,利福平是一种可以抑制RNA聚合酶β亚基的半合成抗生素,耻垢分枝杆菌中ADP-核糖转移酶可以利用NAD作为辅因子将ADP-ribosyl加到利福平上,而利福平的核糖基化会使其失活,导致耻垢分枝杆菌对利福平耐受[4]。近来,Rominski等证明ADP-核糖基化酶MAB_0591对于脓肿分枝杆菌的天然耐药是必需的[33]。MAB_0591的异源表达可以赋予大肠杆菌和结核菌对利福平的抗性。与脓肿分枝杆菌ATCC 19977菌株相比,脓肿分枝杆菌缺失菌株对利福平的MIC值始终要低。当缺失菌株回补后,恢复与野生型菌株相同的表型[33]。
2.5 可能参与靶标位点的改变
赖氨酸琥珀酰化 (Lysine succinylation) 是近年来新发现的一种新型赖氨酸酰化修饰[34]。2014年,我们课题组利用免疫亲和富集和高灵敏度质谱联合方法绘制了中国广泛耐药结核菌临床菌株的全局性琥珀酰化蛋白图谱,发现有686个琥珀酰化蛋白和1 739个琥珀酰化位点[35]。值得注意的是其中有4个抗结核药物靶标蛋白质被琥珀酰化修饰,包括InhA、RopB和GyrA/GyrB。氟喹诺酮 (Fluoroquinolones,FQs) 是治疗多耐药结核病的第二线药物,而由和基因编码的Ⅱ型DNA拓扑异构酶是其靶标蛋白。GyrA和GyrB蛋白均被发现存在琥珀酰化修饰,其中GyrA蛋白有5个琥珀酰化位点,GyrB蛋白有10个琥珀酰化位点。GyrB蛋白中的一个琥珀酰化修饰位点K523在空间上接近自然突变位点 (A515T或A515V),而该突变位点可以导致对抗生素的耐受。RopB蛋白被发现存在12个琥珀酰化修饰位点,其中一个琥珀酰化修饰位点空间上接近自然突变位点 (I561V)。2010年,Prisic等利用磷酸化蛋白质组学方法对结核菌的蛋白质磷酸化进行了全局性分析,他们鉴定了301个参与广泛功能的磷酸化蛋白质和500个磷酸化位点[36]。其中GyrA和RpoB均被发现存在1个磷酸化位点。因此,探究这些靶标蛋白质的翻译后修饰与抗生素耐受之间的关系将是非常有必要的。
3 与抗生素耐受相关的翻译后修饰蛋白底物
结核菌中还存在一些非药物靶标但与抗生素耐受相关的蛋白存在翻译后修饰。有研究发现结核菌中有58个类泛素化蛋白[37],其中负责催化分枝菌酸和脂肪酸合成的关键酶GlmU (UDP-N-乙酰氨基葡萄糖焦磷酸化酶)、MurA (UDP-N-乙酰基葡萄糖羧乙烯基转移酶) 和FabD (丙二酰辅酶A-酰基载体蛋白转酰酶)、KasB (β-酮脂酰-ACP合成酶)、FabG4 (3-酮脂酰-酰基载体蛋白脱氢酶) 以及反应调节子A (MtrA) 被发现存在Pup修饰。结核菌MtrA参与广泛的调节作用,对结核菌的体外生长繁殖是至关重要的。Nguyen等研究证明MtrA、MtrB和脂蛋白LpqB组成了一个系统来共同调控分枝杆菌的天然耐多药性、细胞动力学和细胞壁内稳态过程[38]。此外,组学鉴定发现异烟肼诱导蛋白IniB存在3个磷酸化位点,IniC存在2个磷酸化位点,与分枝杆菌天然耐药相关的PknG存在1个磷酸化位点[36]。探究这些底物的翻译后修饰是否直接参与结核菌抗生素耐受是将来需要解决的问题。
4 负责PTMs的关键酶是否可以作为新型抗结核药物靶标?
2016年,Garzan等[39]高通量筛选得到了乙酰化酶Eis的两种抑制剂甲基四氢呋喃 (3,2-b) 吡咯-5-羧酸酯和3-(1,3-二氧杂环丁烷)-2-吲哚酮,其中后者和KAN联合使用时,可以明显降低KAN耐药结核菌的MICKAN (Minimum inhibitory concentration,MIC)。最近他们又发现了Eis的新型高效抑制剂——吡咯并 (1,5-α) 吡嗪类似物,该化合物对动物细胞没有明显的毒性,有望成为治疗耐药结核病的氨基糖苷类抗生素佐剂[40]。2017年,Xu等鉴定了一种新型蛋白激酶PknB抑制剂IMB-YH-8,该小分子化合物可以特异性抑制PknB自磷酸化以及GarA的磷酸化,但对人的激酶Akt1或结核菌中的其他激酶活性没有抑制作用[41]。值得注意的是,IMB-YH-8不仅可以抑制敏感型和多耐药临床菌株,也可以抑制胞内结核菌生长。以上这些数据均表明负责PTMs的关键酶可成为抗结核药物设计的新靶标[41]。
5 结语
目前PTMs在结核菌耐药方面作用的研究处于起步阶段,各种PTMs的研究例子较少,因此有许多问题需要解决。例如,除了直接对多种氨基糖苷类抗生素乙酰化外,Eis是否可以通过乙酰化修饰某个关键蛋白质来参与抗生素耐受途径呢?因为最近Ghosh等利用免疫共沉淀方法证明了结核菌Eis可以对拟核相关蛋白MtHU的多个赖氨酸残基进行乙酰化修饰,且MtHU的乙酰化会导致拟核蛋白的DNA压缩能力改变[42]。结核菌中是否还存在其他新型PTMs?结核菌中各种PTMs之间是否存在交叉对话,且这些交叉对话网络是否会影响抗生素耐受?MDR和XDR临床结核菌中各种PTMs的定量变化情况是怎样的?虽然目前对PTMs和结核菌耐药网络的研究还不够成熟,但是对PTMs及其关键酶的进一步研究可能为结核病治疗和抗结核新药物的研发提供新的线索。
[1] Organization WH. Global tuberculosis report 2016.
[2] Jarlier V, Nikaido H. Mycobacterial cell wall: structure and role in natural resistance to antibiotics. FEMS Microbiol Lett, 1994, 123(1/2): 11–18.
[3] Warrier T, Kapilashrami K, Argyrou A, et al.-methylation of a bactericidal compound as a resistance mechanism in. Proc Natl Acad Sci USA, 2016, 113(31): E4523–E4530.
[4] Wright GD. Bacterial resistance to antibiotics: enzymatic degradation and modification. Adv Drug Deliv Rev, 2005, 57(10): 1451–1470.
[5] Li GL, Zhang JR, Guo Q, et al. Efflux pump gene expression in multidrug-resistantclinical isolates. PLoS ONE, 2015, 10(2): e0119013.
[6] Gygli SM, Borrell S, Trauner A, et al. Antimicrobial resistance in: mechanistic and evolutionary perspectives. FEMS Microbiol Rev, 2017, 41(3): 354–373.
[7] Broberg CA, Orth K. Tipping the balance by manipulating post-translational modifications. Curr Opin Microbiol, 2010, 13(1): 34–40.
[8] Nakedi KC, Nel AJ, Garnett S, et al. Comparative Ser/Thr/Tyr phosphoproteomics between two mycobacterial species: the fast growing Mycobacterium smegmatis and the slow growingBCG. Front Microbiol, 2015, 6: 237.
[9] Birhanu AG, Yimer SA, Holm-Hansen C, et al. Nε- and O-Acetylation inlineage 7 and lineage 4 strains: proteins involved in bioenergetics, virulence, and antimicrobial resistance are acetylated. J Proteome Res, 2017, 16(11): 4045–4059.
[10] Kandpal M, Aggarwal S, Jamwal S, et al. Emergence of drug resistance inand other bacterial pathogens: the posttranslational modification perspective//Arora G, Sajid A, Kalia V, eds. Drug Resistance in Bacteria, Fungi, Malaria, and Cancer. Cham: Springer, 2017.
[11] Tarrant MK, Cole PA. The chemical biology of protein phosphorylation. Annu Rev Biochem, 2009, 78(1): 797–825.
[12] Wehenkel A, Bellinzoni M, Graña M, et al. Mycobacterial Ser/Thr protein kinases and phosphatases: physiological roles and therapeutic potential. Biochim Biophys Acta-Prot Proteom, 2008, 1784(1): 193–202.
[13] Boshoff HIM, Myers TG, Copp BR, et al. The transcriptional responses ofto inhibitors of metabolism: novel insights into drug mechanisms of action. Jf Biol Chem, 2004, 279(38): 40174–40184.
[14] Kang CM, Abbott DW, Park ST, et al. Theserine/threonine kinases PknA and PknB: substrate identification and regulation of cell shape. Genes Dev, 2005, 19(14): 1692–1704.
[15] Wolff KA, Nguyen HT, Cartabuke RH, et al. Protein kinase G is required for intrinsic antibiotic resistance in mycobacteria. Antimicrob Agents Chemother, 2009, 53(8): 3515–3519.
[16] Hamasha K, Sahana MB, Jani C, et al. The effect of Wag31 phosphorylation on the cells and the cell envelope fraction of wild-type and conditional mutants ofstudied by visible-wavelength Raman spectroscopy. Biochem Biophys Res Commun, 2010, 391(1): 664–668.
[17] Xu WX, Zhang L, Mai JT, et al. The Wag31 protein interacts with AccA3 and coordinates cell wall lipid permeability and lipophilic drug resistance in. Biochem Biophys Res Commun, 2014, 448(3): 255–260.
[18] Veyron-Churlet R, Zanella-Cléon I, Cohen-Gonsaud M, et al. Phosphorylation of theβ-Ketoacyl-Acyl carrier protein reductase MabA regulates mycolic acid biosynthesis. J Biol Chem, 2010, 285(17): 12714–12725.
[19] Ando H, Miyoshi-Akiyama T, Watanabe S, et al. A silent mutation inconfers isoniazid resistance on. Mol Microbiol, 2014, 91(3): 538–547.
[20] Molle V, Kremer L, Girard-Blanc C, et al. An FHA phosphoprotein recognition domain mediates protein EmbR phosphorylation by PknH, a Ser/Thr protein kinase from. Biochemistry, 2003, 42(51): 15300–15309.
[21] Sharma K, Gupta M, Krupa A, et al. EmbR, a regulatory protein with ATPase activity, is a substrate of multiple serine/threonine kinases and phosphatase in. FEBS J, 2006, 273(12): 2711–2721.
[22] Sharma K, Gupta M, Pathak M, et al. Transcriptional control of the mycobacterialoperon by PknH through a regulatory protein, EmbR,. J Bacteriol, 2006, 188(8): 2936–2944.
[23] Carabetta VJ, Cristea IM. The regulation, function, and detection of protein acetylation in bacteria. J Bacteriol, 2017, 199(16): e00107–17.
[24] Gu LX, Chen YL, Wang QT, et al. Functional characterization of sirtuin-like protein in. J Proteome Res, 2015, 14(11): 4441–4449.
[25] Ferrándiz MJ, Martín-Galiano AJ, Arnanz C, et al. Reactive oxygen species contribute to the bactericidal effects of the fluoroquinolone moxifloxacin in Streptococcus pneumoniae. Antimicrob Agents Chemother, 2015, 60(1): 409–417.
[26] Kottur J, Nair DT. Reactive oxygen species play an important role in the bactericidal activity of quinolone antibiotics. Angew Chem Int Ed, 2016, 55(7): 2397–2400.
[27] Darwin KH. Prokaryotic ubiquitin-like protein (Pup), proteasomes and pathogenesis. Nat Rev Microbiol, 2009, 7(7): 485–491.
[28] Li QM, Xie LX, Long QX, et al. Proteasome accessory factor C () is a novel gene involved inintrinsic resistance to broad-spectrum antibiotics–fluoroquinolones. Sci Rep, 2015, 5: 11910.
[29] Olivencia BF, Müller AU, Roschitzki B, et al.PafBC is involved in regulation of DNA damage response. Sci Rep, 2017, 7: 13987.
[30] Zaunbrecher MA, Sikes RD, Metchock B, et al. Overexpression of the chromosomally encoded aminoglycoside acetyltransferase eis confers kanamycin resistance in. Proc Natl Acad Sci USA, 2009, 106(47): 20004–20009.
[31] Gikalo MB, Nosova EY, Krylova LY, et al. The role of eis mutations in the development of kanamycin resistance inisolates from the Moscow region. J Antimicrob Chemother, 2012, 67(9): 2107–2109.
[32] Chen WJ, Biswas T, Porter VR, et al. Unusual regioversatility of acetyltransferase Eis, a cause of drug resistance in XDR-TB. Proc Natl Acad Sci USA, 2011, 108(24): 9804–9808.
[33] Rominski A, Roditscheff A, Selchow P, et al. Intrinsic rifamycin resistance ofis mediated by ADP-ribosyltransferase MAB_0591. J Antimicrob Chemother, 2017, 72(2): 376–384.
[34] Weinert B, Schölz C, Wagner SA, et al. Lysine succinylation is a frequently occurring modification in prokaryotes and eukaryotes and extensively overlaps with acetylation. Cell Rep, 2013, 4(4): 842–851.
[35] Xie LX, Liu W, Li QM, et al. First succinyl-proteome profiling of extensively drug-resistantrevealed involvement of succinylation in cellular physiology. J Proteome Res, 2015, 14(1): 107–119.
[36] Prisic S, Dankwa S, Schwartz D, et al. Extensive phosphorylation with overlapping specificity byserine/threonine protein kinases. Proc Natl Acad Sci USA, 2010, 107(16): 7521–7526.
[37] Pearce MJ, Arora P, Festa RA, et al. Identification of substrates of theproteasome. EMBO J, 2006, 25(22): 5423–5432.
[38] Nguyen HT, Wolff KA, Cartabuke RH, et al. A lipoprotein modulates activity of the MtrAB two-component system to provide intrinsic multidrug resistance, cytokinetic control and cell wall homeostasis in. Mol Microbiol, 2010, 76(2): 348–364.
[39] Garzan A, Willby MJ, Green KD, et al. Discovery and optimization of two Eis inhibitor families as kanamycin adjuvants against drug-resistant. ACS Med Chem Lett, 2016, 7(12): 1219–1221.
[40] Garzan A, Willby MJ, Ngo HX, et al. Combating enhanced intracellular survival (Eis)-mediated kanamycin resistance ofby novel pyrrolo[1, 5-a]pyrazine-based eis inhibitors. ACS Infect Dis, 2017, 3(4): 302–309.
[41] Xu J, Wang JX, Zhou JM, et al. A novel protein kinase inhibitor IMB-YH-8 with anti-tuberculosis activity. Sci Rep, 2017, 7: 5093.
[42] Ghosh S, Padmanabhan B, Anand C, et al. Lysine acetylation of theHU protein modulates its DNA binding and genome organization. Mol Microbiol, 2016, 100(4): 577–588.
(本文责编 郝丽芳)
Post-translational modification and regulatory network ofantibiotic resistance
Longxiang Xie1, Yifang Dang1, and Jianping Xie2
1 Institute of Biomedical Informatics, BasicMedical School, Henan University, Kaifeng 475004, Henan, China 2 Institute of Modern Biopharmaceuticals, School of Life Sciences, Southwest University, Chongqing 400715, China
Currently, there are many studies on the mechanism of antibiotic resistance in(Mtb), but there are few studies on its regulatory mechanism. Post-translational modifications (PTMs) have been recognized for their important role in controlling cellular dynamics such as metabolism and stress response, but the relationship between PTMs and antibiotic resistance gradually attracted the attention of researchers. Here, we summarize the definition of PTMs, and the mechanisms of antibiotic resistance inand discuss how PTMs are involved in antibiotic resistance, in order to provide a new breakthrough for the development of new anti-Ttb drugs.
posttranslational modifications,, antibiotic, antibiotic resistance
December 29, 2017;
March 15, 2018
Doctoral Fund of Ministry of Education of China (No. 2017M62237), National Key Research and Development Program of China (No. 2016YFC0502304).
Jianping Xie. Tel/Fax: +86-23-68367108; E-mail: georgex@swu.edu.cn
中国博士后基金(No. 2017M62237), 国家重点研发项目(No. 2016YFC0502304) 资助。
10.13345/j.cjb.170530

