新型抗结核药物研发:微生物非常规培养与沉默基因激活策略
滕铁山,谢龙祥,谢建平
谢建平 西南大学生命科学学院现代生物医药研究所副所长,博导,研究员 (二级),中国微生物学理事/基础微生物/分子微生物与生物工程专委会副主任、中国微生物学会教学工作委员会委员、中国遗传学会微生物遗传专委会委员、中国医促会结核病防治分会结核病基础组副主任、中华医学会结核病分会基础专委会委员、重庆微生物学会副理事长、重庆免疫学会副理事长,中国毒理学会毒素毒理专委会理事、《遗传》副主编、《生物工程学报》、《药学学报》、《微生物学报》、《中国抗生素杂志》和编委,享受国务院政府特殊津贴专家 (2010),教育部新世纪优秀人才计划入选者 (2011),重庆市缙云英才入选者。主要围绕结核病持续开展研究,先后主持或者主研国家传染病科技重大专项、国家重点研发计划、国家自然科学基金等20余项,近5年以通讯作者发表SCI论文80余篇。主译/副主编《图解微生物学实验指南》、《结核病免疫学》、《分枝杆菌分子生物学》、《微生物学》、《药学细胞生物学》和《DNA科学导论》等教材10余部。《生活中的DNA科学》获教育部精品视频课程 (2014) 和重庆市教学技术成果二等奖 (2016)。“结核病防控新措施的基础研究”获重庆市自然科学三等奖 (2015)。

新型抗结核药物研发:微生物非常规培养与沉默基因激活策略
滕铁山1,2,谢龙祥1,2,谢建平2
1 河南大学 基础医学院 医学生物信息学研究所,河南 开封 475004 2 西南大学 生命科学学院 现代生物医药研究所 三峡库区生态环境与生物资源省部共建国家重点实验室培育基地,重庆 400715

滕铁山, 谢龙祥, 谢建平. 新型抗结核药物研发:微生物非常规培养与沉默基因激活策略.生物工程学报, 2018, 34(8): 1306–1315.Teng TS, Xie LX, Xie JP. Development of new anti-tuberculosis drugs: the strategy of unconventional microbial culture and silencing gene activation. Chin J Biotech, 2018, 34(8): 1306–1315.
由结核分枝杆菌感染引起的结核病是人类重要传染病之一。临床上结核菌耐药性日趋严重,不断出现的耐多药及广泛耐药结核病患者,使现有的一线至五线药物不能满足结核病防控需求。微生物来源的天然产物是药物先导化合物的重要来源。环境中存在大量常规培养条件下未培养微生物,同时微生物基因组中也存在大量未被表达的“沉默代谢途径”。运用各种方法对未培养微生物进行再培养,同时激活微生物的沉默代谢途径,进而获得潜在的新型抗生素药物已成为目前研究热点。文中系统阐述了近年来获取天然化合物所采用的微生物非常规培养技术及沉默代谢途径激活策略,同时总结了利用这两种方法获得的新型抗结核天然产物,并展望了这些方法在抗结核药物进一步研发中的应用前景。
结核分枝杆菌,未培养微生物,沉默基因,单株菌-多次级代谢产物 (OSMAC)
结核病是由结核分枝杆菌 (,以下简称结核菌) 感染引起的、严重危害人类健康的重要传染病[1-2]。耐多药结核病 (Multidrug-resistant tuberculosis,MDR-TB) 的出现使一线抗结核药物不能有效达到治疗目的,此时须使用二线以上抗结核药物进行治疗。二线抗结核药物治疗周期平均在2–4年,且不如一线药物治疗效果好,常伴有严重的不良反应,导致患者的依从性差、复发率高,还可能导致广泛耐药结核病 (Extensively drug-resistant tuberculosis,XDR-TB) 的出现[3]。最近又发现了全耐药结核菌 (Totally drug-resistant tuberculosis,TDR-TB),TDR-TB定义为结核菌在体外对所有的一线和二线抗结核药物都有抗性,这更使得结核病的防治雪上加霜[4]。为了应对这种状况,美国和欧洲药品管理局分别在2012年和2013年推出了两种新型抗结核药物贝塔喹啉(Bedaquiline,又称TMC207) 和迪拉马尼片(Delamanid,又称OPC67683),二者被专一用作耐药结核的治疗[5-6]。但即便如此,临床上已经分离到这两种抗结核药物的耐药菌株。结核病的防控需要发展与现存药物没有交叉抗性的新型药物[7-8]。
微生物次级代谢产物是药物先导化合物的重要来源,目前临床上使用的60%的抗癌药物和70%的抗生素是来源于天然产物及其衍生物[9]。面对病原菌耐药严重的问题,重新发掘具有独特作用机制的天然药物显得势在必行。然而由于对微生物原位生长的环境条件及培养基质方法的缺乏,超过99%的微生物不能通过传统培养方法获得,而这些微生物中蕴含着巨大的生物资源[10]。所以,增强微生物的可培养性、把更多的未培养微生物转变成为可培养微生物将是一件意义重大的工作。其次,在微生物的合成途径中存在大量在传统单一培养条件下不表达的代谢途径,称为“沉默代谢途径”。沉默代谢途径具有合成新型次级代谢产物的潜力,且远远超过已发现的代谢产物数量。在后基因组时代的今天,如何激活这些“沉默代谢途径”,产生结构类型更加多样的微生物次级代谢产物,挖掘和开发潜在的药用资源,成为研究的热点[11]。本文系统阐述了未培养微生物的非常规培养与沉默代谢途径激活的方法,总结了利用这些方法获得的新型抗结核天然产物,并结合本实验室的研究进展,展望了这些方法在抗结核药物研究中的应用前景。
1 微生物来源天然产物的研究方法
通过改变培养基成分、培养条件和添加酶抑制剂等干扰因素,对未培养微生物进行再培养,或激活某些微生物的合成基因簇,挖掘其产生特殊次级代谢产物的能力,可以获得大量骨架新颖、活性高的药物前导化合物[9-12]。在抗结核药物研究中,利用这些方法在土壤和海洋微生物中已经获得了丰富多样的抗结核活性天然产物 (图1)。下述为近年来常用的微生物非常规培养方法及沉默代谢途径的激活策略。
1.1 未培养微生物的非常规培养
使用传统微生物学培养技术对不同生境微生物可培养性的测定来看,海水中微生物可培养的约为0.001%–0.1%,淡水约为0.25%,土壤约为0.3%,活性污泥为1%–15%左右。由于未培养微生物无论是其物种类群还是新陈代谢途径、生理生化反应与产物等,都存在着丰富的新颖性和多样性,因而比以往的可培养微生物具有更为丰富和多样化的、可供人类开发利用的生物资源[13]。如利用原位培养方法,在微生物肯塔基伦茨氏菌中得到的化合物Lassomycin,其对结核菌具有特异的杀菌活性,并具有特殊的活性机制与杀菌效果。继续探索未培养微生物的再培养,对抗结核药物研发具有重要意义。增强微生物的可培养性的方法包括以下几个方面:
(1) 限制性培养策略:传统培养基的营养比较丰富,培养条件相对温和,所以适合优势菌种较快生长。但地球上也同时分布着大量适应贫瘠营养环境的微生物,较丰富的营养基质特别是高浓度的盐及有机物会造成它们复苏障碍[14]。所以需要按培养目的进行条件限制性培养(如厌氧、高温、高盐、寡营养、特殊的培养底物等),才能分离出未培养微生物[15-16]。
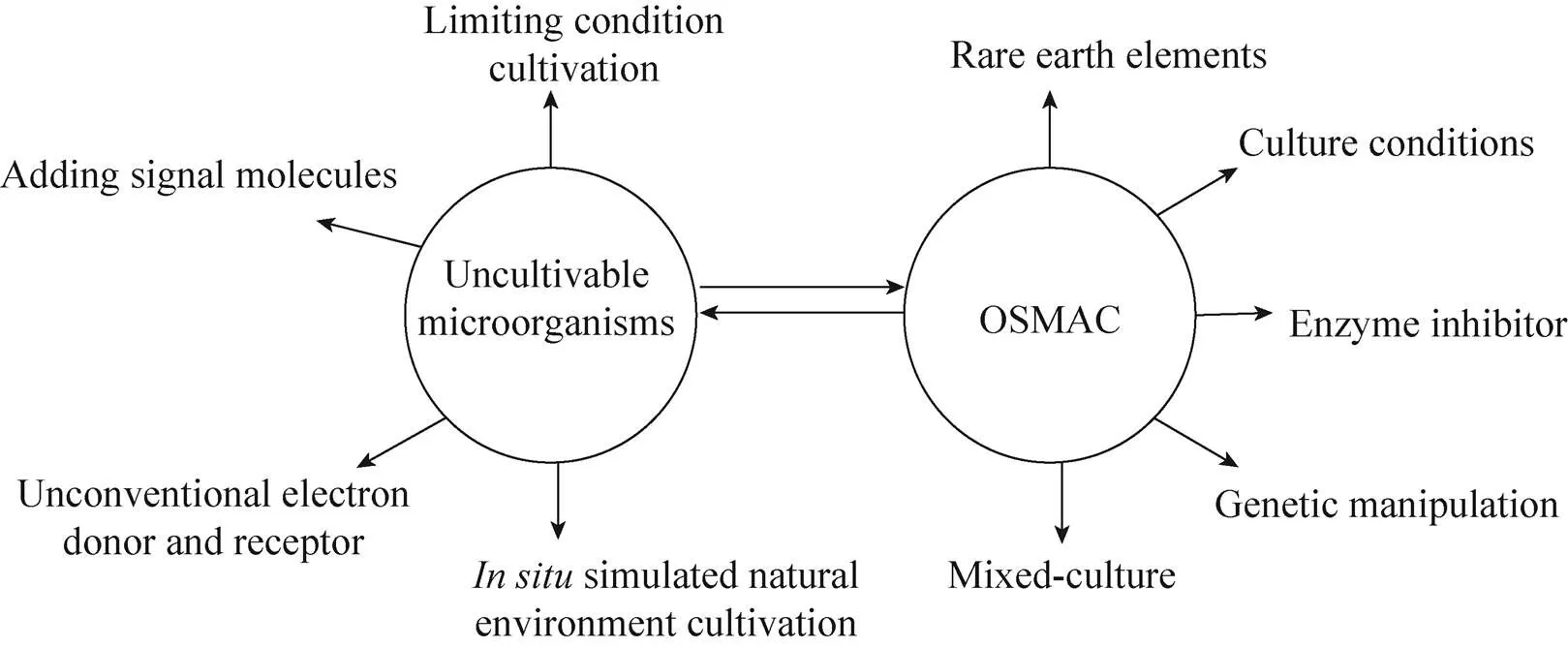
图1 微生物来源天然产物的研究方法 (其中未培养微生物的方法与OSMAC的策略在某些研究中是可以通用的)
(2) 添加信号分子:在培养基中加入微生物相互作用的信号分子,可简单模拟微生物间的相互作用,进而满足微生物生长繁殖的要求。经常向培养基中加入酰基高丝氨酸内酯、cAMP (Cyclic adenosine monophosphate,cAMP) 或ATP (Adenosine triphosphate,ATP) 等微生物之间沟通的信号分子,能促使未培养微生物得到培养。其中与革兰氏阴性菌多种基因调控有关的cAMP是最有效的信号分子,微量的cAMP可使10%的微生物细胞培养出来。
(3) 原位仿生境培养策略:将微生物分散附着或包埋在特殊载体上,固定微生物细胞,但其代谢产物及信号分子可自由扩散并相互利用[17-18]。该方法能最大限度保持微生物的群落特性及细胞之间的相互联系,可以将大量微生物转变为可培养微生物。该方法的优点是既能纯培养又能保留微生物的原生态特性,弥补了传统纯培养的部分弱点。
(4) 采用非常规的电子供体与电子受体:在分离化能自养菌时,添加非常规的电子供体与受体,可以快速达到分离的目的。如以亚砷酸为电子供体,氧为受体,以CO2为唯一碳源,从废弃的金属矿中分离到化能自养菌 (NT-26),后者能产生很多活性天然产物。另外,为了降低培养过程中优势菌种代谢所产生的过氧化物、自由基和一些拮抗物质的毒害作用,在培养基中添加如丙酮酸钠、甜菜碱、超氧化物歧化酶和过氧化氢酶等,可以降解这些毒性成分。超氧化物歧化酶和丙酮酸钠的代谢产物NADH、H+可与超氧化物结合,从而降低了超氧化物对细胞的损伤。
1.2 沉默代谢途径的激活
单株菌-多次级代谢产物 (One strain-many compounds,OSMAC) 策略强调通过改变培养基成分、培养条件和添加酶抑制剂等干扰因素来刺激微生物,激活不同的合成基因簇,挖掘其产生不同次级代谢产物的能力,从而获得骨架新颖、活性高的新化合物[19]。在抗结核药物研发中,OSAMC策略具有独特的优势,可特异性对抗结核化合物进行筛选。如利用OSAMC策略,在海洋细菌链霉菌属sp.的发酵液中获得多肽Cyclomarin A,其对MDR-TB与XDR-TB都有抗菌活性,且其作用靶标为结核菌内的ClPC1蛋白。下文将介绍常用的OSMAC策略。
1.2.1 稀土元素处理
在化工业稀土元素的重要性是众所周知的,其对活细胞的生物作用则鲜为人知。稀土元素包括钪、钇、镧系元素在内共17种元素,它们通过降解pppGpp (或ppGpp) 的方法,能够提高抗生素的产量,并在激活细菌的沉默基因和低表达基因方面有重要作用[20]。pppGpp与ppGpp都是重要的转录调控因子,可参与微生物的严谨反应,其可与RNA聚合酶结合而直接影响RNA聚合酶与DNA形成开放复合体的稳定性,从而抑制rRNA启动子的转录。在温和条件下,稀土金属离子对pppGppp及ppGpp有较好的水解作用,进而可以提高微生物基因表达,并最终会导致某些抗生素合成的增加。
1.2.2 改变微生物生长条件
不同的C/N比对菌体的生长繁殖以及次级代谢产物有很大的影响。碳源过多容易产生较低的pH环境,进而影响微生物生长;碳源不足容易引起微生物自身的衰老和自溶。氮源过多会使微生物生长过旺,影响环境pH偏高,不利次级代谢产物的积累;氮源不足则影响菌体繁殖量,进而影响次级代谢产物的产量。盐浓度也能影响微生物的次级代谢产物。盐浓度的高低直接影响体系渗透压的大小,而适当的渗透压是微生物正常生长的保障。培养体系pH的变化影响微生物的代谢过程,导致次级代谢产物的差异性。体系pH值发生变化不仅对各种酶产生影响,还会影响膜表面电荷的性质及膜的通透性,改变菌体对基质的利用速率,从而影响菌的生长和产物的合成。利用酸胁迫 (pH≤5) 和碱胁迫 (pH≥10),可以激活微生物在极端条件下才进行表达的基因,进而产生一些新的天然化合物。如从陆生及海洋耐酸真菌的酸性培养产物中获得了具有抗甲型流感病毒、抗菌及细胞毒活性的聚酮和吲哚类生物碱[21]。
1.2.3 酶抑制剂
通过酶抑制剂的添加,选择性地抑制代谢途径中相关酶的活性,在抑制某些代谢途径的同时,促进其他通路相关代谢产物的合成。使用靶向核糖体的链霉素与庆大霉素,及靶向RNA聚合酶的抗生素利福平与ppGpp,可以选择性地使得核糖体蛋白S12及RNA聚合酶β亚单元进行自发突变,进而导致激活某些沉默的合成基因。在微生物培养基中添加组氨酸去乙酰化酶抑制剂辛二酰苯胺氧肟酸、丁酸钠与丙戊酸,及DNA甲基转移酶抑制剂5-氮胞苷,可以对DNA进行共价修饰并调控真核基因表达,进而改变全基因组水平的基因表达,并激活沉默的化合物合成基因。添加亚MIC (Minimum inhibitory concentration)浓度的抗生素甲氧苄啶,通过诱导沉默基因的表达,也可以诱导合成新的化合物。
1.2.4 混合培养
微生物的混合培养能够发生协同或者拮抗作用,进而使微生物产生结构新颖、高活性的次级代谢产物,这是近年来OSMAC策略在微生物次级代谢产物研究方面的新思路[22]。天然来源的抗生素多是由于微生物生存环境受到另一物种威胁而产生的竞争化学武器,探索研究这一防御机制是寻找新抗生素资源非常有效的途径。近年来,通过混合培养刺激菌株产生相应的新代谢产物成为OSMAC策略中新的研究思路。
1.2.5 遗传方法
在沉默代谢途径的基因簇中,不仅包括合成基因,还包括一些必需的调控基因。这些调控基因的过量表达或缺失可以显著影响次级代谢产物的生物合成。因此,增加正调控基因的表达或阻断负调控基因的表达都可以有效地激活沉默基因簇表达,获得新型次级代谢产物。如有研究者利用CRISPR-Cas9技术激活链霉菌中沉默的基因簇,并揭示出沉默基因中隐藏着潜在的化合物库[23]。研究人员首先利用计算工具鉴定出沉默的生物合成基因簇,随后利用CRISPR/Cas9技术将一种强启动子序列插入到想要激活的基因前面,促使细菌制造这些基因簇编码的天然产物。利用此种方法,他们分离出一种具有新骨架的Ⅱ型聚酮类化合物并且确定了其分子式为C23H16O8,其具有的非对称环己酮结构在其他聚酮类化合物中还未曾发现。
2 抗结核活性天然产物的分离及其作用靶标
天然产物的研究已进入了一个新的时代,微生物来源的天然产物已成为当前抗结核药研发的热点领域之一[24]。基于上述的研究方法,近年来在新型抗结核药物筛选的研究中取得了很多令人欣喜的成果。如利用原位培养与iCHIP等方法,研究者获得了几个具有独特作用靶标及作用机制的天然产物,这些天然产物具有较强的抗耐药结核菌活性,为新型抗结核药物的筛选提供了模板和思路,我们着重介绍其中的Lassomycin、Ecumicin及泰斯巴汀(Teixobactin) (表1)。
Gavrish等[25]通过原位培养并延长培养时间的方法,从土壤微生物的发酵液中分离出化合物Lassomycin,其显示出较高的抗结核活性,对结核菌包括MDR与XDR菌株的MIC值是0.8–3.0 μg/mL,MBC值为1–4 μg/mL。通过检测抗菌谱发现,Lassomycin可选择性地杀死结核菌,且对其他微生物无杀菌活性。进一步研究发现,Lassomycin在结核菌内的作用靶标是ClpC1P1P2,后者是结核菌生长所必需的ATP依赖的蛋白酶,而且其他微生物中没有编码该蛋白的基因,其只存在于分枝杆菌属的微生物中。Lassomycin可以结合到ClpC1P1P2的N末端口袋状结构中,并激活它的ATP酶活性,进而导致ClpC1P1P2 处于活性蛋白酶状态并开始降解结核菌细胞内的关键蛋白。这种通过激活胞内蛋白酶 (而不是抑制) 而起到抑制病原菌的机制比较少见,预示Lassomycin很有希望作为抗结核药物或药物佐剂进入临床应用,同时也为新抗结核药物的设计提供了思路[26-27]。

表1 抗结核天然产物来源、活性及作用靶标
通过高通量筛选的方法,研究人员从65 000种放线菌的萃取物中发现野野村氏菌sp. MJM5123能产生一种新的抗生素Ecumicin[28],一个环形的13肽结构。Ecumicin对结核菌的最小杀菌浓度为0.34 μmol/L。其对MDR、XDR及潜伏感染的结核菌均有杀灭活性,对其他病原微生物如大肠杆菌、金黄色葡萄球菌、链球菌等没有抑制活性,表明Ecumicin对结核菌也有特异的杀菌活性。与Lassomycin类似,Ecumicin也能解偶联结核菌中ClpC1 ATPase复合物,进而激活蛋白酶ClpP1P2的水解活性,造成结核菌由于蛋白的水解而被杀灭[29]。结核分枝杆菌通过Clp蛋白酶复合体可水解在环境压力下形成的错误折叠、不可逆损伤以及对自身有害的蛋白质,从而保证结核分枝杆菌的稳态,维持其在感染机体后的正常的生存和繁殖[30]。另一个ClPC1抑制剂是Cyclomarin A,其是由链酶菌sp.产生的多肽,共包含7个氨基酸残基,其中3个为稀有氨基酸[31]。Cyclomarin A最早是在1999年分离得到,最初被确认具有抗炎活性,但研究人员证明Cyclomarin A对MDR-TB与XDR-TB都有抗菌活性,其作用靶标为结核菌内的ClPC1蛋白[32]。通过与ClpC1结合导致后者结构改变,并激活其蛋白酶活性,最后导致结核菌内正常蛋白被降解,结核菌由于蛋白稳定被破坏而死亡[33]。上述研究表明抗结核先导化合物的新型作用模式,阐释了ClpC1作为结核菌的治疗靶标的美好研究前景[32]。后续研究人员又对该代谢产物进行了一系列修饰,得到了Cyclomarin B、Cyclomarin C以及最近报道的Desoxycyclomarin C,药物评价结果显示了该类化合物巨大的抗肺结核药物开发潜力[34]。
美国东北大学Kim Lewis研究小组通过对未培养微生物进行原位培养,利用Ichip (Isolation chip,Ichip) 技术从未培养微生物β-变形杆菌的发酵液中分离到一种新型抗生素Teixobactin[31]。与糖肽类抗生素、羊毛硫抗生素及防御素的分子结构不同,Teixobactin的分子结构属于缩酚酸肽,是第一个可以结合lipid Ⅱ的抗生素。利用Teixobactin对的突变库进行筛选,在低倍MIC浓度下依然没有筛选出抗性菌株,表明Teixobactin的作用靶标是结核菌中的必需基因所编码,从而能够避免的抗性产生,因此有较好的应用前景。Teixobactin的作用机制是结合肽聚糖前体lipid Ⅱ和lipid Ⅲ,进而抑制细胞壁肽聚糖的合成并达到抑制微生物的生长的作用。Teixobactin主要通过结合lipid Ⅱ的焦磷酸酯和多糖,从而阻断细菌细胞壁的合成路径。Lipid Ⅱ是一个非蛋白靶点,细菌不易通过突变产生耐药,是目前发现的有效的抗菌靶点[35];另外,lipid Ⅱ是细菌特有的成分,可大大降低lipid II抑制剂对哺乳动物细胞产生毒性的可能性。此外,Teixobactin也作用于lipid Ⅲ,后者是细胞壁磷壁酸的合成前体,当Teixobactin与lipid Ⅲ结合时,可导致自溶素被释放,将肽聚糖层溶解,最终导致细菌死亡。除抑制结核菌以外[36],Teixobactin对其他革兰氏阳性菌也有很好的抗菌活性,如对炭疽杆菌与艰难梭菌的最低抑菌浓度值达到纳克级;另外其对多种耐药菌如耐甲氧西林金黄色葡萄球菌(Methicillin-resistant,MRSA) 和耐万古霉素金黄色葡萄球菌 (Vancomycin resistant,VRE) 等有效,MIC均在1 μg/mL以下[37]。
3 展望
耐药结核菌感染已成为全人类的威胁,除了限制抗生素的滥用外,新型抗生素的研发也在紧锣密鼓地展开。微生物品种繁多、代谢途径多样、遗传可塑性强,是研制新药取之不尽的宝库。挖掘这些微生物资源,需要利用微生物非常规培养与沉默基因激活策略。如利用原位培养、营养限制培养及Ichip技术,从微生物代谢产物中获得的具有抗结核菌活性的天然产物Lassomycin、Ecumicin 和Teixobactin,它们以Clp蛋白复合体或lipid Ⅱ为作用靶点,具有较好的应用前景。我们认为,综合利用上述方法继续在微生物中寻找结构新颖的天然产物,并进一步检测这些化合物的抗结核活性,是抗结核药物研究的一个重要方向。本实验室利用营养限制培养策略从微生物分散泛菌中分离到抗结核活性的化合物PantocinWH-1。PantocinWH-1为多肽结构,分子量为1 927 Da,且对结核菌具有特异的杀灭活性,体内试验也显示出优异的应用前景。
此外,一些新的技术也可以用作未培养微生物与OSMAC策略的探索,如宏基因组学(Metagenomics与稳定同位素(Stable isotope probing,SIP)[47]。宏基因组学是直接提取特定环境中的总DNA,并克隆到可培养的宿主细胞中以构建宏基因组文库,从获得的克隆当中进行功能筛选和序列筛选分析,文库包含了可培养的和未培养的微生物基因,避开了微生物分离培养等问题,极大地扩展了微生物资源的利用空间[48]。宏基因组学是研究未培养微生物、寻找新功能基因和开发获得新活性物质的重要新途径,微生物及其天然产物的探索领域正在宏基因组的帮助下不断扩大[49]。如Gillespie等[50]利用细菌人工染色体载体pBeloBACII构建了两个土壤样品的宏基因组文库,获得了两个具有广谱抗菌作用的新抗生素Turbomycin A和Turbomycin B。可以预期其对抗结核药物研发也具有较好的应用前景。SIP 作为示踪物,主要是标记营养底物来培养微生物,如利用同位素15N或15N-13C标记的氨基酸作为营养底物去培养微生物,进而通过NMR检测使用这些氨基酸合成的新的天然产物,目前多篇最新文献报道了该方法的成功应用[51-52]。鞠建华等从深海放线菌中发现了具有抗结核菌活性的系列物质,通过SIP等技术优化改造获得低细胞毒活性、强抗结核菌活性的化合物——怡莱霉素E (IlamycinE)[46]。怡莱霉素E对结核菌H37Rv菌株的MIC值为9.8 nmol/L,是一线抗结核药物利福平活性的30倍。怡莱霉素E对正常细胞的毒性较低,在抗结核活性和细胞毒性之间的选择性指数为400–1 500,显示出较好的安全性窗口,具有成药潜力。
综上,微生物非常规培养与沉默基因激活策略是发现抗结核活性产物的重要方法。利用上述方法,研究人员已经得到了数个具有应用前景的天然产物,这些天然产物具有成为新型抗结核药物的潜力。另外,随着新的生物新技术方法的应用 (如宏基因组学及SIP技术),微生物来源的天然产物筛选模式也发生了进一步的变化。通过多种生物技术的交叉使用,微生物天然产物开发有望再次成为新药开发的高产资源,为人类解除疾病和增进健康提供更多更好的选择。
[1] Osborne R. First novel anti-tuberculosis drug in 40 years. Nat Biotechnol, 2013, 31(2): 89–90.
[2] O’Neill MB, Mortimer TD, Pepperell CS. Diversity ofacross evolutionary scales. PLoS Pathog, 2015, 11(11): e1005257.
[3] Gunther G. Multidrug-resistant and extensively drug-resistant tuberculosis: a review of current concepts and future challenges. Clin Med (Lond), 2014, 14(3): 279–285.
[4] Parida SK, Axelsson-Robertson R, Rao MV, et al. Totally drug-resistant tuberculosis and adjunct therapies. J Intern Med, 2015, 277(4): 388–405.
[5] Caminero JA, Piubello A, Scardigli A, et al. Bedaquiline: how better to use it. Eur Respir J, 2017, 50(5): 1701670.
[6] Tandon R, Nath M. Tackling drug-resistant tuberculosis: current trends and approaches. Mini Rev Med Chem, 2017, 17(6): 549–570.
[7] Lehar SM, Pillow T, Xu M, et al. Novel antibody-antibiotic conjugate eliminates intracellular. Nature, 2015, 527(7578): 323–328.
[8] Bradley P, Gordon CN, Walker TM, et al. Rapid antibiotic-resistance predictions from genome sequence data forand. Nat Commun, 2015, 6: 10063.
[9] Ortega MA, van der Donk WA. New insights into the biosynthetic logic of ribosomally synthesized and post-translationally modified peptide natural products. Cell Chem Biol, 2016, 23(1): 31–44.
[10] Wilson MC, Mori T, Rückert C, et al. An environmental bacterial taxon with a large and distinct metabolic repertoire. Nature, 2014, 506(7486): 58–62.
[11] Rutledge PJ, Challis GL. Discovery of microbial natural products by activation of silent biosynthetic gene clusters. Nat Rev Microbiol, 2015, 13(8): 509–523.
[12] Clardy J, Fischbach MA, Walsh CT. New antibiotics from bacterial natural products. Nat Biotechnol, 2006, 24(12): 1541–1550.
[13] Kaeberlein T,Lewis K, Epstein SS. Isolating “Uncultivable” microorganisms in pure culture in a simulated natural environment. Science, 2002, 296(5570): 1127–1129.
[14] Buerger S, Spoering A, Gavrish E, et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol, 2012, 78(9): 3229–3233.
[15] Nyonyo T, Shinkai T, Tajima A, et al. Effect of media composition, including gelling agents, on isolation of previously uncultured rumen bacteria. Lett Appl Microbiol, 2013, 56(1): 63–70.
[16] Wrighton KC, Thomas BC, Sharon I, et al. Fermentation, hydrogen, and sulfur metabolism in multiple uncultivated bacterial phyla. Science, 2012, 337(6102): 1661–1665.
[17] Ferrari BC, Binnerup SJ, Gillings M. Microcolony cultivation on a soil substrate membrane system selects for previously uncultured soil bacteria. Appl Environ Microbiol, 2005, 71(12): 8714–8720.
[18] Ben-Dov E, Kramarsky-Winter E, Kushmaro A. Anmethod for cultivating microorganisms using a double encapsulation technique. FEMS Microbiol Ecol, 2009, 68(3): 363–371.
[19] Parish T. Targeting mycobacterial proteolytic complexes with natural products. Chem Biol, 2014, 21(4): 437–438.
[20] Kawai K, Wang GJ, Okamoto S, et al. The rare earth, scandium, causes antibiotic overproduction inspp. FEMS Microbiol Lett, 2007, 274(2): 311–315.
[21] Frisvad JC. Media and growth conditions for induction of secondary metabolite production//Keller N, Turner G, eds. Fungal Secondary Metabolism. Totowa, NJ: Humana Press, 2012, 944: 47–58.
[22] Zarins-Tutt JS, Barberi TT, Gao H, et al. Prospecting for new bacterial metabolites: a glossary of approaches for inducing, activating and upregulating the biosynthesis of bacterial cryptic or silent natural products. Nat Prod Rep, 2015, 33(1): 54–72.
[23] Zhang MM, Wong FT, Wang YJ, et al. CRISPR-Cas9 strategy for activation of silentbiosynthetic gene clusters. Nat Chem Biol, 2017, doi: 10.1038/nchembio.2341.
[24] Hamamoto H, Urai M, Ishii K, et al. Lysocin E is a new antibiotic that targets menaquinone in the bacterial membrane. Nat Chem Biol, 2015, 11(2): 127–133.
[25] Gavrish E, Sit CS, Cao SG, et al. Lassomycin, a ribosomally synthesized cyclic peptide, killsby targeting the ATP-dependent protease ClpC1P1P2. Chem Biol, 2014, 21(4): 509–518.
[26] Al Toma RS, Kuthning A, Exner MP, et al. Site-directed and global incorporation of orthogonal and isostructural noncanonical amino acids into the ribosomal lasso peptide Capistruin. ChemBioChem, 2015, 16(3): 503–509.
[27] Hegemann JD, Zimmermann M, Xie XL, et al. Lasso peptides: an intriguing class of bacterial natural products. Acc Chem Res, 2015, 48(7): 1909–1919.
[28] Gao W, Kim JY, Anderson JR, et al. The Cyclic Peptide Ecumicin targeting ClpC1 is active against. Antimicrob Agents Chemoth, 2015, 59(2): 880–889.
[29] Leodolter J, Warweg J, Weber-Ban E, et al. TheClpP1P2 protease interacts asymmetrically with its ATPase partners ClpX and ClpC1. PLoS ONE, 2015, 10(5): e0125345.
[30] McGillivray A, Golden NA, Kaushal D. TheClp gene regulator is required forreactivation from hypoxia-induced dormancy. J Biol Chem, 2015, 290(4): 2351–2367.
[31] Bürstner N, Roggo S, Ostermann N, et al. Gift from nature: cyclomarin a killsandparasites by distinct modes of action. ChemBioChem, 2015, 16(17): 2433–2436.
[32] Schmitt EK, Riwanto M, Sambandamurthy V, et al. The natural product cyclomarin killsby targeting the ClpC1 subunit of the caseinolytic protease. Angew Chem Int Ed, 2011, 50(26): 5889–5891.
[33] Olivares AO, Baker TA, Sauer RT. Mechanistic insights into bacterial AAA+ proteases and protein-remodelling machines. Nat Rev Microbiol, 2016, 14(1): 33–44.
[34] Barbie P, Kazmaier U. Total synthesis of desoxycyclomarin C and the cyclomarazines A and B. Org Biomol Chem, 2016, 14(25): 6055–6064.
[35] Ng V, Chan WC. New found hope for antibiotic discovery: lipid II inhibitors. Chemistry, 2016, 22(36): 12606–12616.
[36] Rawal T, Butani S. Combating tuberculosis infection: a forbidding challenge. Indian J Pharm Sci, 2016, 78(1): 8–16.
[37] Von Nussbaum F, Sussmuth RD. Multiple attack on bacteria by the new antibiotic teixobactin. Angew Chem Int Ed, 2015, 54(23): 6684–6686.
[38] Jung IP, Ha NR, Kim AR, et al. Mutation analysis of the interactions betweencaseinolytic protease C1 (ClpC1) and ecumicin. Int J Biol Macromol, 2017, 101: 348–357.
[39] Sass P, Josten M, Famulla K, et al. Antibiotic acyldepsipeptides activate ClpP peptidase to degrade the cell division protein FtsZ. Proc Natl Acad Sci USA, 2011, 108(42): 17474–17479.
[40] Ollinger J, O’Malley T, Kesicki EA, et al. Validation of the essential ClpP protease inas a novel drug target. J Bacteriol, 2012, 194(3): 663–668.
[41] Hartkoorn RC, Sala C, Neres J, et al. Towards a new tuberculosis drug: pyridomycin-nature’s isoniazid. EMBO Mol Med, 2012, 4(10): 1032–1042.
[42] Kremer L, Douglas JD, Baulard AR, et al. Thiolactomycin and related analogues as novel anti-mycobacterial agents targeting KasA and KasB condensing enzymes in. J Biol Chem, 2000, 275(22): 16857–16864.
[43] Lee H, Suh JW. Anti-tuberculosis lead molecules from natural products targetingClpC1. J Ind Microbiol Biot, 2016, 43(2/3): 205–212.
[44] Vasudevan D, Rao SPS, Noble CG. Structural basis of mycobacterial inhibition by Cyclomarin A. J Biol Chem, 2013, 288(43): 30883–30891.
[45] Ling LL, Schneider T, Peoples AJ, et al. A new antibiotic kills pathogens without detectable resistance. Nature, 2015, 520(7547): 455–459.
[46] Ma JY, Huang HB, Xie YC, et al. Biosynthesis of ilamycins featuring unusual building blocks and engineered production of enhanced anti-tuberculosis agents. Nat Commun, 2017, 8(1): 391.
[47] Milshteyn A, Schneider JS, Brady SF. Mining the metabiome: identifying novel natural products from microbial communities. Chem Biol, 2014, 21(9): 1211–1223.
[48] Chung EJ, Lim HK, Kim JC, et al. Forest soil metagenome gene cluster involved in antifungal activity expression in. Appl Environ Microbiol, 2008, 74(3): 723–730.
[49] Frias-Lopez J, Shi YM, Tyson GW, et al. Microbial community gene expression in ocean surface waters. Proc Natl Acad Sci USA, 2008, 105(10): 3805–3810.
[50] Kealey C, Creaven CA, Murphy CD, et al. New approaches to antibiotic discovery. Biotechnol Lett, 2017, 39(6): 805–817.
[51] Hungate BA, Mau RL, Schwartz E, et al. Quantitative microbial ecology through stable isotope probing. Appl Environ Microbiol, 2015, 81(21): 7570–7581.
[52] Mazard S, Schäfer H. Stable isotope probing to study functional components of complex microbial ecosystems//Paulsen I, Holmes A, Eds. Environmental Microbiology. Totowa, NJ: Humana Press, 2014, 1096: 169–180.
(本文责编 郝丽芳)
Development of new anti-tuberculosis drugs: the strategy of unconventional microbial culture and silencing gene activation
Tieshan Teng1,2, Longxiang Xie1,2, and Jianping Xie2
1 Institute of Biomedical Informatics, School of Basic Medical Sciences, Henan University, Kaifeng 475004, Henan, China 2 State Key Laboratory Breeding Base of Eco-Environment and Bio-Resource of the Three Gorges Area, Key Laboratory of Eco-environments in Three Gorges Reservoir Region, Ministry of Education, Institute of Modern Biopharmaceuticals, School of Life Sciences, Southwest University, Chongqing 400715, China
Tuberculosis (TB), caused by, has become a major human infectious disease. The existing first-line and second-line TB drugs have poor treatment outcomes in patients with MDR-TB and XDR-TB. There is an urgent need for new and better drugs to treat tuberculosis due to lengthy and complex treatment regimens and a rising problem of drug resistance. Microbial-derived natural products have revealed enormous reservoirs of as yet untapped lead compounds. In this review, we discuss the strategies that have been developed in bacteria and fungi to isolation of non-culturable microorganisms and activation of silent biosynthetic gene clusters involved in the study of microbial-derived natural products. This review also highlights recent advances in microbial-derived natural products with anti-tuberculosis activity using these methods.
, un-cultured microorganisms, silent genes, one strain-many compounds (OSMAC)
December 30, 2017;
March 22, 2018
Natural Science Foundation of Education Department of Henan Province (No. 17A310015).
Jianping Xie. Tel: +86-23-68367108; E-mail: georgex@swu.edu.cn
河南省高等学校重点科研项目 (No. 17A310015) 资助。
10.13345/j.cjb.170534

