细菌耐药性应对策略研究进展
尹业师,陈华海,曹林艳,唐林,何福林
尹业师 博士,教授,湖南科技学院生物工程学科带头人。从事肠道微生物与宿主相互作用研究。主持国家自然科学基金和国家863计划子课题等;以主要作者在、、、、等期刊发表论文60余篇;以第一发明人申报中国发明专利6项。中华医学会消化病学分会微生态组委员、中国微生物学会永久会员、中国生物工程学会终身会员和美国微生物学会会员。、、、、《生物工程学报》和《微生物学通报》等期刊的审稿专家。

细菌耐药性应对策略研究进展
尹业师,陈华海,曹林艳,唐林,何福林
湖南科技学院 化学与生物工程学院 湘南优势植物资源综合利用湖南省重点实验室,湖南 永州 425199

尹业师, 陈华海, 曹林艳, 等. 细菌耐药性应对策略研究进展. 生物工程学报, 2018, 34(8): 1346–1360.Yin YS, Chen HH, Cao LY, et al. Progress in strategies to combat antimicrobial resistance. Chin J Biotech, 2018, 34(8): 1346–1360.
细菌耐药性 (Antimicrobial resistance,AMR) 持续增长,但新上市抗生素数量却持续下降。抗生素耐药基因 (Antimicrobial resistance gene,ARG) 和抗生素耐药菌感染已严重威胁人类健康。因此,需要多方面联合采取措施来应对AMR所带来的各种挑战,包括创新生物医药、改善抗生素使用和抗生素耐药监测系统、减少抗生素耐药基因产生速度、阻止健康护理相关感染和多重抗生素耐药菌传播与扩散、开发微生物学快速诊断方法与设备、减少临床和兽医抗生素滥用等。庆幸的是,AMR已受到各国政要、科学家和企业家等的高度重视与支持,相信随着新技术、新产品的不断问世和管理新措施的不断出台,AMR问题一定会得到控制和缓解。
抗生素,细菌耐药性,抗生素耐药菌,抗生素耐药基因
细菌耐药性 (AMR) 已成为当代医学和制药公司面临的一个严重问题。抗生素耐药细菌 (简称耐药菌) 感染数量的增加已经严重威胁全球公共健康,其发病率和致死率均很高。由耐药菌引起的死亡人数相当于HIV、乳腺癌和前列腺癌致死人数的总和[1-3],这对社会经济学和生态学造成了严重影响。美国每年大约有200万人的感染和23万人的死亡与耐药菌相关 (http://1.usa.gov/1nDmtkJ)。更严重的是,全球每年有约70万人的死亡是耐药菌感染导致的。由于亚洲和非洲抗生素使用量仍然很大,AMR形势将不断恶化,专家们预测,到2050年每年将有1 000万人因此而死亡,其经济损失将超过数万亿美元[4]。
随着临床和非临床抗生素的随意和大量使用,AMR新时代已经到来[5],但药物研发速度远跟不上细菌耐药性产生的速度。1983−1987年期间,美国FDA批准了16种新的抗生素,但这个数字在显著下降,2008−2012年期间总共只批准了2种新抗生素[6],而且已经有超过45年没有批准任何用于治疗革兰氏阴性菌感染的新类别抗生素[7]。因此,应对AMR所带来的严重威胁和挑战,需要国际合作与共同努力,包括加大宣传教育力度、加强抗生素使用管理、研发新的抗菌物质和寻找抗生素替代方法等[8]。
1 加强应对AMR管理
临床耐药菌的出现与传播很大程度上与医院和养殖场抗生素销售的管控不严和随意使用有关[9-10]。目前,当一个新抗生素上市,其耐药菌株很快就可以被找到[11]。可见应对AMR已不能单纯通过研发新型抗生素来解决,必须采取综合治理措施。管控人和动物病原菌AMR需在 One health理念指导下,联合政策制定者、科研工作者、兽医和终端消费者来采取行动,防止耐药性的产生和传播 (图1)。庆幸的是,抗生素的可持续利用已经受到广泛关注。2015年,世界卫生组织 (WHO) 决议WAH68.7强烈要求各成员国制定应对AMR策略[12]。已有超过100个成员国作出了回应并采取了相关措施[12]。近年来,中国作为成员国之一也正在积极采取各项措施应对抗生素耐药[13]。2016年,原国家卫生和计划生育委员会下发了 (《遏制细菌耐药国家行动计划 (2016−2020年)》(http://www.nhfpc.gov.cn/yzygj/ s3593/201608/f1ed26a0c8774e1c8fc89dd481ec84d7.shtml)),提出到2020年实现“争取研发上市全新抗菌药物1−2个,新型诊断仪器设备和试剂5−10项”,“零售药店凭处方销售抗菌药物的比例基本达到全覆盖”等多项行动目标。2017年,原农业部也印发了(《全国遏制动物源细菌耐药行动计划(2017−2020年)》(http://www.moa.gov.cn/govpublic/ SYJ/201706/t20170623_5726086.htm)),提出到2020年,实现“推进兽用抗菌药物规范化使用。省 (区、市) 凭兽医处方销售兽用抗菌药物的比例达到50%”等目标。2016年9月召开的“联合国大会国家元首高级别会议”上,多国科学家联合发文呼吁:提高对AMR的认识;加强对抗生素的监测和评估,制定确实有效的措施促进抗生素合理使用;鼓励国家和社会基金资助应对AMR相关研究;联合多部门开展协调行动,在国家层面协调非政府组织、民间团体和私人机构等,通过各国努力提高抗生素的有效利用,实现WHO全球行动计划[14-15]。
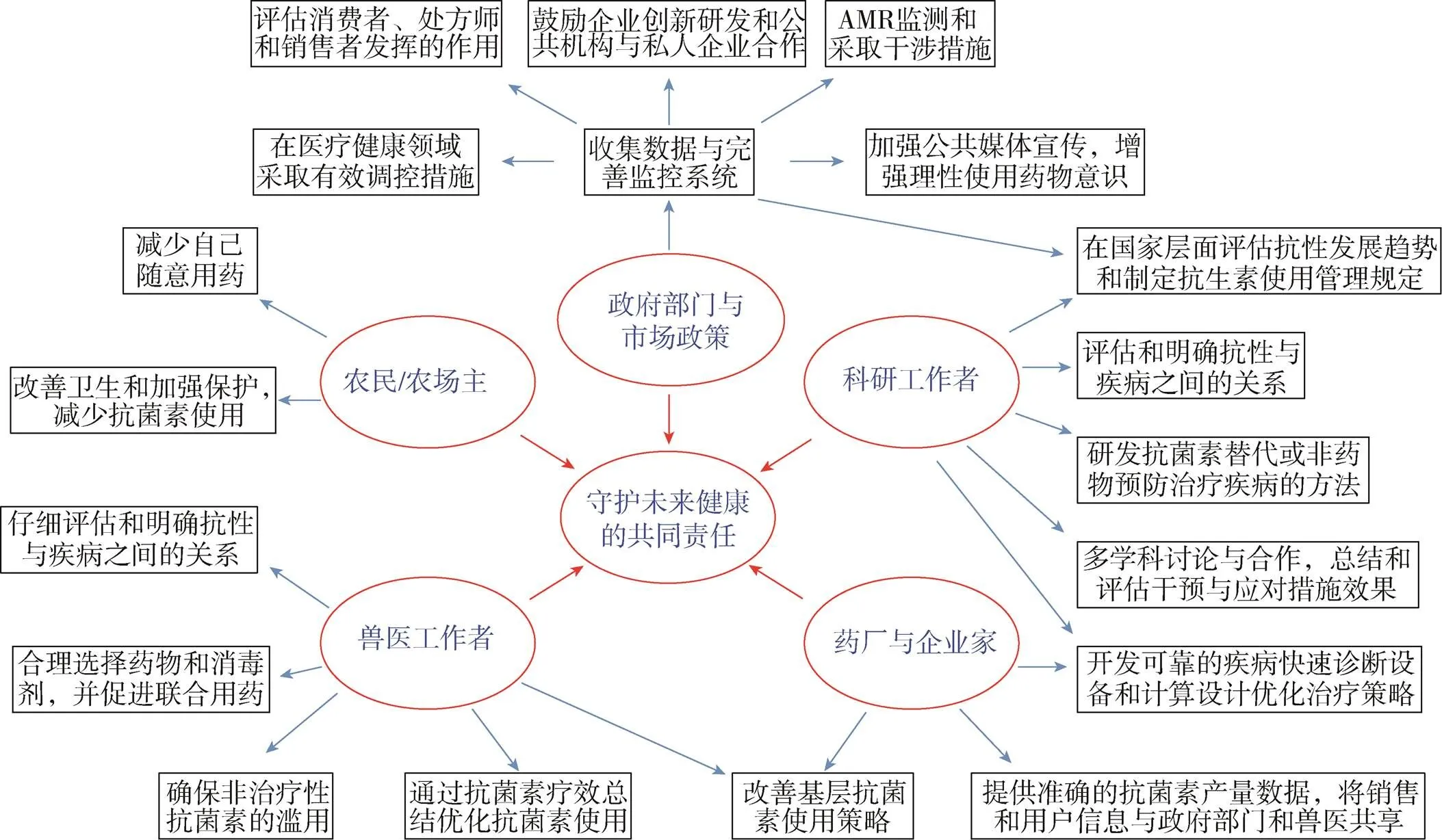
图1 各方协作应对AMR战略合作图(改编自Sharma等的文章[16])
1.1 充分发挥政府的引导和监管功能
政府部门应加强宣传与教育,监控抗生素使用和耐药产生情况,鼓励和投入更多专项经费资助科学家研究AMR产生、传播和进化机制,引导企业研发AMR防治新策略。除了研发抗菌活性更好的新抗生素外,建立完善的抗生素使用和耐药性产生监控体系、及早诊断和预测耐药菌的产生和有针对性地采取应对措施也非常重要。只有建立了强大的监测系统,应用生物信息学和基因组学新进展将全球和当地抗生素在人和动物中的消费情况、耐药机制和细菌表型等整合起来,才能获得更可靠的流行病学数据,才能更好地指导应对AMR危机。如研究发现在4个主要场所AMR进化比较频繁:1) 人和动物微生物组;2) 医院和长期护理单位;3) 污水和其他任何形式的生物残留;4) 土壤及其表面或地下水环境[17]。这将为我们有针对性地防治耐药性指明方向。
1.2 加强临床医生和兽医抗生素使用培训,合理、个性化使用抗生素
WHO将抗生素分为3类——常规抗生素、被监控抗生素和储备抗生素,且对不同类别抗生素的使用作了说明:常规抗生素是可广泛使用、容易获得的抗生素;被监控抗生素是比较容易产生耐药性的抗生素,因此不建议作为大部分感染治疗的首选;储备抗生素耐药性产生较少,被推荐仅用于最后一道防线的治疗[18]。但临床医生在具体操作时会越级或超量使用[15]。由于长期滥用抗生素导致携带多种耐药基因的多种AMR细菌出现[19-21],因此有必要对临床医生加强抗生素使用培训和教育。在中国广西省的2个县、25个乡镇卫生所进行的一项研究中,研究者对基层医务工作者 (医生及护工) 进行了为期9个月的培训,结果发现培训后,抗生素处方率从82%显著下降至40%,而对照组 (未接受培训) 的抗生素处方率未发生显著改变[22]。虽然针对基层医生的培训或可有效减少抗生素滥用,但加强抗生素管理是否能有效阻止AMR还有待进一步研究。因为大部分实验设计都不够完善,不同研究者使用的参数和条件不一致,很难得出证据充分的结论[23]。
加强对兽医工作者的培训和兽用抗生素的管理也非常重要,因为有报道表明抗生素生产总量的近80%被用于动物生产[24]。在动物生产过程中抗生素经常被用于疾病预防和促生长,但动物也存在将耐药菌株传播给人和环境的风险[16]。有研究表明,限制食用动物行业抗生素使用,动物耐药菌减少,人群尤其是直接与食用动物接触的人群耐药菌也有类似减少。但在普通人群中,这种观点还证据不足,有待进一步研究[25]。
1.3 加强水环境综合治理,阻断耐药基因和耐药菌传播
携带耐药基因的耐药菌在环境,尤其是饮用水和污水中的出现将严重影响人类健康。虽然自然选择会引进低水平AMR的出现,但人类活动可导致环境中高水平AMR的传播与流行[26]。工厂、社区、临床医院和农场污水中高浓度耐药基因和耐药菌已严重威胁整个生态圈[27]。在中国17个主要城市的32个污水处理厂采集并分析116个污水样本,发现381个AMR基因在各大城市都普遍存在,并与废水核心菌群和人体肠道菌群显著相关[28]。另外DNA也是污水处理厂中不可忽视的AMR基因来源。在未经处理的废水中,细胞相关耐药基因占主要部分;经过生物处理、污泥沉降、膜过滤、消毒后,细胞相关的耐药基因显著减少,但ARG/16S rRNA的比值在消毒后上升[29]。更让人吃惊的是,在全球25个城市饮用水样本中,共检测到属于16种ARG类型的181种亚型;其中针对杆菌肽、氨基糖苷、磺胺、β-内酰胺等的耐药基因占优势,应警惕ARG在饮用水系统中的潜在水平传播[30]。
2 “老药新用”与新抗菌物质挖掘
2.1 “老药新用”
另外,优化抗生素治疗的药代动力学和药理学可以改善治疗效果、减少毒性和耐药性出现的风险[34]。如有研究表明多粘菌素的副作用并不像以前报道的那样频繁,尤其是在精准靶向用药后[35]。但由于老抗生素很少被纳入监测系统,其相关耐药性产生速度、最小抑菌浓度 (Minimum inhibitory concentration,MIC) 和协同作用等数据比较缺乏[34]。另外,地域性差异导致从局部得出的结果并不一定准确[36],因此需要进一步研究。同时,由于一些老的抗生素 (如磷霉素、甲氧苄啶-磺胺甲氧异恶唑、氯林可霉素、普那霉素、梭链孢酸) 也可以导致耐药基因的水平转移,它们的重新启用需要进行全局的生态学调查。
2.2 联合用药
提高已有抗生素疗效的一个方法是联合用药,联合用药比任何单一药物的效果好很多[37]。实验室研究表明利福平联合达托霉素 (靶向抗万古霉素肠球菌[38])、拉氧头孢联合妥布霉素或者头孢曲松联合妥布霉素 (靶向肠杆菌科[39])、氨基糖苷类联合β-内酰胺类 (靶向革兰氏阴性菌[40])、环丝氨酸联合没食子酸 (靶向耐甲氧西林金黄色葡萄球菌[41]),均对治疗耐药菌感染有较好疗效。另外最近研究报道将传统的头孢类抗生素头孢他啶与β-内酰胺酶抑制剂阿维巴坦联用既保证了抗生素功能,又减少了细菌耐药性[42]。
抗生素与植物提取物联合使用也是较有前景的协同杀菌策略之一[43-45]。多项研究表明,酚类化合物,如玫瑰果中tellimagrandin Ⅰ[46]和熊果中鞣料云实素[47],其生物转化可提高抗生素的抑菌效果。另外,Souto等报道杧果乙醇提取物与四环素和红霉素联合使用可以使其MIC降低4倍[16]。将柚木甲醇提取物与四环素合用,起到协同抗鼠伤寒沙门氏菌和肺炎克雷伯氏菌作用,其MIC浓度分别降低2倍和4倍[16]。
2.3 计算生物学结合合成生物学方法发现新型抗菌物质
细菌、真菌和植物次生代谢产物是主要的抗菌物质来源,当前医药用抗菌物质大约有70%来源于细菌和真菌次级代谢产物。很多次级代谢产物,如聚酮类抗生素、非核糖体多肽、生物碱类化合物、糖类萜类化合物和细菌素等主要由基因簇编码[48],且典型的次生代谢产物产生者其基因组中一般都含有生物合成基因簇(Biosynthetic gene cluster,BGCs)[49]。由于大部分抗生素来源于可培养细菌的天然产物,但细菌可培养率低,大量潜在抗生素难见天日。随着测序技术的发展和宏基因组学研究的不断深入,全球微生物组,包括土壤微生物组和肠道微生物组已被逐渐认为是新药发现的巨大资源宝库[50-51]。Donia等对人类微生物组进行分析,鉴定到了大于14 000个BGCs,并对其中3 000多个进行了比较详细的分析[52],Walsh等从人类微生物组数据库中鉴定到了数十个细菌素基因簇[53]。
令人备受鼓舞的是,有些研究开始使用生物信息学分析结果来指导抗菌物质的发现与合成 (图2)。Hover等根据达托霉素合成基因的保守序列设计引物,构建扩增子文库,依据文库序列预测和查找新型的基因元件,再结合进一步的宏基因组测序和异源表达,从土壤中发现了一种分布广泛、钙离子依赖型、抗MRSA菌的新抗生素,被命名为malacidins;新抗生素在小鼠感染模型中效果极佳,且不产生选择耐药性[54]。Chu等根据分析结果合成了一种新的抗菌物质humimycins,它对临床分离的MRSA具有很好的抑制活性,能提高一些β-内酰胺抗生素的活性,增加感染小鼠的成活率[55]。Vila-Farres等利用生物信息学分析结果指导合成了2个新的抗生素,一个具有抗细菌活性,另一个具有抗真菌活性[56]。
2.4 代谢产物鉴定与化合物筛选方法发现新型抗菌物质
由于环境微生物在实验室条件下比较难实现纯培养,科学家们已经发明了从未培养菌中提取活性物质的方法。Kim研究组通过原位混合菌培养的方法从土壤样品中直接筛选活性分子。通过对原位培养菌抑菌活性代谢产物鉴定,发现了2种新的抗生素Lassomycin和泰斯巴汀 (Teixobactin),这2种新型抗菌物质对MRSA和结核分枝杆菌有较好的抑制活性,且不容易导致耐药性产生[57-58]。
2.选人视野的广阔性。实行竞争性选拔的目的,就是最大限度地“选贤任能”,把各方面优秀人才选拔上来。竞争性选拔干部给所有层面的干部,特别是优秀年轻干部提供了难得的机会和平台。从报名参加竞聘的干部来源上看,所属单位和岗位都各不相同,丰富了源头活水,保证了干部来源的广泛性;从干部所属的层面上看,竞争性选拔特别是公开选拔使各个层面的干部都能参与进来,特别是赋予了广大基层干部以宝贵的机会,从而能够有效地优化干部队伍结构。
化合物文库筛选一直是科学家和制药企业使用的经典方法之一。Kim等在其最新研究中采用秀丽隐杆线虫-MRSA筛选模型,从82 000个合成小分子化合物中筛选发现了185个化合物可明显减少MRSA引起的秀丽隐杆线虫死亡。进一步对其中两个结构相似的合成维甲酸——CD437和CD1530进行研究发现,合成维甲酸通过破坏细菌脂质双层而杀伤生长期和持留MRSA;且在MRSA慢性感染的小鼠模型中,CD437及其类似物均具有良好的疗效[59]。
3 抗菌新策略
尽管抗生素耐药形势仍然严峻,但科学家们已探索研发了很多新的抗菌策略 (图3),以帮助应对可能出现的“无药可用”危机。
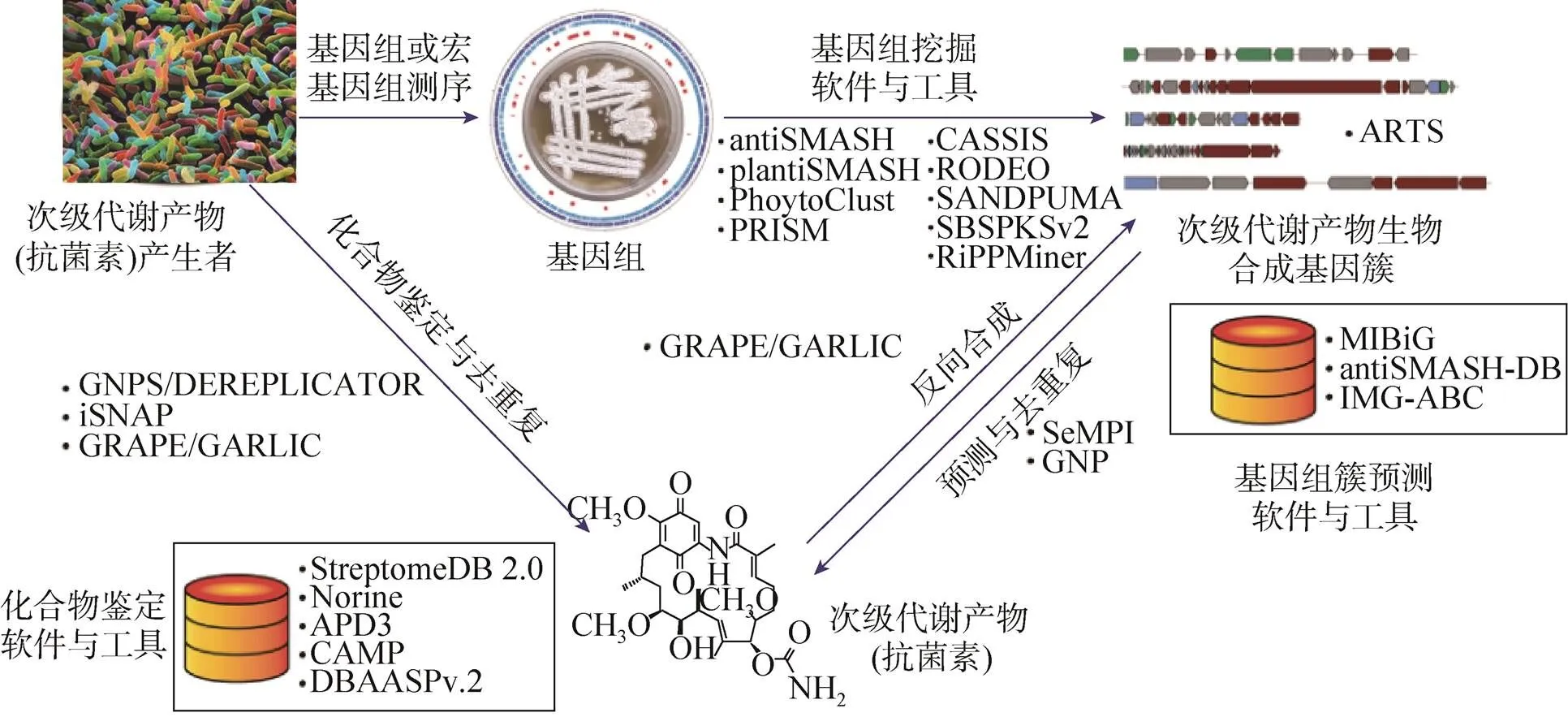
图2 发现新型抗菌物质示意图(改编自Kim等的文章[60])
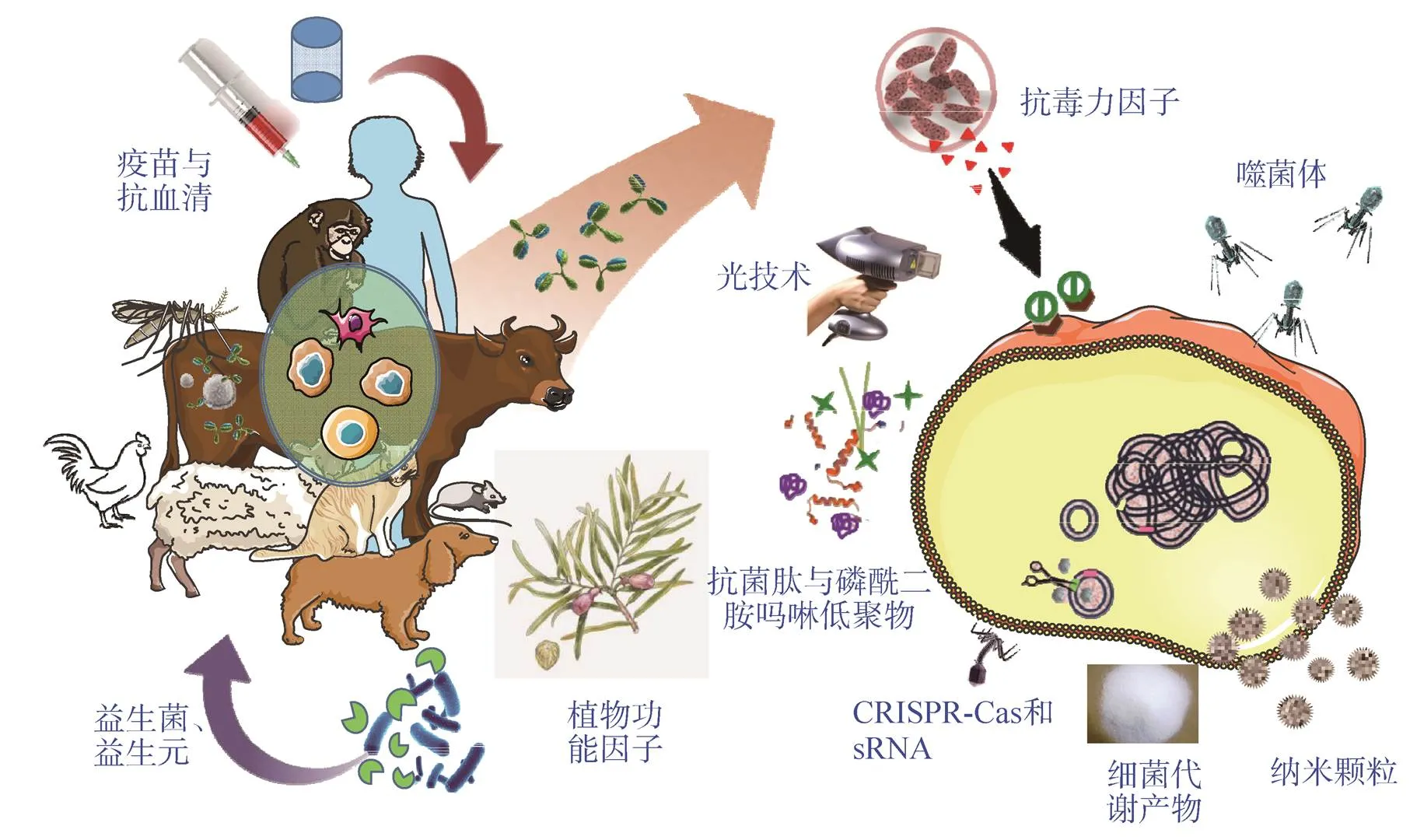
图3 抗菌新策略汇总(改编自Sharma等的文章[16])
3.1 噬菌体疗法
随着多重耐药病原菌的增多,噬菌体疗法越来越受到关注。很多学者使用动物模型对临床相关病原菌,如铜绿假单胞菌、艰难梭菌、耐万古霉素肠球菌、产β内酰胺酶大肠杆菌、鲍氏不动杆菌和金黄色酿脓葡萄球菌进行了研究,结果发现噬菌体对细菌感染或化脓有较好的治疗效果,能明显降低实验动物死亡率[61]。临床实验也表明,噬菌体疗法对耐药铜绿假单胞菌和金黄色酿脓葡萄球菌有较好的抑制作用[62]。最近美国FDA批准的一个紧急案例中,静脉注射噬菌体拯救了一位由多重耐药菌感染而濒临死亡的患者,更是让人对噬菌体疗法充满期待[63]。另外,噬菌体裂解酶、多糖解聚酶等也被报道具有降解细菌荚膜、生物膜和革兰氏阴性菌脂多糖外膜等功能[64-68],具有很好的应用开发前景。
3.2 抗体与疫苗
尽管AMR持续增多,但最近25年来,没有新类抗生素的问世。即使最新的一些鼓励刺激措施可能会促进新抗生素的研发,但也可能很快会出现耐药。淋球菌已经相继对用于治疗的抗生素产生了耐药,已经接近无药可治的地步[69]。疫苗在全球疾病预防中的重要作用已有近2个世纪的历史,应该可以被考虑为防治AMR的重要武器[69]。Laverde等研究发现,TraM免疫或抗TraM抗血清可以靶向4型分泌系统蛋白,对多重耐药性革兰氏阳性致病菌有很好抑制作用,能显著减少小鼠肝脏中肠球菌的菌落数[70]。Sassone-Corsi 等将铁载体蛋白连接到高免疫原性蛋白后免疫小鼠,成功激发了铁载体蛋白特异的IgA,有效降低铁载体蛋白结合铁的效率,进而降低病原体感染[71]。疫苗接种不仅避免了抗生素的初次使用[72],而且减少了细菌继发感染后的二次使用[73]。另外,疫苗的效果可以通过群体免疫扩展至未接种疫苗的人群,从而使细菌耐药性减少[74]。针对肺炎球菌、结核病、伤寒、流感、呼吸道合胞病毒和淋病的研究已表明加速疫苗研发具有明显优势[69,75-76]。
3.3 抗毒力因子治疗
以毒力因子为治疗靶标的精准抗菌治疗已有成功案例。小分子virstatin和toxtazin B是影响霍乱弧菌毒素表达的抗毒力抑制因子,在霍乱弧菌感染动物模型中表现出很好的疗效[77]。抗毒力药物bezlotoxumab是一种抗艰难梭菌毒素TcdB的单克隆抗体,已于2016年被FDA批准用于高复发风险艰难梭菌感染患者的治疗[78]。其他很多抗金黄色葡萄球菌和铜绿假单胞菌的抗毒力因子治疗也在进行临床实验[78]。
3.4 植物功能因子
植物有多种功能特性,主要表现在具有各种生物活性次级代谢产物或植物化合物,如皂苷类、生物碱、花青素、香豆素类、黄酮类、酚类、萜类、醌类、单宁、外源凝集素和多肽等[16]。理论上讲,天然产物应该比普通的抗生素具有更多优势。很多抗微生物中草药同时具有抗细菌、真菌、原虫和病毒的特性。同时,中草药产品具有免疫促进作用,有利于宿主对感染的抵抗。青蒿素是传统医学中分离获得的最著名的抗微生物药物,现已成为主要的抗疟疾药[18]。另外,如从博落回中分离获得的植物药sanguiritrin 已经成功用于临床治疗成人和小孩细菌和真菌感染性耳炎、脓性皮炎、皮肤癣、阴道炎、宫颈糜烂、牙周炎、坏死溃疡性龈炎等[79]。
3.5 益生菌、益生元和合生元
尽管益生菌、益生元和合生元的益生功能主要通过改变肠道环境和增强宿主抵抗力等方面来实现[80],但一些实验数据证实有些益生菌具有直接对抗多种耐药菌的功能。如嗜酸乳杆菌和假链状双歧杆菌SPM1309对临床分离的MDR铜绿假单胞菌具有很强的抑制作用[16,81]。干酪乳杆菌对MDR贺氏 (杆) 菌和大肠杆菌具有很强抑制活性[16,82]。且基于乳酸菌的直接饲喂微生物已经上市,BovamineTM和BovamineDefendTM已经被广泛用于减少大肠杆菌O157:H7感染[83]。另外,粪微生态移植作为一种新的多重耐药细菌感染治疗新手段也已用于临床实验[84]。
3.6 抗菌肽类
细菌素是微生物核糖体合成的一种具有抗菌活性的多肽,研究较多的有nisin和lacticin等。虽然一些细菌素如nisin已被开发用于乳腺炎的抗生素替代治疗[85],但目前其主要用途仍以食品保存和防腐为主[41]。目前科学家们正试图利用结构-功能关系理性设计原理对细菌素进行基因改造[86],以提高其特异性、稳定性和扩大宿主谱,从而扩展其用途[87-89]。
宿主防御肽是哺乳动物为了抵抗病原菌感染而分泌的一种小分子短肽。研究较多的是从人肠道中发现的一种阳离子抗菌肽LL-37。研究表明,抗菌肽LL-37对革兰氏阴性菌 (鲍曼不动杆菌、大肠杆菌、鼠伤寒沙门氏菌、铜绿假单胞菌、嗜麦芽窄食单胞菌、普通变形杆菌、肺炎克雷伯氏菌、淋病奈瑟氏菌) 和革兰氏阳性菌 (葡萄球菌、肠球菌、链球菌、芽孢杆菌、嗜酸乳杆菌、单增李斯特菌、痤疮丙酸杆菌) 均有较好的抑菌效果[90]。另外,β-防御素和新近发现的β抵抗素样分子 (RELMβ) 等,也是肠道重要的抗菌蛋白武器[91]。
肽核酸(Peptide nucleic acids,PNAs) 作为反义分子通过抑制靶基因翻译起到抑菌作用。由于可以抵抗核酸酶、蛋白酶和其他酶的降解,被认为是一种很有前景的体内替代经典抗生素的方法[92]。Mondhe等通过设计靶向不同翻译起始区域的PNAs来改变其抗菌谱,从而使之有更好的靶向性。如肽BS0001被设计用来靶向杀死枯草芽孢杆菌,肽KS0001被设计用来靶向杀死肺炎克雷伯菌等[93]。
3.7 纳米颗粒
纳米颗粒可能通过电荷相互作用粘附到细菌膜表面从而破坏细菌膜完整性,改变细胞壁结构,阻止重要酶信号通路等。纳米颗粒及这些离子诱导的氧化应激可以不可逆地损害细菌细胞组分,从而导致细菌死亡[94]。已有研究报道纳米颗粒可替代抗生素用于对抗流产布鲁氏杆菌、金黄色葡萄球菌和牛皮癣感染等[16,95-96]。同时,不同的纳米材料(有机和无机纳米材料,脂质体,生物分子如多糖、脂类、蛋白/多肽和病毒衣壳)与抗生素联合使用,其抗菌效果更好[97-100]。将抗生素连接到纳米材料将有利于增加感染部位抗生素的浓度和提高抗生素与细菌间的相互作用。同样,纳米颗粒与抗菌肽和香精油联合使用也具有协同抗菌作用[101]。纳米材料可作为新型抗菌药物载体系统,以提高药物的内化[102]。
3.8 促进细菌代谢来对抗AMR
细菌代谢减缓与很多抗生素耐受和耐药相关,促进细菌代谢可增加其敏感性[103-106]。通过研究耐药菌的代谢状态,Peng等发现耐药迟钝爱德华菌其代谢谱与敏感菌有差异,其中心代谢通路出现了一些缺陷。耐药菌株的葡萄糖和丙氨酸含量明显较低[107]。通过添加外源代谢物可以刺激中心代谢和增加药物摄入,从而提高抗生素治疗的敏感性[107]。这与以前报道的耐药嗜麦芽窄养单胞菌和铜绿假单胞菌结果一致[108-109]。Allison等也表明外源添加代谢物 (如葡萄糖、甘露醇、果糖) 可刺激中心代谢,使氨基糖苷抗生素能更好清除大肠杆菌和金黄色葡萄球菌的持留和生物膜生成[103]。
3.9 CRISPR-Cas工具
CRISPR-Cas工具已经用来设计生产靶向特异序列的抗微生物制剂[110-113]。这种方法在体外很有效,可选择性杀死携带耐药基因或毒力基因的靶向细菌。Citorik等利用CRISPR-Cas技术设计特异性抗生素,显著提升了大蜡螟感染小鼠的存活率[110]。Bikard等应用CRISPR-Cas工具设计特异性靶向毒性基因的抗菌物质,特异性杀死有毒金黄色葡萄球菌,而对无毒金黄色葡萄球菌没有影响,且在小鼠体内具有很好疗效[111]。
3.10 基于光的抗感染专利技术
最近十年,基于光的治疗在对抗各种AMR方面取得了重大进展。这种方法包括使用抗微生物蓝光、抗菌光动力疗法和杀菌紫外线照射等。光治疗与传统抗生素治疗相比的优点是可选择性地清除微生物细胞,而对人体细胞和组织无害。这些治疗和装置有很多有趣功能和应用,如牙科、一些眼科和皮肤疾病、很难清洗的表面、内科器官 (如胃) 消毒、仪器设备和房间清毒等[114]。
3.11 使用小RNA来对抗耐药性
小RNAs (sRNAs) 是一种存在于细菌中的长度在50−500 bp的核苷酸[115]。已有研究表明调控sRNAs表达对维持AMR表型和让细菌具有生物优势非常重要[116-122]。一些选择性的菌株暴露在抗生素后其sRNAs表达谱发生改变[116-119]。更重要的是,调控抗生素引起的sRNAs反应可增加细菌对多种抗生素的敏感性[120-121,123]。sRNA 有发展成为新药的巨大潜能。sRNA 激动剂或拮抗剂与传统抗生素同时使用可能是优化与减少耐药菌出现的方法之一[124]。
3.12 磷酰二胺吗啉低聚物(Phosphorodiamidatemorpholino oligomers,PMOs)
与DNA类似,PMOs是一个以合成的磷酰二胺和吗啉为骨架的ATCG低聚物[92]。PMOs在体外和体内均能对临床病原菌如鲍氏不动杆菌、洋葱伯克霍尔德菌和大肠杆菌有较好的抑制作用[125-127],代表了一类新的有前景的精准抗菌物质。最新研究表明,靶向、和等必需基因的PMOs与阳离子抗生素多粘菌素B结合后,可提高2–8倍抗铜绿假单胞菌活性[128];靶向可抑制铜绿假单胞菌生物膜形成;靶向的化合物与托普霉素具有协同作用,可以导致小鼠肺部细菌总量降低3个数量级。
4 前景与展望
尽管抗生素的使用和管理还存在很多漏洞,耐药菌和耐药基因的诊断与监测还并不完善,以上列举的各种抗菌策略也还存在许多不足,大众被警告,一场“抗生素”灾难即将来临,人类即将面临来自无法杀灭细菌的最大威胁;然而,世界末日还未来临,科学家们正在积极应对AMR的威胁,新知识及化学疗法正在保卫我们的未来,未来是光明而不是黑暗的[129]。
科学家和企业家们正在努力寻找理想的抗生素替代物 (无毒且容易从人体中排出,能稳定通过胃肠道,环境友好容易分解,选择性抗某些病原菌,对土著肠道菌群影响很小或没影响,不产生耐药性)。各国政府也正在联合各方力量积极应对AMR,包括:1) 改造已有抗生素;2) 寻找新型抗生素;3) 发展和改善给药系统;4) 优化抗生素给药频率和剂量等[130]。
[1] Lipsky BA, Itani K, Norden C, et al. Treating foot infections in diabetic patients: a randomized, multicenter, open-label trial of linezolid versus ampicillin-sulbactam/amoxicillin-clavulanate. Clin Infect Dis, 2004, 38(1): 17–24.
[2] de Kraker ME, Davey PG, Grundmann H, et al. Mortality and hospital stay associated with resistantandbacteremia: estimating the burden of antibiotic resistance in Europe. PLoS Med, 2011, 8(10): e1001104.
[3] Carlet J, Collignon P, Goldmann D, et al. Society’s failure to protect a precious resource: antibiotics. Lancet, 2011, 378(9788): 369–371.
[4] 由英国政府和惠康基金会 (Wellcome Trust) 资助, 以经济学家Jim O’Neill为主席的细菌耐药性评估委员会发表相关研究成果. [2016-05-16]. https://amr-review.org/sites/default/files/160525_Final%20paper_with%20cover.pdf.
[5] Brown ED, Wright GD. Antibacterial drug discovery in the resistance era. Nature, 2016, 529(7586): 336–343.
[6] Boucher HW, Talbot GH, Benjamin DK Jr, et al. 10 x '20 Progress--development of new drugs active against gram-negative bacilli: an update from the infectious diseases Society of America. Clin Infect Dis, 2013, 56(12): 1685–1694.
[7] de La Fuente-Nunez C, Torres MD, Mojica FJ, et al. Next-generation precision antimicrobials: towards personalized treatment of infectious diseases. Curr Opin Microbiol, 2017, 37: 95–102.
[8] Enioutina EY, Teng LD, Fateeva TV, et al. Phytotherapy as an alternative to conventional antimicrobials: combating microbial resistance. Expert Rev Clin Pharmacol, 2017, 10(11): 1203–1214.
[9] Hao HH, Cheng GY, Iqbal Z, et al. Benefits and risks of antimicrobial use in food-producing animals. Front Microbiol, 2014, 5: 288.
[10] Barriere SL. Clinical, economic and societal impact of antibiotic resistance. Expert Opin Pharmacother, 2015, 16(2): 151–153.
[11] Davies J, Davies D. Origins and evolution of antibiotic resistance. Microbiol Mol Biol Rev, 2010, 74(3): 417–433.
[12] Inoue H, Minghui R. Antimicrobial resistance: translating political commitment into national action. Bull World Health Organ, 2017, 95(4): 242.
[13] Xiao YH, Li LJ. China’s national plan to combat antimicrobial resistance. Lancet Infect Dis, 2016, 16(11): 1216–1218.
[14] Laxminarayan R, Amábile-Cuevas CF, Cars O, et al. UN high-level meeting on antimicrobials—what do we need? Lancet, 2016, 388(10041): 218–220.
[15] Vikesland PJ, Pruden A, Alvarez PJJ, et al. Toward a Comprehensive strategy to mitigate dissemination of environmental sources of antibiotic resistance. Environ Sci Technol, 2017, 51(22): 13061–13069.
[16] Sharma C, Rokana N, Chandra M, et al. Antimicrobial resistance: its surveillance, impact, and alternative management strategies in dairy animals. Front Vet Sci, 2017, 4: 237.
[17] Baquero F, Martínez JL, Cantón R. Antibiotics and antibiotic resistance in water environments. Curr Opin Biotechnol, 2008, 19(3): 260–265.
[18] Cui LW, Su XZ. Discovery, mechanisms of action and combination therapy of artemisinin. Expert Rev Anti Infect Ther, 2009, 7(8): 999–1013.
[19] Icgen B, Yilmaz F. Co-occurrence of antibiotic and heavy metal resistance in Kızılırmak River isolates. Bull Environ Contam Toxicol, 2014, 93(6): 735–743.
[20] Lv L, Yu X, Xu Q, et al. Induction of bacterial antibiotic resistance by mutagenic halogenated nitrogenous disinfection byproducts. Environ Pollut, 2015, 205: 291–298.
[21] Xu YB, Hou MY, Li YF, et al. Distribution of tetracycline resistance genes and AmpC β-lactamase genes in representative non-urban sewage plants and correlations with treatment processes and heavy metals. Chemosphere, 2017, 170: 274–281.
[22] Wong GWK. Reducing antibiotic prescriptions for childhood upper respiratory tract infections. Lancet Glob Health, 2017, 5(12): e1170–e1171.
[23] Bertollo LG, Lutkemeyer DS, Levin AS. Are antimicrobial stewardship programs effective strategies for preventing antibiotic resistance? A systematic review. Am J Infect Control, 2018, doi: 10.1016/j.ajic.2018.01.002.
[24] Zhang QQ, Ying GG, Pan CG, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: source analysis, multimedia modeling, and linkage to bacterial resistance. Environ Sci Technol, 2015, 49(11): 6772–6782.
[25] Suzuki S, Horinouchi T, Furusawa C. Acceleration and suppression of resistance development by antibiotic combinations. BMC Genomics, 2017, 18: 328.
[26] Rodriguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river. Water Res, 2015, 69: 234–242.
[27] Devarajan N, Laffite A, Graham ND, et al. Accumulation of clinically relevant antibiotic-resistance genes, bacterial load, and metals in freshwater lake sediments in Central Europe. Environ Sci Technol, 2015, 49(11): 6528–6537.
[28] Su JQ, An XL, Li B, et al. Metagenomics of urban sewage identifies an extensively shared antibiotic resistome in China. Microbiome, 2017, 5: 84.
[29] Zhang Y, Li AL, Dai TJ, et al. Cell-free DNA: a neglected source for antibiotic resistance genes spreading from WWTPs. Environ Sci Technol, 2018, 52(1): 248–257.
[30] Ma LP, Li B, Jiang XT, et al. Catalogue of antibiotic resistome and host-tracking in drinking water deciphered by a large scale survey. Microbiome, 2017, 5: 154.
[31] Pulcini C, Bush K, Craig WA, et al. Forgotten antibiotics: an inventory in Europe, the United States, Canada, and Australia. Clin Infect Dis, 2012, 54(2): 268–274.
[32] Cassir N, Rolain JM, Brouqui P. A new strategy to fight antimicrobial resistance: the revival of old antibiotics. Front Microbiol, 2014, 5: 551.
[33] Chen FF, Di HX, Wang YX, et al. Small-molecule targeting of a diapophytoene desaturase inhibitsvirulence. Nat Chem Biol, 2016, 12(3): 174–179.
[34] Mouton JW, Ambrose PG, Canton R, et al. Conserving antibiotics for the future: new ways to use old and new drugs from a pharmacokinetic and pharmacodynamic perspective. Drug Resist Updat, 2011, 14(2): 107–117.
[35] Falagas ME, Kasiakou SK. Toxicity of polymyxins: a systematic review of the evidence from old and recent studies. Crit Care, 2006, 10(1): R27.
[36] Deshpande LM, Fritsche TR, Moet GJ, et al. Antimicrobial resistance and molecular epidemiology of vancomycin-resistant enterococci from North America and Europe: a report from the SENTRY antimicrobial surveillance program. Diagn Microbiol Infect Dis, 2007, 58(2): 163–170.
[37] Loewe S. The problem of synergism and antagonism of combined drugs. Arzneimittelforschung, 1953, 3(6): 285–290.
[38] Rand KH, Houck H. Daptomycin synergy with rifampicin and ampicillin against vancomycin-resistant enterococci. J Antimicrob Chemother, 2004, 53(3): 530–532.
[39] Fass RJ. Comparativeactivities of beta-lactam-tobramycin combinations againstand multidrug-resistant Gram-negative enteric bacilli. Antimicrob Agents Chemother, 1982, 21(6): 1003–1006.
[40] Scudamore RA, Goldner M. Penetration of the outer membrane ofby synergistic combinations of beta-lactam and aminoglycoside antibiotics. Antimicrob Agents Chemother, 1982, 21(6): 1007–1010.
[41] Zhao WH, Hu ZQ, Okubo S, et al. Mechanism of synergy between epigallocatechin gallate and β-lactams against methicillin-resistant. Antimicrob Agents Chemother, 2001, 45(6): 1737–1742.
[42] Wright GD. Empowering older antibiotics. Cell, 2016, 167(2): 301.
[43] Darwish RM, Aburjai TA. Effect of ethnomedicinal plants used in folklore medicine in Jordan as antibiotic resistant inhibitors on. BMC Complement Altern Med, 2010, 10: 9.
[44] Dudhatra GB, Mody SK, Awale MM, et al. A comprehensive review on pharmacotherapeutics of herbal bioenhancers. Sci World J, 2012, 2012: 637953.
[45] Kumar-Sarangi M, Chandra-Joshi B, Ritchie B. Natural bioenhancers in drug delivery: an overview. P R Health Sci J, 2018, 37(1): 12–18.
[46] Shiota S, Shimizu M, Mizusima T, et al. Restoration of effectiveness of beta-lactams on methicillin-resistantby tellimagrandin I from rose red. FEMS Microbiol Lett, 2000, 185(2): 135–138.
[47] Shimizu M, Shiota S, Mizushima T, et al. Marked potentiation of activity of β-lactams against methicillin-resistantby corilagin. Antimicrob Agents Chemother, 2001, 45(11): 3198–3201.
[48] Cotter PD, Ross RP, Hill C. Bacteriocins-a viable alternative to antibiotics? Nat Rev Microbiol, 2013, 11(2): 95–105.
[49] Cimermancic P, Medema MH, Claesen J, et al. Insights into secondary metabolism from a global analysis of prokaryotic biosynthetic gene clusters. Cell, 2014, 158(2): 412–421.
[50] Lam YC, Crawford JM. Discovering antibiotics from the global microbiome. Nat Microbiol, 2018, 3(4): 392–293.
[51] Garcia-Gutierrez E, Mayer MJ, Cotter PD, et al. Gut microbiota as a source of novel antimicrobials. Gut Microbes, 2018, doi: 10.1080/19490976.2018.1455790.
[52] Donia MS, Cimermancic P, Schulze CJ, et al. A systematic analysis of biosynthetic gene clusters in the human microbiome reveals a common family of antibiotics. Cell, 2014, 158(6): 1402–1414.
[53] Walsh CJ, Guinane CM, Hill C, et al.identification of bacteriocin gene clusters in the gastrointestinal tract, based on the Human Microbiome Project’s reference genome database. BMC Microbiol, 2015, 15: 183.
[54] Hover BM, Kim SH, Katz M, et al. Culture-independent discovery of the malacidins as calcium-dependent antibiotics with activity against multidrug-resistant Gram-positive pathogens. Nat Microbiol, 2018, 3(4): 415–422.
[55] Chu J, Vila-Farres X, Inoyama D, et al. Discovery of MRSA active antibiotics using primary sequence from the human microbiome. Nat Chem Biol, 2016, 12(12): 1004–1006.
[56] Vila-Farres X, Chu J, Inoyama D, et al. Antimicrobials inspired by nonribosomal peptide synthetase gene clusters. J Am Chem Soc, 2017, 139(4): 1404–1407.
[57] Ling LL, Schneider T, Peoples AJ, et al. A new antibiotic kills pathogens without detectable resistance. Nature, 2015, 517(7535): 455–459.
[58] Gavrish E, Sit CS, Cao SG, et al. Lassomycin, a ribosomally synthesized cyclic peptide, killsby targeting the ATP-dependent protease ClpC1P1P2. Chem Biol, 2014, 21(4): 509–518.
[59] Kim W, Zhu WP, Hendricks GL, et al. A new class of synthetic retinoid antibiotics effective against bacterial persisters. Nature, 2018, 556(7699): 103–107.
[60] Kim HU, Blin K, Lee SY, et al. Recent development of computational resources for new antibiotics discovery. Curr Opin Microbiol, 2017, 39: 113–120.
[61] Lin DM, Koskella B, Lin HC. Phage therapy: An alternative to antibiotics in the age of multi-drug resistance. World J Gastrointest Pharmacol Ther, 2017, 8(3): 162–173.
[62] Abedon ST. Bacteriophage clinical use as antibacterial “Drugs”: utility and precedent. Microbiol Spectr, 2017, 5(4), doi: 10.1128/microbiolspec.BAD- 0003-2016.
[63] Forde A, Hill C. Phages of life - the path to pharma. Br J Pharmacol, 2018, 175(3): 412–418.
[64] Czaplewski L, Bax R, Clokie M, et al. Alternatives to antibiotics-a pipeline portfolio review. Lancet Infect Dis, 2016, 16(2): 239–251.
[65] Yan JL, Mao JX, Xie JP. Bacteriophage polysaccharide depolymerases and biomedical applications. BioDrugs, 2014, 28(3): 265–274.
[66] Drulis-Kawa Z, Majkowska-Skrobek G, Maciejewska B. Bacteriophages and phage-derived proteins—applicationapproaches. Curr Med Chem, 2015, 22(14): 1757–1773.
[67] Pires DP, Oliveira H, Melo LD, et al. Bacteriophage-encoded depolymerases: their diversity and biotechnological applications. Appl Microbiol Biotechnol, 2016, 100(5): 2141–2151.
[68] Latka A, Maciejewska B, Majkowska-Skrobek G, et al. Bacteriophage-encoded virion-associated enzymes to overcome the carbohydrate barriers during the infection process. Appl Microbiol Biotechnol, 2017, 101(8): 3103–3119.
[69] Clift C, Salisbury DM. Enhancing the role of vaccines in combatting antimicrobial resistance. Vaccine, 2017, 35(48 Pt B): 6591–6593.
[70] Laverde D, Probst I, Romero-Saavedra F, et al. Targeting type IV secretion system proteins to combat multidrug-resistant gram-positive pathogens. J Infect Dis, 2017, 215(12): 1836–1845.
[71] Sassone-Corsi M, Chairatana P, Zheng TF, et al. Siderophore-based immunization strategy to inhibit growth of enteric pathogens. Proc Natl Acad Sci USA, 2016, 113(47): 13462–13467.
[72] Fleming-Dutra KE, Hersh AL, Shapiro DJ, et al. Prevalence of inappropriate antibiotic prescriptions among US ambulatory care visits, 2010–2011. JAMA, 2016, 315(17): 1864–1873.
[73] Kwong JC, Maaten S, Upshur RE, et al. The effect of universal influenza immunization on antibiotic prescriptions: an ecological study. Clin Infect Dis, 2009, 49(5): 750–756.
[74] Lipsitch M, Siber GR. How can vaccines contribute to solving the antimicrobial resistance problem? mBio, 2016, 7(3): e00428-16.
[75] Tomczyk S, Lynfield R, Schaffner W, et al. Prevention of antibiotic-nonsusceptible invasive pneumococcal disease with the 13-valent pneumococcal conjugate vaccine. Clin Infect Dis, 2016, 62(9): 1119–1125.
[76] von Gottberg A, de Gouveia L, Tempia S, et al. Effects of vaccination on invasive pneumococcal disease in South Africa. N Engl J Med, 2014, 371(20): 1889–1899.
[77] Anthouard R, DiRita VJ. Chemical biology applied to the study of bacterial pathogens. Infect Immun, 2015, 83(2): 456–469.
[78] Dickey SW, Cheung GYC, Otto M. Different drugs for bad bugs: antivirulence strategies in the age of antibiotic resistance. Nat Rev Drug Discov, 2017, 16(7): 457–471.
[79] Gupta PD, Birdi TJ. Development of botanicals to combat antibiotic resistance. J Ayurveda Integr Med, 2017, 8(4): 266–275.
[80] Markowiak P, Śliżewska K. Effects of probiotics, prebiotics, and synbiotics on human health. Nutrients, 2017, 9(9): 1021.
[81] Jamalifar H, Rahimi HR, Samadi N, et al. Antimicrobial activity of differentspecies against multi-drug resistant clinical isolates of. Iran J Microbiol, 2011, 3(1): 21–25.
[82] Mirnejad R, Vahdati AR, Rashidiani J, et al. The antimicrobial effect ofculture supernatant against multiple drug resistant clinical isolates ofand. Iran Red Crescent Med J, 2013, 15(2): 122–126.
[83] Téllez G, Lauková A, Latorre JD, et al. Food-producing animals and their health in relation to human health. Microb Ecol Health Dis, 2015, 26: 25876, doi: 10.3402/mehd.v26.25876.
[84] Manges AR, Steiner TS, Wright AJ. Fecal microbiota transplantation for the intestinal decolonization of extensively antimicrobial-resistant opportunistic pathogens: a review. Infect Dis (Lond), 2016, 48(8): 587–592.
[85] Pieterse R, Todorov SD, Dicks MT. Mode of action andsusceptibility of mastitis pathogens to macedocin ST91KM and preparation of a teat seal containing the bacteriocin. Braz J Microbiol, 2010, 41(1): 133–145.
[86] Nes IF, Johnsborg O. Exploration of antimicrobial potential in LAB by genomics. Curr Opin Biotechnol, 2004, 15(2): 100–104.
[87] Kuwano K, Tanaka N, Shimizu T, et al. Dual antibacterial mechanisms of nisin Z against Gram-positive and Gram-negative bacteria. Int J Antimicrob Agents, 2005, 26(5): 396–402.
[88] Field D, O’Connor R, Cotter PD, et al.activities of nisin and nisin derivatives alone and in combination with antibiotics againstbiofilms. Front Microbiol, 2016, 7: 508.
[89] Field D, Gaudin N, Lyons F, et al. A bioengineered nisin derivative to control biofilms of. PLoS ONE, 2015, 10(3): e0119684.
[90] Xhindoli D, Pacor S, Benincasa M, et al. The human cathelicidin LL-37—A pore-forming antibacterial peptide and host-cell modulator. Biochim Biophys Acta (BBA)-Biomemb, 2016, 1858(3): 546–566.
[91] Propheter DC, Chara AL, Harris TA, et al. Resistin-like molecule β is a bactericidal protein that promotes spatial segregation of the microbiota and the colonic epithelium. Proc Natl Acad Sci USA, 2017, 114(42): 11027–11033.
[92] Sully EK, Geller BL. Antisense antimicrobial therapeutics. Curr Opin Microbiol, 2016, 33: 47–55.
[93] Mondhe M, Chessher A, Goh S, et al. Species-selective killing of bacteria by antimicrobial peptide-PNAs. PLoS ONE, 2014, 9(2): e89082.
[94] Hajipour MJ, Fromm KM, Ashkarran AA, et al. Antibacterial properties of nanoparticles. Trends Biotechnol, 2012, 30(10): 499–511.
[95] Seil JT, Webster TJ. Antimicrobial applications of nanotechnology: methods and literature. Int J Nanomedicine, 2012, 7: 2767–2781.
[96] Gomes F, Henriques M. Control of bovine mastitis: old and recent therapeutic approaches. Curr Microbiol, 2016, 72(4): 377–382.
[97] Veerapandian M, Yun K. Functionalization of biomolecules on nanoparticles: specialized for antibacterial applications. Appl Microbiol Biotechnol, 2011, 90(5): 1655–1667.
[98] Faraji AH, Wipf P. Nanoparticles in cellular drug delivery. Bioorg Med Chem, 2009, 17(8): 2950–2962.
[99] Gaucher G, Marchessault RH, Leroux JC. Polyester-based micelles and nanoparticles for the parenteral delivery of taxanes. J Control Release, 2010, 143: 2–12.
[100] Subbiah R, Veerapandian M, Yun KS. Nanoparticles: functionalization and multifunctional applications in biomedical sciences. Curr Med Chem, 2010, 17(36): 4559–4577.
[101] Allahverdiyev AM, Kon KV, Abamor ES, et al. Coping with antibiotic resistance: combining nanoparticles with antibiotics and other antimicrobial agents. Expert Rev Anti Infect Ther, 2011, 9(11): 1035–1052.
[102] Santos RS, Figueiredo C, Azevedo NF, et al. Nanomaterials and molecular transporters to overcome the bacterial envelope barrier: Towards advanced delivery of antibiotics. Adv Drug Deliv Rev, 2017, doi: 10.1016/j.addr.2017.12.010.
[103] Allison KR, Brynildsen MP, Collins JJ. Metabolite-enabled eradication of bacterial persisters by aminoglycosides. Nature, 2011, 473(7346): 216–220.
[104] Bryan LE, van den Elzen HM. Effects of membrane-energy mutations and cations on streptomycin and gentamicin accumulation by bacteria: a model for entry of streptomycin and gentamicin in susceptible and resistant bacteria. Antimicrob Agents Chemother, 1977, 12(2): 163–177.
[105] Kohanski MA, Dwyer DJ, Hayete B, et al. A common mechanism of cellular death induced by bactericidal antibiotics. Cell, 2007, 130(5): 797–810.
[106] Martínez JL, Rojo F. Metabolic regulation of antibiotic resistance. FEMS Microbiol Rev, 2011, 35(5): 768–789.
[107] Peng B, Su YB, Li H, et al. Exogenous alanine and/or glucose plus kanamycin kills antibiotic-resistant bacteria. Cell Metab, 2015, 21(2): 249–262.
[108] Alonso A, Morales G, Escalante R, et al. Overexpression of the multidrug efflux pump SmeDEF impairsphysiology. J Antimicrob Chemother, 2004, 53(3): 432–434.
[109] Stickland HG, Davenport PW, Lilley KS, et al. Mutation ofcauses global changes in the physiology and metabolism of. J Proteome Res, 2010, 9(6): 2957–2967.
[110] Citorik RJ, Mimee M, Lu TK. Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases. Nat Biotechnol, 2014, 32(11): 1141–1145.
[111] Bikard D, Euler CW, Jiang WY, et al. Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials. Nat Biotechnol, 2014, 32(11): 1146–1150.
[112] Gomaa AA, Klumpe HE, Luo ML, et al. Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems. mBio, 2014, 5(1): e00928–13.
[113] Yosef I, Manor M, Kiro R, et al. Temperate and lytic bacteriophages programmed to sensitize and kill antibiotic-resistant bacteria. Proc Natl Acad Sci USA, 2015, 112(23): 7267–7272.
[114] Ahmed I, Fang Y, Lu M, et al. Recent patents on light-based anti-infective approaches. Recent Pat Antiinfect Drug Discov, 2017, doi: 10.2174/1872213X11666171108104104.
[115] Kwenda S, Gorshkov V, Ramesh AM, et al. Discovery and profiling of small RNAs responsive to stress conditions in the plant pathogen Pectobacterium atrosepticum. BMC Genomics, 2016, 17: 47.
[116] Molina-Santiago C, Daddaoua A, Gómez-Lozano M, et al. Differential transcriptional response to antibiotics byDOT-T1E. Environ Microbiol, 2015, 17(9): 3251–3262.
[117] Howden BP, Beaume M, Harrison PF, et al. Analysis of the small RNA transcriptional response in multidrug-resistantafter antimicrobial exposure. Antimicrob Agents Chemother, 2013, 57(8): 3864–3874.
[118] Yu J, Schneiders T. Tigecycline challenge triggers sRNA production in Salmonella enterica serovar Typhimurium. BMC Microbiol, 2012, 12: 195.
[119] Chen Y, Indurthi DC, Jones SW, et al. Small RNAs in the genus. mBio, 2011, 2(1): e00340–10.
[120] Kim T, Bak G, Lee J, et al. Systematic analysis of the role of bacterial Hfq-interacting sRNAs in the response to antibiotics. J Antimicrob Chemother, 2015, 70(6): 1659–1668.
[121] Ramos CG, Grilo AM, Sousa SA, et al. Regulation of Hfq mRNA and protein levels inandby theMtvR sRNA. PLoS ONE, 2014, 9(6): e98813.
[122] Jeeves RE, Marriott AAN, Pullan ST, et al.is resistant to isoniazid at a slow growth rate by single nucleotide polymorphisms incodon Ser315. PLoS ONE, 2015, 10(9): e0138253.
[123] Kim JN, Kwon YM. Genetic and phenotypic characterization of the RyhB regulon intyphimurium. Microbiol Res, 2013, 168(1): 41–49.
[124] Chan H, Ho J, Liu XD, et al. Potential and use of bacterial small RNAs to combat drug resistance: a systematic review. Infect Drug Resist, 2017, 10: 521–532.
[125] Greenberg DE, Marshall-Batty KR, Brinster LR, et al. Antisense phosphorodiamidate morpholino oligomers targeted to an essential gene inhibitcomplex. J Infect Dis, 2010, 201(12): 1822–1830.
[126] Geller BL, Deere JD, Stein DA, et al. Inhibition of gene expression inby antisense phosphorodiamidate morpholino oligomers. Antimicrob Agents Chemother, 2003, 47(10): 3233–3239.
[127] Geller BL, Marshall-Batty K, Schnell FJ, et al. Gene-silencing antisense oligomers inhibitgrowthand. J Infect Dis, 2013, 208(10): 1553–1560.
[128] Howard JJ, Sturge CR, Moustafa DA, et al. Inhibition ofby peptide-conjugated phosphorodiamidate morpholino oligomers. Antimicrob Agents Chemother, 2017, 61(4), doi: 10.1128/AAC.01938-16.
[129] Cox JAG, Worthington T. The ‘antibiotic apocalypse’-scaremongering or scientific reporting? Trends Microbiol, 2017, 25(3): 167–169.
[130] Sharma A, Kumar AD, Dua M, et al. Nano-technology for targeted drug delivery to combat antibiotic resistance. Expert Opin Drug Deliv, 2012, 9(11): 1325–1332.
(本文责编 陈宏宇)
Progress in strategies to combat antimicrobial resistance
Yeshi Yin, Huahai Chen, Linyan Cao, Lin Tang, and Fulin He
Key Laboratory of Comprehensive Utilization of Advantage Plants Resources in Hunan South, College of Chemistry and Bioengineering, Hunan University of Science and Engineering, Yongzhou 425199, Hunan, China
Antimicrobial resistance is on the rise while the number of antibiotics being brought to market continues to drop. Drug-resistant genes and drug-resistant bacteria infection have seriously threatened human health. Therefore, antimicrobial resistance presents an ongoing challenge that requires multifaceted approaches including: biomedical innovation; improved surveillance of antibiotic consumption and antimicrobial resistance generated rates; prevention of health-care-associated infections and transmission of multidrug-resistant bacteria and environmental dissemination; rapid microbiological diagnosis; and curtailed clinical and veterinary misuse. Fortunately, combating antimicrobial resistance has been highly valued and supported by the government, scientists and entrepreneurs of various countries. With the continuous introduction of new technologies, new products, and new management measures, the problem of antimicrobial resistance must be controlled and alleviated.
antibiotic, antimicrobial resistance, antibiotic-resistant bacteria, antibiotic-resistant gene
May 26, 2018;
Jun 14, 2018
National Natural Science Foundation of China (No. 31741109), Natural Science Foundation of Hunan Province (No. 2018JJ2146).
Yeshi Yin. Tel/Fax: +86-746-2382989; E-mail: yinyeshi@126.com Fulin He. Tel/Fax: +86-746-6381164; E-mail: 2339695475@qq.com
国家自然科学基金 (No. 31741109),湖南省自然科学基金 (No. 2018JJ2146) 资助。
10.13345/j.cjb.180223

