基于混池测序的抗玉米灰斑病QTL定位
崔凤超, 王平喜, 刘小刚, 邹 枨
中国农业科学院作物科学研究所, 北京 100081
玉米(ZeamayesL.)是世界范围内重要的粮食作物之一,在工业原料、食品加工和饲料等方面应用广泛[1]。玉米灰斑病对玉米产量危害严重,危害程度受环境条件和玉米种质的影响,在美国,玉米灰斑病高发地带玉米产量降低50%以上[2];在巴西中部,玉米灰斑病高发地带玉米产量降低20%~50%[3];在南非,玉米灰斑病高发地带玉米产量降低20%~60%[4]。玉米灰斑病是由玉米尾孢菌(Cercosporazeine)[5,6]和玉蜀黍尾孢菌(Cercosporazeae-maydis)[7]引起的真菌性病害,主要侵染玉米叶片,也可侵染苞叶和叶鞘。1925年,玉米灰斑病在美国的伊利诺伊州和亚历山大州被首次报道[7],随后开始逐渐蔓延[8~10]。1991年,玉米灰斑病在我国首次发生于辽宁省丹东市[11],近年来,在我国的西南地区、黄淮海地区和东北地区等玉米主产区均有发生,并呈现逐渐加重的态势[12,13]。因此,开展对玉米灰斑病的研究具有重要意义。
20世纪90年代以来,科研人员一直对玉米灰斑病的抗性位点进行挖掘。Lehmensiek等[14]将分离群体混合分析法[15,16]与RFLP标记结合,在1、3和5号染色体上共鉴定了4个抗玉米灰斑病QTL(quantitative trait loci),其中1号染色体的表型变异解释率最高,为37%。Zhang等[17]以玉米灰斑病抗病自交系Q11和感病自交系Y32构建分离群体,在5号和8号染色体上分别鉴定出一个主效QTL,并将8号染色体的QTLqRgls1精细定位到1.4 Mb的区间;随后,Xu等[18]在Zhang等研究的基础上,将5号染色体的QTLqRgls2精细定位到约1Mb的区间。Shi等[19]通过元分析方法整合了57个玉米灰斑病抗病QTL,共鉴定了26个“真实”QTL和7个“热点”QTL。王平喜等[20]以IBM2 2008为参考图谱,利用元分析重新整合65个抗玉米灰斑病QTL,获得11个“一致性”QTL区间,通过与拟南芥和水稻的基因组同源比对分析,最终确定了10个玉米灰斑病抗性基因。
分离群体混合分析是基因定位的有效手段之一。20世纪末,研究人员将具有多态性的分子标记与极端表型材料的DNA混池结合,能够成功鉴定与目标性状相关联的位点[15,16];随后,Sun等[21]发现,群体大小、标记密度和极端表型选择比例等是分离群体混合分析中重要的影响因素。随着生物技术的不断发展,研究人员将分离群体混合分析与高通量测序技术相结合,开发混池测序方法,该方法已在水稻、大豆等作物中得到广泛应用[22,23]。然而,混池测序方法在植物病害候选基因挖掘方面应用较少。
本文采用混池测序方法对玉米灰斑病进行研究,利用BC1F1群体中的极端表型,构建两个极端表型的DNA混池并完成高通量测序。数据分析后得到高质量单核苷酸多态性标记(single nucleotide polymorphism,SNP),并确定玉米灰斑病抗病区间。最后,通过抗病区间基因提取、注释、同源分析等方法挖掘玉米灰斑病抗性基因。相比传统基因定位的方法,混池测序方法简单、快速,无需筛选大量多态性标记,为基因定位提供了新的思路。
1 材料与方法
1.1 试验材料及群体构建
本实验中,选择玉米灰斑病抗病自交系Suwan1为供体亲本,感病自交系HM01为受体亲本。2013年夏季在河南新乡,将两个亲本杂交产生F1群体;2013年冬季在海南三亚,以HM01为轮回亲本构建BC1F1群体;2014年夏季在云南德宏,种植BC1F1群体,群体内包括464份家系,在自然发病条件下对玉米单株进行玉米灰斑病抗性鉴定,并以该BC1F1群体进行后续研究。
1.2 表型鉴定
在玉米授粉后7 d、14 d和21 d分别对BC1F1群体进行玉米灰斑病抗病性鉴定。当感病自交系丹340发病程度达到7级时,开始进行田间调查,调查部位主要为玉米棒三叶(穗位叶片、穗位上方叶片和穗位下方叶片)。调查的标准如下:1级为高抗,叶片上无病斑或穗位下方叶片有极少量病斑,病斑占叶面积小于等于5%;3级为抗病,叶片上有少量病斑,占叶面积6%~10%;5级为中抗,穗位下方叶片上病斑较多,占叶面积11%~30%;7级为感病,穗位下方叶片上有大量病斑,占叶面积31%~70%;9级为高感,玉米植株基本被病斑所覆盖或枯死,病斑占叶面积71%以上[24]。除上述情况,玉米植株还会出现非玉米灰斑病致死现象,这种情况需要额外记录。
1.3 混池测序
在BC1F1群体玉米灰斑病抗病性鉴定后,分别选择30株抗病和30株感病植株构建混池。具体步骤主要有:首先,利用CTAB法[25]分别提取玉米单株DNA;然后,在DNA浓度检测后,将抗病植株DNA等量混合构成抗病混池,感病植株DNA等量混合构成感病混池;最后,将两个DNA混池样品送往测序公司(北京贝瑞和康生物技术有限公司),公司质量检测合格后,构建插入片段为200 bp的测序文库,利用Illumina Hiseq 2500测序仪、双端125 bp策略完成基因组测序。
1.4 数据分析
1.4.1数据质量控制 通常使用FastQC软件(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)对原始数据进行质量评估,分别查看单碱基测序质量分布、序列测序质量分布和序列GC含量分布等方面信息,若某评估项目不合格,需要依据情况进行过滤,直到评估合格后进行后续数据分析。
1.4.2序列比对参考基因组 为了提高序列比对的准确性,本研究使用FLASH软件[26]对双端reads进行合并。之后,使用BBMap软件(http://sourceforge.net/projects/bbmap/)将序列比对到玉米参考基因组B73(RefGen_v3),将唯一比对的变异信息用于后续分析。随后,运用SAM tools[27]检测SNP变异位点,只保留SNP测序深度为20以上、比对质量值大于等于30的结果。
1.4.3抗玉米灰斑病相关位点挖掘 针对SNP标记在两个混池中的测序深度差异进行卡方检验,结果中P<0.01的标记作为可能的信号位点,将全基因组1Mb距离范围内大于等于3个信号的区段定义为玉米灰斑病抗性QTL的可能位点。依据此方法,充分挖掘可能的SNP变异位点。利用MaizeGDB[28]和Phytozome(http://www.phytozome.net/)网站,将抗病区间内的基因与拟南芥和水稻基因组进行同源比对,最终确定候选基因。
2 结果与分析
2.1 表型分析
2014年夏季,在云南德宏试验基地,对BC1F1群体中玉米灰斑病抗性进行鉴定。具体依据图1A(彩图见图版四)进行病害分级,该群体病害等级呈连续性分布(图1B),7级数量最多,由此可见,玉米灰斑病抗性是多基因控制的数量性状,与前人研究结果一致[29,30]。
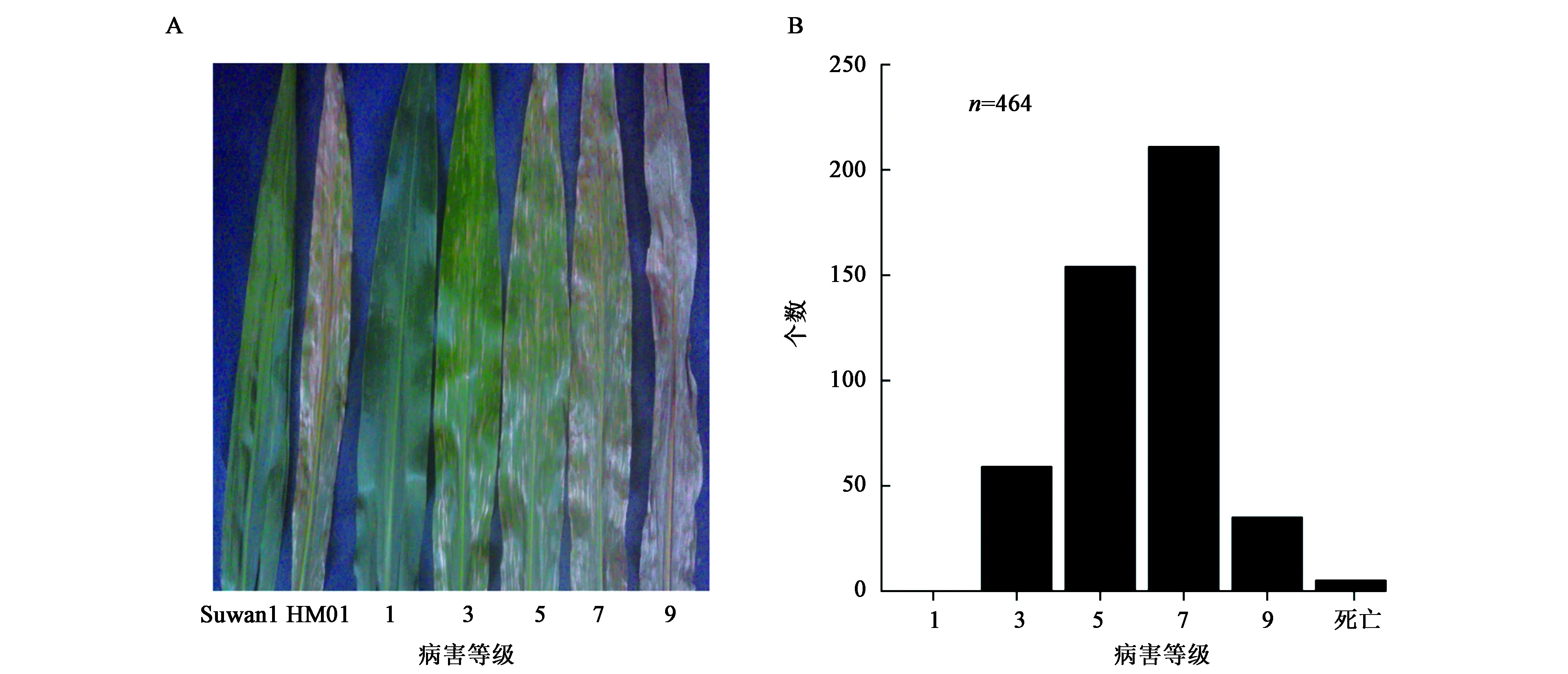
图1 BC1F1群体玉米灰斑病抗性鉴定Fig.1 The identification of GLS resistance in the BC1F1 population in maize.
2.2 混池测序数据分析
根据公司提供的测序数据资料,运行FastQC软件进行数据质量评估,结果表明,90%以上单碱基的测序质量值在30以上,插入片段文库分布集中在200 bp左右,与测序试验设计相吻合。整体评估结果表明,此次测序数据较好,可以用于后续数据分析。生物信息学软件分析获得高质量的SNP标记,最终依据抗病池和感病池测序深度差异的卡方检验,确定29个抗病QTL区间(表1)。从表1中可以看出,玉米灰斑病抗性相关位点在全基因组范围内分布广泛,在玉米的10条染色体中,仅在8号染色体上未检测到抗病位点。此外,检测到的29个QTL区间中,最小的QTL区间可达43 bp。
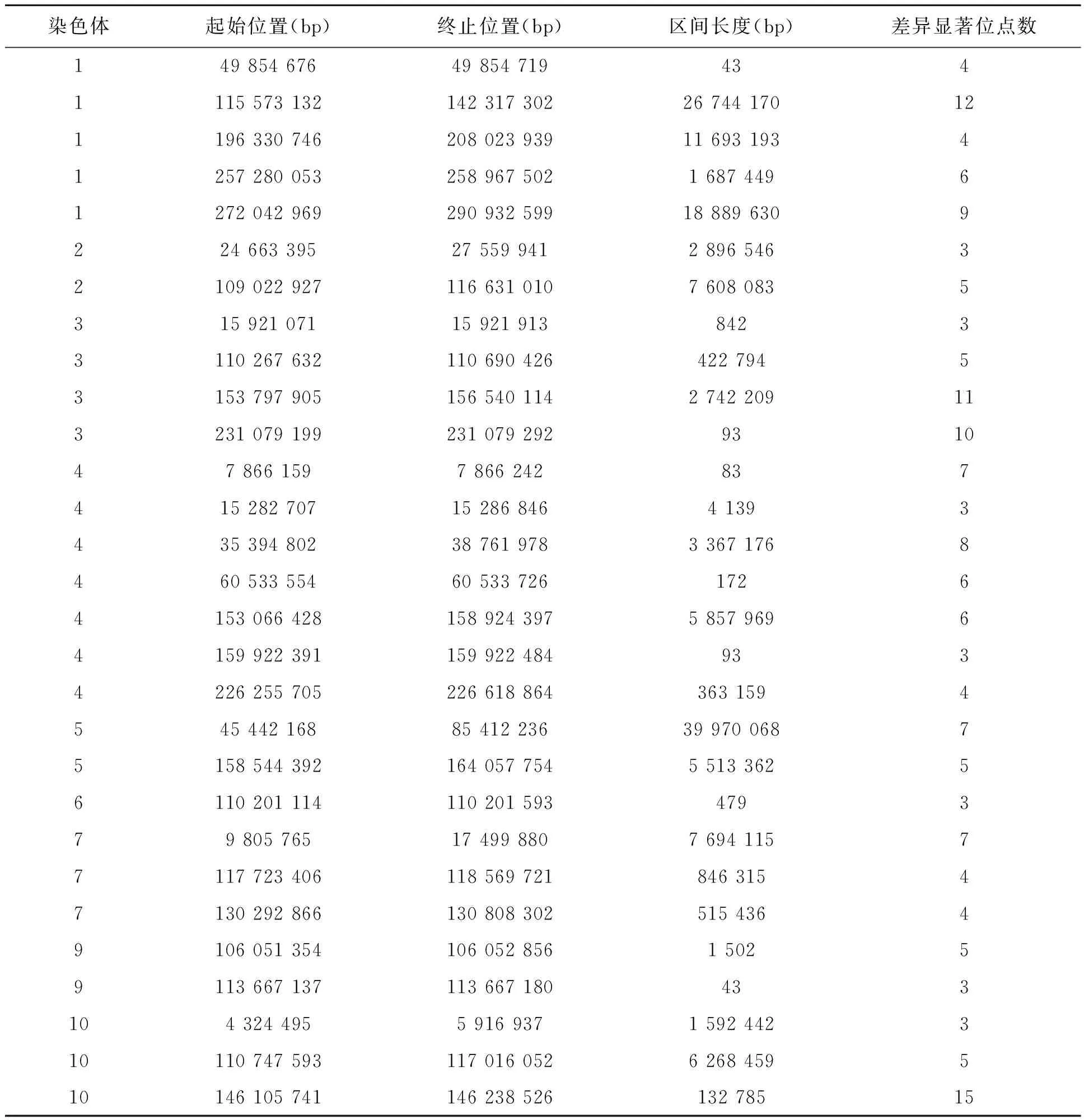
表1 玉米灰斑病抗病QTL区间Table 1 Resistant QTL intervals to GLS in maize.
针对表1中抗玉米灰斑病的QTL区间,共提取2 768基因,结合MaizeGDB和Phytozome网站进行基因功能分析。由于抗病基因编码的蛋白质具有高度保守的结构域,如NBL-LRR(nucleotide-binding site leucine-rich repeat)类型的抗病蛋白、含有螺旋卷曲结构域(coiled-coil)或蛋白激酶结构域(protein kinase)的抗病蛋白等[31,32],根据这些结构域特征,在表1中Chr1:196 330 746~208 023 939、Chr5:45 442 168~85 412 236和Chr10:4 324 495~5 916 937区间内分别确定1个候选基因GRMZM2G322748、GRMZM2G091696和GRMZM2G350841(表2)。在表2中,同源比对分析表明,GRMZM2G322748基因在拟南芥和水稻中的同源基因分别为AT3G14460.1、LOC_Os08g43000;GRMZM2G091696基因的同源基因分别为AT3G07040.1、LOC_Os06g48520.1;GRMZM2G350841基因的同源基因分别为AT1G59780.1、LOC_Os11g38580.1。此外,3个候选基因的注释信息包含LRR、NB-ARC等抗病基因保守结构域,这些结构域对植物抗病蛋白的活性调节具有重要作用[33]。综上所述,这些基因对玉米灰斑病抗性的解析具有重要意义,可用于后续研究。

表2 玉米灰斑病抗性相关候选基因Table 2 Candidate associated genes to GLS resistance in maize.
3 讨论
3.1 玉米灰斑病表型鉴定
玉米灰斑病发病情况主要分为人工接种和自然发病两种情况,对于人工接种而言,需要建立玉米灰斑病病原菌培养及接种体系[34]。此外,田间气候条件对人工接种的菌株和鉴定等方面影响很大,人工接种的成功率也有待进一步提高。对于自然发病而言,则有效避免了人工接种中出现的问题,但是需要选定合适的种植区域,以保证玉米植株能够稳定发病,因此,我们选择玉米灰斑病发病适宜的云南德宏地区进行BC1F1群体的病害鉴定。一般选择在授粉2~3周开始玉米灰斑病调查,因为这是病害发病严重时期,是病害评价的理想阶段[18]。在玉米灰斑病鉴定过程中,Zhang等[17]尝试使用玉米穗位叶病斑扫描的方法进行病害鉴定,但其研究表明这种方法并不理想,主要原因可能是单个叶片扫描结果很难代表整个植株的发病情况。在自然发病的条件下,一般采用人工目测的方法鉴定玉米灰斑病病害等级。
3.2 混池测序方法的应用
本研究通过混池测序方法确定了3个玉米灰斑病抗性候选基因GRMZM2G322748、GRMZM2G0911696和GRMZM2G350841。其中GRMZM2G322748基因位于bin1.06区域内,该区域与Lehmensiek等[14]玉米灰斑病研究结果一致;GRMZM2G0911696基因位于bin5.03区域内,与Xu等[18]玉米灰斑病抗病区间定位结果一致。同时bin1.06和bin5.03两个区域与王平喜等[20]元分析的结果一致,这两个区域都是玉米灰斑病抗性相关的热点区域。GRMZM2G350841基因位于bin10.01~bin10.02区间内,是新发掘的抗病位点,需要进一步探究。以上结果表明混池测序方法是基因挖掘的有力手段,具有操作简单、试验周期短的特点。相比传统的QTL定位,混池测序方法不需要筛选大量的分子标记,也不需要构建特定的近等基因系。通过高通量测序,成千上万的SNP标记可以被一次性捕获,这些变异信息既可以鉴定性状关联基因或QTL,也可以用于开发分子标记。后期我们将进行基因功能解析,以期培育玉米灰斑病的抗病品种,进而提升玉米材料抗病性等,这在遗传育种方面具有重要意义。
3.3 混池测序方法的影响因素
混池测序结果的分析对玉米灰斑病抗性位点的鉴定至关重要,其影响因素较多。通常,精准的田间表型鉴定是混池测序的基本要求,若表型鉴定不准确,会降低混池测序的分析效果;除此之外,目前的测序技术仍存在一定程度的测序错误[35],因此,需要选择合适的测序深度,对于30个样本的混池,混池测序深度通常大于等于30。在混池测序数据分析前期阶段,首先应该对测序数据的质量进行评估,若数据质量存在问题,应该进行适当处理。对于数据分析过程也需要依据试验整体情况选择合适的软件和参数,这样才会得到更加优化的结果。
混池测序可以应用于多种植物和性状,通过本研究,我们得到了玉米灰斑病抗性相关的基因,进一步证实了混池测序的实用性。现在比较常用的混池测序分析方法是基于等位基因频率变化的QTL-seq[22,36,37]、MutMap[38~40]等,但群体结构、连锁不平衡等因素并没有被考虑进去,所以在未来研究中,对混池测序分析方法的优化仍然是值得探究的问题。

