利用癌症基因组图谱对乳腺癌患者CDH1基因突变分析*
林 琳 倪 楠 赵海鹰 姜晓峰 涂 巍*
近年来,乳腺癌在我国城市地区的发病率逐年上升,居女性癌症的前3位[2]。乳腺癌的病因尚不明确,可能与脂肪及酒精摄入、女性初潮年龄普遍提前及停经年龄推后、初产妇年龄增大及生产胎数下降等因素密切相关。有研究表明,基因突变与乳腺癌的发生、发展以及预后都密切相关[3-7]。
E-钙黏蛋白(E-cadherin,CDH1)是Ca2+依赖性细胞粘附分子,编码CDH1基因在1995年首次被克隆,其位于染色体16q22.1,基因全长约100 kb,由16个外显子和15个内含子组成,在物种间高度保守,其编码蛋白包含882个氨基酸,蛋白通过介导细胞粘附和信号转导,参与细胞形态发生和组织分化等生物学行为[8]。有研究发现,在遗传性弥漫型胃癌、卵巢癌中都发现CDH1基因突变病例[9-11]。而CDH1突变与乳腺癌的相关性研究鲜有报道。为此,本研究通过癌症基因组图谱(the cancer genome atlas,TCGA)公共数据集,研究乳腺癌中CDH1突变的发生,为乳腺癌的诊断和治疗提供有价值的线索。
1 材料和方法
1.1 一般资料
选取TCGA数据库中1988-2013年诊断的乳腺癌病例,数据的使用符合TCGA官方网站(https://tcga-data.nci.nih.gov/docs/publications/tcga/)的出版要求。入选的1098例患者,按照CDH1是否发生突变分为野生型CDH1基因未突变组(986例),年龄26~90岁,中位年龄58岁;突变型CDH1基因突变组(112例),年龄34~90岁,中位年龄61岁,肿瘤位置、美国癌症联合委员会(AJCC)肿瘤分期、发病人种等临床资料见表1。
1.2 观察与评价指标
应用cBioPortal网站(http://www.cbioportal.org/)分析TCGA数据集中CDH1突变情况、CDH1基因拷贝数与信使核糖核酸(messenger ribonucleic acid,mRNA)表达的关系及基因共表达分析。
1.3 统计学方法
生存分析采用cBioPortal网站提供的Kaplan-Meier和log-rank检验法,以P<0.05为差异有统计学意义。
2 结果
2.1 CDH1突变
1098个病例中,CDH1突变112例,占比10.4%,其中Q23*突变最多,共6例,P127Afs*41和R63*突变各4例,E243K、R335*及X722_splice各3例,N166Mfs*49、Q195*及X646_splice各2例,另有72个病例突变位点各不相同(见表2)。

表1 1098例乳腺癌患者临床特征(例)
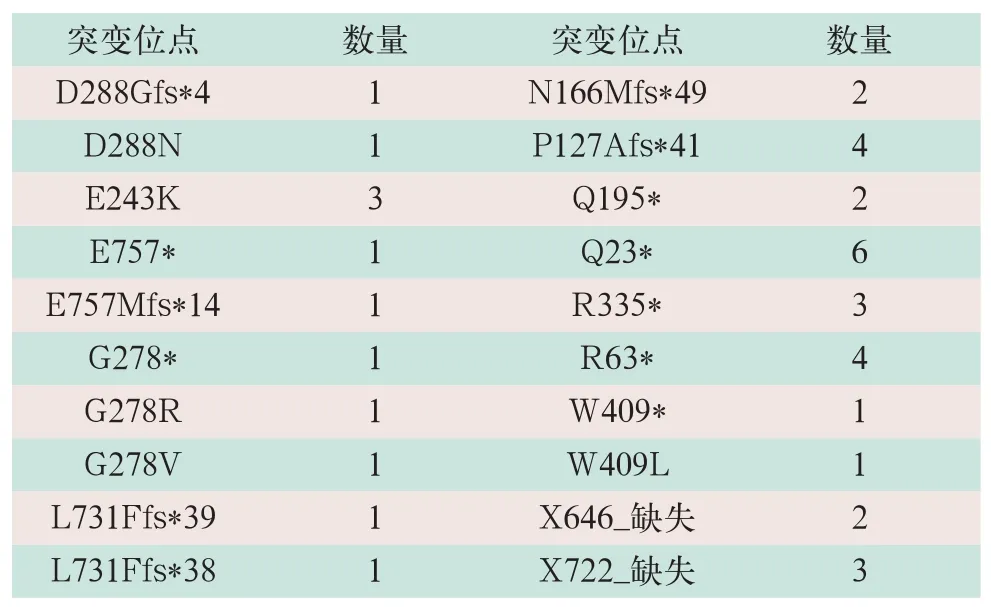
表2 112例CDH1突变患者的CDH1突变结果(例)
2.2 CDH1基因拷贝数与mRNA表达的关系
应用cBioPortal网站分析乳腺癌TCGA数据集,研究CDH1基因mRNA表达水平与其基因拷贝数量和突变的变化,基因拷贝数量的增加,其mRNA表达增多,但拷贝数到达一定程度后,mRNA表达水平变化不明显,同时,突变会导致CDH1 mRNA表达的下降(如图1所示)。
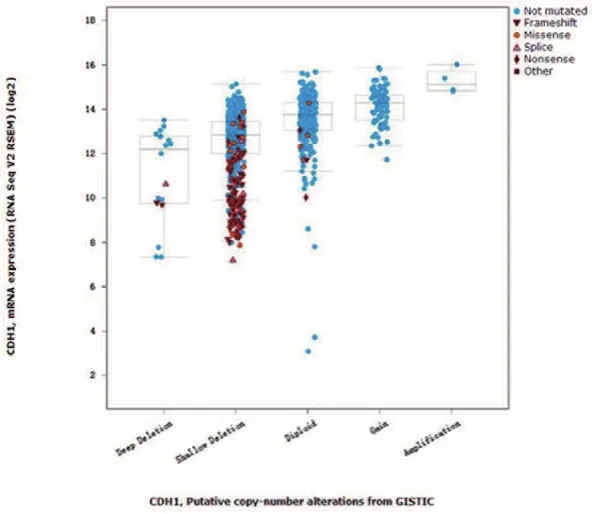
图1 CDH1基因拷贝数与mRNA表达关系示图
2.3 基因共表达分析
应用cBioPortal网站分析乳腺癌TCGA数据集,进行基因共表达研究结果显示,AP1G1、CTCF、IST1、C16ORF70等基因mRNA与CDH1表达呈现一定程度的正相关(如图2所示)。
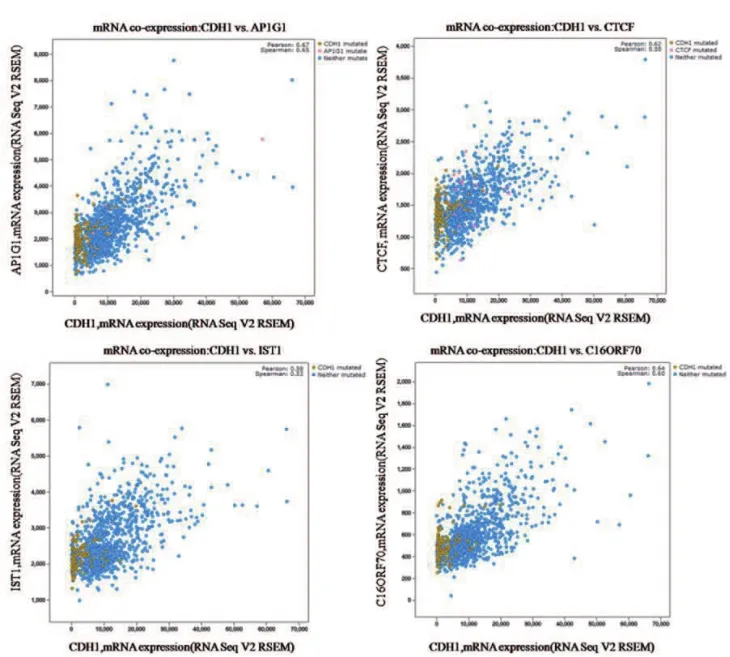
图2 CDH1 基因共表达分析示图
2.4 患者生存期分析
应用cBioPortal网站分析乳腺癌TCGA数据集,采用Kaplan-MeierPlotter在线数据进行患者生存分析结果显示,CDH1突变组与未突变组生存期差异无统计学意义(如图3、图4所示)。

图3 乳腺癌患者的总生存期

图4 乳腺癌患者的无病生存期
2.5 调控网络分析
cBioPortal整合来自人参考蛋白质数据库(human reference protein database,HRPD),Reactome,国立癌症研究所(National Cancer Insititue,NCI)自然版(Nature)和斯隆-凯特琳癌症中心(The Memorial Sloan-Kettering Cancer Center,MSKCC)Cancer Cell Map的数据进行分析,此调控网络中包含51节点,包含CDH1和其50个相关基因,可见CDH1主要通过PI3K/AKT发挥生物学作用,分析结果如图5所示。
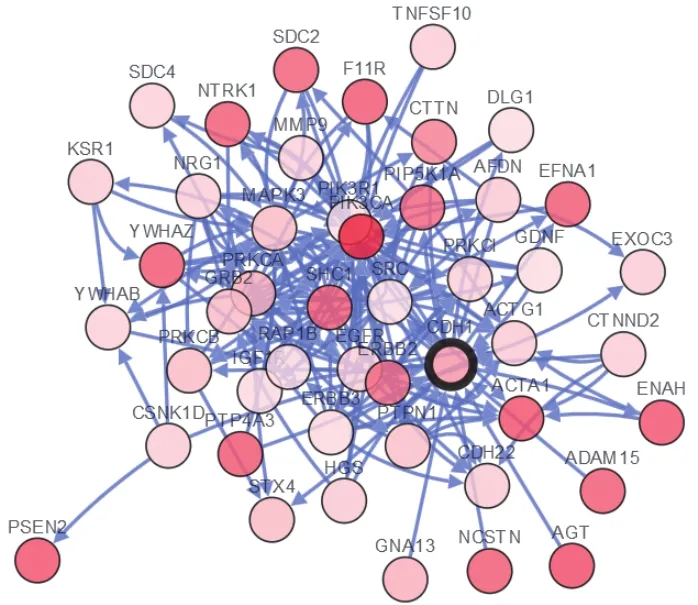
图5 调控网络示图
3 讨论
CDH1是钙黏蛋白家族的重要成员之一,介导基底膜细胞间的连接,其异常表达会降低细胞间的粘附作用,使细胞易于与周围组织分离,与肿瘤细胞转移密切相关,其相关机制主要为:①上皮间充质细胞转化;②Wnt信号通路;③Jak-STAT信号途径;④EGFR信号通路。同时,CDH1基因突变、单核苷酸多态性、启动子甲基化及转录异常调控与与肿瘤的发生、发展及转移的关系密切[12-14]。
CDH1的缺失会促进原发肿瘤的早期生长和继发肿瘤的增殖生长,并能够引起肿瘤的侵袭转移[15]。在多种肿瘤中,CDH1的缺失与肿瘤的预后、肿瘤进展和转移都密切相关[16]。有研究发现,CDH1基因突变会引起癌细胞中CDH1表达缺失,对肿瘤的侵袭和转移扩散产生重要的影响[17]。近年来研究表明,乳腺癌组织中,CDH1的表达调控与其增殖、浸润和转移密切相关,CDH1对乳腺癌的侵袭、转移起到促进作用[18-20]。CDH1生成减少是细胞发生上皮-间质转换(epithelial-mesenchymal transition,EMT)的关键步骤,细胞发生EMT时,可导致细胞间的紧密连接减弱,上皮细胞拥有间质细胞的特性从而获得侵袭和转移能力,此时,上皮细胞表型的标志物CDH1等的表达减少,间质细胞表型的标志物N-钙黏素和波形蛋白等的表达增多。
本研究显示,CDH1突变与乳腺癌的发生有一定程度的相关性。结果表明,CDH1突变总数较多,但是突变位点并不集中,这些突变的发生皆导致CDH1表达的下调或缺失,导致CDH1表达下降,从而对乳腺癌的发生和发展起到一定的促进作用。基因共表达研究还发现AP1G1、CTCF、IST1及C16ORF70等基因mRNA与CDH1表达呈现一定程度的正相关。已有研究表明,CTCF与乳腺癌细胞的凋亡和转移相关[21-22]。AP1G1、IST1及C16ORF70这3个基因目前尚无报道称其与乳腺癌发病相关,相关机制还需要进一步研究。
乳腺癌根据分型采取不同的治疗方案,但目前尚无个体化以及更有针对性的治疗方案,这些初步研究为进一步探讨CDH1基因突变与乳腺癌的发生和发展奠定了一定的基础,为乳腺癌发病机制以及靶向治疗提供新的思路。
[1]Siegel RL,Miller KD,Jemal A.Cancer statistics,2017[J].CA Cancer J Clin,2017,67(1):7-30.
[2]Linos E,Spanos D,Rosner BA,et al.Effects of reproductive and demographic changes on breast cancer incidence in China:a modeling analysis[J].J Natl Cancer Inst,2008,100(19):1352-1360.
[3]王洪义,吕有勇.乳腺癌p53基因突变规律及其预后意义[J].实用肿瘤杂志,2000,15(5):307-309.
[4]赖春宁,王建安,黎燕,等.186例乳腺癌患者BRCA1基因突变检测[J].中华肿瘤杂志,2001,23(6):483-485.
[5]陈营,朱旬,肖莉,等.乳腺癌PIK3CA基因热点突变的研究[J].中华肿瘤防治杂志,2013,20(7):505-508.
[6]马志萍,王雯,张巍.三阴性乳腺癌33例患者BRCA1/2基因突变状态及临床病理学特征[J].中华病理学杂志,2016,45(6):397-400.
[7]邓粤敏,徐韫健.PIK3CA基因突变状态在乳腺癌不同临床病理特征中的研究[J].国际检验医学杂志,2016,37(15):2110-2111.
[8]Berx G,Staes K,van Hengel J,et al.Cloning and characterization of the human invasion suppressor gene E-cadherin(CDH1)[J].Genomics,1995,26(2):281-289.
[9]Kaurah P,MacMillan A,Boyd N,et al.Founder and recurrent CDH1 mutations in families with hereditary diffuse gastric cancer[J].JAMA,2007,297(21):2360-2372.
[10]姜勇,万远廉,王振军,等.遗传性弥漫型胃癌上皮型钙黏素基因种系突变的检测[J].中华外科杂志,2004,42(15):914-917.
[11]姜伶俐,辛晓燕,周洁晶,等.上皮性卵巢癌中CDH1基因突变/甲基化对上皮型钙黏附素表达的影响[J].山西医科大学学报,2010,41(3):214-218.
[12]谭勇川,贾钰铭,雷开键.CDH1基因与卵巢癌[J].川北医学院学报,2015(6):896-901.
[13]Shen ZS,Zhou CC,Li JY,et al.The association,clinicopathological significance,and diagnostic value of CDH1 promoter methylation in head and neck squamous cell carcinoma:a meta-analysis of 23 studies[J].Onco Targets Ther,2016,9:6763-6773.
[14]Cardoso MFS,Castelletti CHM,Lima-Filho JL,et al.Putative biomarkers for cervical cancer:SNVs,methylation and expression profiles[J].Mutat Res,2017,773:161-173.
[15]Beavon IR.The E-cadherin-catenin complex in tumour metastasis:structure,function and regulation[J].Eur J Cancer,2000,36(13 Spec No):1607-1620.
[16]Kowalski PJ,Rubin MA,Kleer CG.E-cadherin expression in primary carcinomas of the breast and its distant metastases[J].Breast Cancer Res,2003,5(6):R217-R222.
[17]Becker KF,Atkinson MJ,Reich U,et al.E-cadherin gene mutations provide clues to diffuse type gastric carcinomas[J].Cancer Res,1994,54(14):3845-3852.
[18]Li P,Sun T,Yuan Q,et al.The expressions of NEDD9 and E-cadherin correlate with metastasis and poor prognosis in triplenegative breast cancer patients[J].Onco Targets Ther,2016,9:5751-5759.
[19]Wang R,Li Z,Guo H,et al.Caveolin 1 knockdown inhibits the proliferation,migration and invasion of human breast cancer BT474 cells[J].Mol Med Rep,2014,9(5):1723-1728.
[20]Palen K,Weber J,Dwinell MB,et al.E-cadherin re-expression shows in vivo evidence for mesenchymal to epithelial transition in clonal metastatic breast tumor cells[J].Oncotarget,2016,7(28):43363-43375.
[21]Venkatraman B,Klenova E.Role of CTCF poly(ADP-Ribosyl)ation in the regulation of apoptosis in breast cancer cells[J].Indian J Med Paediatr Oncol,2015,36(1):49-54.
[22]Mustafa M,Lee JY,Kim MH.CTCF negatively regulates HOXA10 expression in breast cancer cells[J].Biochem Biophys Res Commun,2015,467(4):828-834.

