建兰花色形成的分子调控机理研究
李文建,冯 景,贾文庆,沈永宝*
(1 南京林业大学 风景园林学院,南京 210037;2 河南城建学院 生命科学与工程学院,河南平顶山 467036;3 南京林业大学 南方现代林业协同创新中心,南京 210037;4 河南科技学院 园艺园林学院,河南新乡 453003)
建兰(Cymbidiumensifolium)属于兰科兰属,多年生地生植物,栽培历史悠久。建兰花色繁多,花香幽玄,花姿挺拔,具备极高的美学价值、经济价值和药用价值[1]。前人从芬芳美、色彩美、形态美和意蕴美等方面进行兰花鉴赏[2]。花色是决定园林植物观赏性的关键因素,兰花花色以黄绿色为主,另有红色、粉色、黄色、紫色、褐色甚至碧绿、白色等纯色(素心)或复色(彩心)品种。
植物花色是多种因子协同作用的结果,与花瓣色素种类和含量、生理环境如pH、金属离子等多种因素密切关系[3-4]。花色变化受多因素影响,其中色素种类和含量的时空组合对植物花色起决定性作用[5]。花色色素主要包括类黄酮、类胡萝卜素和生物碱3类。类胡萝卜素存在于质体内,产生月季、郁金香、百合等的黄色及橙色;生物碱类色素有小蘖碱、婴粟碱、甜菜碱等,小蘖碱使小蘖属植物呈深紫色,婴粟碱使婴粟属和绿绒篙属植物呈黄色;类黄酮(黄酮类化合物),存在于液泡,有形成黄色的黄酮、二氢黄酮、查尔酮,以及形成红色、紫色和蓝色等花色的花青素(类黄酮类)[6-8]。研究表明兰花的花色素主要有类黄酮、类胡萝卜素和叶绿素等,其中花青素(类黄酮类)存在最为广泛[9-10]。
建兰作为重要的经济花卉,具有极高的观赏价值和应用前景。国内外对观赏建兰的研究主要集中于种质资源分析、栽培种植技术、繁殖技术和成分工艺等方面。涉及基因和基因调控方式等植物花色形成的成分分析已有较多报道,但关于建兰此种特色花卉的花色形成机理尚未见报道,限制了建兰基因工程育种和种质资源收集与相关研究。本研究通过高通量High-seq测序技术,进行比较性转录组文库构建,分析花色物质的代谢通路及关键调控基因的转录活性,从基因分子水平探索建兰花色形成的相关机理。以期为今后建兰基因工程改变花色和品质、培育新的花色品种奠定基础并提供可靠的信息分析。
1 材料和方法
1.1 材 料
建兰植株采购于四川省成都市温江区,品种为“龙福星蝶”,采购时正为盛花期,花瓣为典型星蝶式复色,颜色以黄绿色为主,花瓣正面边缘红色,正面内侧有红色斑点。实验材料取自同一植株完全开放花朵,分别为红色花瓣(H, 3个重复)、黄绿色花瓣(L, 3个重复),取样前采用无水乙醇拭擦花瓣,使花瓣完整洁净,再采用解剖刀分取裁剪同一花瓣不同颜色部位的组织,立即冻存于-80 ℃超低温冰箱,用于总RNA提取。
1.2 方 法
1.2.1RNA提取用Trizol法提取2种颜色花瓣总RNA,每个样品提取3次,再分别采用Nanodrop、Qubit 2.0、Aglient 2100方法检测RNA样品的纯度、浓度和完整性。
1.2.2反转录与建库总RNA提取完成后,采用带有Oligo(dT)的磁珠富集mRNA,加入裂解缓冲液随机打断,反转录为cDNA链,再进行纯化、末端修复、加A尾、连接测序接头,构建测序文库。
1.2.3转录组测序与组装基于边合成边测序(Sequencing By Synthesis, SBS)技术,使用HiSeq X-ten高通量测序平台对cDNA文库进行测序,得到原始数据(Raw Data)。对Raw Data进行数据过滤,去除其中接头序列及引物序列,过滤低质量Reads,最终获得高质量的Clean Data。用Trinity[11]软件对Clean Data进行序列组装,获得该物种的Unigene库,并与NR、Swiss-Prot、GO、COG、KOG、eggNOG4.5、KEGG数据库比对,得到Unigene功能注释信息。
1.2.4差异表达分析采用DESeq[12]进行样品组间的差异表达分析,获得2个条件之间的差异表达基因集。Unigene的表达丰度可通过FPKM方法进行估算,差异表达基因(differentially expressed genes, DEGs)筛选标准:False Discovery Rate FDR(false discovery rate)< 0.05且log2|FC| ≥2,其中FC(fold change)表示两样品组间表达量之比值。对差异表达基因做GO功能富集分析、COG分类、KEGG注释及通路富集分析。
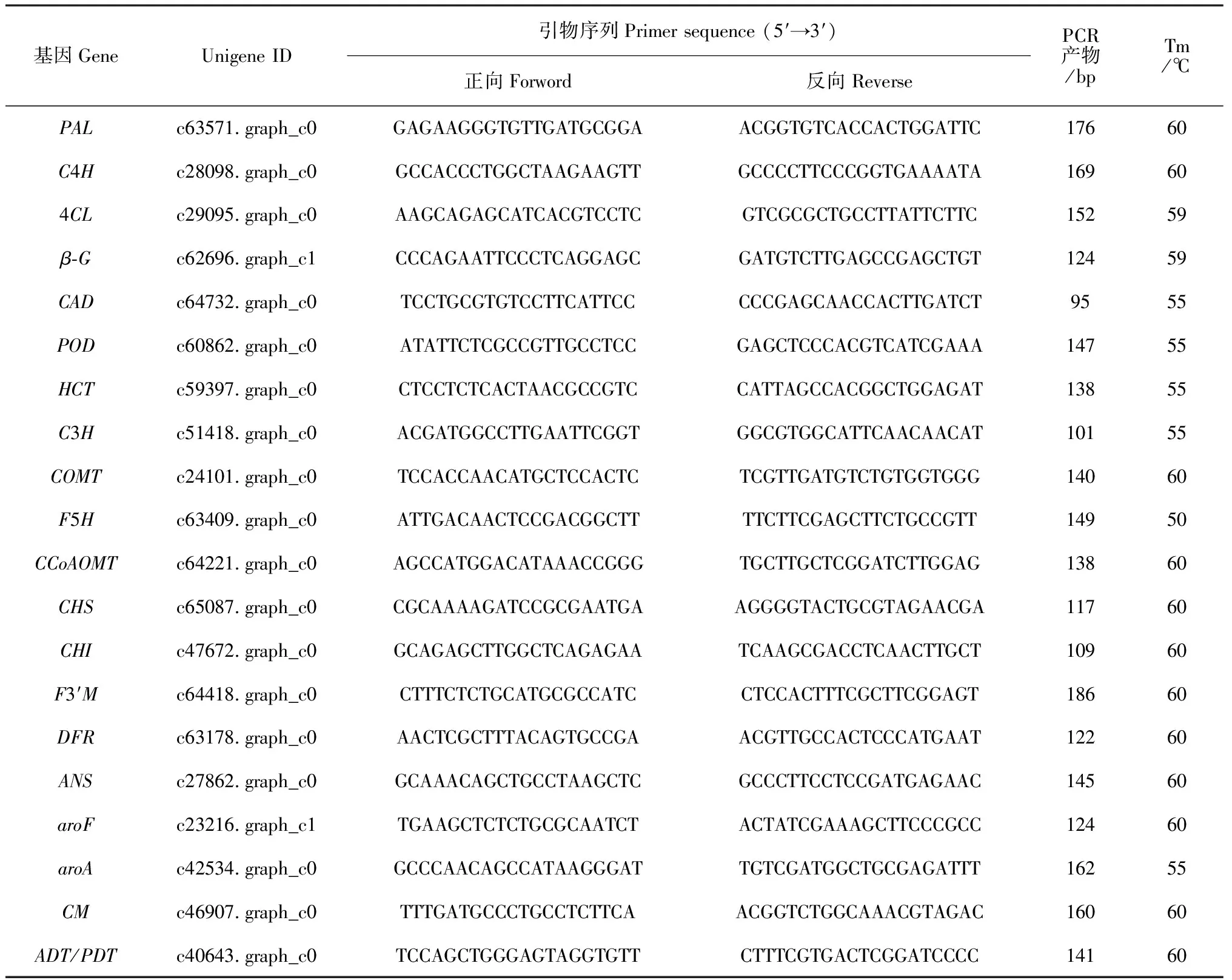
表1 建兰关键差异表达基因QRT-PCR引物信息
1.2.5基因转录模式验证用荧光定量PCR(QRT-PCR)法对建兰花瓣转录组中关键通路基因序列的转录活性进行验证,以肌动蛋白Actin为内参,所涉基因引物信息见表1。在Bio-Rad CFX 1 000 Real-Time PCR平台上,以建兰花瓣cDNA样品为模板,PCR体系为20 μL:10 μL SsoFastTMEvaGreen Supermix (Bio-Rad, USA)、1.0 μL 引物(10 nmol/L)、适量模板与双蒸水。PCR程序:94 ℃孵育20 s, 45个循环(94 ℃ 5 s、Tm退火与延伸共20 s),溶解曲线(65~95 ℃, 0.5 ℃/s)。设置3次重复,用2-ΔΔCt方法进行定量数据计算。
2 结果与分析
2.1 花瓣转录组测序及组装
花瓣总RNA提取量达4.1~6.5 μg,RNI值≥7.9,28S/18S>1.8,且OD260/280>2.0,OD260/230>1.8,表明RNA质量较高。由Illumina HiSeq X-ten高通量测序及原始数据过滤后,6个建兰花瓣样品中共获得131 110 030个高质量碱基序列,碱基数为39 087 158 498 bp。经Trinity组装共得到206 312条转录本和106 479条Unigene,其中长度1 kb以上的Unigene有17 875条(图1)。Transcript与Unigene的N50分别为2 127和1 087,平均长度分别为1 150.14和655.25,组装完整性较高。2种样品Mapped Reads在Clean Reads中所占比例分别为81.86%(L)、76.61%(H),表明转录组测序实验结果质量较高。

图1 建兰花瓣转录组Unigene长度分布图Fig.1 The unigene length of genes in Cymbidium ensifolium petal transcriptomes
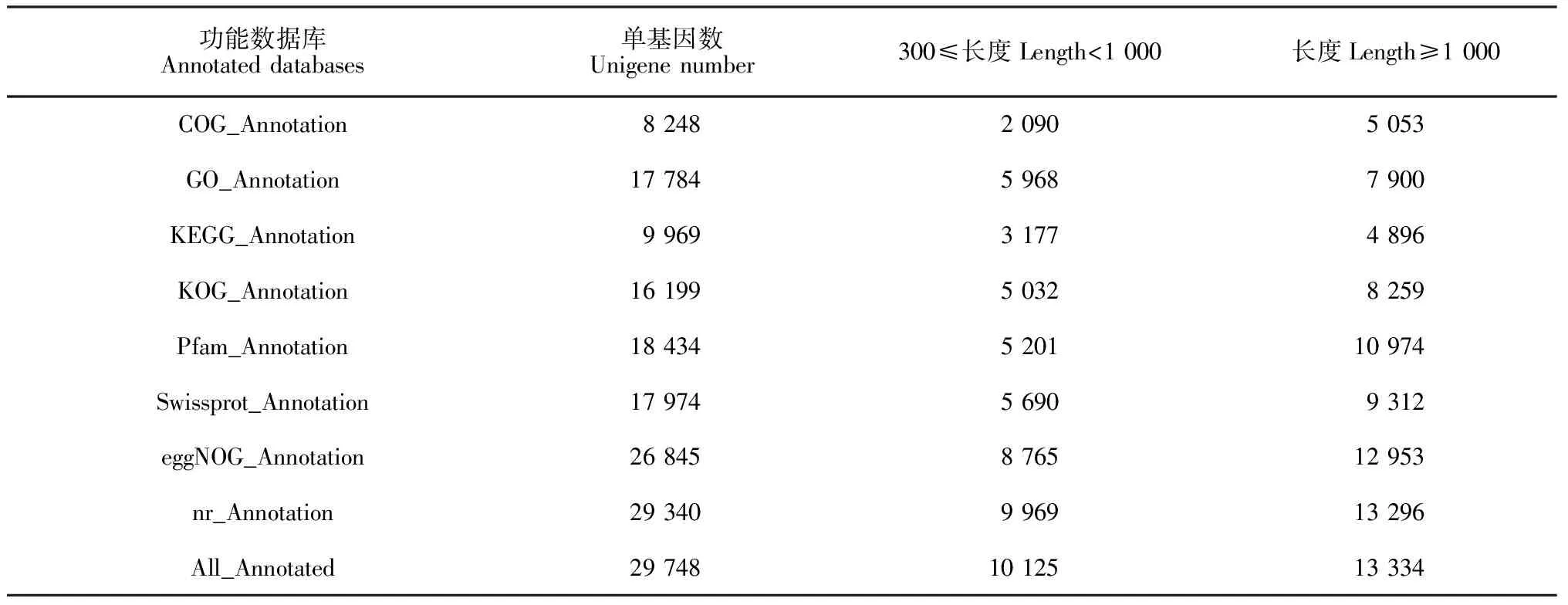
功能数据库Annotated databases单基因数Unigene number300≤长度Length<1 000长度Length≥1 000COG_Annotation8 2482 0905 053GO_Annotation17 7845 9687 900KEGG_Annotation9 9693 1774 896KOG_Annotation16 1995 0328 259Pfam_Annotation18 4345 20110 974Swissprot_Annotation17 9745 6909 312eggNOG_Annotation26 8458 76512 953nr_Annotation29 3409 96913 296All_Annotated29 74810 12513 334
2.2 功能注释
建兰基因组尚未公布,转录组数据注释只能借助公共数据库,故难度增大而准确度降低。本研究比对结果,共获得29 748个有注释信息的Unigene,占Unigene总数的27.94%(表2),注释率不足三分之一,明显偏低。KEGG数据库主要特点是系统分析基因产物细胞代谢途径及其功能,其注释量仅为9 969条(9.36%),表明建兰合成代谢通路信息不完善,有待更全面深入进行基因功能研究。
2.3 花瓣转录组差异表达分析
基因表达具有时间和空间特异性,不同内、外因条件单独或协同作用都会导致基因差异表达。基于总体表达量的FPKM数据分析表明,红色花瓣较黄绿色花瓣存在1 365个DEGs(差异表达基因),其中776个基因转录表达量上升,589个基因转录表达量下降(图2)。基于基因在不同样品中表达量,对识别到的差异表达基因进行功能注释,结果表明建兰花瓣差异表达Unigene共有1 175个得到注释结果,其中692 DEGs被GO数据库注释,424 DEGs被KEGG数据库注释。
2.4 花瓣差异表达基因GO功能富集
GO分析过程,共692个DEG被注释到生物学过程、细胞组分、分子功能等GO分类的54项功能分类中(图3)。在生物学过程一类中,差异表达基因主要分布于代谢过程(metabolic process, 392 DEGs)、细胞转化(cellular process, 385 DEGs)、单一的生物过程(single-organism process, 329 DEGs)、细胞代谢(cellular metabolic process, 300 DEGs)等功能分类中;在细胞组分一类中,与细胞(cell part, cell, intracellular, intracellular part)、细胞器(organelle)等相关的DEG富集较多;在分子功能一类中,主要与催化活性(catalytic activity)、结合(binding, heterocyclic compound binding, organic cyclic compound binding)等功能相关的DEG较多。
2.5 花瓣差异表达基因KEGG通路富集分析
与黄绿色花瓣相比,红色花瓣中776个基因转录表达量上升,589个基因转录表达量下降,其中424个DEG被KEGG数据库注释,涉及93条代谢通路。KEGG通路富集分析结果(图4)表明,DEGs主要分布于黄酮类化合物的生物合成(Flavonoid biosynthesis, 16 Unigenes)、苯丙素的生物合成(Phenylpropanoid biosynthesis, 31 Unigenes)、 苯丙氨酸代谢(Phenylalanine metabolism, 21 Unigenes)、脂肪酸延长(Fatty acid elongation, 10 Uni-genes)、光合天线蛋白(Photosynthesis-antenna proteins, 11 Unigenes)等代谢通路。

图2 差异表达基因火山图Fig.2 The volcanic diagram of differential gene expression
2.6 类黄酮合成代谢相关差异表达基因分析
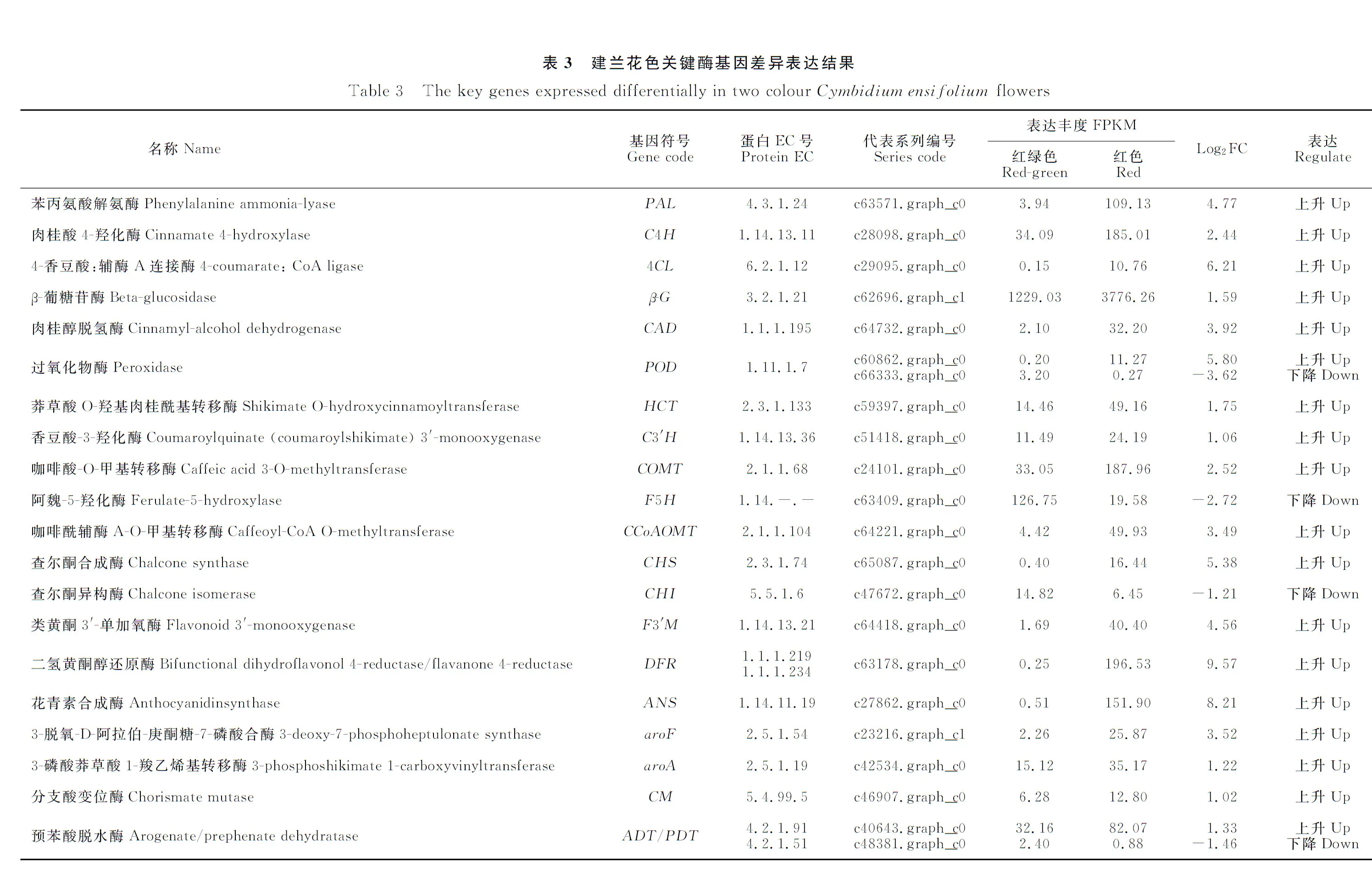
本研究分析发现,建兰黄绿色花瓣与红色花瓣中包括20个类黄酮合成代谢相关差异表达基因,涉及苯丙素的生物合成(Phenylpropanoid biosynthesis, 31 unigenes)、黄酮类化合物生物合成(Flavonoid biosynthesis, 16 unigenes)、苯丙氨酸代谢(Phenylalanine metabolism, 21 unigenes)、黄酮和黄酮醇生物合成(Flavone and flavonol biosynthesis, 3 unigenes)、芪类化合物的合成及姜辣素(Stilbenoid, diarylheptanoid and gingerol biosynthesis, 5 unigenes)、苯丙氨酸、酪氨酸和色氨酸生物合成(Phenylalanine, tyrosine and tryptophan biosynthesis, 7 unigenes)。与黄绿色花瓣相比,各代谢通路中差异表达基因(表3),除阿魏酸-5-羟化酶(ferulate-5-hydroxylase,F5H)和查尔酮异构酶(chalcone isomerase,CHI)基因表达下调,过氧化物酶(peroxidase,POD)、预苯酸脱水酶(arogenate/prephenate dehydratase,ADT/PDT)基因表达既有上升又有下降,其他DEGs表达均发生上调。所有关键差异表达基因中DFR、ANS、4CL、CHS差异最为显著。
2.7 差异转录基因的转录活性验证
用荧光定量PCR法,对建兰花瓣转录组中苯丙烷代谢途径、类黄酮代谢途径中关键基因进行转录活性验证,结果显示,所选基因在两种颜色花瓣中表达量比值趋势与转录组FPKM比值趋势一致(图5),说明本研究获得的转录组数据具有较高的参考价值。

图3 差异表达基因GO功能富集Fig.3 GO function enrichment of differentially expressed genes

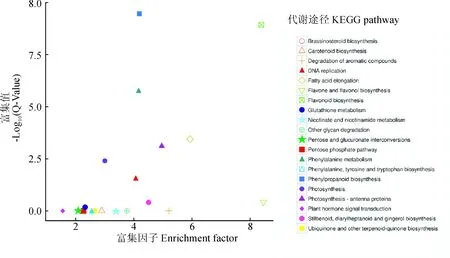
图4 差异表达基因KEGG通路富集散点图Fig.4 Scatter plots of differentially expressed genes in KEGG pathway

图5 建兰花瓣转录组基因表达水平验证Fig.5 Verification of expressed genes in Cymbidium ensifolium petals
3 讨 论
转录组测序技术利用高通量测序技术进行cDNA测序,能全面快速获得材料特定组织某一状态下所有mRNA几乎所有的转录本信息[13]。因而在植物花色机理中的应用研究越来越多。Chen等[14]通过454转录组测序技术分析桃花不同花色花蕾,解析了包括C4H、CHS、CHI及F3H等桃花花色色素合成相关的关键差异基因;Yang等[15]通过转录组测序技术研究不同温度下莲花的开花机理;郑涛等[16]构建了2个花色印度野牡丹不同花期6份花瓣样品的转录组数据库,为野牡丹属植物调控花色相关功能基因开发和利用奠定了基础;徐凌云等[17]喜盐鸢尾内轮花被转录组测序结果对花色形成关键基因进行克隆,初步分析不同花色喜盐鸢尾花色差异的主要原因。
本研究KEGG通路富集分析发现,不同花色建兰转录组数据差异集中在类黄酮合成代谢相关信号通路,类黄酮是重要花色物质,呈现从浅黄到深黄等多种颜色,其在植物花朵中种类和数量相当丰富,如黄酮醇、黄酮、查尔酮和花色素苷等都是花色形成过程中辅助色素成分[18]。本研究发现,相较于黄绿色花瓣,在红色花瓣中,分支酸变位酶(chorismate mutase, CM)、苯丙氨酸解氨酶(Phenylalanine ammonia-lyase, PAL)、肉桂酸4-羟化酶(Cinnamate 4-hydroxylase, C4H)、4-香豆酸:辅酶A连接酶(4-coumarate:CoA ligase, 4CL)、肉桂醇脱氢酶(cinnamyl-alcohol dehydrogenase, CAD)等类黄酮生物合成前体,以及查尔酮合成酶(chalcone synthase, CHS)、二氢黄酮醇还原酶(dihydroflavonol 4-reductase, DFR)、花青素合成酶(leucoanthocyanidin dioxygenase, ANS)等催化查尔酮、花青素化合物合成的关键基因表达均发生上调。
莽草酸通过CM、PDT作用形成苯丙氨酸,进入类黄酮合成代谢途径。苯丙氨酸是花青素类化合物(类黄酮)生物合成的最初前体,被PAL、C4H、4CL催化生成4-香豆酰CoA,再与3-丙二酰CoA反应,为类黄酮提供基本碳骨架,经一系列酶促反应和不同的羟基化、甲基化、糖基化、酰基化修饰后被转运到液泡中积累[10-19]。因此,本研究红色花瓣中CM、PAL、C4H、4CL等基因表达上调可能与积累4-香豆酰CoA物质相关,有利于类黄酮物质生成。
CHS催化4-香豆酰CoA与3-丙二酰CoA合成黄色查尔酮,同时为下游反应提供C14骨架,是植物黄酮类物质合成限速酶,其表达受限将抑制花青素和几乎所有其他类黄酮合成[20]。文心兰OgCHS 5′上游启动子区域甲基化修饰导致OgCHS基因不活跃,导致花中不能积累花青素苷,微粒轰击OgCHS瞬时表达花青素苷,花青素积累利于提高花朵红色程度[21]。Nakatsuka 等[22]研究表明,抑制龙胆CHS表达使植物花青素苷水平降低,导致花色变浅或变白,显示CHS与植物花色相关。DFR和ANS是花青素合成过程关键酶,能将无色二氢黄酮醇转化为无色花青素而合成未修饰的有色花色素[23-24]。多数情况下,花色或果实呈鲜艳颜色,DFR和ANS均高表达[25-26]。香石竹DFR、ANS基因转录水平随花色从粉至红颜色加深而提高[27]。丁灵[10]对18个花色不同石斛兰品种花色相关基因表达模式分析,发现DFR、ANS是白色、黄色和粉红色花色形成的决定性作用酶基因。
建兰是五大国兰品种之一,花色是评价建兰品质关键之一。本研究发现,黄绿色花瓣与红色花瓣转录水平差异基因集中在类黄酮合成代谢途径方面,红色花瓣中多种基因表达上调,特别是CHS、DFR和ANS等3个基因在2种颜色花瓣中差异最为显著;CHS、DFR和ANS在黄绿色花瓣中表达量极低,而在红色花瓣中表达量明显提升;该结果与之前报道的CHS、DFR和ANS是花青素合成的关键基因一致,提示其可能是建兰花色调控的关键基因。
参考文献:
[1] 黄佩璐. 六个建兰品种的叶绿素荧光特性与园林应用研究[D]. 福州:福建农林大学, 2016.
[2] 谭福台. 兰艺的鉴赏[J]. 广东园林, 1996, (3): 2-8.
TAN F T. Appreciation of Lanyi[J].GuangdongGarden, 1996, (3): 2-8.
[3] PAECH K. Colour development in flowers[J].Organization, 1955,12(6): 401-435.
[4] MOL J, CORNISH E, MASON J,etal. Novel coloured flowers[J].CurrentOpinioninBiotechnology, 1999,10(2): 198-201.
[5] 徐怡倩, 袁 媛, 陶秀花,等. 小苍兰花瓣主要花色苷组分研究[J]. 植物研究, 2016,36(2): 184-189.
XU Y Q, YUAN Y, TAO X H,etal. Main anthocyanin profiles in petals ofFreesiahybrida[J].BulletinofBotanicalResearch, 2016,36(2): 184-189.
[6] 郑 智. 木芙蓉花色形成的表型研究[D]. 长沙:湖南农业大学, 2009.
[7] 郑志亮. 花卉作物的花色基因工程[J]. 北方园艺, 1994,3(1): 37-38.
ZHENG Z L. Genetic engineering of flowers and colors in flower crops[J].NorthernHorticulture, 1994,3(1): 37-38.
[8] 陈 建, 吕长平, 陈晨甜,等. 不同花色非洲菊品种花色素成分初步分析[J]. 湖南农业大学学报(自科版), 2009,35(1): 73-76.
CHEN J, LÜ C P, CHEN C T,etal. Components of flower pigments in the petals of different colorGerberajamesonii[J].JournalofHunanAgriculturalUniversity(Nature Science), 2009,35(1): 73-76.
[9] HEW C S, YONG J W H. The Physiology of Tropical Orchids in Relation to the Industry[M]. World Scientific, 1997.
[10] 丁 灵. 蝴蝶石斛兰花色和花香形成相关基因的表达与分析[D]. 海口:海南大学, 2016.
[11] GRABHERR M G, HAAS B J, YASSOUR M,etal. Full length transcriptome assembly from RNA Seq data without a reference genome[J].NatureBiotechnologyItalic, 2011,(29): 644-652
[12] LENG N, DAWSON J A, THOMSON J A,etal. EBSeq: An empirical Bayes hierarchical model for inference in RNA-seq experiments[J].Bioinformatics, 2013.29(8): 1 035-1 043.
[13] FIAMMETTA A, NUNZIO D, LAURA T,etal. Comparative 454 pyrosequencing of transcripts from two olive genotypes during fruit development[J].BMCGenomics, 2009,10(1): 399.
[14] CHEN Y, MAO Y, LIU H,etal. Transcriptome analysis of differentially expressed genes relevant to variegation in peach flowers[J].PLoSOne, 2014,9(3): e90842.
[15] YANG M, ZHU L, XU L,etal. Comparative transcriptomic analysis of the regulation of flowering in temperate and tropical lotus (Nelumbonucifera) by RNA-Seq[J].AnnalsofAppliedBiology, 2014,165(1): 73-95.
[16] 郑 涛, 林艺华, 林秀香,等. 两个花色印度野牡丹转录组De novo数据分析[J]. 分子植物育种, 2017,15(2): 513-518.
ZHENG T, LIN Y H, LIN X X,etal. Transcriptome de novo data analysis ofMelastomamalabathricumwith two flower colors[J].MolecularPlantBreeding, 2017,15(2): 513-518.
[17] 徐凌云, 王俊丽, 周宜君. 喜盐鸢尾花色形成关键基因的克隆及表达分析[J]. 植物遗传资源学报, 2017,18(2): 340-348.
XU L Y, WANG J L, ZHOU Y J. Cloning and Expression Analysis of Anthocyanin Biosynthetic Genes fromIrishalophila(Iridaceae)[J].JournalofPlantGeneticResources, 2017,18(2): 340-348.
[19] 石少川, 高亦珂, 张秀海,等. 植物花青素生物合成相关基因的研究及应用[J]. 植物研究, 2011,31(5): 633-640.
SHI S C, GAO Y K, ZHANG X H,etal. Progress on plant genes involved in biosynthetic pathway of anthocyanins[J].BulletinofBotanicalResearch, 2011,31(5): 633-640.
[20] CLARK S T, VERWOERD W S. A systems approach to identifying correlated gene targets for the loss of colour pigmentation in plants[J].BMCBioinformatics, 2011,12(1): 1-18.
[21] LIU X J, CHUANG Y N, CHIOU C Y,etal. Methylation effect on chalcone synthase gene expression determines anthocyanin pigmentation in floral tissues of twoOncidium, orchid cultivars[J].Planta, 2012,236(2): 401-409.
[22] NAKATSUKA T, KEI-LCHIRO M, ABE Y,etal. Flower color modification of gentian plants by RNAi-mediated gene silencing[J].PlantBiotechnology, 2008,25(1): 61-68.
[23] JOHNSON E T, RYU S, YI H,etal. Alteration of a single amino acid changes the substrate specificity of dihydroflavonol 4-reductase[J].ThePlantJournal, 2001,25(3): 325-333.
[24] CHEN W H, HSU C Y, CHENG H Y,etal. Downregulation of putative UDP-glucose: flavonoid 3-O-glucosyl transferase gene alters flower coloring inPhalaenopsis[J].PlantCellReports, 2011,30(6): 1 007-1 017.
[25] MONTEFIORI M, ESPLEY R V, STEVENSON D,etal. Identification and characterisation of F3GT1 and F3GGT1, two glycosyl transferases responsible for anthocyanin biosynthesis in red-fleshed kiwifruit (Actinidiachinensis)[J].ThePlantJournal, 2011,65(1): 106-118.
[26] HAN Y, VIMOLMANGKANG S, SORIAGUERRA R E,etal. Introduction of apple ANR genes into tobacco inhibits expression of both CHI and DFR genes in flowers, leading to loss of anthocyanin[J].JournalofExperimentalBotany, 2012,63(7):2 437-2 447.
[27] MATO M, ONOZAKI T, OZEKI Y,etal. Flavonoid biosynthesis in pink-flowered cultivars derived from ‘William Sim’ carnation (Dianthuscaryophyllus)[J].JournaloftheJapaneseSocietyforHorticulturalScience, 2001,70(3):315-319.

