洋葱CMS-T型育性分子标记的筛选与鉴定
潘美红,杨海峰,惠林冲,薛 萍,张新圣,缪美华,陈振泰
(连云港市农业科学院,江苏连云港 222000)
洋葱(AlliumcepaL.)属单子叶天门冬目(Asparagale)百合科(Liliaceae)葱属(Allium)的重要园艺作物[1],已有4 000多年的栽培历史,被世界各地广泛栽培[2]。联合国粮食与农业组织2012调查数据显示:中国是生产洋葱最多的国家,达到2.05×107t[3],主要用于外贸出口和国内消费。目前国内洋葱杂交种主要以进口日本和韩国为主,缺乏自主杂交品种。
洋葱是2 a生植物,在杂交育种中,1个世代需跨越3个年份,异花授粉,伞状花序,花小,结籽率低,人工去雄困难,所以利用洋葱细胞质雄性不育(CMS,cytoplasmic male sterility)系选育杂交种尤为重要。首次报道在洋葱品种‘Italian Red’[4]和‘Jaune paille des Vertus’[5]中分别发现CMS-S和CMS-T 2种细胞质雄性不育类型。利用现代DNA分子标记技术,PCR扩增能够快速鉴定洋葱雄性细胞质不育类型;Sato[6]利用cob标记能够区分CMS-S型,而不能区分正常可育株(N)和CMS-T;Engelke等[7]利用 orfA501标记在洋葱不育株中扩增出473 bp条带,可育株没有或很弱,与cob标记相结合区分洋葱可育株N、CMS-S和CMS-T,陈沁滨等[8]和吴海涛等[9]利用此标记将洋葱材料101A鉴定为CMS-S和将8A鉴定为CMS-T。Havey[10]在洋葱叶绿体DNA中开发出Cp-cytotype标记,Kim等[11]利用线粒体DNA中的cob-type2和 atp6标记都能将CMS-S型鉴定,但不能区分正常可育株N和CMS-T,而Kim等[12]利用洋葱线粒体DNA中相关细胞色素c氧化酶亚基I(coxI,cytochromecoxidase subunit I)基因,该基因连接到洋葱 orfA501同系物序列上,与韭菜 orfA501序列相比,其含有 atp9基因的5’序列,由coxI和 orfA501同源物组成2 175 bp连续开放阅读框的嵌合序列,并开发出新的标记命名为 orf725,能够将洋葱3种类型完全区分开。洋葱的细胞质雄性不育表现为母系遗传,主要是由线粒体和叶绿体基因中片段差异造成的[13-14]。目前,针对洋葱的N、CMS-S和CMS-T 3种类型叶绿体DNA基因组已公布,研究表明,可育株N(GenBank数据中登录号:KM088013,下同)和CMS-S(KM088014)核苷酸之间有352个SNPs和141处片段插入或缺失,多态性丰富,而N和CMS-T(KM088015)核苷酸之间有4个SNPs和2处片段插入,根据petN和psbM基因之间的差异,设计Cpp1分子标记,能够利用简单的PCR将洋葱的N、CMS-S和CMS-T 3种类型完全区分开[15]。随后Kim等[16]发表洋葱CMS-S线粒体DNA基因组,序列全长316 363 bp,GenBank数据中登录号:KU318712。洋葱叶绿体和线粒体DNA基因组的公开发表,将更有利于洋葱细胞质雄性不育机制的研究。
国外已经利用恢复基因型(MsMs)、保持系(msms)和不育系(msms)生产洋葱F1杂交种。洋葱不育株可以通过有无花粉判断,而保持系则需要不断地杂交、回交来选择,其随机性大、选育周期长。近年来,RAPD[17]、RFLP[18]、AFLP等[19]分子标记研究洋葱细胞质雄性不育和可育材料的多态性,并开发鉴定洋葱育性遗传位点的分子标记。Jones等[4]研究认为CMS-S不育由1对隐性细胞核基因(msms)控制,而Schweisguth[20]认为CMS-T可能是3个独立的细胞核基因共同控制,杂交后代分离比较复杂,Kim[21]认为洋葱2种不育类型都是由单核基因Ms位点控制,开发出jnurf13分子标记,适合CMS-S和CMS-T 2种类型,并且与Ms位点紧密连锁。所以,通过开发分子标记鉴定纯合的恢复基因型(MsMs)、保持系(msms)和不育系(msms)的基因型对杂交制种尤为重要,能够显著缩短育种年限和减少工作量。目前,国内洋葱杂交种生产相关报道很少,尤其是中日照洋葱品种。本试验拟用cob(s)、cob(n)、 orfA501、Cpp1、 orf725 、jnurf13、AcSKP1、DNF-566、RNS-357、OPT和PsaO共11个与洋葱育性相关的分子标记,对连云港市农业科学院蔬菜课题组(以下简称本课题组)选育的洋葱不育系材料进行验证,筛选出简单、高效的分子标记,为洋葱育种奠定基础。
1 材料与方法
1.1 试验材料
洋葱试验材料的生态类型为中日照,全部来自于连云港市农业科学院蔬菜研究室,共34份,分别编号为1~34号,供试材料名称和特点见表1,D9和d9、A1和a1、A3和a3、A-2和a-2、A-3和a-3、T和t分别为相配套的不育系和保持系,6套不育系材料引种名称、来源、单位和时间见表2,其中D9和d9、A1和a1为2套稳定的不育系。表1中13号材料是母本黄皮不育株A-2与父本红皮保持株t杂交的粉皮F1(A-2×t),14号是母本粉皮不育株13号与父本红皮保持系t进行回交的红皮BC1[(A-2×t)×t]。红皮洋葱材料406中筛选出4株不育株,并与406材料中可育株t2、t5、t6、t11分别进行杂交获得F1,分别命名为T-2-2、T-1-5、T-2-6和T-7-11;同时对可育株t2、t5、t6、t11进行自交获得F1,即表1中t-2、t-5、t-6和t-11;512、728、562、B1、H1、911、930、932和948为常规资源材料;T8×512、T10×728、T2×562 3份 材料为杂交种F1。
1.2 试验方法
试验材料在连云港市农业科学院蔬菜试验基地种植,1~14号材料在开花期分别取其1~2片嫩叶,并调查统计是否有花粉,套袋自交,15~34号为随机选取的洋葱鳞茎球,取5~10 g鳞茎片,液氮保存,用北京天根DNA试剂盒(型号:DP350-03)提取洋葱基因组DNA,引物由苏州金唯智生物有限公司合成(表3),PCR扩增试剂购置于TAKARA公司,反应体系为25 μL,包括:2 μL DNA(0.1 μg),2.5 μL 10× PCR buffer,1.25 μL(10 μmol/L)正向引物,1.25 μL(10 μmol/L)反向引物,2 μL dNTPs(10 mmol/L),0.25 μLTaq酶和15.8 mL ddH2O。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min[22],AcSKP1采用MK886、MK898、MK620、和MK628四对引物混合PCR 扩增,反应条件参考Huo等[23],jnurf13的PCR反应条件参考Kim[21]的方法。cob(s)、cob(n)、 orfA501、 orf725、DNF-566和RNS-357的PCR产物采用质量浓度为10 g/L的琼脂糖凝胶进行检测,Cpp1、jnurf13、AcSKP1、OPT和PsaO的PCR产物采用8%的聚丙烯酰氨凝胶(464 mL/L H2O,250 mL/L 1 mmol/L Tris-HCl (pH 8.8),266 mL/L 30% Acrylamide,10 mL/L 10% SDS,10 mL/L 10%(NH4)2S2O8,0.6 mL/L TEMED)电泳检测,电压100 V,电泳3 h,硝酸银染色,采用佳能EOS6D相机在胶片灯上拍照记录。
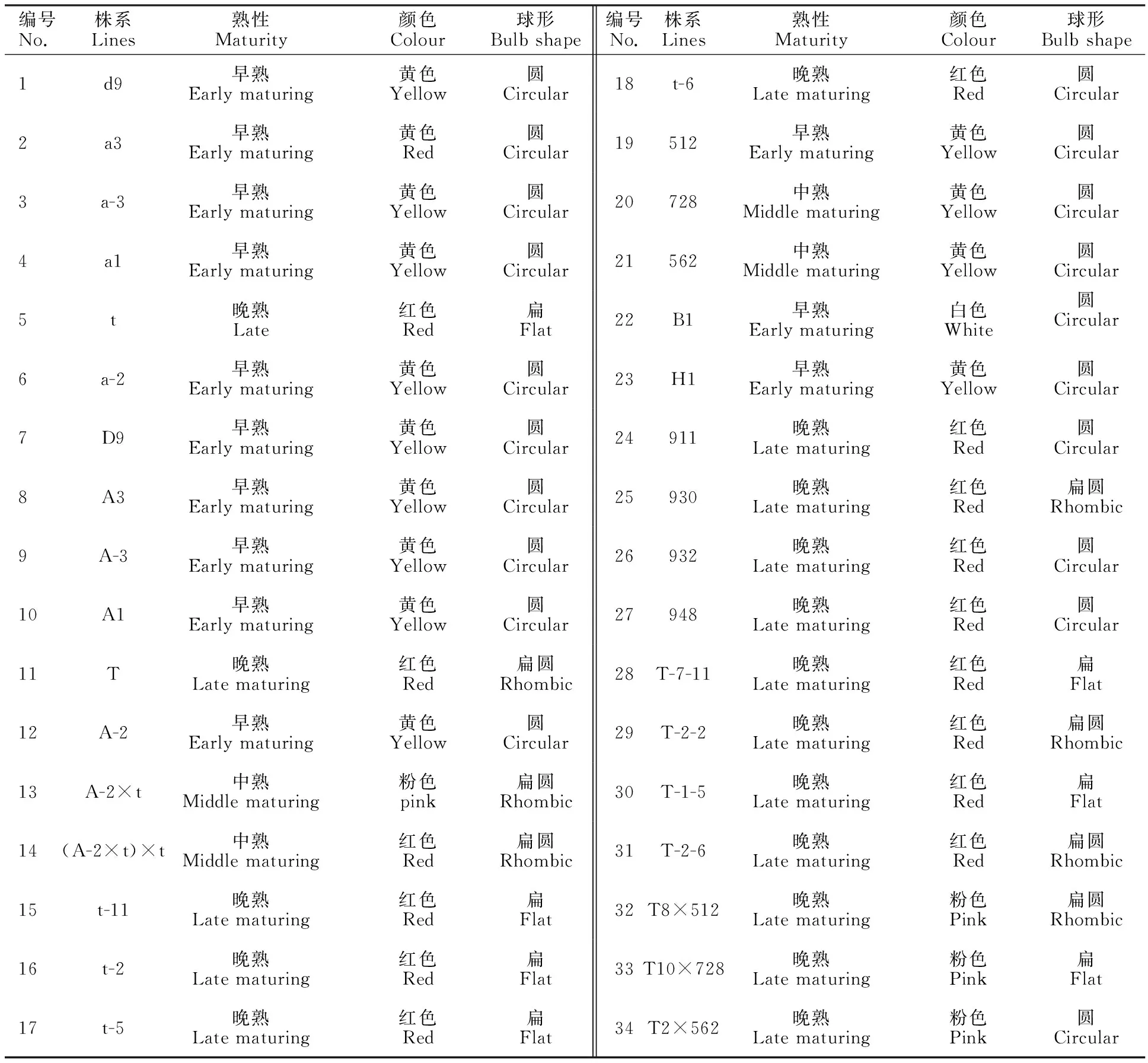
表1 供试洋葱材料编号、株系及特点Table 1 Code,lines and characteristics of tested onion materials

表2 洋葱6份不育材料来源及关系Table 2 Sources and relationship of six onion materials

表3 洋葱分子标记引物Table 3 Primer sequences of molecular markers in onion
2 结果与分析
2.1 洋葱细胞质雄性不育类型鉴定
2.1.1 洋葱不育株表型观察 当洋葱伞状花序上的花朵开裂达到40%~ 60%时,观察结果显示,正常可育株花朵中雄蕊饱满,花药和花粉囊开裂后能够正常弹出花粉(图1-A),且套袋自交后能够正常结籽。洋葱的不育株花朵开裂后,雄蕊较弱,花药在发育过程中渐渐萎缩干枯,无花粉粒弹出,套袋自交不结籽(图1-B)。对洋葱材料1~14 号进行表型观察和套袋后,1~6号洋葱花药有花粉弹出,套袋自交结籽,说明是可育株;7~14 号洋葱花药无花粉弹出,套袋自交未结籽,说明是不育株。

图1 洋葱可育株(A)和不育株(B)花序形态特性Fig.1 Morphological characteristics of fertile flowers (A) and sterile flowers (B)
2.1.2 洋葱不育类型鉴定 采用cob标记对1~14 号洋葱材料进行PCR扩增,cob(s)标记结果显示在414 bp大小处没有条带或很弱,初步说明1~14号都不是S型不育(图2-A),而cob(n)标记结果显示只在180 bp大小处条带很亮,说明1~14 号是可育或T型细胞质不育;用 orfA501标记引物只能在不育系中扩增出473 bp大小的条带,而可育系中没有或者很弱,结果显示1~6号没有条带或很弱,而7~14号扩增出473 bp,条带清晰,结果与Sato等[6]和Engelke等[7]报道一致,以上分子标记结果表明:洋葱材料1~6 号是可育株,7~14号为CMS-T不育株。 orf725[12]能够在可育株中扩增出1条833 bp的条带,T型中扩增出833 bp和628 bp 2条带,而在S型中只能扩增出1条628 bp的条带,结果显示本试验材料1~6号PCR扩增出1条带,说明是可育株,7~ 14号扩增出2条带,为CMS-T型不育株。Cpp1[15]标记是根据洋葱叶绿体基因组DNA核苷酸序列的差异开发出的标记,能够在可育株中扩增出1条264 bp的条带,T型中扩增出1条307 bp的条带,而在S型中扩增出1条254 bp的条带,由于3条带大小差异只有10~53 bp,PCR产物采用聚丙烯酰氨凝胶电泳(PAGE)检测,从图2-E中看出1条307 bp很亮的条带和1条264 bp亮度较弱的条带,不能有效区分本试验材料的不育类型。

株系1 Breeding lines 1.d9;2.a3;3.a-3;4.a1;5.t;6.a-2;7.D9;8.A3;9.A-3;10.A1;11.T;12.A-2;13.A-2×t;14.(A-2×t)×t;A.cob(s);B.cob(n);C. orfA501;D. orf725;E.Cpp1
图2不同引物对洋葱不育类型鉴定
Fig.2Identificationofonionsteriletypewithdifferentprimers
2.2 洋葱育性恢复基因型(Ms)验证
本课题组早期通过传统的杂交、回交在洋葱同一品种中筛选出稳定的保持系和不育系d9和D9、a1和A1,选用前人报道的DNF-566、RNS-357、jnurf13、PsaO、OPT和SKP1[23](MK886、MK898、MK620、MK628)共6个分子标记对1~34号材料进行验证,DNF-566标记能够在含有Ms位点中扩增出556 bp,RNS-357标记只在含有ms位点的洋葱中扩增出1条357 bp的条带,而含有Msms中能同时扩增出2条带。结果表明1~4、6~19、23、28、29、31和33号材料显示只有1条357 bp的条带,说明该材料基因型为msms;21、24和26号扩增出1条566 bp的条带,基因型为MsMs;22、25、30、32和34号扩增出2条带,其中25和32号2条带较弱,基因型为Msms;而5、20和27 3个材料2个标记均无条带(图3-A和图3-B)。
OPT标记能够在含有Ms位点中扩增出1条659 bp的条带,在含有ms位点中扩增出1条357 bp的条带,而含有Msms中能同时扩增出2条带,同样PsaO能够在含有Ms位点中扩增出1条490 bp的条带,在含有ms位点中扩增出1条437 bp的条带,而含有Msms中能扩增出2条带。结果显示OPT分子标记鉴定结果与PsaO分子标记互相矛盾,如11、14、22和24号,OPT标记鉴定为MsMs,而PsaO鉴定为Msms;16、27、30、31和34号,OPT标记鉴定为Msms,而PsaO鉴定为MsMs;2个分子标记鉴定结果相同的为5、15、20、28、29和33号,基因型为MsMs,23和25号为Msms,其他均不同(图3-C和图3-D),所以,OPT和PsaO标记不适合作为本试验材料的筛选Ms遗传位点。
为了能够准确鉴定洋葱可育株N、CMS-S和CMS-T 3种类型,Kim[21]报道的jnurf13分子标记可以用于区分3种类型,能够在含有Ms位点中扩增出1条241 bp的条带,在含有ms位点中扩增出1条229 bp的条带,而含有Msms中能扩增出2条带。Huo等[23]开发出的SKP1标记能够区分洋葱可育株N和CMS-S 2种类型,能够在含有Ms位点中扩增出1条898 bp的条带,在含有ms位点中扩增出1条628 bp的条带,而含有Msms中能扩增出2条带,但未对T型细胞质雄性不育类型进行报道。本试验材料中包括CMS-T类型,结果发现jnurf13和SKP1标记的结果完全一致,2个标记显示7~14号基因型为msms,且为不育株,与表型观察的结果相符合,20、21、24、26和27号5个材料基因型为MsMs,5、22、23、25、30、33和34号7个材料基因型为Msms,其他22个材料基因型为msms。对6个标记结果分析表明,DNF-566、RNS-357、jnurf13和SKP1中7~14号符合Ms遗传规律(表4),基因型为纯合msms不育株,初步分析表明:jnurf13和SKP1标记适合本材料筛选Ms遗传位点。

不同洋葱材料1 Different onion materials 1.d9;2.a3;3.a-3;4.a1;5.t;6.a-2;7.D9;8.A3;9.A-3;10.A1;11.T;12.A-2;13.A-2×t;14.(A-2×t)×t;15.t-11;16.t-2;17.t-5;18.t-6;19.512;20.728;21.562;22.B1;23.H1;24.911;25.930;26.932;27.948;28.T-7-11;29.T-2-2;30.T-1-5;31.T-2-6;32.T8×512;33.T10×728;34.T2×562。A.DNF-566;B.RNS-357;C.OPT;D.PsaO;E.jnurf13;F.SPK1
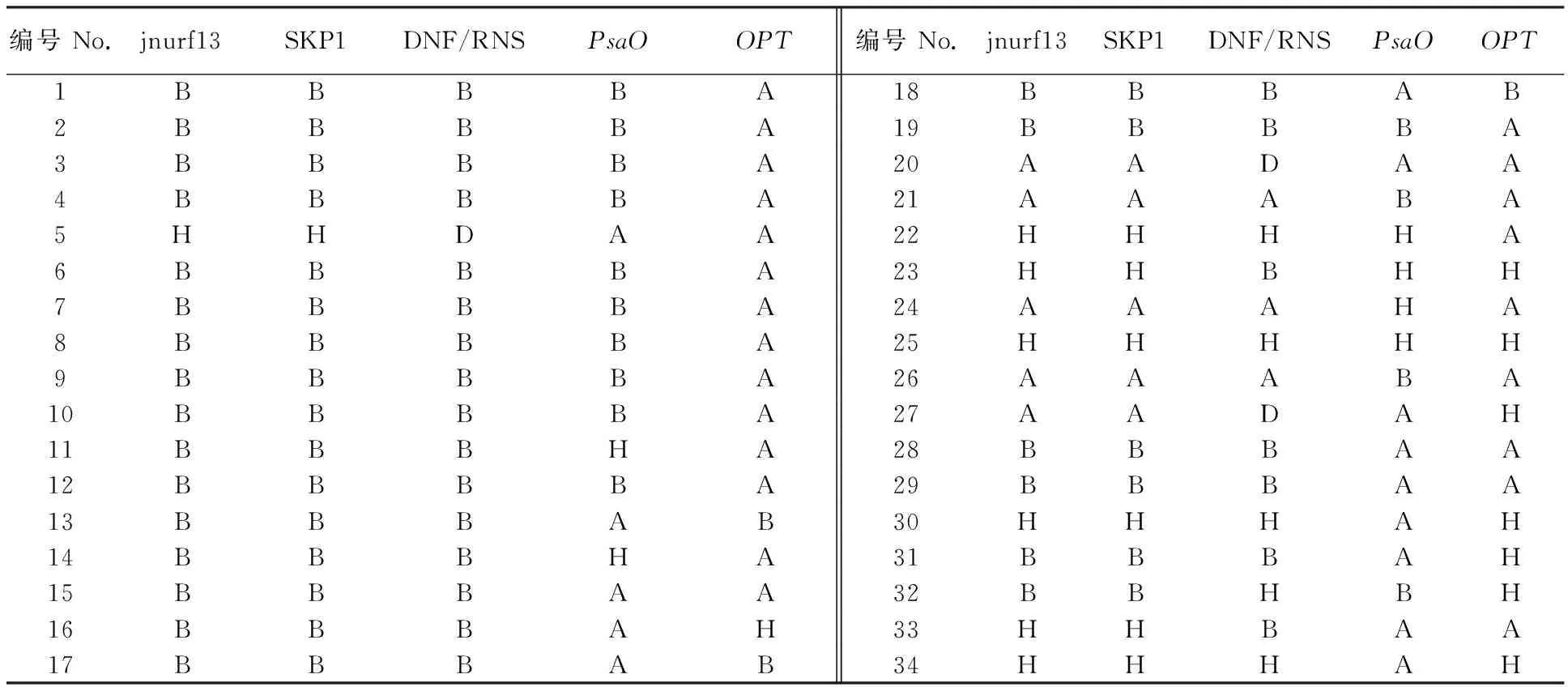
图3 洋葱Ms位点分子标记PCR验证Fig.3 Identification of simple PCR markers linked to the Ms locus in onion
注:A.显性纯合子(MsMs);H.杂合子(Msms);B.隐性纯合子(msms);D.显隐性纯合子或杂合子(MsMs,Msms,msms)。
Note:A.homozygous dominant (MsMs); H.heterozygous (Msms); B.homozygous recessive (msms);D.homozygous dominant,homozygous recessive,or heterozygous(MsMs,Msms,msms).
2.3 洋葱不同标记实用性比较
综合比较洋葱不育系鉴定相关分子标记cob(s)、cob(n)、 orfA501、 orf725和Cpp1,其中 orf725能够简单、快速的鉴定洋葱N、CMS-S和CMS-T 3种类型。与洋葱育性相关的6个分子标记jnurf13、AcSKP1、DNF-566、RNS-357、OPT和PsaO中(表5),其中jnurf13和AcSKP1能够准确鉴定本试验材料,包括CMS-T类型,AcSKP1采用琼脂糖凝胶电泳检测PCR产物,用时少、速度快,但其采用混合PCR扩增比jnurf13普通PCR扩增所用试剂成本高,jnurf13标记试剂成本较低,比较适合生产应用。

表5 洋葱不同标记实用性比较分析Table 5 Comparison and analysis of different marks of onion
3 讨 论
洋葱细胞质雄性不育系是解决洋葱杂交制种最有效的途径之一,在生产实践中,通过田间观察,易发现不育株,进一步通过分子标记来验证不育类型。自Jones等[4]和Berninger等[5]报道洋葱CMS-S和CMS-T,很多分子标记被用来区分不育类型,Cp-cytotype[25]、cob[6]、atp6和cob-type2[11]都只能区分CMS-S型,且有的PCR扩增后还需要进行后续酶切反应。orf501[7]只能区分不育和可育株,需要和上述鉴定S型的标记联合分析,才能将洋葱N、CMS-S和CMS-T区分开(图2-A、图2-B、图2-C), orf725[12]和Cpp1[15]可以用简单的PCR区分洋葱3种类型,本试验材料中用 orf725分子标记能够区分N和CMS-T类型,与cob和orf501结果一致,而Kim等[15]利用叶绿体基因组DNA开发的Cpp1标记能够区分3种类型,Cpp1标记分析130份来源于不同国家地区的栽培种,发现83份N型中含有54 bp的插入,与CMS-T完全一致,但无法区分N和CMS-T 2种类型。本试验中1~14号为N与CMS-T类型,PCR扩增条带大小完全一致,不能有效地进行区分,与Kim报道结果一致,其同时提出新的模型,即正常可育株N分为N-Type1,含有43 bp插入的为N-Type2,CMS-T分为CMS-T-Type1,含有1 bp 的SNP的插入类型为CMS-T-Type2。本试验中的1~6号按此分类对应是N-Type2,其他还需要进一步测序分析。虽然洋葱CMS-S型线粒体基因组DNA已发布,但还需要进一步研究分析SNP三者之间的差异。比较上述分子标记在随机材料中验证不育株的类型,结果表明 orf725分子标记最为简单、经济实用。
控制洋葱育性相关的基因研究,最早由Jones等[4]提出CMS-S由Ms基因控制,Schweisguth等[20]认为CMS-T由3个独立的细胞核基因共同控制,随着分子标记的广泛应用,ACms.1100[17]、jnurf05和jnurf17[26]、jnurf12和jnurf13[21]、OPT和PsaO[27]、WHR240[28]、PMS1[29]、DNF-566和RNS-357[19]、Ms[30]等与Ms位点相关标记被开发应用,12个分子标记中,只有jnurf12和jnurf13标记报道3种类型(N、CMS-S和CMS-T),其他10个标记只报道N、CMS-S 2种材料,其中ACms.1100和PMS1标记都以‘506L’为CMS材料,并未指出S或T型,而本试验不育材料都为CMS-T型。ACms.1100标记位于jnurf17和jnurf06标记之间, jnurf12标记没有与任何重组体的Ms基因座完美连接,比位于0.05 cM距离的jnurf05标记更接近于Ms基因座,然而,当用jnurf12标记分析不同来源的CMS-S时,标记基因型出现差异。jnurf13是位于jnurf12标记侧翼的约5.5 kb序列,依据有12 bp的插入,开发设计出jnurf13分子标记适合洋葱3种不同类型的鉴定[21]。jnurf13分子标记验证1~34号材料,7~14号为不育系,33和34号为杂交种,验证结果与表型完全一致,所以,本试验结果支持Kim提出的洋葱3种类型(N、CMS-S和CMS-T)是由Ms位点控制,同时,AcSKP1分子标记验证结果与jnurf13标记完全一致,首次报道AcSKP1标记适合本试验提供的细胞质雄性不育T型。DNF-566和RNS-357标记只能分别鉴定Ms和ms,2个标记结合分析,5、20和27号经重复PCR扩增试验后无条带(图3-A、图3-B),排除人为操作或试剂仪器等问题,其他31份材料中,只有23、32和33号3个材料结果与jnurf13、SKP1不一致,其他完全一致,尤其是7~14号CMS-T材料,3个标记结果完全一致,更进一步支持Kim的观点。而Huo等[27]报道的PsaO和OPT标记,基于‘506L’和‘H6’ 2个品种的F1和F2,而对于田间随机的CMS-S材料就不能完全适应,发现2个标记与其他3个标记结果有很多不同之处,所以,PsaO和OPT标记不适合作为本试验材料的Ms育性相关标记的验证。
另外,在洋葱中报道的标记ACms.1100,不能确定是连锁标记或功能标记[17];WHR240标记基于洋葱花蕾期cDNA序列,操作繁琐[28];Ms标记能够在田间直接应用筛选S型不育系和保持系[31],但CMS-T是否适合并未报道。而最新研究报道基于洋葱材料混合群体分离分析法(BSA)和转录组测序(RNA-seq)得到30个与Ms遗传位点相关候选基因,其中14个连锁不平衡,并对其cDNA全长测序,在可育株和不育株间进行氨基酸比对,发现有4个基因的氨基酸有差异,其中的 PMS1基因组DNA中有错配修复,是最好的候选基因标记,并对其进行相关验证[29],但是否适合本试验材料还需进一步验证。尽管jnurf13和AcSKP1标记适合本试验材料,但是从生产应用选育恢复基因型(MsMs)、保持系(msms)和不育系(msms)角度考量,AcSKP1分子标记采用的是混合PCR,试剂耗材成本相对高,而jnurf13分子标记适用于简单PCR验证,所以,jnurf13分子标记更适合在洋葱大田生产中应用。
参考文献Reference:
[1] 朱德蔚,王德槟,李锡香.中国作物及其野生近缘植物[M].蔬菜卷下册.北京:中国农业出版社,2008:947-950.
ZHU D W,WANG D B,LI X X.Chinese Crops and Their Wild Relatives [M].Vegetable Volume 2.Beijing: China Agricultural Publishing House,2008:947-950.
[2] MICHAELl J H.On the origin and distribution of normal cytoplasm of onion [J].GeneticResourcesandCropEvolution,1997,44 (4):307-313.
[3] FAO.Major food and agricultural commodities and producers- countries by commodity [DB/OL]//[2016-08-14].http://faostat.fao.org/site/567/DesktopDefault.aspx?PageID=567#ancor.
[4] JONES H A,EMSWELLER S L.A male-sterile onion [J].ProceedingsofAmericanSocirtyforHorticulturalScience,1936,34:582-585.
[5] BERNINGER E.Contribution à l’étude de la sterilité mle del’oignon (AlliumcepaL.) [J].AnnalsofAméliorPlantes,1965,15:183-199.
[6] SATO Y.PCR amplification of CMS-specific mitochondrial nucleotide sequences to identify cytoplasmic genotypes of onion (AlliumcepaL.) [J].TheoreticalandAppliedGenetics,1998,96:367-370.
[7] ENGELKE T,TEREFE D,TATLIOGLU T.A PCR-based marker system monitoring CMS-(S),CMS-(T) and (N)-cytoplasm in the onion (AlliumcepaL.) [J].TheoreticalandAppliedGenetics,2003,107:162-167.
[8] 陈沁滨,侯喜林,王建军,等.洋葱细胞质雄性不育系101A的分子鉴定[J].西北植物学报,2006,26(12):2430-2433.
CHEN Q B,HOU X L,WANG J J,etal.Molecular identification of cytoplasmic male sterility line 101A of onion [J].ActaBotaniceBoreali-occidentaliaSinica,2006,26(12):2430-2433.
[9] 吴海涛,王建军,侯喜林,等.两个洋葱雄性不育系细胞质类型的鉴定与分析[J].园艺学报,2009,36(5):723-726.
WU H T,WANG J J,HOU X L,etal.Identification and analysis of cytoplasmic male sterility in onion (AlliumcepaL.) origin from Chinese cultivars [J].ActaHorticulturaeSinica,2009,36(5):723-726.
[10] HAVEY M J.Identification of cytoplasms using the polymerase chain reaction to aid in the extraction of maintainer lines from open-pollinated populations of onion [J].TheoreticalandAppliedGenetics,1995,90:263-268.
[11] KIM S,YOON M.Comparison of mitochondrial and chloroplast genome segments from three onion (AlliumcepaL.) cytoplasm types and identification of a trans-splicing intron of cox2 [J].CurrentGenetics,2010,56: 177-188.
[12] KIM S,LEE E,CHO D Y,etal.Identification of a novel chimeric gene, orf725,and its use in development of a molecular marker for distinguishing three cytoplasm types in onion (AlliumcepaL.) [J].TheoreticalandAppliedGenetics,2009,118:433-441.
[13] KIM S.Identification of hypervariable chloroplast intergenic sequences in onion (AlliumcepaL.) and their use to analyse the origins of male-sterile onion cytotypes [J].JournalofHorticulturalScience&Biotechnology,2013,88:187-194.
[14] LILLY J W,HAVEY M J.Sequence analysis of a chloroplast intergenic spacer for phylogenetic estimates inAlliumsection Cepa and a PCR-based polymorphism detecting mixtures of male-fertile and male-sterile cytoplasmic onion [J].TheoreticalandAppliedGenetics,2001,102(1):78-82.
[15] KIM S,PARKB J Y,YANGB T.Comparative analysis of the complete chloroplast genome sequences of a normal male-fertile cytoplasm and two different cytoplasms conferring cytoplasmic male sterility in onion (AlliumcepaL.) [J].TheJournalofHorticulturalScienceandBiotechnology,2015,90 (4):459-468.
[16] KIM B,KIM K,YANG T J,etal.Completion of the mitochondrial genome sequence of onion (AlliumcepaL.) containing the CMS-S male-sterile cytoplasm and identification of an independent event of theccmFNgene split [J].CurrentGenetics,2016,62(4):873-885.
[17] BANG H,KIM S,PARK S O,etal.Development of a codominant CAPS marker linked to the Ms locus controlling fertility restoration in onion (AlliumcepaL.) [J].ScientiaHorticulturae,2013,153:42-49.
[18] MARTIN W J,MCCALLUM J,SHIGYO M,etal.Genetic mapping of expressed sequences in onion and in silico comparisons with rice show scant collinearity [J].MolecularGeneticsandGenomics,2005,274 (3):197-204.
[19] YANG Y Y,HUO Y M,MIAO J,etal.Identification of two SCAR markers co-segregated with the dominantMsand recessivemsalleles in onion (AlliumcepaL.) [J].Euphytica,2013,190 (2):267-277.
[20] SCHWEISGUTH B .Étude d’un nouveau type de stérilité male chez l’oignon,AlliumcepaL.[J].AnnualAméliorerPlantes,1973,23:221-233.
[21] KIM S.A codominant molecular marker in linkage disequilibrium with a restorer-of-fertility gene(Ms) and its application in reevaluation of inheritance of fertility restoration in onions [J].MolecularBreeding,2014,34:769-778.
[22] 惠林冲.莲藕NnDREB2A克隆,表达分析和功能验证[D].江苏扬州:扬州大学,2015:33-34.
HUI L CH.Molecular cloning,expression analysis and function identification of NnDREB2A from lotus root (NelumbonuciferaGaertn.) [D].Yangzhou Jiangsu:Yangzhou University,2015:33-34.
[23] HUO Y M,LIU B J,YANG Y Y,etal.AcSKP1,a multiplex PCR-based co-dominant marker in complete linkage disequilibrium with the male-fertility restoration(Ms) locus,and its application in open-pollinated populations of onion [J].Euphytica,2015,204 (3):711-722.
[24] BANG H,CHO D Y,YOO K S,etal.Development of simple PCR-based markers linked to theMslocus,a restorer-of-fertility gene in onion (AlliumcepaL.) [J].Euphytica,2011,179 (3):439-449.
[25] HAVEY M J.Identification of cytoplasms using the polymerase chain reaction to aid in the extraction of maintainer lines from open-pollinated populations of onion [J].TheoreticalandAppliedGenetics,1995,90:263-268.
[26] PARK J,BANG H,CHO D Y,etal.Construction of high-resolution linkage map of theMslocus,a restorer-of-fertility gene in onion (AlliumcepaL.) [J].Euphytica,2013,192(2):267-278.
[27] HUO Y,MIAO J,LIU B,etal.The expression of pectin methylesterase in onion flower buds is associated with the dominant male-fertility restoration allele [J].PlantBreeding,2012,131:211-216.
[28] KIM S,KIM C W,MINKYU P,etal.Identification of candidate genes associated with fertility restoration of cytoplasmic male-sterility in onion (AlliumcepaL.) using a combination of bulked segregant analysis and RNA-seq [J].TheoreticalandAppliedGenetics,2015,128 (11):2289-2299.
[29] GOKCE A F,HAVEY M J.Linkage equilibrium among tightly linked RFLPs and theMslocus in open-pollinated onion populations [J].JournaloftheAmericanSocietyforHorticulturalScience,2002,127 (6):944-946.
[30] GOKCE A F,MCCALLUM J,SATO Y,etal.Molecular tagging of theMslocus in onion [J].JournaloftheAmericanSocietyforHorticulturalScience,2002,127(4):576-582.

