长期施肥对酸性土壤氨氧化微生物群落的影响
王欣丽,朱 飞,姚 静,姜永见,王 芸,任丽英*
(1 临沂大学资源环境学院,山东临沂 276000;2 山东省水土保持与环境保育重点实验室,山东临沂 276000;3 临沂市农业局,山东临沂 276000)
硝化作用一直是氮转化过程中一个备受关注的环节,分为自养硝化和异样硝化[1]。自养硝化是指微生物以无机碳为碳源,将NH3氧化为亚硝酸盐或硝酸盐的过程。异样硝化指微生物以有机碳或有机氮化合物为碳源和能源进行的硝化作用。农田以自养硝化作用为主[2]。氨氧化作用是自养硝化作用的第一步,也是限速步骤[3]。氨氧化古菌 (ammoniaoxidizing archaea,AOA) 和氨氧化细菌 (ammoniaoxidizing bacteria,AOB) 是氨氧化作用的主要驱动者。许多研究表明,土壤pH[4–5]、NH3浓度[6]、土壤深度[7–8]、田间施肥管理[9–11]、土壤类型[12–13]等均可以改变氨氧化微生物的群落结构。
研究表明,长期施肥对土壤氨氧化微生物的丰度和群落结构产生了显著的影响[6,10,14–15]。pH值是影响土壤硝化作用最重要的环境因子之一,最能解释AOA和AOB的生态位分异[16]。pH值不仅影响着硝化作用底物的化学形态 (如NH3+ H+↔ NH4+) 和生物可利用性,也影响着AOA和AOB生长和功能活性。He等[9]和Chen等[17]在对长期定位施肥的酸性旱地和水稻土进行的研究发现,不同施肥处理间AOB的群落几乎没有变化,AOA的群落结构变化较大,认为AOA的群落组成对土壤理化性质的变化产生了比较明显的响应。Shen等[10]和Wang等[14]在中性及碱性土壤的研究发现,施肥尤其是施用高量氮肥,会明显改变AOB的群落组成并增加其丰度,而对AOA的影响不明显。Song等[18]在研究过量施氮肥导致土壤不断酸化时发现,AOA和AOB的群落结构均发生了变化,AOB的丰度受到土壤酸化的强烈影响。施用有机肥可提高酸性红壤的pH值至近中性,尚未见到AOB丰度和群落发生了怎样的变化的研究报道。为此,我们选择了肥料长期定位试验的土壤,研究其理化性质与氨氧化微生物丰度和群落结构的关系,进一步了解氨氧化微生物对酸性土壤环境变化的响应,以期为提高肥料的利用效率、保护红壤旱地土质量提供科学依据。
1 材料与方法
1.1 试验点概况和试验设计
土壤样品采集自江西省红壤站长期定位试验田(116°20′24″N,28°15′30″E)。该试验始建于1986年,位于江西省进贤县,属于典型的亚热带季风气候,年降水量为1537 mm,蒸发量1100~1200 mm,年平均气温17.5℃。试验田建立前是表层土壤已经被完全腐蚀的荒地,有机碳含量非常低,土壤由第四纪红粘土发育而来。轮作方式是早晚两季玉米,种植时间从4月上旬到11月,其他为冬闲时间。选择不施肥 (CK)、施氮肥 (N)、施氮磷钾肥(NPK) 和施有机肥 (OM) 4个处理,每个处理3次重复,每个小区面积为22.22 m2。供试肥料为尿素[N 120 kg/(hm2·a)]、过磷酸钙 [P2O560 kg/(hm2·a)]、氯化钾 [K2O 120 kg/(hm2·a)]和猪粪[2000 kg/(hm2·a)]。无机肥和有机肥等量分两季施用,每季玉米播前作为基肥一次性施用。
土壤样品于2013年4月在各处理小区采集0—15 cm耕层土壤,理化性质测定参考鲁如坤方法,土壤NH3的浓度根据化学方程式:25℃时pKa=9.25,基于含水量、铵的浓度和pH计算而得[20]。
1.2 土壤DNA提取和amoA基因丰度测定
不同处理土壤微生物DNA采用FastDNA® SPIN Kit for soil (MP Biomedicals, USA) 试剂盒提取。amoA基因丰度测定采用SYBR Premix Ex TaqTM Kit(TaKaRa) 试剂盒,在CFX96 Real-Time PCR System(Bio-rad, USA) 实时荧光定量PCR仪上扩增,分别以Arch-amoAF/Arch-amoAR[21]和amoA-1F/amoA-2R[22]为引物,定量分析古菌和细菌的amoA基因拷贝数。20 μL qPCR 反应体系包括 10 μL 2 × SYBR Premix Ex PCR buffer、上下游引物各0.5 μmol/L及1 μL DNA模板。基因扩增条件如下:95℃预变性3.0 min;95℃变性10 s;55℃退火30 s;72℃延伸30 s,83℃读板5 s,40个循环。采用融解曲线分析确定PCR产物的特异性,其程序为从65℃到95℃,每循环增加0.5℃后读取荧光值。以氨氧化细菌和古菌amoA基因的重组质粒作为标准DNA模板,经过10倍连续稀释,细菌和古菌amoA基因拷贝数标准曲线浓度范围分别为1.16 × 102~1.16 ×108和 1.15 × 102~1.15 × 108。PCR 的扩增效率为 96.8%,R2值为 0.995。
1.3 amoA的DGGE分析、克隆测序及系统发育分析
古菌和细菌amoA基因的PCR-DGGE引物分别为Arch-amoAF/Arch-amoAR-GC和amoA-1FGC/amoA-2R[10]。PCR反应体系包括5 μL含有20 mmol/L Mg2+的 10 × HotStar-Taqbuffer (TaKaRa)、0.5 μL (浓度为 20 μmol/L) 的上下游引物、2.5 U 的HotStar-Taqpolymerase (TaKaRa) 和 1 μL 的 DNA 模板,用无菌ddH2O补足至50 μL。反应程序如下:94℃预变性 5 min;94℃变性30 s,55℃退火45 s,72℃延伸45 s,35个循环;最后72℃延伸7 min。CK、N、NPK处理细菌amoA基因扩增产物浓度过低,不适于后续DGGE分析,只对OM处理AOB进行了DGGE分析。
PCR产物分别加入到6% (AOA) 和8% (AOB) 聚丙烯酰胺凝胶中,变性梯度分别为30%~65%(AOA) 和45%~75% (AOB)。在60℃、80 V条件下电泳17 h后,SYBR green I染色30 min,染色结果用凝胶成像系统 (Gel Doc XR+,Bio-Rad) 进行紫外成像。
1.4 数据分析
利用SPSS 16.0进行数据处理,Tukey单因素方差分析比较各处理之间的差异显著性,采用Pearson相关分析评估土壤特性和氨氧化微生物丰度之间的关系。DGGE图谱利用BioRad公司的Quantity One 4.4分析。利用MEGA 4.0软件邻接法(neighbor-joining) 构建氨氧化古菌和细菌的系统发育树,并用Bootstrap对进化树进行1000次可信度分析。采用CANOCO 4.5.1软件 (icrocomputer Power,Ithaca, USA) 分析AOA群落结构和土壤理化因子间的关系。
2 结果与分析
2.1 不同施肥处理土壤的理化性质
表1表明,不同施肥处理对酸性红壤的影响差异显著。与对照的pH 5.03相比,长期施用猪粪显著提高了土壤的pH值 (6.40),単施氮肥、氮磷钾化肥则显著降低至4.03、4.66, 加剧了土壤的酸化 (P<0.05)。与对照和化肥处理 (N、NPK) 相比,施用猪粪显著提高了土壤有机质碳、全氮、铵态氮含量;两个化肥处理间差异不显著,NPK处理与对照的差异也达到显著水平。硝态氮则表现出不同的规律,单施氮处理在测定年份的含量显著高于NPK和猪粪处理,后两者又显著高于对照。土壤NH3作为自养硝化作用的底物,其浓度在OM处理最高,并显著高于其他三个处理。
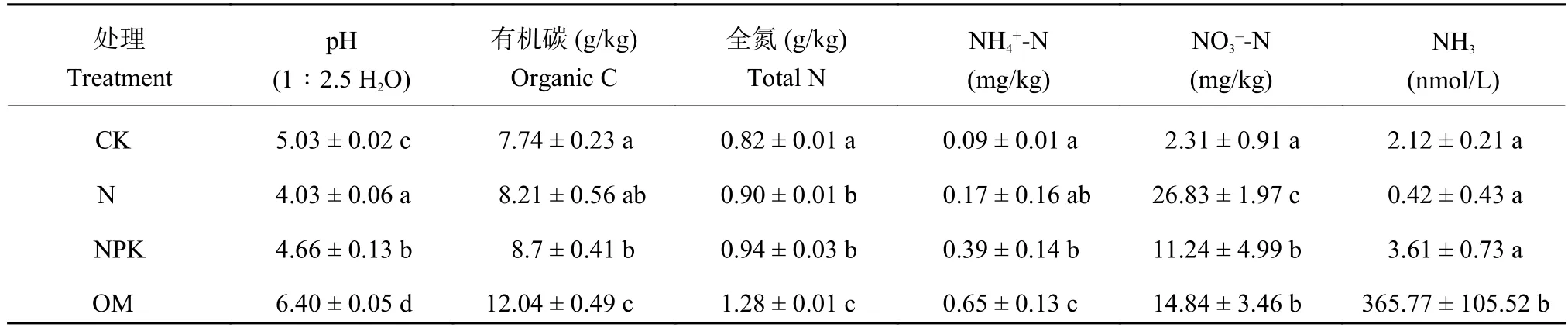
表 1 不同施肥处理土壤的理化性质Table 1 Soil characteristics under different fertilizer treatments
2.2 不同处理土壤氨氧化古菌 (AOA) 和氨氧化细菌 (AOB) 丰度及其与环境因子的相关性分析
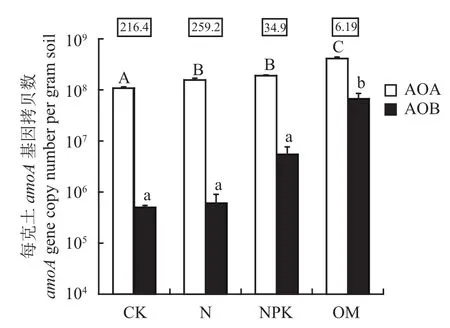
图 1 不同施肥处理土壤古菌和细菌amoA基因丰度Fig. 1 Abundance of archaeal and bacterial amoA genes in soils under different treatments
由图1可以看出,所有土壤AOA的amoA基因丰度显著高于AOB的amoA基因丰度。猪粪处理的土壤AOA和AOB丰度均显著高于其他三个处理,N和NPK处理的AOA丰度显著高于CK(P< 0.05),但AOB丰度三者间差异不显著。说明施肥促进了AOA的生长。猪粪处理土壤AOB丰度显著高于其他施肥处理土壤 (P< 0.05)。对照和氮肥处理 (N)AOA/AOB丰度比相当,NPK和猪粪处理降低了AOA/AOB丰度比。
Pearson相关分析表明 (表2),土壤AOA丰度与土壤有机质碳、全氮呈极显著正相关 (P< 0.01),与土壤铵态氮、NH3浓度呈显著正相关 (P< 0.05),与土壤pH、硝态氮关系不显著 (P> 0.05);AOB丰度与土壤NH3浓度呈极显著正相关 (P< 0.01),与有机质碳、全氮呈显著正相关关系 (P< 0.05),与土壤pH、铵态氮、硝态氮关系均不显著 (P> 0.05)。
2.3 不同施肥处理土壤氨氧化微生物群落组成及驱动因子分析
对不同施肥处理土壤的AOAamoA基因进行PCR-DGGE分析 (图2),结果显示,不同处理间土壤的古菌amoA基因DGGE条带具有显著差异,尤其是猪粪处理与其他三种处理土壤差别最大。CK和N、NPK处理共有条带较多,分别为Band A4、A7、A10、A11、A15。其中Band A4、A7是四种处理土壤的共有条带。Band A1、A5、A6、A8是猪粪处理土壤特有的条带。系统发育分析显示 (图3),Band A1~A8、Band A10、A11 和 Band A13~A15属于Group 1.1b类群,Band A9和Band A12属

表 2 不同施肥处理土壤的理化性质与AOA和AOB丰度的Pearson相关分析 (n = 12)Table 2 Pearson correlation analyses with soil characteristics and abundance of archaeal and bacterial amoA genes

图 2 不同施肥处理土壤古菌amoA的DGGE图谱Fig. 2 DGGE patterns of archaeal amoA gene under different fertilizer treatments

图 3 不同施肥处理土壤古菌amoA基因系统发育树Fig. 3 Phylogenetic tree based on archaeal amoA gene in DGGE bands under different fertilization treatments
于Group 1.1a-associated类群,因此大部分条带所代表的种类属于Group 1.1b类群。另外,对丰度较高的OM处理的AOBamoA做了PCR-DGGE分析 (图4a)。结果分析 (图4b) 表明,猪粪处理土壤AOB属于两个类群,分别是NitrosospiraCluster 9 (Band B1、B2) 和NitrosospiraCluster 3a (Band B3~B6)。因此,大部分条带所属类群是NitrosospiraCluster 3a。
以四种不同处理土壤DGGE条带作为响应变量,用土壤pH、有机质、总氮、铵态氮、硝态氮、土壤中NH3浓度作为解释变量进行冗余分析(RDA),如图5所示。在RDA分析中,向量的长度代表环境变量与AOA之间的相关程度;向量指向的方向相同表示正相关,反之表示负相关;向量两两之间的角度指示两个土壤性质之间的相关性。
RDA分析显示,OM处理与CK处理AOA群落结构在第一典型变量明显分开,说明长期施有机肥显著改变了AOA的群落结构,且环境因子分析显示土壤pH、有机质、总氮、铵态氮、土壤中NH3浓度是导致AOA群落变化的主要环境因子。在第二典型变量上,N和CK处理土壤AOA群落结构明显分开,且硝态氮含量可能是导致这一变化的主要因子。CK和NPK处理AOA群落结构没有明显分开。第一排序轴 (Axis 1) 解释了样本中85%的变异,第二排序轴 (Axis 2) 解释了样本8.9%的变异,两者合并解释了样本93.9%的总变异。

图 5 不同施肥处理土壤古菌amoA基因DGGE图谱对于各环境因子的冗余分析Fig. 5 Redundancy analysis between the DGGE pattern of archaeal amoA gene and soil characteristics under different fertilization treatments
3 讨论
本研究中酸性土壤施化肥后对AOA的丰度和群落结构均产生了一定的影响,施有机肥后对AOA和AOB的组成均产生了强烈影响。
NH3的氧化为缓慢生长的氨氧化微生物提供唯一的能量,在酸性土壤中,铵能够解离出的NH3浓度较低,有利于对NH3具有高亲和力的AOA生长[23];土壤pH值越低,NH4+能够解离出的NH3越少,对AOA的生长越有利。反之,NH3浓度越高对AOB的生长越有利。长期施有机肥显著促进了AOA的生长,前人的研究中也有类似的报道[17]。Chen等[17]通过分析,认为AOA的丰度与土壤总氮、有机质和NH4+-N含量呈正相关关系,肥料本身作为底物供应AOA生长比土壤pH值发挥了更重要的作用。本研究结果分析表明,施有机肥同时促进了AOA和AOB的生长,而化肥显著促进了AOA的生长,对AOB的生长没有显著影响。驱动土壤AOA和AOB丰度变化的因子分析表明,土壤总氮、有机质碳、NH4+-N和NH3含量均与AOA丰度呈显著正相关,与土壤pH相关不显著。从长期施肥处理的土壤的理化性状分析可以看出,施用有机肥提高土壤有机碳、全氮和铵态氮的效果显著高于化肥和对照,化肥处理的相对于对照虽有所增加,并没有全部达到显著水平。
本研究中长期施有机肥显著促进了AOB的生长,而化肥处理提高AOB生长的效果不显著。相关性分析表明,总氮、有机质和NH3均与AOB丰度呈显著正相关,有机肥提高土壤有机态碳、氮含量和NH3浓度的效果是促进AOB显著增加的原因。长期施无机肥导致土壤pH值进一步下降,过低的pH不适合AOB的生长[24],AOB在有机肥和化肥处理间的显著差异应该是土壤酸度作用的结果。而酸性土壤长期施有机肥之后,土壤pH值接近中性,更利于AOB的生长。这种丰度变化与土壤介质的NH3浓度有关[18,25]。但OM处理土壤中AOA丰度仍然有较大幅度的增长,可能的原因是土壤有机质含量的提高促进了 AOA 的异养生长[26–27]。
系统发育树显示,除了Band A9和Band A12属于Group 1.1a-associated类群,其他均属于Group 1.1b类群。Band A5、A6、A8是OM中特有的优势条带,说明OM处理近中性pH值环境更适合这三个种生存;Band A11是CK和N、NPK中最亮的条带,它所代表的种在低pH值的环境中更容易生存。长期施肥尤其是有机肥显著改变了AOA的群落结构,可能的原因是AOA可以进行自养、兼养和异养生长[26–27]。对AOB丰度较高的OM处理土壤基于amoA基因做了PCR-DGGE分析,Band B3、B4、B5是优势条带,属于NitrosospiraCluster 3a类群,说明OM处理的近中性红壤中适合这三个种的生存,这与以往中性农田土中NitrosospiraCluster 3占主导的观点[28]相同。
4 结论
基于amoA基因利用qPCR和PCR-DGGE技术,对27年不同施肥处理酸性玉米地土壤的氨氧化微生物群落进行了分析,发现长期定位施肥,尤其是长期施加有机肥,改变了酸性土壤氨氧化微生物的丰度和群落结构。
参 考 文 献:
[ 1 ]Zhang J B, Sun W J, Zhong W H, Cai Z C. The substrate is an important factor in controlling the significance of heterotrophic nitrification in acidic forest soils[J]. Soil Biology and Biochemistry,2014, 76: 143–148.
[ 2 ]王敬, 程谊, 蔡祖聪, 等. 长期施肥对农田土壤氮素关键转化过程的影响[J]. 土壤学报, 2016, 53(2): 292–304.
Wang J, Cheng Y, Cai Z C,et al. Effects of long-term fertilization on key processes of soil nitrogen cycling in agricultural soil: a review[J].Acta Pedologica Sinica, 2016, 53(2): 292–304.
[ 3 ]Kowalchuk G A, Stephen J R. Ammonia-oxidizing bacteria: a model for molecular microbial ecology[J]. Annual Review of Microbiology,2001, 55: 485–529.
[ 4 ]Nicol G W, Leininger S, Schleper C,et al. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria[J]. Environmental Microbiology,2008, 10: 2966–2978.
[ 5 ]Li H, Weng B S, Huang F Y,et al. pH regulates ammonia-oxidizing bacteria and archaea in paddy soils in Southern China[J]. Applied Microbiology and Biotechnology, 2015, 99(14): 6113–6123.
[ 6 ]Verhamme D T, Prosser J I, Nicol G W. Ammonia concentration determines differential growth of ammonia-oxidising archaea and bacteria in soil microcosms[J]. The ISME Journal, 2011, 5(6):1067–1071.
[ 7 ]Onodera Y, Nakagawa T, Takahashi R,et al. Seasonal change in vertical distribution of ammonia-oxidizing archaea and bacteria and their nitrification in temperate forest soil[J]. Microbes and Environments, 2010, 25: 28–35.
[ 8 ]卢圣鄂, 王蓥燕, 陈勇, 等. 不同施肥制度对石灰性紫色水稻土中氨氧化古菌群落结构的影响[J]. 生态学报, 2016, 36(21): 6919–6927.Lu S E, Wang Y Y, Chen Y,et al. Impact of different long-term fertilization systems on ammonia oxidation Archaea communitystructures in Calcareous Purple Paddy soil[J]. Acta Ecologica Sinica, 2016, 36(21): 6919–6927.
[ 9 ]He J Z, Shen J P, Zhang L M,et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under longterm fertilization practices[J]. Environmental Microbiology, 2007, 9:2364–2374.
[10]Shen J P, Zhang L M, Zhu Y G,et al. Abundance and composition of ammonia-oxidizing bacteria and ammonia oxidizing archaea communities of an alkaline sandy loam[J]. Environmental Microbiology, 2008, 10(6): 1601–1611.
[11]Xue C, Zhang X, Zhu C,et al. Quantitative and compositional responses of ammonia–oxidizing archaea and bacteria to long-term field fertilization [J]. Scientific Reports, 2016, doi: 10.1038/srep 28981.
[12]Chen X, Zhang L M, Shen J P,et al. Soil type determines the abundance and community structure of ammonia-oxidizing bacteria and archaea in flooded paddy soils[J]. Journal of Soils and Sediments,2010, 10: 1510–1516.
[13]Shen J P, Xu Z H, He J Z. Frontiers in the microbial processes of ammonia oxidation in soils and sediments[J]. Journal of Soils and Sediments, 2014, 14: 1023–1029.
[14]Wang Y N, Ke X B, Wu L Q,et al. Community composition of ammonia-oxidizing bacteria and archaea in rice field soil as affected by nitrogen fertilization[J]. Systematic and Applied Microbiology,2009, 32(1): 27–36.
[15]杨亚东, 张明才, 胡君蔚, 等. 施氮肥对华北平原土壤氨氧化细菌和古菌数量及群落结构的影响[J]. 生态学报, 2017, 37(11):3636–3646.Yang Y D, Zhang M C, Hu J W,et al. Effects of nitrogen fertilizer application on abundance and community structure of ammonia oxidizing bacteria and archaea in a North China agricultural soil[J].Acta Ecologica Sinica, 2017, 37(11): 3636–3646.
[16]Hu H W, Zhang L M, Dai Y,et al. pH-dependent distribution of soil ammonia oxidizers across a large geographical scale as revealed by high-throughput pyrosequencing[J]. Journal of Soils and Sediments,2013, 13(8): 1439–1449.
[17]Chen X, Zhang L M, Shen J P,et al. Abundance and community structure of ammonia-oxidizing archaea and bacteria in an acid paddy soil[J]. Biology and Fertility of Soils, 2011, 47(3): 323–331.
[18]Song H, Che Z, Cao W,et al. Changing roles of ammonia-oxidizing bacteria and archaea in a continuously acidifying soil caused by overfertilization with nitrogen[J]. Environmental Science and Pollution Research, 2016, 23(12): 11964–11974.
[19]鲁如坤. 土壤农业化学分析方法[M]. 北京: 中国农业科技出版社,2000.Lu R K. Methods of soil agricultural chemical analysis [M]. Beijing:China Agriculture Science and Technology Press, 2000.
[20]Lu L, Han W, Zhang J B,et al. Nitrification of archaeal ammonia oxidizers in acid soils is supported by hydrolysis of urea[J]. The ISME Journal, 2012, 6(10): 1978–1984.
[21]Francis C A, Roberts K J, Beman J M,et al. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102: 14683–14688.
[22]Rotthauwe J, Witzel K, Liesack W. The ammonia monooxygenase structural geneamoAas a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704–4712.
[23]Martens-Habbena W, Berube P M, Urakawa H,et al. Ammonia oxidation kinetics determine niche separation of nitrifying Archaea and Bacteria[J]. Nature, 2009, 461: 976–979.
[24]Prosser J I. Autotrophic nitrification in bacteria[J]. Advances in Microbial Physiology, 1989, 30: 125–181.
[25]He J Z, Hu H W, Zhang L M. Current insights into the autotrophic thaumarchaeal ammonia oxidation in acidic soils[J]. Soil Biology and Biochemistry, 2012, 55: 146–154.
[26]Jia Z J, Conrad R. Bacteria rather than archaea dominate microbial ammonia oxidation in an agricultural soil[J]. Environmental Microbiology, 2009, 11: 1658–1671.
[27]Wessén E, Nyberg K, Jansson J K,et al. Responses of bacterial and archaeal ammonia oxidizers to soil organic and fertilizer amendments under long-term management[J]. Applied Soil Ecology, 2010, 45(3):193–200.
[28]Stephen J R, McCaig A E, Smith Z,et al. Molecular diversity of soil and marine 16S rRNA gene sequences related to beta-subgroup ammonia-oxidizing bacteria[J]. Applied and Environmental Microbiology, 1996, 62(11): 4147–4154.

