红三叶遗传图谱构建及抗白粉病基因QTL定位
蒲小剑,田久胜,田新会,杜文华
(甘肃农业大学草业学院,草业生态系统教育部重点实验室,中-美草地畜牧业可持续发展研究中心, 甘肃 兰州730070)
红三叶(Trifoliumpratense)为豆科三叶草属中最重要的一种牧草,其青干草中富含异黄酮,具有调节人体新陈代谢,抗肿瘤、心血管疾病、骨质疏松症、早老年性痴呆症、妇女更年期综合征、机体免疫力下降等作用[1],在预防骨质疏松症、更年期综合征及治疗胃溃疡、胃癌、乳腺癌、肠癌和前列腺癌等方面具有特殊功效,是保健食品业和医药生产中提取异黄酮的重要原料[2-3]。
岷山红三叶(T.pratensecv. Minshan)是1944年从美国引进,经过多年栽培驯化,选育出的适宜于甘肃省高寒阴湿区种植的特有牧草种质资源。其青干草中粗蛋白含量为19.75%,异黄酮含量为4.796 μg·mg-1,是大豆(Glycinemax)的8~10倍[2]。近年来,其种植面积不断扩大[4]。但在长期种植过程中,由于品种更新缓慢、退化严重,重度感染白粉病(感病率达70%),对单位面积草产量和异黄酮提取量影响极大[5]。因此,培育适宜于该区种植的抗白粉病红三叶新品种,成为农牧业生产中亟待解决的问题。
杂交育种、诱变育种和杂种优势利用等常规育种技术均能达到培育新品种的目的。转基因育种技术能有效弥补传统育种的不足,打破物种间的界限,使品种选育快速、高效、定向[6],能够有针对性地改良和选育作物新品种[7]。遗传图谱构建和相关性状基因定位是基因克隆和转基因育种的前提[8]。
分子遗传图谱构建作图群体主要分临时性和永久性两大类,常用的标记有RFLP、RAPD、AFLP、SSR和SRAP等。构建某一植物的图谱时,具体选用何种标记,要依据具体研究情况决定[8]。国内外学者利用不同标记构建了不同植物的遗传图谱,就是同一种植物,也可以利用不同分子标记构建遗传图谱[9-12]。AFLP标记是一项结合了RFLP和PCR技术特点的全基因组分子标记。它具有RFLP技术的可靠性和PCR技术的高效性,只需要极少量DNA材料,而且不需要预知基因组DNA序列特征,不需要Southern杂交,试验结果稳定可靠,可以快速获得大量信息,而且再现性高,重复性好,因而非常适合于品种指纹图谱绘制,遗传连锁图谱构建及遗传多样性研究,很快便成为植物分子遗传图谱构建的主要标记[13]。在一些多态性极少,而且待测样品较少的情况下,用AFLP分析能达到满意结果。徐志等[9]利用AFLP标记构建了小麦(Triticumaestivum)白粉菌遗传连锁图谱。其他学者也先后利用AFLP标记构建了水果[14]、蔬菜[15-17]、棉花[18]、大豆[19]等作物的遗传图谱。AFLP也是高密度基因图谱绘制或对某个基因所在区域进行精细定位较为理想的方法[9]。AFLP是目前农牧业生产中构建遗传图谱和基因定位应用最多的分子标记[19]。
岷山红三叶为异花授粉植物,品种的遗传背景不一致,群体内存在遗传多样性。白粉病抗性由单基因(质量性状遗传位点)或多基因(数量性状遗传位点)控制[5]。目前国内外在植物中共检测到白粉病QTLs44个,这些抗白粉病QTLs分布在A、B和D染色体组里,在同源群间,第5群中检测到QTL最多[11]。对抗白粉病基因的定位主要集中在小麦、黄瓜(Cucumissativus)、甜瓜(Cucumismelo)和草莓(Fragaria×ananassa)等植物上,小麦抗白粉病基因定位方面的研究尤为突出[12]。国际小麦基因命名委员会正式命名了来自39个位点的55个抗白粉病基因(Pm1~Pm39),其中30个已找到连锁的分子标记[11]。这些众多的分子标记类型中,以SSR、AFLP和SRAP应用最为广泛。刘联正等[11]利用SSR标记将小麦抗白粉病基因PmWP6192定位于2AL染色体上,刘素兰等[12]利用SSR技术将小麦新种质N9628-2的抗白粉病基因定位在6AS上,李春鑫等[20]利用SSR和SRAP标记将抗小麦白粉病新基因PmHNK定位于3BL,Alfandi等[21]利用AFLP标记将黄瓜中的抗白粉病基因定位在5BL上,Fernando等[22]利用SSR和AFLP标记将甜瓜的抗病基因定位在2AL染色体上。目前国内外利用AFLP标记构建红三叶遗传图谱及抗白粉病QTL定位方面的报道较少。本研究拟以红三叶抗、感白粉病材料杂交得到的F1代为作图群体,利用AFLP标记构建红三叶高密度遗传图谱,并对抗白粉病QTL进行定位,为抗白粉病基因克隆和转基因等分子辅助育种奠定基础。
1 材料与方法
1.1 试验地概况
本试验田间部分在甘肃省临洮县临洮农校农场进行,E 103°87′,N 35°37′。海拔1892 m,降水量562 mm,无霜期80~190 d,年平均气温7.0 ℃(最高气温34.6 ℃,最低气温-29.5 ℃),土壤为黑麻土,有灌溉条件,试验地肥力均匀。前茬作物为玉米(Zeamays)。
1.2 试验材料鉴定及接种
以重度感染白粉病岷山红三叶为父本(male parent,M)、甘肃农业大学培育的高抗白粉病红三叶新品系“甘农PR1”(据任继周[23]病害调查与研究方法分别确定为重感和高抗,数据未发表,其中新品系“甘农RP1”为育种用试验材料)为母本(female parent,F)杂交形成F1代群体以及亲本。2015年3月下旬将杂交F1代种子点播,株距20 cm,行距30 cm,播种深度1~2 cm,得到F1代群体。利用项目组前期研究筛选出的11对AFLP引物对提取自红三叶亲本及杂交F1代群体的基因组DNA进行电泳(JY-ECP3000高压电泳仪-北京君意东方电泳设备有限公司;JY-SPFT水平电泳槽-北京君意东方电泳设备有限公司)。根据电泳图谱条带特征,淘汰假杂种,保留真杂种,用于构建遗传图谱。参照文献[5]的白粉菌接种方法和侵染型记载标准,于2015年6月中旬为F1代群体和父母本苗期人工接种白粉菌,待岷山红三叶6月底充分发病后,记载F1代群体中每个单株的侵染型。从F1代群体中分别随机选取15株抗病和感病单株,采用改良CTAB(cetyl trimethyl ammonium bromide)法[9]分别提取DNA(TGL16M型台式高速冷冻离心机-湖南凯达科学仪器有限公司;水浴锅),并用1.0%琼脂糖凝胶电泳和紫外分光光度计检测其浓度和纯度(PCR扩增仪-BioRad-MyCycler;紫外凝胶成像仪-UVP GDS-8000;超微量紫外分光光度计-Quawell-Q5000;相机),OD260/280为1.8±0.1,符合试验要求。提纯合格后的DNA样置于4 °C冰箱内保存备用。5次重复进行实验。
1.3 试剂
MseI、EcoRI和T4DNA连接酶均购自Thermo Scientific;2×PCR Master Mix,DNA Marker均购自BBI Life Sciences;引物及接头(表1)由上海生工生物工程(上海)有限公司人工合成;其他试剂均为国药分析纯。

表1 接头和引物序列Table 1 The sequence of adapter and primer
1.4 AFLP分析
参照Vos等[24]的方法,略作调整。分步法酶切体系:200~300 ng DNA 9.0 μL、10×Tag Buffer 3 μL、8 UEcoRI,6 UMseI,ddH2O补齐20 μL。37 ℃水浴5 h、65 ℃水浴15 min,-20 ℃保存。连接体系:单链人工接头加ddH2O稀释至50 μmol·L-1,经PCR反应合成双链,加入3 U T4DNA连接酶,于22 ℃下连接过夜。接头制作:将合成的人工单链稀释至50 μmol·L-1,经PCR反应94 ℃,5 min;65 ℃,10 min;37 ℃,10 min;25 ℃,10 min;4 ℃保存。合成双链接头,-20 ℃保存备用。预扩增体系:连接产物模板DNA 4 μL、0.8 μLEcoRI,0.6 μLMseI预扩增引物,15 μL 2×PCR Master Mix,然后ddH2O补平,离心混匀进行PCR反应。PCR反应程序:94 ℃ 2 min进行变性;94 ℃ 30 s,56 ℃ 1 min,72 ℃ 1 min,进行扩增,共35个循环;72 ℃ 10 min进行延伸,-20 ℃保存备用。选择性扩增体系:2.0 μL稀释30倍的预扩增产物,Primer E,0.8 μL;Primer M,1.0 μL;2×PCR Master Mix,10 μL;ddH2O,6.2 μL。PCR反应程序:94 ℃ 2 min进行变性;65 ℃ 30 s,72 ℃ 1 min,每个循环开始温度下降0.7 ℃,共扩增13个循环;94 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,共扩增23个循环;-20 ℃保存备用。
1.5 扩增产物检测
制备4%变性聚丙烯酰胺凝胶,待胶聚合后恒电压1800 V预电泳30 min。将PCR扩增产物(2 μL)与上样Buffer混合均匀,经90 ℃变性3 min后,置于4 ℃保存等待点样。点样后1500 V恒功率电泳1.5 h左右。采用Carlos银染方法[25],胶版微干后拍照、并统计数据。
1.6 统计与分析
以30个F1群体真杂种个体及双亲共32个单株的基因组DNA为PCR模板,用11对AFLP引物进行PCR扩增。读带的原则:记录清晰、明确的条带;模糊、不易辨认的条带不做纪录。同一位点有带记为“H”,无带记为“A”,缺失用“-”表示。AFLP标记名称由引物组合编号和多态性条带序号组合而成。由同1对引物组合扩增出多个多态性条带,分别记为引物组合名-01、-02,依此类推如E3M4-01,E3M7-05等。最后按软件JoinMap 4.0的作图格式要求对获得的标记基因型数据进行整理和编辑。
利用JoinMap 4.0软件[26]构建遗传图谱的过程:File—New Project—Load Data,分别将基因型数据导入JoinMap 4.0作图软件;然后运行Data—Locus Genot. Freq.—Calculate Dudo,对标记进行卡方检验,剔除偏分离标记;接着运行LOD Groupings (tree)窗口下的Calculate命令,对各标记间的连锁关系进行分组,图距也由Calculate命令计算出来;然后根据计算后不同的分组,运行Population—Create Groups using the Groupings Tree,选择适宜的LOD值(2~10)在Population菜单下分别构建各个基因连锁群;然后运行Group—Calculate Map命令对每个连锁群创建连锁图;最后用Export命令导出结果。
利用本试验构建的红三叶AFLP图谱和MAPQTL 6.0软件[27]对抗白粉病有关QTL进行定位分析,并计算各位点性状的育种值和贡献率。
2 结果与分析
2.1 红三叶遗传图谱构建
基于149个AFLP标记构建的红三叶基因连锁图包含7个连锁群(LG1~LG7)(图1,表2)。所获得连锁图谱总距离为640.5 cM,标记间平均距离4.3 cM。各连锁群的距离为55.2~140.6 cM,标记数为10~33,标记间的距离为1.8~9.4 cM。其中,连锁群LG1的遗传距离(140.6 cM)最大,包含15个AFLP标记,标记间平均距离为9.4 cM;LG2的遗传距离为70.6 cM,包含19个AFLP标记,标记间平均距离为3.7 cM;LG3的遗传距离为110.3 cM,包含23个AFLP标记,标记间平均距离为4.8 cM;LG4的遗传距离(55.2 cM)是所有连锁群中最小的,但包含31个AFLP标记,标记间平均距离为1.8 cM;LG5的遗传距离为83.2 cM,包含10个AFLP标记,标记间平均距离为8.3 cM;LG6的遗传距离为122.9 cM,包含的AFLP标记最多(33个),标记间平均距离为3.7 cM;LG7的遗传距离为57.7 cM,包含18个AFLP标记,标记间平均距离为3.2 cM。
2.2 红三叶抗白粉病基因QTL分析
利用MAPQTL 6.0对红三叶抗白粉病QTL进行定位分析(图1)。LOD≥2.0时,检测到5个与红三叶抗白粉病性状有关的QTL,分别定位于遗传图谱的LG4和LG5连锁群(图1中阴影部分)。

图1 红三叶遗传图谱及抗白粉病QTL定位Fig.1 Linkage map and QTL analysis on powdery mildew resistance in red clover 图中LG4和LG5的阴影为红三叶抗白粉病QTL位点。Shadows on LG4 and LG5 mean QTLs resistant to the powdery mildew in red clover.
5个与红三叶抗白粉病相关的QTL暂命名qrp-1,qrp-2,qrp-3,qrp-4和qrp-5,其中qrp-1、qrp-2、qrp-3和qrp-4位于LG4连锁群上,qrp-5位于LG5连锁群上。5个QTL位点的加性效应均为负值,对抗白粉病表现增效加性效应,其中qrp-1最小(-2.22),qrp-4最大(-1.11)(表3)。在5个QTL位点中,qrp-1的遗传距离为4.6 cM,包含的AFLP标记数最多(3个),其LOD值(10.45)和对红三叶白粉病抗性的贡献率(90%)最大,为主效基因;qrp-2的LOD值(3.28)次之,包含2个AFLP标记,其加性效应和贡献率分别为-1.48和39%;qrp-5的遗传距离最大(6.1 cM),但包含的位点数最少(0),LOD值(2.44)和贡献率(31%)相对较低,其也是唯一一个不在LG4连锁群上的QTL位点。
3 讨论
3.1 作图群体的选择
作图群体的选择是影响遗传图谱构建和图谱质量好坏的主要因素之一,常见遗传连锁图谱作图群体有临时性作图群体和永久性分离群体,临时性作图群体包括杂交F1代、F2代和F4代群体[28-30]。永久性分离群体包括近等基因系群体、双单倍体群体、重组自交系群体、回交自交系群体[31-34]。如果植物的亲本基因型杂合度较高,则F1分离作图群体为适宜作图群体[28]。包括红三叶在内的大多数多年生牧草为异花授粉植物,亲本高度杂合,自交不亲和,选育高纯度自交系相对较难,构建回交群体有一定阻碍[28]。杂交F1代是大多数牧草分子遗传连锁图谱构建的常用作图群体,已应用于紫花苜蓿(Medicagosativa)[35]、红三叶[36]、鸭茅(Dactylisglomerata)[37]、黑麦草(Loliumperenne)[38]和高羊茅(Festucaelata)[39]等牧草。
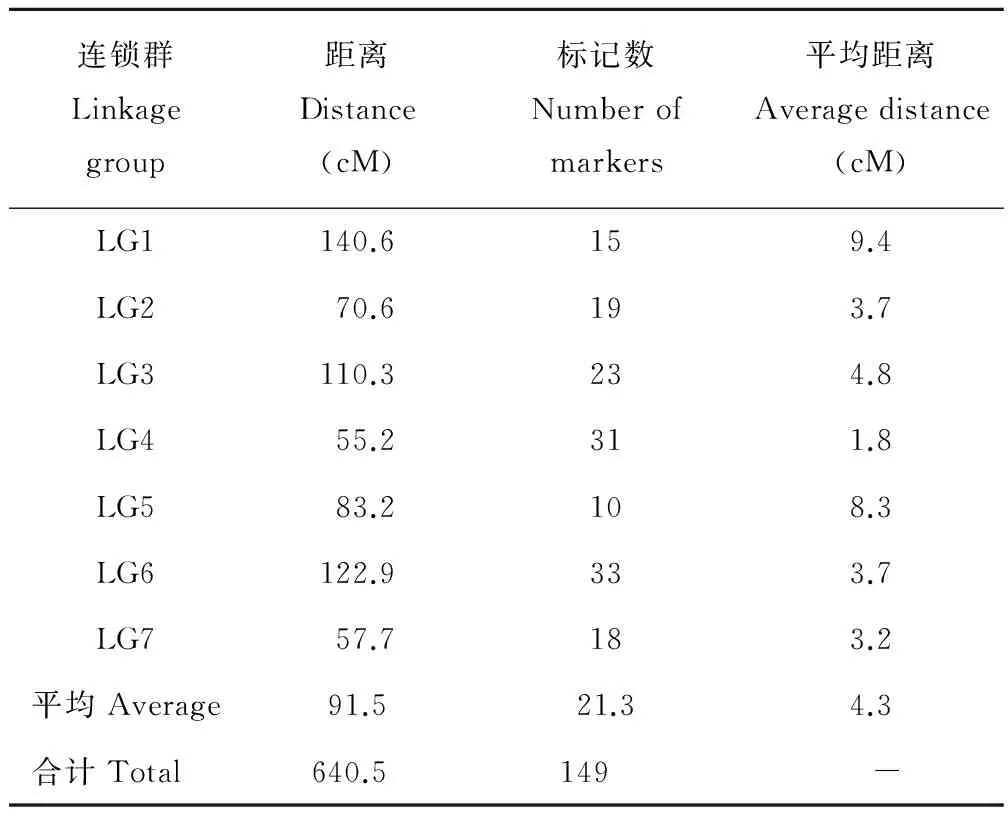
表2 红三叶遗传连锁图谱中各连锁群的图距和标记分布Table 2 Genetic distance and distribution of markers in the linkage groups of red clover
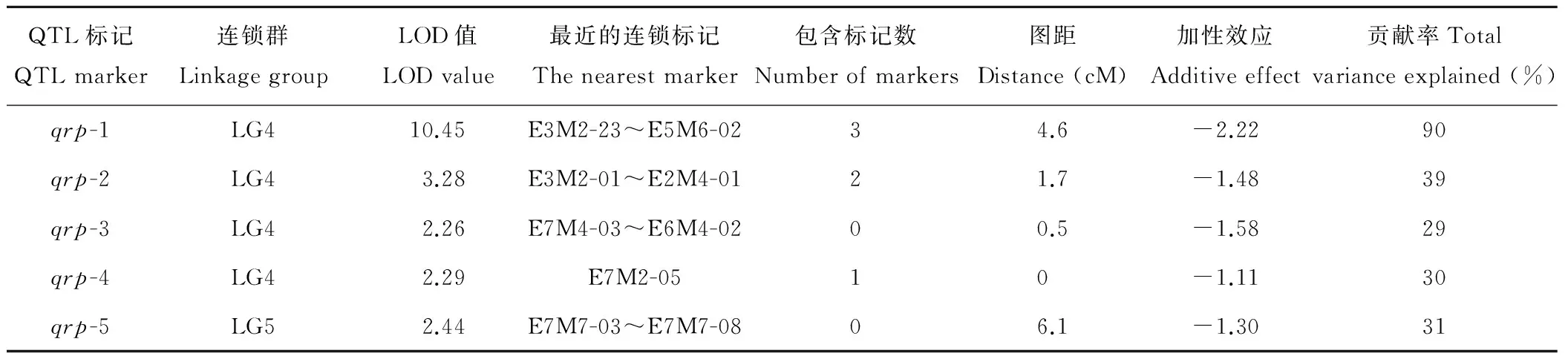
表3 红三叶抗白粉病性状的QTL分析Table 3 QTL analysis of powdery mildew resistance in red clover
3.2 作图标记的选择
DNA分子标记直接体现植物DNA水平上的遗传多样性,因为其易操作、不易受环境影响和可重复性强等优点,在牧草遗传图谱构建研究中应用广泛。现阶段,SRAP、AFLP、SSR、RFLP和RAPD等分子标记是遗传作图的主要方法。遗传图谱构建的关键在于选取适宜的标记系统。实践证明,AFLP的多态性条带在图谱上定位的比例非常高,能够满足构建遗传连锁图谱要求[40-42]。荷兰Keygene公司和PE公司共同构建了拟南芥(Arabidopsisthaliana)遗传连锁图谱,共有700多个AFLP位点,拟南芥的全部基因组在图谱中体现的非常好[43-44]。Jaime等[45]利用AFLP结合27个形态学性状,对马铃薯(Solanumtuberosum)多态性水平及其亲缘关系进行了研究。
3.3 遗传图谱构建
牧草的草产量、品质和非生物抗性等性状往往具有连续的表型变异,并且由多个数量性状位点调控[46-48]。前人主要利用回交法,对具有优良性状的亲本进行改良,其育种时间较长,效率十分低[46]。分子遗传连锁图谱不仅能够在分子层面分析,而且可以对主要农艺性状的基因进行标记、定位和基因克隆,并可以将目标基因转入到特定植物中[48]。利用转基因等分子标记辅助育种,可加快牧草育种进程[49]。
一般而言,连锁框架图要求标记间平均间隔要小于20 cM。如果标记间的平均距离为10~20 cM或更小时,就可以进行主基因定位[50]。本试验由149个AFLP标记构建的遗传图谱,全长640.5 cM,标记间平均距离为4.3 cM,平均间距1.8~9.4 cM。图谱标记分布均匀,可用于QTL定位。
3.4 抗白粉病QTL定位
近年来QTL定位研究是国内外高度重视的一种方法,其发展非常迅猛。最初使用传统的单标记分析法[51],然后发明了利用两个侧邻标记的区间作图法[52],后来发展为在模型中加入其他类型标记,以减少遗传背景效应误差的复合区间作图法[36],目前QTL定位最先进的方法是多重区间作图法[53]。多重区间作图法能够对单一QTL进行快速、有效定位[37,51]。本研究以抗、感白粉病红三叶材料杂交形成的F1群体为作图群体,构建AFLP遗传图谱,并利用多重区间作图法对抗白粉病QTL进行定位分析,可以为红三叶分子辅助育种奠定基础。
4 结论
本试验利用红三叶杂交F1代群体为作图群体,由149个AFLP标记构建得到7个连锁群(LG1,LG2,LG3,LG4,LG5,LG6和LG7)。所获得遗传图谱的总距离和标记间平均距离分别为640.5和4.3 cM。在所得连锁图谱中,LG4遗传距离最小(55.2 cM),包含的标记数高达31个,标记间平均距离最小(1.8 cM),是所有连锁图谱中最密集的。
应用区间作图法对红三叶抗白粉病基因进行QTL分析定位,共检测到5个与抗白粉病相关的QTL位点(qrp-1,qrp-2,qrp-3,qrp-4和qrp-5),分布在LG4和LG5连锁群上,其中LG4上含有4个QTL位点。qrp-1对红三叶白粉病抗性的贡献率最大(90%),为主效QTL。
参考文献References:
[1] Jiang Y B, Wang C Z, Cui G W. Effect of red clover isoflavone on immune organs, igs and anti-oxidation activity of broiler chicks. Acta Agrestia Sinica, 2011, 19(3): 520-524.
姜义宝, 王成章, 崔国文. 红车轴草异黄酮对肉鸡免疫器官、免疫球蛋白及抗氧化性能的影响. 草地学报, 2011, 19(3): 520-524.
[2] Du W H. Red clover. Lanzhou: Gansu Science and Technology Press, 2006: 7-8.
杜文华. 红三叶. 兰州: 甘肃科学技术出版社, 2006: 7-8.
[3] Liu X L, Du W H, Song C. Effects of nitrogen and phosphorus fertilization on isoflavone content in red clover. Acta Agriculture Boreali-occidentalis Sinica, 2010, 19(7): 159-163.
刘晓玲, 杜文华, 宋超. 氮磷肥施用量对红三叶中异黄酮含量的影响. 西北农业学报, 2010, 19(7): 159-163.
[4] Wang Z M, Yue M Q, Du W H,etal. Excellent legume for feeding and medicine-MinshanTrifoliumpretense. Pratacultural Science, 2005, 22(4): 33-35.
王志明, 岳民勤, 杜文华, 等. 集饲用和药用价值于一体的牧草新秀——岷山红三叶. 草业科学, 2005, 22(4): 33-35.
[5] Liu X L, Song C, Du W H. Study on mechanism of resistance to powdery mildew in Minshan red clover. Grassland and Turf, 2011, 31(3): 73-76.
刘晓玲, 宋超, 杜文华. 红三叶对白粉病抗性机理研究. 草原与草坪, 2011, 31(3): 73-76.
[6] Wang Z H, Zhu D L, Yu C. Application in germplasm improvement of medical plants via biotechnology. Bulletin of Science and Technology, 2010, 26(1): 88-92.
王忠华, 朱东亮, 俞超. 现代育种技术在药用植物品种改良中的应用. 科技通报, 2010, 26(1): 88-92.
[7] Jia X X, Qi E F, Ma S,etal. Analysis of drought tolerance and herbicide resistance in transgenic potato plants over-expressingDREB1A/Bar. Acta Prataculturae Sinica, 2015, 24(11): 58-64.
贾小霞, 齐恩芳, 马胜, 等. 转DREB1A/Bar双价基因马铃薯的耐旱性及除草剂抗性分析. 草业学报, 2015, 24(11): 58-64.
[8] Li D M. Constructions on the linkage genetic map and QTL mapping for forage yield and rust resistance related traits in triticale. Lanzhou: Gansu Agricultural University, 2016.
李冬梅. 饲草型小黑麦的遗传图谱构建及草产量和抗锈病相关基因的QTL定位. 兰州: 甘肃农业大学, 2016.
[9] Xu Z, Mei L H, Li Z Y,etal. A genetic linkage map ofBlumeriagramf.sp. tritici based on AFLP molecular markers and a virulence genes. Acta Phytopathologica Sinica, 2009, 39(1): 16-22.
徐志, 梅丽宏, 李志英, 等. 利用AFLP分子标记和无毒基因构建小麦白粉菌遗传连锁图谱. 植物病理学报, 2009, 39(1): 16-22.
[10] Xu J Q, He F, Cui F,etal. Powdery mildew resistant locus inTritileymustranslocation line shannong 6343 mapped by molecular markers. Molecular Plant Breeding, 2012, 10(1): 86-91.
徐金秋, 何方, 崔法, 等. 小滨麦易位系山农6343抗白粉病基因的分子标记定位. 分子植物育种, 2012, 10(1): 86-91.
[11] Liu L Z, Li H, Yang R,etal. Mapping of gene resistance to wheat powdery mildew using SSR markers. Acta Botanica Boreali-Occidentalia Sinica, 2012, 32(7): 1322-1327.
刘联正, 李华, 杨睿, 等. 小麦抗白粉病基因SSR标记定位. 西北植物学报, 2012, 32(7): 1322-1327.
[12] Liu S L, Wang C Y, Wang Q Y,etal. SSR analysis of powdery mildew resistance gene in a new GERMPLASM N9628-2 ofTriticumaestivumL. Acta Agronomica Sinica, 2008, 34(1): 84-88.
刘素兰, 王长有, 王秋英, 等. 小麦新种质N9628-2抗白粉病基因的SSR分析. 作物学报, 2008, 34(1): 84-88.
[13] Li Q Y, Zhang X L, Zhang Z G,etal. Advance of studies on constructing linkage map in Chinese cabbage using molecular marker. Biotechnology Bulletin, 2012, (6): 18-24.
李巧云, 张晓亮, 张志刚, 等. 大白菜分子遗传图谱构建研究进展. 生物技术通报, 2012, (6): 18-24.
[14] Zhu J, Wang T, Zhao Y J,etal. Identification of apple varieties with AFLP molecular markers. Acta Horticulturae Sinica, 2000, 27(2): 102-106.
祝军, 王涛, 赵玉军, 等. 应用AFLP分子标记鉴定苹果品种. 园艺学报, 2000, 27(2): 102-106.
[15] Gao J Q, Zhou J C, Wang X C,etal. Construction of AFLP fingerprint of partial high resistance to low phosphorus stress genotypic in Sugarbeet (BetavulgarisL.). Jiangsu Agricultural Sciences, 2017, 45(6): 36-38.
高金秋, 周建朝, 王孝纯, 等. 部分高抗低磷胁迫基因型甜菜AFLP指纹图谱的构建. 江苏农业科学, 2017, 45(6): 36-38.
[16] Wang D, Yu X X, Yu Z,etal. AFLP analysis of genetic differences among elite hybrid potato lines. Acta Prataculturae Sinica, 2016, 25(9): 117-124.
王丹, 于肖夏, 于卓, 等. 马铃薯杂种优良株系遗传差异的AFLP分析. 草业学报, 2016, 25(9): 117-124.
[17] Xie L B, Wang X, Chen L X,etal. QTL mapping of Vitamin C in pepper fruit based on AFLP and SSR. Chinese Agricultural Science Bulletin, 2017, 33(16): 41-45.
谢立波, 王雪, 陈立新, 等. 基于AFLP和SSR技术对辣椒果实维生素C含量的QTL定位. 中国农学通报, 2017, 33(16): 41-45.
[18] Wang Z W, Wei L M, Xie X M,etal. Establishment of cotton genetic map and QTL analysis of fiber quality traits by using AFLP. Jiangsu Agricultural Sciences, 2015, 43(7): 71-74.
王志伟, 魏利民, 谢晓美, 等. 利用AFLP构建棉花遗传图谱及纤维品质性状QTL分析. 江苏农业科学, 2015, 43(7): 71-74.
[19] Wang X Z, Zhou R, Zhang X J,etal. Map construction and QTL analysis of oil content in soybean. Chinese Journal of Oil Crop Sciences, 2008, 30(3): 272-278.
王贤智, 周蓉, 张晓娟, 等. 大豆遗传图谱的构建和含油量的QTL分析. 中国油料作物学报, 2008, 30(3): 272-278.
[20] Li C X, Xu W G, Wang G S,etal. Molecular marker mapping and genetic analysis of a novel powdery mildew resistance genePmHNK. Scientia Agricultura Sinica, 2009, 42(8): 2771-2777.
李春鑫, 许为钢, 王根松, 等. 小麦白粉病抗病新基因PmHNK的遗传分析和分子标记定位. 中国农业科学, 2009, 42(8): 2771-2777.
[21] Alfandi M, Ji Y, Shen L P,etal. Construction of genetic linkage map and localization of QTLs for powdery mildew resistance in cucumber (Cucumissativus). IV International Symposium on Cucurbits, 2010, 871: 33-41.
[22] Fernando Y L, Carmen C, Emilio S,etal. Genetic linkage map of melon (CucumismelonL.) and localization of a major QTL for powdery mildew resistance. Molecular Breeding, 2011, 27(2): 181-192.
[23] Ren J Z. Scientific research methods of pratacultural science. Beijing: China Agricultural Press, 1998: 214-236.
任继周. 草业科学研究方法. 北京: 中国农业出版社, 1998: 214-236.
[24] Vos P, Hogers R, Bleeker M,etal. AFLP a new technique for DNA fingerprinting. Nucleic Acids Research, 1995, 23: 4407-4414.
[25] Liu S X. Practical biological histological techniques. Beijing: Science Press, 2003: 222-224.
刘世新. 实用生物组织学技术. 北京: 科学出版社, 2003: 222-224.
[26] Jiang Z Y, Yu X X, Yu Z,etal. Construction of a genetic linkage map for tetraploid hybrid wheat grass using a SSR molecular marker. Acta Prataculturae Sinica, 2016, 25(6): 94-101.
姜志艳, 于肖夏, 于卓, 等. 利用SSR分子标记构建四倍体杂交冰草的遗传连锁图谱. 草业学报, 2016, 25(6): 94-101.
[27] Ding C L, Liu Y, Xu N X,etal. QTL analysis of traits relating to cold resistance ofZoysiajaponica. Acta Agrestia Sinica, 2010, 18(5): 703-707.
丁成龙, 刘颖, 许能祥, 等. 日本结缕草抗寒相关性状的QTL分析. 草地学报, 2010, 18(5): 703-707.
[28] Cai C F, Liu G X, Cheng F Y,etal. Selecting optimal F1segregation population for genetic linkage mapping in tree peony. Journal of Beijing Forestry University, 2015, 37(3): 140-147.
蔡长福, 刘改秀, 成仿云, 等. 牡丹遗传作图最适F1分离群体的选择. 北京林业大学学报, 2015, 37(3): 140-147.
[29] Guo J B, Huang L, Cheng L Q,etal. An integrated genetic linkage map from three F2population of cultivated peanut (ArachishypogaeaL.). Acta Agronomica Sinica, 2016, 42(2): 159-169.
郭建斌, 黄莉, 成良强, 等. 利用3个F2群体整合高密度栽培种花生遗传连锁图. 作物学报, 2016, 42(2): 159-169.
[30] Du X L, Zhang Z W, Wu B,etal. Construction and analysis of genetic linkage map in tartary buckwheat (Fagopyrumtataricum) using SSR. Chinese Agricultural Science Bulletin, 2013, 29(21): 61-65.
杜晓磊, 张宗文, 吴斌, 等. 苦荞SSR分子遗传图谱的构建及分析. 中国农学通报, 2013, 29(21): 61-65.
[31] Jiang Z Y, Yu X X, Yu Z,etal. Construction of an AFLP-based genetic linkage map of tetraploid hybrid wheatgrass. Journal of Triticeae Crops, 2015, 35(4): 457-463.
姜志艳, 于肖夏, 于卓, 等. 四倍体杂交冰草AFLP遗传连锁图谱的构建. 麦类作物学报, 2015, 35(4): 457-463.
[32] Fang Y Y, Yu X X, Yu Z,etal. QTL mapping analysis of 10 important traits inSorghum-Sudangrass hybrid. Journal of Northwest A&F University (Nature Science Edition), 2015, 43(4): 26-34, 43.
房永雨, 于肖夏, 于卓, 等. 高丹草10个重要性状的QTL定位分析. 西北农林科技大学学报(自然科学版), 2015, 43(4): 26-34, 43.
[33] Li L, Chen Z W, Du Z Z,etal. Genetic map preliminary structure of low-nitrogen tolerance used DH population in barley. Journal of Triticeae Crops, 2012, 32(6): 1021-1025.
李梁, 陈志伟, 杜志钊, 等. 大麦DH群体在耐低氮相关遗传连锁图谱初步构建中的运用. 麦类作物学报, 2012, 32(6): 1021-1025.
[34] Studer B, Jensen L B, Hentrup S,etal. Genetic characterisation of seed yield and fertility traits in perennial ryegrass (LoliumperenneL.). Theoretical and Applied Genetics, 2008, 117(5): 781-791.
[35] Wang M Y, Zhang T J, Long R C,etal. Preliminary construction of genetic map in tetraploid alfalfa. Acta Agrestia Sinica, 2015, 23(6): 1247-1251.
王梦颖, 张铁军, 龙瑞才, 等. 四倍体紫花苜蓿遗传图谱的初步构建. 草地学报, 2015, 23(6): 1247-1251.
[36] Isobe S, Kölliker R, Hisano H,etal. Construction of a consensus linkage map for red clover (Trifoliumpretense L.). BMC Plant Biology, 2009, (9): 57.
[37] Xie W G. Genetic linkage map and flowering time gene mapping in orchard grass (DactylisglomerataL.). Ya’an: Sichuan Agricultural University, 2012.
谢文刚. 鸭茅分子遗传连锁图谱构建及开花基因定位. 雅安: 四川农业大学, 2012.
[38] Luo Y C, Ma X, Zhang X Q. DNA fingerprinting of annual ryegrass (LoliummultiflorumLam.) varieties using SSR makers. Journal of Agricultural Biotechnology, 2013, 21(7): 799-810.
罗永聪, 马啸, 张新全. 利用SSR技术构建多花黑麦草品种指纹图谱. 农业生物技术学报, 2013, 21(7): 799-810.
[39] Zhong L, Yang C Y, Shu J H,etal. Research progress on genetic breeding of tall fescue (FestucaarundinaceaSchreb.). Agricultural Science & Technology, 2014, 15(2): 265-268.
[40] Li S, Zhao G F. AFLP molecular marker and its application. Acta Botanica Boreali-Occidentalia Sinica, 2003, 23(5): 830-836.
李珊, 赵桂仿. AFLP分子标记及其应用. 西北植物学报, 2003, 23(5): 830-836.
[41] Hu Z H, Guo H Y, Hu X L,etal. Genetic diversity ofCannabissativaL. based on AFLP analysis. Journal of Plant Genetic Resources, 2012, 13(4): 555-561.
胡尊红, 郭鸿彦, 胡学礼, 等. 大麻品种遗传多样性的AFLP分析. 植物遗传资源学报, 2012, 13(4): 555-561.
[42] Li H Y, Li Z Y, Xin X,etal. Genetic diversity inMedicagoruthenica(L.) Trautv. populations using AFLP marker. Journal of Plant Genetic Resources, 2016, 17(1): 78-83.
李鸿雁, 李志勇, 辛霞, 等. 野生扁蓿豆种质资源AFLP 遗传多样性的分析. 植物遗传资源学报, 2016, 17(1): 78-83.
[43] Wang B, Weng M L. The principle and application of AFLP. Hybrid Rice, 1996, (5): 27-30.
王斌, 翁曼丽. AFLP的原理及其应用. 杂交水稻, 1996, (5): 27-30.
[44] Bert P F, Charmet G, Sourdille P,etal. A high-density molecular map for ryegrass (Loliumperenne) using AFLP markers. Theoretical and Applied Genetics, 1999, 99(3): 445-452.
[45] Jaime S S, Daniza M U, Leonardo A R. Molecular description and similarity relationships among native germplasm potatoes (Solanumtuberosumssp.tuberosumL.) using morphological data and AFLP markers. Electronic Journal of Biotechnology, 2007, 10(3): 436-443.
[46] Xie W G, Liu W X, Zhang J Q,etal. Molecular genetic linkage map and its application in forage crops. Pratacultural Science, 2014, 31(6): 1147-1159.
谢文刚, 刘文献, 张建全, 等. 牧草分子遗传连锁图谱及其应用. 草业科学, 2014, 31(6): 1147-1159.
[47] Jing Z B, Yu L, Wei L,etal. Research advances in genomics and transgenic of forage in abroad. Biotechnology Bulletin, 2011, (11): 12-25.
井赵斌, 俞靓, 魏琳, 等. 国外牧草基因组学和转基因技术研究进展. 生物技术通报, 2011, (11): 12-25.
[48] Zhu Y W, Lin Y R, Chen L. Research progress of rice molecular breeding in China. Journal of Xiamen University (Natural Science), 2016, 55(5): 661-671.
朱义旺, 林雅容, 陈亮. 我国水稻分子育种研究进展. 厦门大学学报(自然科学版), 2016, 55(5): 661-671.
[49] Hirata M, Cai H W, Inoue M,etal. Development of simple sequence repeat (SSR) markers and construction of an SSR-based linkage map in Italian ryegrass (LoliummultiflorumLam.). Theoretical and Applied Genetics, 2006, 113(2): 270-279.
[50] Tian L B. Constructing of genetic maps and QTL mapping of powdery mildew resistance in bitter melon. Shenyang: Shenyang Agricultural University, 2015.
田丽波. 苦瓜遗传图谱构建及白粉病抗性的QTL定位. 沈阳: 沈阳农业大学, 2015.
[51] Shang L G. Genetic analysis of heterosis using backcross population in upland cotton (GossypiumhirsutumL.). Beijing: China Agricultural University, 2016.
商连光. 陆地棉回交群体杂种优势遗传基础研究. 北京: 中国农业大学, 2016.
[52] Zhang J. Construction of high-density genetic map and QTL analysis of ornamental traits in Mei. Beijing: Beijing Forestry University, 2016.
张杰. 梅花高密度遗传图谱构建及部分观赏性状QTL分析. 北京: 北京林业大学, 2016.
[53] Wang L. Construction of wheat molecular genetic map and QTL analysis for agronomic and quanlity traits. Taian: Shandong Agricultural University, 2012.
王霖. 小麦遗传连锁图谱构建及主要农艺和品质性状QTL定位. 泰安: 山东农业大学, 2012.

