TYMS基因在前列腺癌细胞中的表达水平及其临床预后意义
宋章兴 崔应东 饶雪飞
前列腺癌(prostate cancer, PCa)是中老年男性中第二常见的恶性肿瘤[1]。据报道,每10万人中就有128人罹患PCa,全球每年有110万新诊断的PCa,且每年有超过3万人死于PCa[1-3]。此外,随着人口的老年化和诊断技术的提高,PCa的发病率逐年上升[4]。虽然局限性PCa一般发展比较缓慢,但其容易发生远处转移,尤其是骨转移最为常见。有研究表明PCa的骨转移率高达90%,一旦PCa发生远处转移或骨转移,患者的临床预后则极差[5]。目前对于前列腺的相关检查手段主要包括直肠指诊、经直肠超声、血清前列腺特异性抗原(prostate specific antigen, PSA)检测及前列腺穿刺活检[3,5-7]。直肠指诊和经直肠超声检查主要与检查者的经验有关,其主观性比较强;血清PSA检测虽然可以重复进行,但是PSA水平容易受到其他因素的影响,其特异性不高;前列腺穿刺活检属有创检查,且有一定的假阴性率。因此,亟待开发出新的检测手段,对于PCa的早期诊断和精准治疗具有重要的意义。
胸苷酸合成酶(thymidylate synthase, TYMS)在嘧啶代谢中发挥着重要作用,是DNA合成的关键酶[8]。有报道认为其与体内多种肿瘤细胞的增殖密切相关[9-12],然而目前尚未有报道其与PCa的发病及进展之间的关系。因此,本研究通过分析TYMS基因表达与PCa的关系,以期为PCa的早期诊断和治疗提供参考。
对象与方法
一、研究材料及方法
GEO(gene expression omnibus)(https://www.ncbi.nlm.nih.gov/geo/)数据库是由美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)建设并维护的基因表达谱储存数据库,截止2018年2月该数据库已经收集4 348 项数据集共2 397 211例样本的基因表达谱数据,我们从中下载4项独立的PCa基因表达数据集GSE32448[13]、GSE16560[14]、GSE17951[15-16]以及GSE79957[17]进行相关分析。GSE32448包含40例PCa患者的临床样本和40正常前列腺组织的临床样本,我们利用该数据集比较TYMS基因在PCa组织中和正常前列腺组织中的表达差异;GSE16560包含了281例PCa患者的基因表达数据,同时还附带对应PCa患者的临床病史特点数据(包括患者的发病年龄、Gleason评分及随访时间等),我们利用该数据集分析TYMS基因表达高低与PCa患者的发病年龄及总生存期的相关性。GSE79957包含了260例PCa患者临床样本的基因表达数据,同样也包含比较详细的PCa患者临床病史数据(包括患者的发病年龄、TNM分期及PSA水平等),我们利用该数据集分析TYMS基因表达与PCa患者TNM分期及血清PSA水平的相关性;GSE17951包含了154例PCa患者临床样本的基因表达数据,我们利用该数据集进行基于TYMS基因表达水平的基因集富集分析(gene set enrichment analysis, GSEA),以明确TYMS对PCa可能的作用机制。
二、统计学分析
TYMS在PCa和正常前列腺组织中的表达水平采用均数±标准差的形式呈现,并采用两独立样本t检验分析正常前列腺组织和PCa组织中TYMS基因表达差异。以TYMS在GSE16560和GSE79957中表达水平的中位数将PCa患者临床样本分为TYMS高表达组和TYMS低表达组。使用卡方检验分析TYMS高表达组和TYMS低表达组PCa患者临床病理特点的差异。采用Log-rank法对TYMS高表达组和TYMS低表达组PCa患者进行生存分析。以上所有统计分析采用SPSS 21.0软件完成,P<0.05作为差异具有统计学意义的判别依据。采用GraphPad 5.0作为绘图工具。此外,我们采用GSEA2-2.2.4[18-19]进行GSEA分析,以hallmark 基因集作为参考基因集,置换次数为1 000次,以P<0.05、错误发现率(false discovery rates, FDR)<0.25作为显著性富集的筛选依据。
结 果
一、TYMS在正常前列腺组织和PCa组织中的表达情况
TYMS在GSE32448中所对应的探针为1554696_s_at。如图1所示,正常前列腺组织中TYMS的表达水平明显低于PCa组织中的表达水平[(16.04±9.54)vs(45.57±91.07),P<0.044 8]。
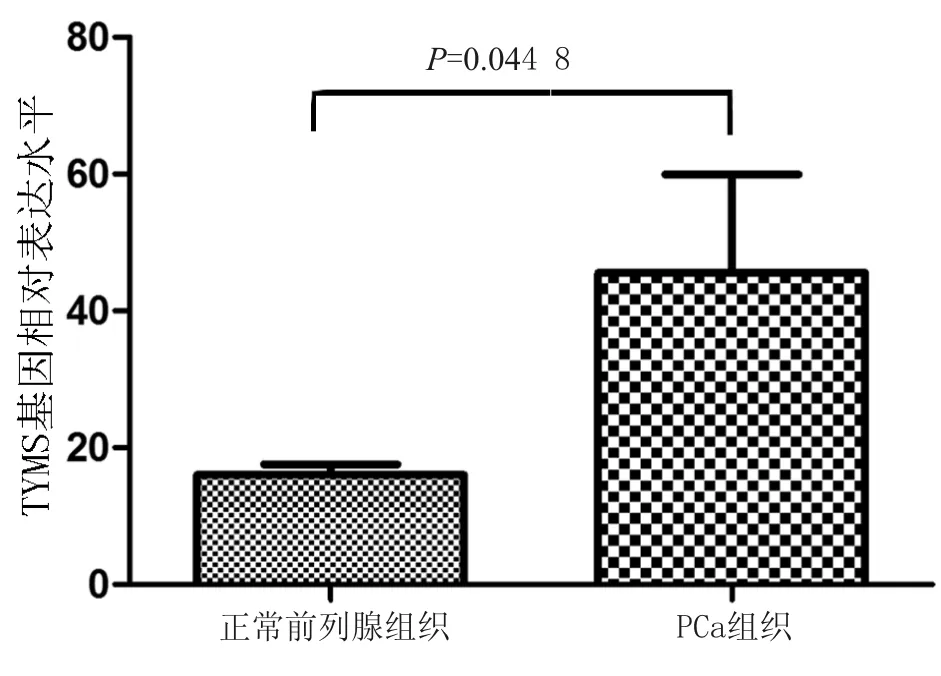
图1正常前列腺组织和PCa组织中TYMS表达水平
二、TYMS表达与PCa患者临床病理相关性分析
为了进一步探讨TYMS表达水平与PCa的发病、进展之间的关系,我们分析了PCa患者的年龄、Gleason评分、TNM分期、血清PSA水平在TYMS高表达组和TYMS低表达组中的分布差异。如表1所示,TYMS高表达组和TYMS低表达组患者在年龄和血清PSA水平分布上无明显差异。TYMS低表达组中Gleason评分为6的患者数目明显高于TYMS高表达组(P<0.000 1),TYMS高表达组中肿瘤分期T3的患者数明显多于TYMS低表达组(P=0.002)。

表1 TYMS基因表达水平与PCa患者临床病理特征的相关性
﹡TYMS高表达患者中有3例未报道患者TNM分期
三、TYMS对PCa患者预后的影响
为了进一步明确TYMS与PCa患者总生存期的关系,我们分析了GSE16560数据集中TYMS高表达组(n=140)和TYMS低表达组(n=141)患者的总生存期。如图2所示,TYMS低表达组PCa患者总生存期明显优于TYMS高表达组(P=0.005 6)。
四、基于TYMS基因高表达的GSEA分析
为了推测TYMS影响PCa患者临床预后可能的作用机制,我们以hallmark基因集为参考基因集,对TYMS进行GSEA分析,结果如表2所示,TYMS高表达组PCa样本主要富集了E2F信号通路、MYC通路、有丝分裂纺锤体、DNA修复、G2M检查点以及糖酵解等生物学过程,提示TYMS可能通过以上信号通路或分子生物学过程影响PCa细胞的增殖。

图2TYMS基因低表达组患者总生存期明显优于TYMS高表达组患者

表2 基于TYMS基因高表达组PCa样本的GSEA分析
讨 论
PCa是欧美国家男性患者最为常见的恶性肿瘤,占男性患者癌症相关死亡的第二位[1]。虽然以PSA为基础的早期诊断明显改善了PCa患者的生存期,但是对于PCa过度诊断和过度治疗也随之上升,进而限制了血清PSA检测在临床上的应用价值[6-7]。另一方面,一旦PCa发生转移,患者的中位生存期则不到5年;去势抵抗的PCa患者的中位生存期则不到2年[3]。因此,寻找特异性更高的PCa早期诊断方法具有重要意义。
本研究表明,PCa组织中TYMS的表达水平明显高于正常前列腺组织中的表达水平。低表达TYMS的PCa患者Gleason评分和TNM分期明显优于高表达TYMS的PCa患者。同时低表达TYMS的PCa患者的总生存期明显优于高表达TYMS的PCa患者,提示TYMS与PCa患者的不良临床特点密切相关。此外,TYMS高表达组和TYMS低表达组患者血清PSA表达水平无明显差异,这可能与PSA检测的特异性不高有关。
有多篇研究报道TYMS基因能够影响体内多种肿瘤细胞的增殖。王明等[9]研究表明TYMS基因低表达与直肠癌患者近期疗效具有明显的相关性,TYMS低表达的直肠癌患者放疗敏感性更高。Weekes等[10]的研究表明携带有TYMS基因突变的胰腺癌患者化疗过程中不良反应更少。俞永康等[11]的研究表明ERCC1、TYMS、TUBB3、RRM1和TOP2A是影响食管鳞癌临床病理特征的部分关键基因。罗春英等[12]的研究表明低表达TYMS基因的非小细胞肺癌患者化疗反应更好、生存期更长。这些研究与我们的结论是相似的,即TYMS低表达的PCa患者临床病理特点更好,其生存期也更长。
GSEA分析结果表明,TYMS可能通过多种信号通路(E2F、MYC)、细胞周期(有丝分裂、G2M检查点)以及细胞代谢(糖酵解)等途径影响PCa细胞的增殖。
综上所述,TYMS在PCa患者中的表达水平明显高于正常前列腺组织,低表达TYMS的PCa患者在T分期、Gleason评分方面优于TYMS高表达的PCa患者,同时低表达TYMS基因的PCa患者总生存期长于高表达TYMS基因的PCa患者。以上结果提示TYMS可能是PCa患者独立的不良预后因素。
[1]Benedetti M, Zona A, Beccaloni E, et al. Incidence of Breast, Prostate, Testicular, and Thyroid Cancer in Italian Contaminated Sites with Presence of Substances with Endocrine Disrupting Properties[J]. Int J Environ Res Public Health,2017,14(4).pii:E355.
[2]Kapoor A, Hotte SJ. Localized prostate cancer[J]. Can Urol Assoc J,2016,10(7-8 Suppl3):S138-S139.
[3]Attard G, Parker C, Eeles RA, et al. Prostate cancer[J]. Lancet,2016,387(10013):70-82.
[4]Pervaiz R, Tulay P, Faisal F, et al. Incidence of cancer in the Turkish Republic of Northern Cyprus[J]. Turk J Med Sci,2017,47(2):523-530.
[5]Klotz L. Activesurveillance for prostate cancer: a review[J]. Curr Urol Rep,2010,11(3):165-171.
[6]Cabarkapa S, Perera M, McGrath S, et al. Prostate cancer screening with prostate-specific antigen: A guide to the guidelines[J]. Prostate Int,2016,4(4):125-129.
[7]Pezaro C, Woo HH, Davis ID. Prostate cancer: measuring PSA[J]. Int Med J,2014,44(5):433-440.
[8]Lima A, Azevedo R, Sousa H, et al. Current approaches for TYMS polymorphisms and their importance in molecular epidemiology and pharmacogenetics[J]. Pharmacogenomics,2013,14(11):1337-1351
[9]王明,李兴德,朱中成. DPYD、TYMS及MTHFR基因表达与直肠癌放化疗敏感性的关系[J]. 现代肿瘤医学,2015,23(12):1697-1700.
[10]Weekes CD, Nallapareddy S, Rudek MA, et al. Thymidylate synthase (TYMS) enhancer region genotype-directed phase II trial of oral capecitabine for 2nd line treatment of advanced pancreatic cancer[J]. Invest New Drugs,2011,29(5):1057-1065.
[11]俞永康,官廷华,姜建青. 食管鳞癌中多基因表达的层次聚类分析[J]. 广东医学,2013,34(21):3305-3307
[12]罗春英,王璇,时姗姗,等. 非小细胞肺癌化疗药物敏感基因表达及其临床病理特征分析[J]. 医学研究生学报,2013,26(5):481-484.
[13]Derosa CA, Furusato B, Shaheduzzaman S, et al. Elevated osteonectin/SPARC expression in primary prostate cancer predicts metastatic progression[J]. Prostate Cancer Prostatic Dis,2011,15(2):150-156.
[14]Sboner A, Demichelis F, Calza S, et al. Molecular sampling of prostate cancer: a dilemma for predicting disease progression[J]. BMC Med Genomics,2010,3:8.
[15]Wang Y, Xia XQ, Jia Z, et al. In silico estimates of tissue components in surgical samples based on expression profiling data[J]. Cancer Res,2010,70(16):6448.
[16]Jia Z, Wang Y, Sawyers A, et al. Diagnosis of prostate cancer using differentially expressed genes in stroma[J]. Cancer Res,2011,71(7):2476-2487.
[17]Tosoian JJ, Druskin SC, Andreas D, et al. Use of the Prostate Health Index for detection of prostate cancer: results from a large academic practice[J]. Prostate Cancer Prostatic Dis,2017,20(2):228-233.
[18]Subramanian A, Tamayo P, Mootha VK, et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles[J]. Proc Natl Acad Sci U S A,2005,102(43):15545-15550.
[19] Mootha VK, Lindgren CM, Eriksson KF, et al. PGC-1alpha-responsive genes involved in oxidative phosphorylation are coordinately downregulated in human diabetes[J]. Nat Genet,2003,34(3):267-273.

