植物3-羟基-3-甲基戊二酰辅酶A还原酶基因研究进展
褚 蔚, 刘洋洋, 李永波, 楚秀生*
1.山东师范大学生命科学学院, 济南 250014;2.山东省农业科学院作物研究所, 农业部黄淮北部小麦生物学与遗传育种重点实验室; 小麦玉米国家工程实验室, 济南 250100
植物体内的异戊二烯类代谢产物如细胞分裂素、赤霉素、脱落酸、叶绿素、萜类、辅酶Q、甾醇和一些植物毒素等是维持植物生长发育及应对环境胁迫(生物胁迫[1~3]和非生物胁迫[4])所必需的重要物质。这些产物中有些还是重要的商业用化合物如医用药物、橡胶、香水及调料等。植物中的异戊二烯类代谢产物主要是通过细胞内甲羟戊酸代谢途径中一系列酶催化合成的,其中3-羟基-3-甲基戊二酰辅酶A还原酶(HMGR;EC:1.1.1.34)是这一代谢途径中的第一个关键限速酶,能够将乙酰辅酶A转化为特别重要的中间产物甲羟戊酸。本文对植物HMGR基因的cDNA克隆、酶结构和功能分析、基因组织表达及调控等方面进行了综述,旨在为其在重要农作物的遗传改良、代谢产物工程植物创制以及植物亲缘关系分析中的应用等研究提供理论依据。
1 植物HMGR基因cDNA的结构和信息
植物HMGR基因的cDNA最早从拟南芥[5]中分离克隆出来,随后从番茄[6]、巴西橡胶树[7]、长春花[8]、马铃薯[9]、香瓜[10]、水稻[11]、小麦[12]等植物中陆续克隆出来。目前,HMGR基因的cDNA已从80多种植物中分离克隆出来,其中拟南芥、巴西橡胶、马铃薯、苜蓿等植物HMGR基因家族的所有成员均已被克隆出来(表1)。
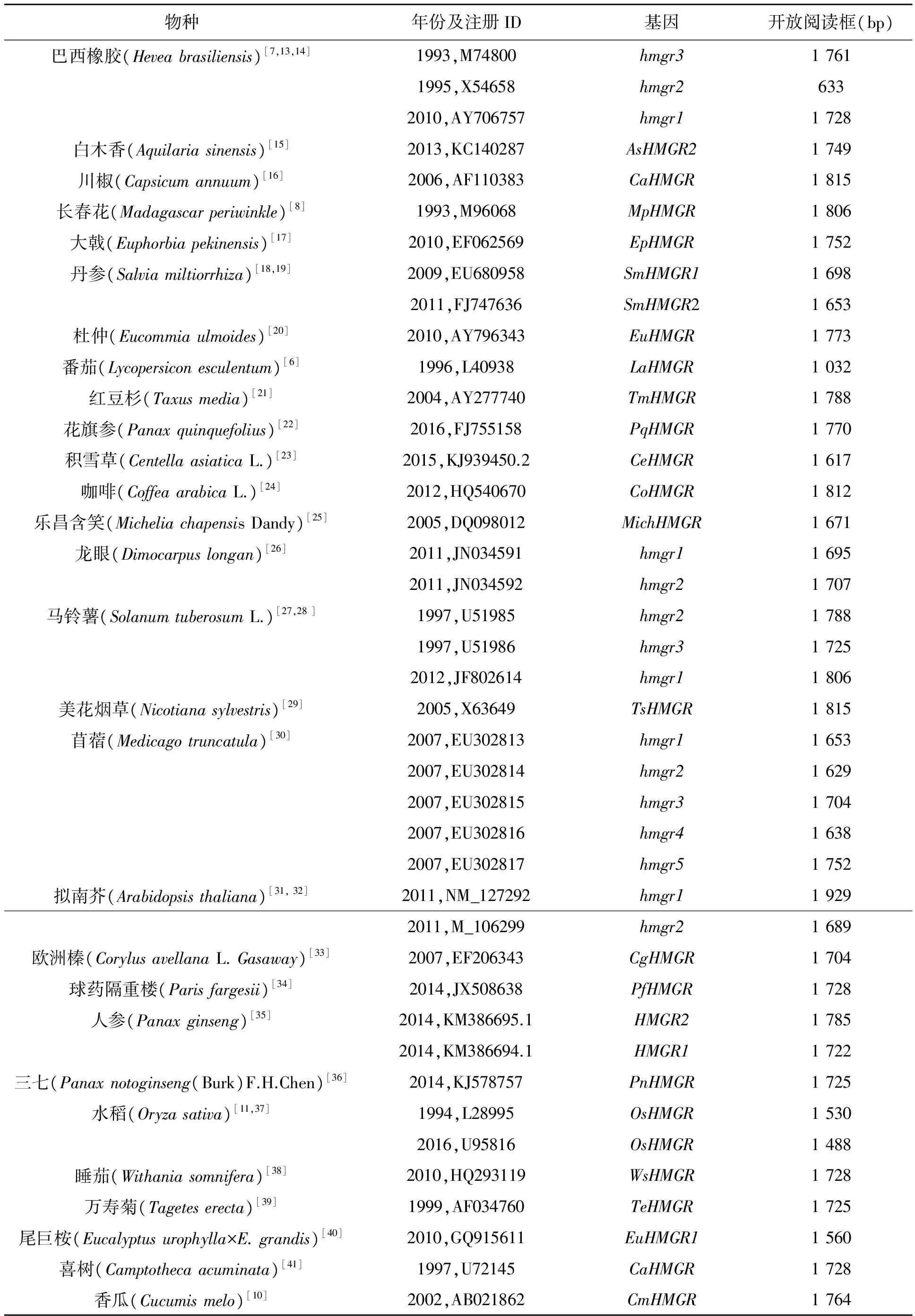
表1 部分植物HMGR基因信息一览表Table 1 Information of partial HMGR genes cloned from plants.
对已克隆的植物HMGR基因的cDNA进行综合分析,发现其编码HMGR的开放阅读框长度在1~2 kb之间不等,多数集中在1.7 kb左右,编码的氨基酸数目在500~600个氨基酸不等。对植物HMGR基因开放阅读框编码的氨基酸序列进行比对,发现多数植物HMGR具有7个保守区域(图1)。Motif1和Motif2为跨膜区,包含20多个氨基酸,多数为脂肪族和芳香族氨基酸交替组合,如大戟EpHMGR的2个跨膜区分别位于多肽链的A40(P)和A62(L)之间、A83(I)和A105(V)之间[17];Motif3和Motif4是HMG-CoA结合区域。Motif3的氨基酸序列为EMPIGYVQIP,在水稻、玉米和小麦等单子叶植物中的第二位氨基酸为亮氨酸(L),双子叶植物中则是蛋氨酸(M)。Motif4非常保守为TTEGCLVA;Motif5和Motif6非常保守,是NADPH结合区域,Motif5和Motif6的氨基酸序列分别为DAMGMNM和GTVGGGTQ。多数植物HMGR末尾还存在Motif7,其中第一个丝氨酸(S)含有磷酸化位点,可能与翻译后调控有关。磷酸化是在类异戊二烯生物合成前几步的酶中确认的主要蛋白质修饰[44]。

图1 8种植物HMGR氨基酸序列比对中的7个保守区域Fig.1 Seven conservative motifs of eight plant HMGR amino acid sequences.注:OsHMGR:水稻(Oryza sativa)AAD08820.1;AtHMGR:拟南芥(Arabidopsis thaliana)NP_179329.1;EpHMGR:大戟(Euphorbia pekinensis)ABK56831.1;HbHMGR:橡胶树(Hevea brasiliensis)AAQ63055.1;GbHMGR:银杏(Ginkgo biloba)AAU89123.1;ZmHMGR:玉米(Zea mays)CAA70440.1;ATHMGR:节节麦(Aegilops tauschii)EMT29532;TaHMGR:小麦(Triticum aestivum)HMGR;“*”和“·”分别表示氨基酸完全相同和半保守。
2 植物HMGR酶的结构
植物HMGR由N-末端区、跨膜区、连接区和C-末端区组成(图2),C-末端序列较N-末端序列保守[36],对截取C端35个氨基酸的短角蒲公英Tbhmgrc3瞬间表达,发现HMGR活性依赖于部分形成一个螺旋的C末端最后30~40个氨基酸残基[46]。高度保守的N-末端氨基酸残基和行使催化功能的C-末端氨基酸残基暴露于内质网膜外侧的细胞质中,如黄花蒿HMGR的N末端和C末端,通过2个跨膜双螺旋被定位在细胞质中[47]。镶嵌在内质网膜上的跨膜区由2个富含疏水氨基酸残基的跨膜结构域构成,由一段短的位于内质网腔内的亲水区域相连。研究表明,跨膜区有助于内质网的形态发生[48]。所有植物HMGR催化区都含有4个保守的区域,即2个HMG-CoA结合区域和2个NADP(H)结合区域,这些残基单元与底物识别和结合有关。
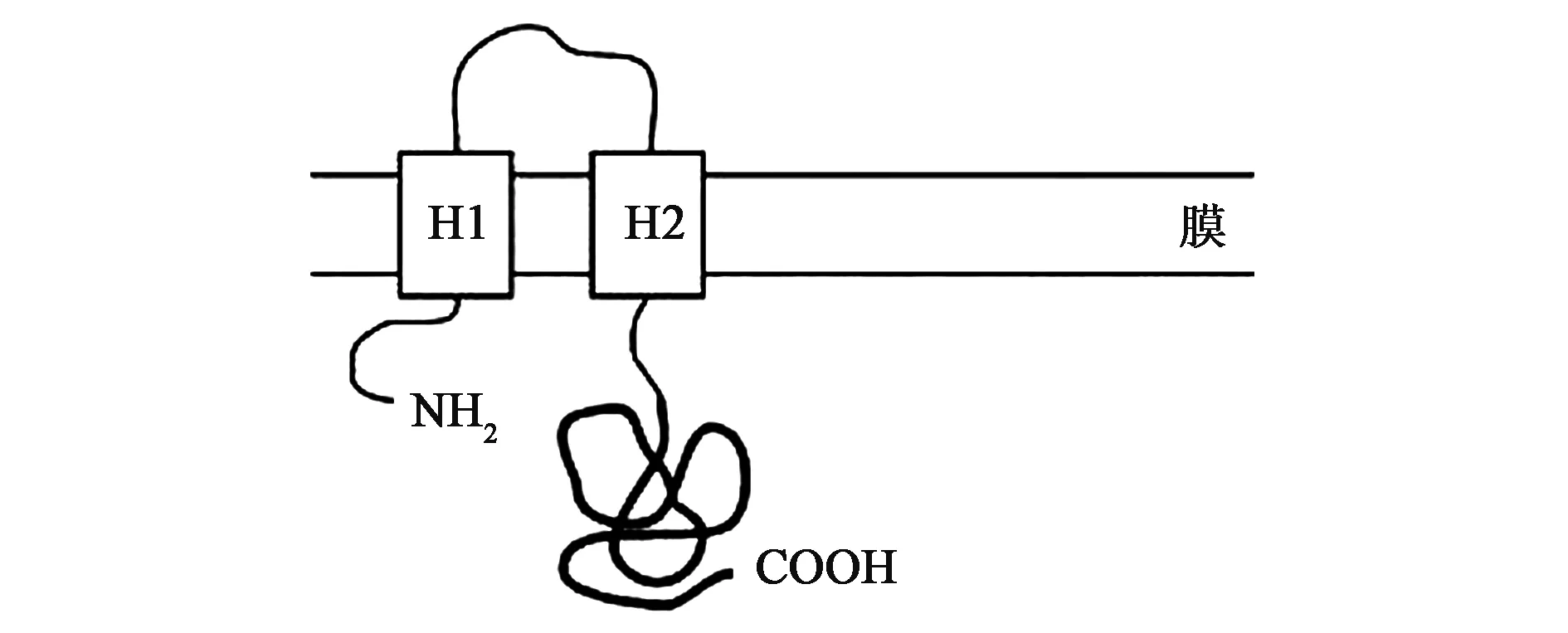
图2 植物HMGR酶结构及其与膜结合示意图[45]Fig.2 Diagram of the plant HMGR structure and bounding to membrane[45]注:H1和H2代表跨膜疏水区,粗线代表催化区。
植物HMGR蛋白的三维结构可分成3个结构域:①N-结构域:N-末端的小螺旋结构域;②L-结构域:包含2个HMG-CoA结合序列(EMPIGYVQIP和TTEGCLVA)和1个NADP(H)结合序列(GTVGGGT),该结构域在立体结构上呈棱镜状,由1个大α螺旋构成其结构的中心元件;③S-结构域:包含另1个NADP(H)结合区域(DAMGMN)的小螺旋结构域。植物HMGR三维结构相似性很高,其空间结构整体呈现一个“V”字型,N-结构域和S-结构域位于“V”型的上端,L-结构域在“V”型下部。图3(彩图见图版一)显示大戟EpHMGR的三级结构,其催化区含有L、N和S 3个结构域,L结构域最大,具有2个HMG-CoA结合区和1个NADP(H)结合区,而小的螺旋状的S结构域包含另1个NADP(H)结合区[17]。

图3 大戟(E. pekinensis)EpHMGR的三维 空间结构图[17]Fig.3 The 3D structure of E. pekinensis EpHMGR[17]. (彩图见图版一)
3 植物HMGR功能分析
HMGR参与的催化反应是以1分子的3-羟基-3-甲基戊二酰辅酶A(HMG-CoA)和2分子的还原型辅酶NADPH为底物,首先,1分子NADPH脱H,加到HMG-CoA中与CoA结合的酰基氧上,随后CoA分子脱离HMG,形成中间体甲羟戊醛和CoASH,最后另1分子NADPH的H加到甲羟戊醛的酰基氧上,形成甲羟戊酸,NADPH还原为2分子NADP+。Maurey 等[49]利用一种单细胞植物藻(Ochromonasmalhamensis),通过对底物进行14C同位素标记,液体闪烁计数器测定HMGR的催化产物,证明了HMGR能够催化3-羟基-3-甲基戊二酰辅酶A形成产物甲羟戊酸。研究表明,对植物HMGR同源或异源过量表达,能够促进代谢途径中类异戊二烯物质含量的增加,然而利用酶抑制剂抑制该酶活性,则类异戊二烯物质含量明显减少,并引起植物表型的一些变化,说明该酶在类异戊二烯合成中具有非常重要的作用,这为利用转基因技术大量合成有重要商业价值的类异戊二烯物质及农作物遗传改良研究奠定了坚实的理论基础。
3.1 植物HMGR酶动力学研究
植物HMGR酶动力学研究是体外验证所克隆的基因编码的酶是否具有催化活性的重要环节,通过分析酶动力学参数,可以获得酶与底物的结合能力以及催化速率等重要信息,为该酶体内过表达中催化效率的功能验证奠定了基础。目前有关植物HMGR酶动力学研究的文献报道相对较少,植物HMGR对底物HMG-CoA和NADPH的Km值是不同的。Km值是酶与底物结合能力的重要体现,Km值越小,酶与底物的结合力越强,反之酶与底物的结合力越弱。由表2可以看出,萝卜HMGR对2个底物的结合力最强,其他4种植物HMGR对HMG-CoA的结合力相似,而玉米HMGR对NADPH的结合力最弱。说明不同植物HMGR对底物的结合力不同,其催化速率也不同。

表2 几种已分离纯化植物HMGR的Km值Table 2 Km values of several plant HMGR purified.
3.2 HMGR体内功能验证
分离克隆的植物HMGR基因编码的酶是否能够在植物体内代谢途径中发挥作用,需要对其进行体内功能验证。通常将植物HMGR基因转入大肠杆菌中,查看其对细菌中甲羟戊酸代谢途径终产物的影响,或通过该酶抑制剂的抑制作用,测定植物体内类异戊二烯物质含量及其对植物表型的影响,达到体内验证其代谢功能的目的。
研究发现,大戟EpHMGR[17]、乐昌含笑MichHMGR[25]和睡茄WsHMGR基因[38]能够促进大肠杆菌中β-胡萝卜素的生物合成;将短角蒲公英TbHMGR1、TbHMGR2和TbHMGR3分别转入一种利用内源MEP(methylerythritol phosphate)途径不能合成IPP的缺陷型大肠杆菌中,发现转入HMGR催化区域基因的大肠杆菌可以存活,证明了3种HMGR都含有相应的催化活性,可以重启MVA途径[45]。
酶抑制剂是验证酶活性的重要化合物。他汀类化合物是动植物HMGR的有效竞争性抑制剂,可以抑制HMGR的活性,进而减少甾醇类物质的合成,影响植物正常生长。研究表明,用30 μmol/L抑制剂lovastatin处理短角蒲公英10 d,其根缩短超过38%,侧根数量也减少,这是由于IPP的生物合成不足而影响植物根长[45];用抑制剂mevinolin处理4周大的人参1 d,可使其不定根中人参皂苷的含量比对照减少34%,人参皂苷的代谢波动与HMGR的酶活性高低相关[35]。拟南芥HMGR1和HMGR2基因缺失突变株对洛伐他汀敏感程度均比野生型植株要高,而且缺失HMGR1基因会导致拟南芥植株矮小、早衰、雄性不育,且甾醇含量比野生型植株低[54]。
3.3 HMGR基因过表达
植物HMGR是甲羟戊酸代谢途径上游的一个关键限速酶,对控制代谢途径中碳源流向起到非常重要的作用,该酶基因表达上调明显促进植物类异戊二烯物质含量的增加,同源及异源基因过表达研究显示,转基因植物中甾醇、萜类等类异戊二烯物质含量明显提高。
研究发现,橡胶树HMGR1基因使转基因烟草总甾醇含量提高了6倍,而且中间代谢产物也在组织中累积[55];青蒿hmgr转基因青蒿中的最高表达量是野生型的2.8倍,而且最高青蒿素含量约是对照植株的1.8倍[56];长春花HMGR基因使黄花蒿中萜类化合物含量提高22.5%[57],使青蒿素含量比受体植株高出38.9%,且酶活性也比受体植株高[58],部分青蒿素含量是受体植株的7.65倍[59];阳春砂AvpHMGR促进了转基因烟草光合色素的合成[60];短角蒲公英TbHMGR1和TbHMGR2的瞬间表达,导致烟草甾醇前体物的较强积累[45];人参PgHMGR基因使转基因桔梗毛状根中的皂苷水平提高1.5~2.5倍,植物固醇水平提高1.1~1.6倍[61];过表达丹参SmHMGR2基因,丹参毛根中双萜和三萜等的含量显著提高[19]。橡胶树hmgr1基因表达导致转基因拟南芥植株表型与野生型明显不同,使叶片增大50%,植株生长更健壮[14];芥菜BjHMGS1基因及其突变体S359A和H188N/S359A基因在拟南芥中过表达,则上调HMGR、SMT2(固醇甲基转移酶基因)、DWF1(δ-24固醇还原酶基因)、CYP710A1(C-22固醇去饱和酶基因)和BR6OX2(油菜素甾醇-6-氧化酶基因)基因的表达,并导致叶片和苗中整体甾醇含量的增加,表现为种子萌发较对照提前、H2O2诱导细胞死亡减少以及依赖水杨酸发病相关基因(PR1/未知蛋白、PR2/β-1、3葡聚糖酶和PR5/类甜蛋白)的组成型表达,导致对灰葡萄孢菌抗性增强,其中转基因拟南芥OE-S359A抗性最高[62]。另外,在转基因棉花中过表达棉铃虫HMGR基因的dsRNA,成功下调棉铃虫HMGR基因,并使目标昆虫的发育和生长受损[63]。
4 植物HMGR基因的组织特异性表达
4.1 在细胞分裂旺盛器官中的表达
HMGR基因在植物的根、茎、叶、花及果实中的表达明显不同,细胞分裂比较旺盛的器官中的表达相对较强。香瓜果实体积大小是由果实早期发育过程中果皮细胞的分裂增殖决定的,早期果实生长过程中CmHMGR基因表达和酶活性非常关键[64];荔枝LcHMG1在果实发育早期阶段表达水平最高,这与高水平细胞分裂有关,并参与调控果实大小,与小果类型相比,LcHMG1在大果类型中的表达水平较高,且表达持续时间较长,而LcHMG2主要在果实发育后期阶段高表达,这与后期细胞伸长需要的类异戊二烯物质的生物合成有关[65];番茄HMGR基因在果实发育早期其mRNA转录水平和酶的活性水平非常高[66];拟南芥HMGR1的mRNA在所有组织中都能检测到,而HMGR2只在分生组织和花中表达水平较高[54];大戟EpHMGR基因在根中高表达,而在茎、叶中低表达[17];乐昌含笑MichHMGR只在叶片中表达,在茎和根中没有表达[25];咖啡CaHMGR1表现暂时性和组织特异性表达,而CaHMGR2则是组成型表达,前者只在发育初始阶段的果实组织(果肉、外胚乳和胚乳)、花芽和叶片中表达,而后者在所有组织(花芽、叶片、树枝和根)及整个发育阶段不同果实组织中都有表达[24];丹参SmHMGR3在根茎叶中均有表达,但在叶中表达量最高[67];人参PgHMGR2在花中表达量最高,其次为叶和根,茎中表达量最低[68];睡茄WsHMGR表达量由多到少依次是花>根>果实>茎和叶,而且在幼叶中的表达量比在成熟叶片中的表达量高[38];白木香AsHMGR2基因主要在根和茎尖中表达,其次是茎,叶中的表达量最低[15];露水草CaHMGR的mRNA在茎、根和叶片中富集[69];球药隔重楼PfHMGR基因在根和茎中的表达比在叶片中强[34];玉米ZmHMG6在种子中较高表达,ZmHMGR5只在种子的胚乳中专一性表达,而大豆GmHMGR4在种子发育后期高表达[70]。由此可见,植物不同器官中,不同HMGR在植物发育不同阶段担负的功能是不同的。
4.2 在代谢产物富集器官中的表达
不同器官中类异戊二烯含量与HMGR表达密切相关。欧洲榛树CgHMGR在根中的表达水平最高,叶片次之,茎中表达量最少[33],这与榛子根部紫杉酚(为双萜物质,一种有效的抗癌物质)含量最高相一致;巴西橡胶树HbHMGR1和HbHMGR3在橡胶树红色幼叶、浅绿幼叶、成熟叶、乳汁液、韧皮部和木质部中都有表达信号,但在橡胶树乳胶液中表达量最高,是调控橡胶合成的主效基因[71];短角蒲公英TbHMGR1在所有组织中都表达,但在乳胶组织中表达量最高[45];尾巨桉EuHMGR1在枝组织中表达水平最高,其次是叶,根中基本没有表达,与桉树萜类精油合成代谢旺盛程度是相一致的[40];人参PgHMGR1和PgHMGR2基因在种子、叶片、茎和花中表达相对较低,但在幼苗叶柄和根中表达较强,PgHMGR2基因在6龄人参根中的转录是3龄人参根中转录的5倍[35];罗汉果HMGR在茎和果实中表达量较高,在叶片中表达量较低,在果实不同发育时期,表达量呈现先升高再降低,然后再升高再降低的波动式变化,与苷V合成积累变化规律相似[72];积雪草CaHMGR在节和叶片中表达最强,而在根中表达较低,节对萜类物质合成非常重要[23]。重要类异戊二烯物质在植物不同器官的累积与HMGR基因表达的一致性、为创制富集类异戊二烯物质的工程植物奠定了坚实的理论基础。
5 植物HMGR基因表达的调控
5.1 化学因子的调控
茉莉酸甲酯(methyl jasmonate,MeJA)可以使HMGR基因表达上调,从而促进植物次生代谢物的积累。100 μmol/L的MeJA能显著提高紫衫细胞悬浮培养液中紫杉醇含量[73];100 mmol/L的MeJA使丹参发根12 h后HMGR表达量是未处理的3.5倍,而96 h后比对照提高了50%[19];MeJA使睡茄WsHMGR基因表达24 h后达到高峰[38];0.2 mmol/L的MeJA诱发的露水草悬浮细胞培养中20-羟基蜕皮酮(20E)的含量是对照的8倍[69]。水杨酸使睡茄WsHMGR基因在24 h时表达增加[38]。化学伤害对白木香AsHMGR2基因表达的诱导强度在8 h时达到最大[15]。Ag+使丹参SmHMGR基因的转录水平24 h时迅速提高[67];25 μmol/L Ag+使露水草悬浮细胞培养中20E的含量是对照的6倍[69]。
5.2 物理因子的调控
拟南芥HMGR1基因表达直接受光照调控,但存在组织特异性。在光照条件下,成熟拟南芥叶子只有极其微弱的GUS显色,而无光条件下幼嫩叶片和叶柄以及成熟叶片边缘都有很深的GUS染色;根部HMGR1启动子的表达只在伸长区,且不受光照影响[74]。切割和真菌感染使马铃薯块茎和甘薯根组织中HMGR表达量上升,进而导致半倍萜烯类植物抗毒素含量增多[75]。物理伤害导致睡茄WsHMGR基因表达显著上调,在24 h时达到高峰[38],而白木香AsHMGR2基因的表达缓慢升高, 6 h 时达到最高表达水平[15]。
此外,植物HMGR基因还有其他的调控方式,生物因素如微生物使假马齿苋HMGR基因表达上调[4],病菌强烈诱导棉花HMGR基因的表达[3];内源代谢因素如甾醇含量的减少可引起关键酶HMGR的反馈调节,烟草细胞鲨烯合酶和角鲨烯环氧化酶的抑制导致HMGR的表达上调,其活性可达到平时的7倍[76];翻译后修饰如丝氨酸磷酸化、N-糖基化修饰以及蛋白质水解等可对酶活性进行调控[44]。
6 展望
植物HMGR基因主要在细胞分裂比较旺盛的器官以及类异戊二烯产物富集的器官中表达强度高,且该基因在转基因受体植株中过表达,能够显著促进类异戊二烯物质含量的增加。研究证明,香瓜CmHMGR基因的表达水平决定了果实体积的大小[64],同样,荔枝LcHMG1在大果类型中的表达水平较高,且持续时间较长[65]。若通过定点诱变技术,创制该酶催化速率提高的基因突变体,转化以类异戊二烯次生代谢产物为主的经济植物,可以进一步提高这些植物体内相应物质的含量,获取更大的商业价值;若转化重要农作物并在体内过量表达,一方面可使作物体内细胞分裂素、赤霉素等植物激素含量水平得到提高,从而加速果实(籽粒)细胞的分裂和伸长,增大作物籽粒的库容,另一方面可加速作物植株中叶绿素的生物合成,进而提高植物的光合效率,增加植株中可溶性糖的含量及籽粒中的淀粉积累,从而提高作物的产量水平。研究表明,细胞分裂素是决定小麦籽粒产量的关键因素[77],降低细胞分裂素氧化酶活性(cytokinin oxidase,CKX)能够显著提高小麦的穗粒数、千粒重和根重[78];将GA3溶液喷洒在小麦赤霉素应答矮杆基因突变体(Rht12)植株的叶片上,能够显著促进植株地上部分的生长[79]。此外,通过该酶基因过表达,某些萜类物质含量的增加可能对作物籽粒品质及食品口感有所改善,或通过加速植物毒素合成,使病虫害防御能力得到进一步提高;对该酶基因结构进行深入剖析,研究其内含子、外显子和启动子等元件,可用于开发代谢途径中关键酶基因的分子标记,进行分子标记辅助育种选择。
另外,根据植物HMGR氨基酸序列构建分子进化树,发现整个植物界HMGR基因家族的功能和进化是相当保守的,该酶基因起源于一个祖先基因,最后演化成4个不同群,单子叶植物和双子叶植物各两个群[48,70]。由于植物HMGR基因是甲羟戊酸途径中的一个相对保守的基因,可以作为生物遗传分化和分子进化研究工作中的重要因子,对于判断物种间亲缘关系的远近具有一定的参考价值。
[1] Mosquera T, Alvarez M F, Jiménez-Gómez J M,etal.. Targeted and untargeted approaches unravel novel candidate genes and diagnostic SNPs for quantitative resistance of the potato (SolanumtuberosumL.) toPhytophthorainfestanscausing the late blight disease[J]. PLoS ONE, 2016, 11(6):e0156254.
[2] Tian X, Ruan J, Huang J,etal.. Gossypol: phytoalexin of cotton[J]. Sci. China(Life Sci.), 2016, 59(2):122-129.
[3] Gupta R, Singh A, Srivastava M,etal.. Microbial modulation of bacoside A biosynthetic pathway and systemic defense mechanism inBacopamonnieriunderMeloidogyneincognitastress[J]. Sci. Rep., 2017, 7:41867,1-11.
[4] 陆 平,田跃胜,王明雪,等. 枸杞脱落酸生物合成关键酶基因NCED的克隆及表达分析[J].植物遗传资源学报,2013,14(2):303-310.
[5] Caelles C, Ferrer A, Balcells L,etal.. Isolation and structural characterization of a cDNA encodingArabidopsisthaliana3-hydroxy-3-methylglutaryl coenzyme A reductase[J]. Plant Mol. Biol., 1989, 13(6): 627-638.
[6] Denbow C J, Lång S, Cramer C L. The N-terminal domain of tomato 3-hydroxy-3-methylglutaryl-CoA reductases sequence microsomal targeting, and glycosylation[J]. J. Biol. Chem., 1996, 271(16): 9710-9715.
[7] Chye M L, Kush A, Tan C T,etal.. Characterization of cDNA and genomic clones encoding 3-hydroxy-3-methylglutaryl-coenzyme A reductase fromHeveabrasiliensis[J]. Plant Mol. Biol., 1991, 16(4): 567-577.
[8] Maldonado-Mendoza I E, Burnett R J, Nessler C L. Nucleotide sequence of a cDNA encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase fromCatharanthusroseus[J]. Plant Physiol., 1992, 100(3):1613-1614.
[9] Choi D, Ward B L, Bostock R M. Differential induction and suppression of potato 3-hydroxy-3-methylglutaryl coenzyme A reductase genes in response toPhytophthorainfestansand to its elicitor arachidonic acid[J]. Plant Cell Online, 1992, 4(10): 1333-1344.
[10] Kato-Emori S, Higashi K, Hosoya K,etal.. Cloning and characterization of the gene encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase in melon (CucumismeloL. reticulatus)[J]. Mol. Genet. Genomics, 2001, 265:135-142.
[11] Nelson A J, Doerner P W, Zhu Q,etal.. Isolation of a monocot 3-hydroxy-3-methylglutaryl coenzyme A reductase gene that is elicitor-inducible[J]. Plant Mol. Biol., 1994, 25(3): 401-412.
[12] 刘洋洋.小麦3-羟基-3-甲基戊二酰辅酶A还原酶(HMGR)基因克隆及功能分析[D].济南:山东师范大学,硕士学位论文,2015.
[13] Chye M L, Tan C T, Chua N H. Three genes encode 3-hydroxy-3-methylglutaryl-coenzyme A reductase inHeveabrasiliensis: hmg1 and hmg3 are differentially expressed[J]. Plant Mol. Biol., 1992 , 19 (3):473-484.
[14] Venkatachalam P, Priya P, Jayashree R,etal.. Molecular cloning and characterization of a 3-hydroxy-3-methylglutaryl-coenzyme A reductase 1 (hmgr1) gene from rubber tree (HeveabrasiliensisMuell. Arg.): A key gene involved in isoprenoid biosynthesis[J]. Physiol. Mol. Biol. Planta, 2009, 15(2):133-143.
[15] 徐艳红,杨 欣,张 争,等.白木香3-羟基-3-甲基戊二酰辅酶A 还原酶基因AsHMGR2的克隆及表达分析[J].药学学报, 2013, 48 (6): 953-959.
[16] Ha S H, Kim J B, Hwang Y S,etal.. Molecular characterization of three 3-hydroxy-3-methylglutaryl-CoA reductase genes including pathogen-induced Hmg2 from pepper (Capsicumannuum)[J]. Biochim. Biophys. Acta, 2003, 1625(3):253-260.
[17] Cao X, Zong Z, Ju X,etal.. Molecular cloning, characterization and function analysis of the gene encoding HMG-CoA reductase fromEuphorbiapekinensisRupr[J]. Mol. Biol. Rep., 2010, 37(3):1559-1567.
[18] Liao P, Zhou W, Zhang L,etal.. Molecular cloning, characterization and expression analysis of a new gene encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase fromSalviamiltiorrhiza[J]. Acta Physiol. Plantarum., 2009, 31(3):565-572.
[19] Dai Z, Cui G, Zhou S F,etal.. Cloning and characterization of a novel 3-hydroxy-3-methylglutaryl coenzyme A reductase gene fromSalviamiltiorrhizainvolved in diterpenoid tanshinone accumulation[J]. J. Plant Physiol., 2011, 168(2):148-157.
[20] Jiang J, Kai G, Cao X,etal.. Molecular cloning of a HMG-CoA reductase gene fromEucommiaulmoidesOliver[J]. Biosci. Rep., 2006, 26(2):171-181.
[21] Liao Z H, Tan Q M, Chai Y R,etal.. Cloning and characterisation of the gene encoding HMG-CoA reductase fromTaxusmediaand its functional identification in yeast [J]. Funct. Plant Biol., 2004, 31(1):73-81.
[22] Wu Q, Song J Y, Sun Y Q,etal.. Transcript profiles ofPanaxquinquefolius, from flower, leaf and root bring new insights into genes related to ginsenosides biosynthesis and transcriptional regulation[J]. Physiol. Plant, 2010, 138(2):134-149.
[23] Kalita R, Patar L, Shasany A K,etal.. Molecular cloning, characterization and expression analysis of 3-hydroxy-3-methylglutaryl coenzyme A reductase gene fromCentellaasiaticaL.[J]. Mol. Biol. Rep., 2015, 42:1431-1439.
[24] Tiski I, Marraccini P, Pot D,etal.. Characterization and expression of two cDNA encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase isoforms in coffee (CoffeaarabicaL.)[J]. J. Integr. Biol., 2011, 15(10):719-727.
[25] Cao X Y, Li C G, Miao Q,etal.. Molecular cloning and expression analysis of a leaf-specific expressing 3-hydroxy-3-methylglutaryl-CoA (HMG-CoA) reductase gene fromMicheliachapensisDandy[J]. J. Med. Plants Res., 2011,5(16):3868-3875.
[26] Xia R, Lu W J, Wang Z H,etal.. Cloning and characterisation of two genes for 3-hydroxy-3-methylglutaryl coenzyme A reductase and their possible roles during fruit development inDimocarpuslongan[J]. J. Hortic. Sci. Biotechnol., 2011, 86(1):25-30.
[27] Korth K L, Stermer B A, Bhattacharyya M K,etal.. HMG-CoA reductase gene families that differentially accumulate transcripts in potato tubers are developmentally expressed in floral tissues[J]. Plant Mol. Biol., 1997, 33(3):545-551.
[28] Ginzberg I, Thippeswamy M, Fogelman E,etal.. Induction of potato steroidal glycoalkaloid biosynthetic pathway by overexpression of cDNA encoding primary metabolism HMG-CoA reductase and squalene synthase [J]. Planta, 2012, 235(6):1341-1353.
[29] Genschik P, Criqui M C, Parmentier Y,etal.. Isolation and characterization of a cDNA encoding a 3-hydroxy-3-methylglutaryl coenzyme A reductase fromNicotianasylvestris[J]. Plant Mol. Biol., 1992, 20(2):337-341.
[30] Kevei Z, Lougnon G, Mergaert P,etal.. 3-hydroxy-3-methylglutaryl coenzyme A reductase1 interacts with NORK and is crucial for nodulation inMedicagotruncatula[J]. Plant Cell, 2007, 19(12):3974-3989.
[31] Lin X, Kaul S, Rounsley S,etal.. Sequence and analysis of chromosome 2 of the plantArabidopsisthaliana[J]. Nature, 1999, 402 (6763):761-768.
[32] Theologis A, Ecker J R, Palm C J,etal.. Sequence and analysis of chromosome 1 of the plantArabidopsisthaliana[J]. Nature, 2000, 408(6814):816-820.
[33] Wang Y, Guo B, Zhang F,etal.. Molecular cloning and functional analysis of the gene encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase from hazel (CorylusavellanaL. Gasaway) [J]. J. Biochem. Mol. Biol., 2007, 40(6):861-869.
[34] Liang Y, Jiang X, Hu Q,etal.. Cloning and characterization of 3-hydroxy-3-methylglutaryl-CoA reductase (HMGR) gene fromParisfargesiiFranch[J]. Ind. J. Biochem. Biol., 2014, 51:201-206.
[35] Kim Y J, Lee O R, Oh J Y,etal.. Functional analysis of 3-hydroxy-3-methylglutaryl coenzyme A reductase encoding genes in triterpene saponin-producing ginseng[J]. Plant Physiol., 2014, 165(1):373-387.
[36] 张 萍,刘迪秋,葛 锋,等. 三七3-羟基-3-甲基戊二酸单酰辅酶A还原酶基因的克隆和生物信息学分析[J].中草药,2014, 45(18):2684-2690.
[37] Ha S H, Lee S W, Kim Y M,etal.. Molecular characterization of Hmg2 gene encoding a 3-hydroxy-methylglutaryl-CoA reductase in rice[J]. Mol. Cells, 2001, 11(3):295-302.
[38] Akhtar N, Gupta P, Sangwan N S,etal.. Cloning and functional characterization of 3-hydroxy-3-methylglutaryl coenzyme A reductase gene fromWithaniasomnifera: An important medicinal plant[J]. Protoplasma, 2013, 250(2):613-622.
[39] Villar-Martínez A D, Ma R, Paredes-López O. Molecular characterization of cDNAs encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase in marigold (Tageteserecta)[J]. J. Plant Physiol., 1999, 155(2):205-211.
[40] 彭 江,孙一铭,宋 锋,等.尾巨桉3-羟基3-甲基戊二酰CoA还原酶基因的克隆及表达分析[J].林业科学,2011, 47(2):164-168.
[41] Maldonado-Mendoza I E, Vincent R M, Nessler C L. Molecular characterization of three differentially expressed members of theCamptothecaacuminata3-hydroxy-3-methylglutaryl CoA reductase (HMGR) gene family[J]. Plant Mol. Biol., 1997, 34(5):781-790.
[42] 陈小保,李帅广,易善泽,等.阳春砂HMGR基因cDNA克隆及生物信息学分析[J].暨南大学学报, 2016, 37 (4), 285-292.
[43] Shen G, Pang Y, Wu W,etal.. Cloning and characterization of a root-specific expressing gene encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase fromGinkgobiloba[J]. Mol. Biol. Rep., 2006, 33(2):117-127.
[44] Hemmerlin A. Post-translational events and modifications regulating plant enzymes involved in isoprenoid precursor biosynthesis[J]. Plant Sci., 2013, 203-204:41-54.
[45] Campos N, Boronat A. Targeting and topology in the membrane of plant 3-hydroxy-3-methylglutaryl coenzyme A reductase[J]. Plant Cell, 1995, 7:2163-2174.
[46] Deenen N V, Bachmann A L, Schmidt T,etal.. Molecular cloning of mevalonate pathway genes fromTaraxacumbrevicorniculatumand functional characterisation of the key enzyme 3-hydroxy-3-methylglutaryl-coenzyme A reductase[J]. Mol. Biol. Rep., 2012, 39:4337-4349.
[47] Kiran U, Ram M, Khan M A,etal.. Structural and functional characterization of HMG-COA reductase fromArtemisiaannua[J]. Bioinformation, 2010, 5(4):146-149.
[48] Ferrero S, Grados-Torrez R E, Leivar P,etal.. Proliferation and morphogenesis of the endoplasmic reticulum driven by the membrane domain of 3-hydroxy-3-methylglutaryl coenzyme A reductase in plant cells[J]. Plant Physiol., 2015, 168:899-914.
[49] Maurey K, Wolf F, Golbeck J. 3-hydroxy-3-methylglutaryl coenzyme A reductase activity inOchromonasmalhamensis[J]. Plant Physiol., 1986, 82:523-527.
[50] Bach T J, Rogers D H, Rudney H. Detergent-solubilization, purification, and characterization of membrane-bound 3-hydroxy-3-methylglutaryl coenzyme A reductase from radish seedlings[J]. Eur. J. Biochem., 1986, 154(1):103-111.
[51] Dale S, Arró M, Becerra B,etal.. Bacterial expression of the catalytic domain of 3-hydroxy-3-methylglutaryl-CoA reductase (isoform HMGR1) fromArabidopsisthaliana, and its inactivation by phosphorylation at Ser577 byBrassicaoleracea3-hydroxy-3-methylglutaryl-CoA reductase kinase[J]. Eur. J. Biochem., 1995, 233(2):506-513.
[52] Bach T J, Weber T, Motel A. Some properties of enzymes involved in the biosynthesis and metabolism of 3-hydroxy-3-methylglutaryl-CoA in plants[A]. In: Biochemistry of the Mevalonic Acid Pathway to Terpenoids[M]. Springer, 1990,1-82.
[53] Wititsuwannakul R, Wititsuwannakul D, Suwanmanee P. 3-Hydroxy-3-methylglutaryl coenzyme a reductase from the latex ofHeveabrasiliensis[J]. Phytochemistry, 1990, 29(5):1401-1403.
[54] Suzuki M, Kamide Y, Nagata N,etal.. Loss of function of 3-hydroxy-3-methylglutaryl coenzyme A reductase 1 (HMG1) inArabidopsisleads to dwarfing, early senescence and male sterility, and reduced sterol levels[J]. Plant J., 2004, 37(5):750-761.
[55] Schaller H, Grausem B, Benveniste P,etal.. Expression of theHeveabrasiliensis(HBK) Mull. Arg. 3-hydroxy-3-methylglutaryl-coenzyme A reductase 1 in tobacco results in sterol overproduction[J]. Plant Physiol., 1995, 109(3):761-770.
[56] Wang Y, Jing F, Yu S,etal.. Co-overexpression of the HMGR and FPS genes enhances artemisinin content inArtemisiaannuaL. [J]. J. Med. Plants Res., 2011, 5(15):3396-3403.
[57] Aquil S, Husaini A M, Abdin M Z,etal.. Overexpression of the HMG-CoA reductase gene leads to enhanced artemisinin biosynthesis in transgenicArterrdsiaannuaplants[J]. Planta Med., 2009, 75(13):1453-1458.
[58] Nafis T, Akmal M, Ram M,etal.. Enhancement of artemisinin content by constitutive expression of the HMG-CoA reductase gene in high-yielding strain ofArtemisiaannuaL.[J]. Plant Biotechnol. Rep., 2011, 5(1):53-60.
[59] Alam P, Abdin M. Over-expression of HMG-CoA reductase and amorpha-4, 11-diene synthase genes inArtemisiaannuaL. and its influence on artemisinin content[J]. Plant Cell Rep., 2011, 30(10):1919-1928.
[60] 刘 卉,杨锦芬,魏洁书,等. 阳春砂HMGR、DXR超表达提高相应酶活性及光合色素含量[A]. 见:第三届全国植物生物技术及其产业化大会论文摘要集[C]. 福州,2011, 142.
[61] Kim Y K, Kim J K, Kim Y B,etal.. Enhanced accumulation of phytosterol and triterpene in hairy root cultures ofPlatycodongrandiflorumby overexpression ofPanaxginseng3-hydroxy-3-methylglutaryl-coenzyme A reductase[J]. J. Agric. Food Chem., 2013, 61(8):1928-1934.
[62] Wang H, Nagegowda D A, Rawat R,etal.. Overexpression ofBrassicajunceawild-type and mutant HMG-CoA synthase 1 inArabidopsisup-regulates genes in sterol biosynthesis and enhances sterol production and stress tolerance[J]. Plant Biotechnol. J., 2012, 10:31-42.
[63] Tian G, Cheng L, Qi X,etal.. Transgenic cotton plants expressing double-stranded RNAs target HMG-CoAreductase(HMGR) gene inhibits the growth, development and survival of cotton bollworms[J]. Int. J. Biol. Sci., 2015, 11:1296-1305.
[64] Kobayashi T, Kato-Emori S, Tomita K,etal.. Detection of 3-hydroxy-3-methylglutaryl-coenzyme A reductase protein Cm-HMGR during fruit development in melon (CucumismeloL.)[J]. Theor. Appl. Genet., 2002, 104(5):779-785.
[65] Xia R, Li C, Lu W,etal.. 3-Hydroxy-3-methylglutaryl coenzyme A reductase 1 (HMG1) is highly associated with the cell division during the early stage of fruit development which determines the final fruit size inLitchichinensis[J].Gene, 2012, 498:28-35.
[66] Narita O J, Gruissem W. Tomato hydroxymethylglutaryl-CoA reductase is required early in fruit development but not during ripening[J]. Plant Cell, 1989, 1:81-190.
[67] 张夏楠,郭 娟,申 业,等. 一个新的丹参3-羟基-3-甲基戊二酰辅酶A还原酶3 基因的克隆及其表达分析[J]. 中国中药杂志,2012, 37(16):2378-2382.
[68] 罗红梅,宋经元,李雪莹,等.人参皂苷合成生物学关键元件HMGR基因克隆与表达分析[J].药学学报, 2013, 48 (2): 219-227.
[69] Wang Q J, Zheng L P, Zhao P F,etal.. Cloning and characterization of an elicitor-responsive gene encoding 3-hydroxy-3-methylglutaryl coenzyme A reductase involved in 20-hydroxyecdysone production in cell cultures ofCyanotisarachnoidea[J]. Plant Physiol. Biochnol., 2014, 84:1-9.
[70] Li W, Liu W, Wei H,etal.. Species-specific expansion and molecular evolution of the 3-hydroxy-3-methylglutaryl coenzyme A reductase (HMGR) gene family in plants[J]. PLoS ONE, 2014, 9(4),e94172:1-10.
[71] Sando T, Takaoka C, Mukai Y,etal.. Cloning and characterization of mevalonate pathway genes in a natural rubber producing plant,Heveabrasiliensis[J]. Biosci. Biotechnol. Biochem., 2008, 72(8):2049-2060.
[72] 郭茜茜,马小军,白隆华,等. 罗汉果内参基因筛选和3-羟基3-甲基戊二酰辅酶A还原酶时空表达分析[J]. 中草药,2014, 45(15):2224-2229.
[73] Yukimune Y, Tabata H, Higashi Y,etal.. Methyl jasmonate-induced overproduction of paclitaxel and baccatin III in Taxus cell suspension cultures[J]. Nat. Biotechnol., 1996, 14(9):1129-1132.
[74] Learned R M, Connolly E L. Light modulates the spatial patterns of 3‐hydroxy-3-methylglutaryl coenzyme A reductase gene expression inArabidopsisthaliana[J]. Plant J., 1997, 11(3):499-511.
[75] Kondo K, Uritani I, Oba K. Induction mechanism of 3-hydroxy-3-methylglutaryl-CoA reductase in potato tuber and sweet potato root tissues[J]. Biosci. Biotech. Biochem., 2003, 67(5):1007-1017.
[76] Wentzinger L F, Bach T J, Hartmann M A. Inhibition of squalene synthase and squalene epoxidase in tobacco cells triggers an up-regulation of 3-hydroxy-3-methylglutaryl coenzyme A reductase[J]. Plant Physiol., 2002, 130(1):334-346.
[77] Song J, Jiang L, Jameson P E. Co-ordinate regulation of cytokinin gene family members during flag leaf and reproductive development in wheat[J]. BMC Plant Biol., 2012, 12: 1-16.
[78] Zalewski W, Galuszka P, Gasparis S,etal.. Silencing of theHvCKX1 gene decreases the cytokinin oxidase/dehydrogenase level in barley and leads to higher plant productivity[J]. J. Exp. Bot., 2010, 61(6):1839-1851.
[79] Chen L, Hao L, Condon A G,etal.. Exogenous GA3application can compensate the morphogenetic effects of the GA-responsive dwarfing geneRht12 in bread wheat[J]. PLoS ONE, 2014, 9(1): e86431.

