酰胺生物降解机制的分子模拟研究
张 娱, 刘智峰, 陈 明, 刘世军, 许洪波, 唐志书*
1.陕西中医药大学陕西省中药资源产业化协同创新中心, 陕西 咸阳 712083;2.湖南大学环境科学与工程学院, 长沙 410082
随着生物化工业、精细化工业的不断发展,微生物催化的各种反应因具有产物纯度高和对环境友好等优势而表现出越来越广阔的应用前景[1]。酰胺酶催化酰胺生成酸就属于这一类,它催化各种酰胺生成相应的酸,产物酸有比较活泼的羧基而更利于修饰或者合成其他更复杂的制药或者化工中间体,但酰胺酶的催化底物范围受限,对简单的、无取代基的直链底物酶活较高,对芳香、杂环,尤其是存在邻位取代基的酰胺催化效率很低,这是电子效应与空间效应共同作用的结果[2,3]。因此,通过筛选菌株或者对酶进行改造提高酰胺的降解效率成为亟待解决的难题。报道称大多数研究主要集中在新菌种的筛选上,而对酶的改造很少关注,若能理解酰胺酶的性质究竟对其底物的特异性起到了何种作用,就可以研究如何改变酶催化中心的构型从而使酶活性中心的柔性增强,扩展酰胺酶的底物适用范围[4,5]。
Kohyama等[6]用RhodococcuspyridinivoransS85-2产腈水合酶和BrevundimonasdiminutaAM 10-C-1产酰胺酶的级联反应将乙腈降解为乙酸,耐受的乙腈浓度达6 mol/L,10 h反应后的转化率高达90%。该工艺简单高效,适用于含乙腈污染物的定点处理。Li等[7]用制药废水处理厂的活性污泥将乙腈作为唯一的碳源和氮源富集培养得到能降解乙腈、丙烯腈、苯甲腈的菌群,这在多种腈类的降解方面有良好的应用前景。
酰胺酶催化水解的反应机制和酰基转移反应相同,底物水的存在给反应机制研究造成了困难。1986年,Macstracci等[8]用乙酰胺作底物,羟胺为共有底物,发现RhodococcusR312酰胺酶是双底物乒乓反应机制,并证实酰胺水解反应也属于这一机制,即酰胺先与酶结合生成酰基-酶复合物,然后酰基转给共底物水或者羟胺形成羧酸或者氧肟酸。羟胺和水并存时,共同争夺酰基-酶复合物,该复合物去酰基化就成了整个反应的限速步骤。因为羟胺比水更亲核,所以酰基转移反应比水解反应更快。迄今为止,还没有从分子水平上(用分子对接技术等方法)对酰胺与酰胺酶之间的相互作用原理的报道。因此本研究通过软件Molegro Virtual Docker(MVD)和LPC/CSU Server,用分子对接技术等方法研究酰胺与它们的降解酶之间的相互作用,以期揭示降解酶的微观降解机制。
1 材料与方法
1.1 受体的准备
本研究采用的晶体结构取自PDB(ProteinData Bank)结构数据库(http://www.pdb.org/pdb),PDB ID:3A1K(来自Rhodococcussp. N-771,简称为RhAmidase)。其3D结构如图1(彩图见图版六)所示。

图1 Rhodococcus sp. N771酰胺酶的3D结构Fig.1 Crystal structure of Amidase from Rhodococcus sp. N771. (彩图见图版六)
1.2 配体的准备
用于分子对接计算的酰胺化合物取自数据库ChemSpider,它建立的目的是将化学结构式与其相关信息整合在一起并将其编入索引在一个单一并可供搜索的数据库中,且所有的使用者都可以免费使用。所选底物的2D和3D结构从ChemSpider(http://www.chemspider.com/)检索,汇总在表1中。
1.3 分子对接方法
本研究采用MVD进行分子对接计算。MVD是跨平台(Linux、Windows和Mac)的预测蛋白质跟化学小分子结合的软件,其对接精度高达87%[9],可以提供准确性较高的对接结果[10]。
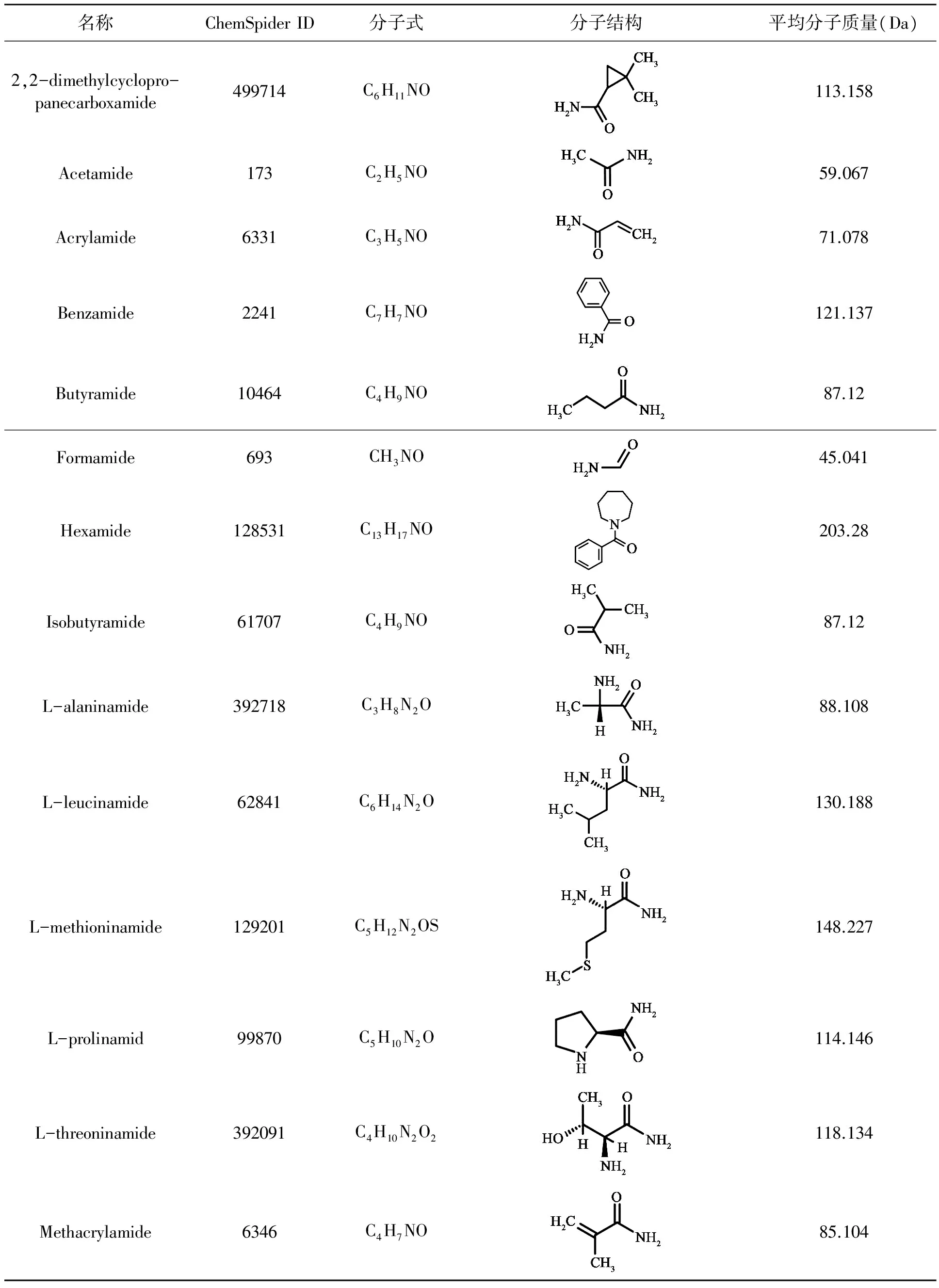
表1 各种化合物的分子结构和特征Table 1 Molecule structure and characteristic of compounds.
对接前去除酰胺酶的水分子、辅因子和结合的配合基。用MVD进行对接时,选择MolDock Score[GRID]为打分函数,栅格分辨率为0.30Å,搜索算法选择MolDock SE,最大迭代次数为1 500,最大群体大小为50。MVD对接结果会提供很多可能的符合构型,基于打分函数与二次打分函数最低原则筛选出最佳结合构象。对接获得的最佳结合构象应用LPC/CSU Server软件进行进一步探讨。LPC/CSU Server是研究配体与受体的相互作用情况的软件,主要预测蛋白质与配体之间的氢键作用、疏水作用以及亲水-疏水作用等[11]。
2 结果与分析
2.1 RhAmidase与酰胺的分子对接结果
研究中选择的酰胺酶来自Rhodococcussp. N-771 (简写为RhAmidase)。选择的酰胺有2,2-dimethylcyclopropanecarboxamide、acetamide、acrylamide、benzamide、butyramide、formamide、hexamide、isobutyramide、L-alaninamide、L-leucinamide、L-methioninamide、L-prolinamid、L-threoninamide、methacrylamide、nicotinamide和propanamide(表1)。至今还没有研究报道这些酰胺与RhAmidase之间的结合模式。这里首次用MVD通过分子对接研究它们之间的结合(图2,彩图见图版六)。

图2 RhAmidase和酰胺之间的结合模式Fig.2 Binding modes between RhAmidase and amides.注:RhAmidase的3D结构用卡通体表示;酰胺的3D结构用绿色条状模型表示。A:RhAmidase 和 acetamide之间的结合模式;B:RhAmidase 和 formamide之间的结合模式;C:RhAmidase 和 nicotinamide之间的结合模式;D:RhAmidase 和 benzamide之间的结合模式;E:RhAmidase 和 propanamide之间的结合模式;F:RhAmidase 和 acrylamide之间的结合模式;G:RhAmidase 和 methacrylamide之间的结合模式;H:RhAmidase 和 butyramide之间的结合模式;I:RhAmidase 和 hexamide之间的结合模式;J:RhAmidase 和 isobutyramide之间的结合模式;K:RhAmidase 和 L-leucinamide之间的结合模式;L:RhAmidase和L-prolinamid之间的结合模式;M:RhAmidase 和L-methioninamide之间的结合模式;N:RhAmidase 和 L-threoninamide之间的结合模式;O:RhAmidase 和 L-alaninamide之间的结合模式;P:RhAmidase 和 2,2-dimethylcyclopropanecarboxamide之间的结合模式。 (彩图见图版六)
2.2 RhAmidase和酰胺最佳结合模式的筛选
在用MVD软件进行分子对接的过程中,采用准确性较高的打分算法MolDock对酰胺酶与选择的底物酰胺的相互作用模式进行打分,从计算返回的10个优势构象中通过比较RMSD、能量打分和基于经验判断选择最佳构象,最终获得底物酰胺与酰胺酶的最佳结合模式。最低的重排得分构象作为首选结果。表2列出了MVD生成的最佳结合构象的MolDock 得分和 Re-Rank 得分(表2)。有研究者观察到RhAmidase 对丙烯酰胺有微弱的水解活性,一定程度上可能是由于RhAmidase 和丙烯酰胺之间的弱相互作用[12]。RhAmidase与L-methioninamide对接时二次打分函数为-76.022 4,较低的打分函数说明配体与蛋白质匹配程度高,复合物稳定性高。由MVD预测的最佳构象经LPC/CSU Server分析结果见图3(彩图见图版七)和表3。由表3可知,RhAmidase与L-methioninamide之间的氢键作用有7种,疏水作用有13种,亲水-疏水作用有10种,芳香-芳香作用有1种。说明RhAmidase与L-methioninamide之间以疏水作用数量最多。

表2 RhAmidase和酰胺之间分子对接构象打分Table 2 Molecular docking pose scores between RhAmidase and amides.
2.3 RhAmidase和L-methioninamide之间的相互作用
高酰胺结合率可以延长酰胺酶活动的持续时间,从而增强反应效果。本研究选取的酰胺酶属于酰胺酶标志(AS)家族,这个家族中Ser-cisSer-Lys 催化三联体守恒[13]。 然而这3种氨基酸在结合L-methioninamide 时并不起重要作用(表3)。
2.4 RhAmidase/L-methioninamide复合物间的相互作用
酰胺酶来源不同,底物特异性也各异。一些酰胺酶只水解脂肪族酰胺,有些只水解芳香族酰胺,还有一些酰胺酶能水解氨基酰胺。大部分酰胺酶只对无环或简单芳香类酰胺有催化能力,对复杂的芳香类或杂环类尤其是邻位有取代基的酰胺活力不强。有研究者认为这种现象是空间位阻和电子效应共同作用的结果。
RhAmidase和L-methioninamide之间的氢键作用由残基GLY219 A、TYR346 A、LEU353 A、ASN253 A、THR218 A、TYR217 A和ALA220 A形成。PRO255 A、ALA350 A、TYR346 A、LEU353 A、ARG225 A和PRO222 A与配体L-methioninamide形成疏水作用。疏水作用在RhAmidase和L-methioninamide之间大量存在,可能是产生RhAmidase/L-methioninamide复合体的主动力。6个残基ARG256 A、LEU353 A、TYR346 A、ARG225 A、THR218 A和PRO222 A在RhAmidase以亲水-疏水作用结合到L-methioninamide 时起重要作用。
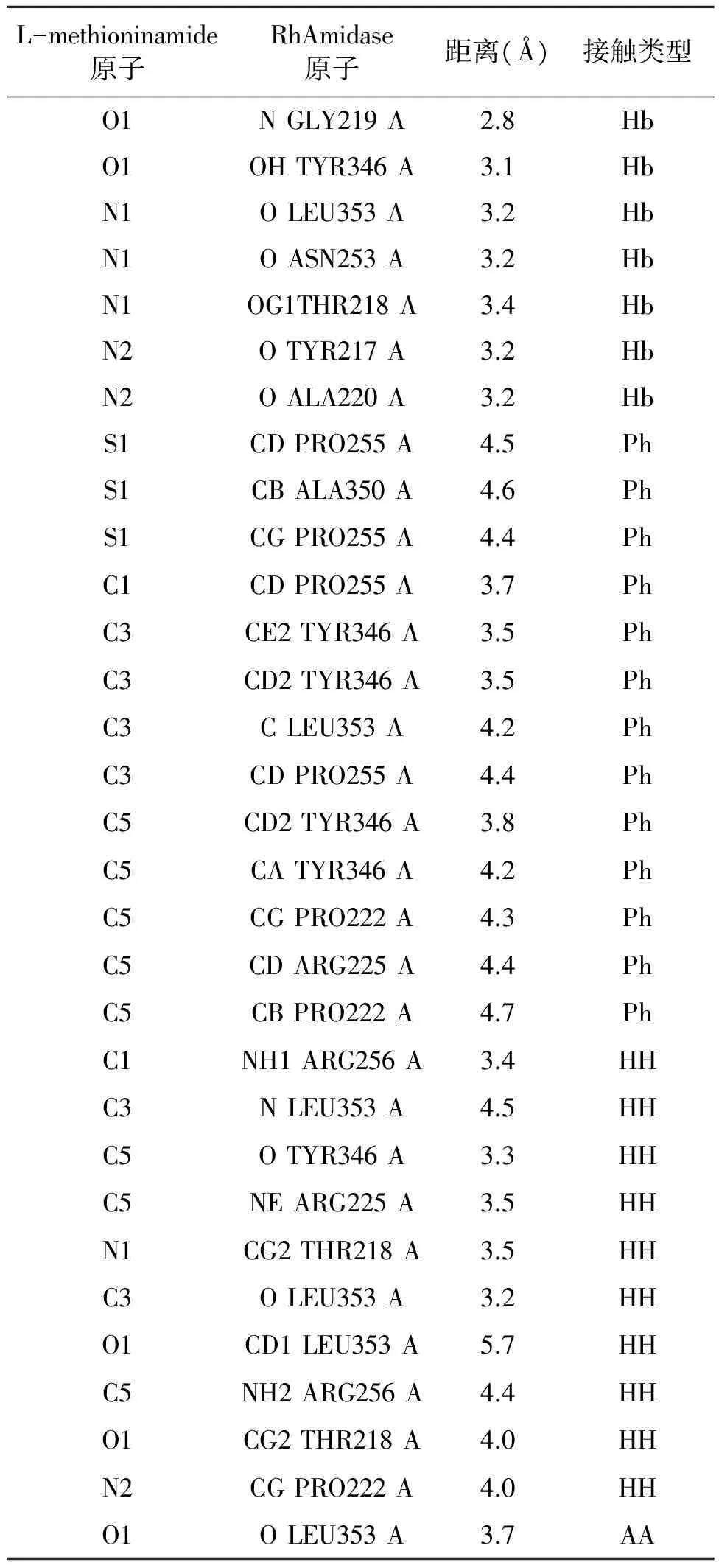
表3 RhAmidase 和L-methioninamide之间的相互作用Table 3 Interactional profile of RhAmidase with L-methioninamide.
注:Hb:氢键; Ph:疏水作用; HH:亲水-疏水作用; AA:芳香-芳香作用。

图3 RhAmidase/L-methioninamide复合物间的 相互作用Fig.3 Interactions of RhAmidase/L-methioninamide complex.注:RhAmidase的3D结构用黄色条状模型表示; L-methioninamide的3D结构用绿色条状模型表示。相互作用用虚线显示。 (彩图见图版七)
腈水合酶的立体选择性较低,酰胺酶的立体选择性较高。在生物细胞中,腈水合酶和酰胺酶的结构基因处于同一个操纵子上,腈水合酶的合成时常伴随着酰胺酶的存在,利用细胞中的腈水合酶和酰胺酶的级联反应可以实现良好的立体选择性转化,利用纯的腈水合酶或者抑制细胞中酰胺酶活性可以实现酰胺的生产。在腈水合酶和酰胺酶的作用下,具光学性质的腈不但能转化成光活性的羧酸和酰胺,还能获得很高的对映体过量值(ee)。
RhAmidase有3个结构域、1个N端α螺旋结构、1个小域和1个大域。N端α螺旋结构域在其他的酰胺酶家族中不存在,这个区域与二聚物结构形成有关,和小域一起形成1个窄的底物结合通道。大域与其他酰胺酶家族在结构上表现出高度的相似性。Ser-cisSer-Lys催化三联体位于大域。但RhAmidase的底物结合口袋相对较窄,由于小结构域中螺旋α13的存在。来自小域的疏水性残基参与底物识别。小域可能参与了底物识别,它与底物特异性的家族酰胺酶之间的差异有关。
3 讨论
酰胺类化合物在工业上用途广泛,也是有机合成的重要中间体。微生物修复技术是解决这类环境问题的有效途径之一。本研究分析了酰胺酶RhAmidase 与底物间的结合模式,并且成功确定了复合体RhAmidase/L-methioninamide的相互作用剖面图。RhAmidase/底物复合体之间的相互作用有氢键、疏水作用、亲水-疏水作用,出现在每种酰胺底物结合到相应的酰胺酶时。RhAmidase和L-methioninamide 之间的氢键作用由残基GLY219 A、TYR346 A、LEU353 A、ASN253 A、THR218 A、TYR217 A 和ALA220 A形成。PRO255 A、ALA350 A、TYR346 A、LEU353 A、ARG225 A 和PRO222 A 与配体L-methioninamide形成疏水作用。疏水作用在 RhAmidase和L-methioninamide之间大量存在,可能是产生RhAmidase/L-methioninamide复合体的主动力。6个残基ARG256 A、LEU353 A、TYR346 A、ARG225 A、THR218 A和PRO222 A在RhAmidase以亲水-疏水作用结合到L-methioninamide 时起重要作用。这些数据在今后设计对酰胺有更好的氧化活性的酰胺酶突变体时非常有用。
分子对接是计算机模拟分子间结合模式的一种重要方法,最初应用于计算机辅助药物设计。目前有研究者将其引入环境科学领域,用于研究生物降解中酶和污染物的相互作用,揭示酶的作用机理。陈明等[14]将分子对接和分子模拟的方法引入了固体废弃物堆肥研究中,探索了木质素过氧化物酶、锰过氧化物酶和漆酶这3种木质素降解酶和木质素小分子之间的结合模式;林玉珍等[15]用分子对接方法模拟了滴滴涕与漆酶、毒死蜱与有机磷水解酶的相互作用,发现滴滴涕与漆酶之间以疏水作用数量最多,毒死蜱与有机磷水解酶以氢键和疏水作用数量最多。这些研究为提高有机污染物的降解效率奠定了理论基础。
在腈水合酶和酰胺酶的作用下,具有光学性质的腈能转化成光活性的羧酸和酰胺。α-芳基丙腈类衍生物水解产生的α-芳基丙酸具有光学活性,是拟除虫菊酯和非甾体抗炎药的重要中间体。Rhodococcussp. CGMCC 0497催化α-芳基丙腈水解成光活性的S-羧酸和R-酰胺,α-芳基丙腈上间位和对位取代的底物具有较好的对应选择性。王梅祥等[16]报道Rhodococcussp. AJ270产生的腈水合酶和酰胺酶能催化外消旋2-甲基-3-苯基环氧乙烷腈、2-芳基-4-戊烯腈类、α-氨基腈和α-甲基(α-乙基、α-异丙基)苯乙腈转化成相应的光活性酰胺和羧酸,ee值高达99%。Christian等[17]发现Klebsiellaoxytoca38.1.2产生的腈水合酶能立体选择性催化光学活性的2-苯基丙腈、2-苯基甘氨酸腈和苯乙醇腈生成对应的S-型酰胺。此外,假单胞菌属(Pseudomonas)、红球菌属(Rhodococcus)等多种菌所产腈水合酶和酰胺酶都表现出较高的立体选择性[5]。
[1] 何庆林,白艳芬,周 威,等.利用糖基转移酶UGT73B6在大肠杆菌中生物催化酚苷类天然产物[J].生物技术通报,2017,33(11):1-6.
[2] Santoshkumar M, Ismailsab M, Anand S N,etal.. Purification and characterization of amidase fromParacoccussp. SKG: Utilization of amidase-inhibited whole cells for bioconversion of acrylonitrile to acrylamide[J].Biocatal. Agric. Biotechnol., 2017,10: 256-263.
[3] Divya M, Benny T, Devaky K S. Phosphonate TSA-built macromatric polymer catalysts as chymotrypsin mimics for the amidolysis of amino acid p-nitroanilides: Effect of the nature and extent of crosslinker on amidase activities[J].Polymer,2017,111:285-296.
[4] 蒲 桃,王宏宇,刘 英,等.新型2-甲基-3-呋喃甲酰胺类化合物的设计、合成及抑菌活性研究[J].四川大学学报(自然科学版),2017,1:178-184.
[5] Fang S M, An X J, Liu H Y,etal.. Enzymatic degradation of aliphatic nitriles byRhodococcusrhodochrousBX2, a versatile nitrile-degrading bacterium[J].Bioresour. Technol.,2015,185:28-34.
[6] Kohyama E, Yoshimura A, Aoshima D,etal.. Convenient treatment of acetonitrile-containing wastes using the tendem combination of nitrile hydratase and amidase-producing microorganism[J].Appl. Microbiol. Biotechnol.,2006,72: 600-606.
[7] Li T, Liu J, Bai R,etal.. Biodegradation of organonitriles by adapted activated sludge consortium with acetonitrile-degrading microorganisms[J]. Water Res.,2007,41(15):3465-3473.
[8] Maestracci M, Thiery A, Arnaud A,etal.. A study of the mechanism of the reactions catalyzed by the amidaseBrevibacteriumsp. R312[J]. Agric. Biol. Chem., 1986,50:2237-2241.
[9] Thomsen R, Christensen M H. MolDock: A new technique for high-accuracy molecular docking[J]. J. Med. Chem.,2006,49(11): 3315-3321.
[10] DeAzevedo W F J. MolDock applied to structure-based virtual screening[J]. Curr. Drug Targets,2010,11(3):327-334.
[11] Sobolev V,Sorokine A,Prilusky J,etal.. Automated analysis of interatomic contacts in proteins[J]. Bioinformatics,1999,15(4): 327-332.
[12] Ohtaki A, Murata K, Sato Y,etal.. Structure and characterization of amidase fromRhodococcussp. N-771: Insight into the molecular mechanism of substrate recognition[J]. Biochim. Biophys. Acta,2010, 1804: 184-192.
[13] Lee S, Park E H, Ko H J,etal.. Crystal structure analysis of a bacterial aryl acylamidase belonging to the amidase signature enzyme family[J]. Biochem. Biophys. Res. Commun.,2015, 467(2): 268-274.
[14] Chen M,Zeng G M,Tan Z Y,etal.. Understanding lignin-degrading reactions of ligninolytic enzymes: Binding affinity and interactional profile[J].PLoS ONE, 2011,6 (9): e25647.
[15] 林玉珍,曾光明,张 娱,等.有机农药滴滴涕和毒死蜂生物降解机制的分子模拟研究[J]. 环境科学, 2012,33(3):1015-1019.
[16] Wang M X, Lu G, Ji G J,etal.. ChemInform abstract: Enantioselective biotransformations of racemic α-substituted phenylacetonitriles and phenylacetamides usingRhodococcussp. AJ270 [J]. Tetrahed. Asym.,2000,11(5):1123-1135.
[17] Christian E, Sabine L W, Lutz F. Enantioselective conversion of α-arylnitriles byKlebsiellaoxytoca[J]. Tetrahed. Asym.,2008,19(22):2573-2578.

