SMILES表达式的子结构关系检测算法
彭 彬,刘 钊,顾进广
(1.武汉科技大学 计算机科学与技术学院,湖北 武汉 430065;2.武汉科技大学 智能信息处理与实时工业系统湖北省重点实验室,湖北 武汉 430065)
0 引 言
寻找一种方法能精确简洁地获取化合物结构信息是化合物检索研究领域首先需要解决的问题[1]。当前研究人员提出了许多研究方案,对结构检索做出了较大的贡献。其主流的研究方向有两种,线性码、连接表。线性码的应用主要有SMILES编码[2,3]和SMARTS编码[4],连接表的研究主要有Ullmarm算法[5]与VF算法[6]。其中基于线性码的SMARTS编码,作为一种有效化合物结构查询方法,被人们广泛使用。
SMARTS编码结构检索的机制是通过输入的SMILES重构化学式,再搜索子图的同形,而不是直接通过SMILES的对比完成的。通过对SMILES编码改造的方式达到检索目的,虽然搜索结果准确性较高,但这种避开分析SMILES编码去重新构造新的编码的方式并不能解决某些场合下的问题。如SMILES表达式可以被一些化学式编辑软件如Marvin导入并转换成二维图形或三维分子模型,若判断编辑的化学式之间的结构关系,则需要分析SMILES表达式的结构信息;再如SMILES表达式与MOL[7]文件都是存储化合物的结构信息,且SMILES表达式能够与MOL文件进行相互转换,通过分析SMILES表达式之间的结构关系可以判断MOL文件间的关系,为之后MOL文件的查找跟分类的研究提供参考。本文以上述研究为基础,提出一种检测SMILES表达式之间子结构关系的算法,通过分析SMILES表达式来获取子结构关系信息。
1 算法思路
在引言中指出,一些应用场景如以SMILES作为结构信息载体的化合物结构匹配,MOL文件分类,需要通过分析SMILES表达式所包含的结构信息去获取化合物之间的结构关系,但通过分析SMILES表达式来检测化合物之间的结构关系,至今还没有一种好的方法。考虑到化合物结构的特殊性和复杂性,子结构查询中所涉及到的子串不能通过简单的子串匹配获取结构关系[8,9]。
针对上述问题,本文的思路是研究一种能确定SMILES表达式之间子结构关系的算法,该算法包括:定义并存储常见原子、化学键和原子间的支链关系;定义切片最小粒度:相邻原子与之间的化学键关系与支链关系作为最小粒度单位;定义起始原子和终止原子,其中起始原子为所述最小粒度对应的第一个原子,终止原子为所述最小粒度对应的第二个原子;基于常见原子、化学键和原子间的支链关系对获取的SMILES表达式进行分析,将SMILES表达式切割成一个个原子对,统计SMILES表达式包含的环个数及原子对的种类、每种原子对的个数并存储到哈希表中;根据原子对的种类、每种原子对的个数、环的个数来确定SMILES表达式之间子结构关系。
2 算法介绍
该算法的目标是确定SMILES表达式之间是否具有子结构关系。该算法包括:
(1)定义并存储常见原子、化学键和支链关系。
(2)定义切片最小粒度:相邻原子和原子之间的化学键关系以及支链关系三者共同决定最小粒度单位。
(3)定义起始原子和终止原子,其中起始原子为所述最小粒度对应的第一个原子;终止原子为所述最小粒度对应的第二个原子。
(4)基于常见原子、化学键和支链关系对获取的SMILES表达式进行分析。将SMILES表达式切割成一个个原子对,统计SMILES表达式包含的环的个数及原子对的种类、每种原子对的个数并将统计结果存储到哈希表中,其中所述原子对根据相邻原子与之间的化学键关系与支链关系确定。
(5)根据原子对的种类、每种原子对的个数、环的个数确定SMILES表达式之间是否具有子结构关系。
算法的主要步骤、切片过程、获取原子、匹配过程4个部分的详细说明如下:
2.1 算法主要步骤
(1)确定常见原子、化学键、支链关系。常见的原子(Atom):H,C,N,O,S,F,Cl,Br,[N+],[N-],[O+],[O-]。常见的化学键(Bond):单键,双键,三键,楔型向上键,楔形向下键,离域键,顺反不确定键,空心键,不确定键。
(2)分析SMILES表达式包含的信息。SMILES存储的信息主要包含原子的种类、原子之间的化学键、支链关系。
(3)定义切片最小粒度:相邻原子、相邻原子之间的化学键与支链关系作为最小粒度单位。决定化学式分子式结构的主要因素有原子的种类,原子之间的化学键,支链关系。
(4)定义起始原子和终止原子。其中起始原子为所述最小粒度对应的第一个原子;终止原子为所述最小粒度对应的第二个原子。
(5)切片处理。将待匹配的SMILES表达式进行切割成一个个原子对。统计原子对的种类、个数,并存储到哈希表中。
(6)匹配判断。根据切片种类、切片对应的数量来判断SMILES表达式之间是否具有子结构匹配关系。
本算法会设置3个常用的变量来存储切片结果。分别为切片种类(Kh),每种切片的数量(Ch),环结构数量(circle)。
2.2 切片过程
根据算法步骤(5)中提到,需要将SMILES表达式按照预定义的最小粒度进行切割。考虑到SMILES表达式环结构是用数字表示,故切片第一步需要获取环结构信息,去掉SMILES表达式中的数字,以简化后续的切片过程。本文将切片过程分为两个子步骤:获取环结构信息,获取切片信息。其中获取环结构信息算法描述如下:
步骤1 预处理。因为此次匹配流程是不考虑异构SMILES情况,所以首先会对那些存储异构 SMILES的字符串剔除异构字符。如"/",""。
步骤2 统计环的个数与环边拆分位置的原子对。其详细过程如下:
(1)遍历SMILES表达式每一个字符,观察是否有数字字符。
(2)若包含数字,记住该数字的位置,并从该位置后继续遍历直到找到包含同样数字字符,环个数加1。存储环边拆分位置的原子对。过滤掉这两个位置的数字,组成新的字符串。
(3)对新组成的字符串进行遍历寻找下一个数字。
(4)当新组成的字符串无数字字符,存储环个数,则环个数统计结束。
算法1:获取环结构信息
Algorithm 1getCircleInfo()
Input:
Setstr←smiles;h=0;Kh={φ};Ch={φ};circle=0
(1)str⟸preHandle(str)
(2)fori=0;i (3)ifisNumber(str[i])then (4)num=str[i] (5)forj=i;j (6)ifstr[i]==str[j]then (7)ifisBond(str[j-1])then (8)piece=getPrevAtom(str[i])+str[j-1] (9)piece=piece+getNextAtom(str[j]) (10)ifpiece∈Kh,piece==Kh[t]then (11)Ch[t]←Ch[t]+1 (12)else (13)Kh+1[t]←Kh[t]∪{piece};Ch+1[t]←Ch[t]∪{φ} (14)h←h+1 (15)endifendifendifendfor (16)circle←circle+1 (17)endifendfor (18)str⟸removeNumber(str) Output: str,h,Kh,Ch,circle SMILES表达式经过算法getCircleInfo(algorithm 1)处理后,将输出参数作为算法getPiece(algorithm 2)的输入参数。其获取切片信息的算法描述如下: 步骤3 对经过预处理与去环结构后的SMILES字符串进行循环遍历,判断str[i]的值情况。 步骤4 若为"=",则化学键="="。起始原子为该位置前的第一个原子,终止原子为该位置后的第一个原子。 步骤5 若为"#",则化学键="#"。起始原子为该位置前的第一个原子,终止原子为该位置后的第一个原子。 步骤6 若为"(",该位置前的第一个原子也就是起始原子与左括号内的内容,组成新的字符串,设置为str1;起始原子与对应右括号后的内容,组成str2;将str1,str2作为新的 SMILES表达式进行切片流程处理。此方式运用的迭代算法。 步骤7 若为")",位置指针向左寻找,找到与该括号匹配的左括号位置,并将左括号左边的第一个原子作为起始原子,位置为起始位置。 步骤8 若为其它字符,则化学键=""。起始原子为该位置前的第一个原子,终止原子为该位置后的第一个原子。 步骤9 根据原子对=startAtom+bond+stopAtom,得到原子对的值,并存储到哈希表中。 步骤10 将此次切片结果存储到哈希表中,key值为片段名,value存储为该片段的个数。切片结果展示的是SMILES结构式被切片后的切片种类与每种切片对应的个数。 算法2:切片过程 Algorithm 2getPiece() Input: Setstr,h,Kh,Ch,circlefromgetCircleInfo() (1)fori=0;i (2)ifstr[i]=="("then (3)str←s1(s2)s3;getPiece(s1(s2));getPiece(s1s3) (4)elseifstr[i]==")"then (5)i←i+1 (6)else (7)ifstr[i]=="#"‖str[i]=="="then (8)bond←str[i] (9)end if (10)startAtom,start⟸getPrevAtom(i) (11)stopAtom,stop⟸getNextAtom(start) (12)i←stop;piece=startAtom+bond+stopAtom (13)ifpiece∈Kh,piece==Kh[t]then (14)Ch[t]←Ch[t]+1 (15)else (16)Kh+1[t]←Kh[t]∪{piece};Ch+1[t]←Ch[t]∪{φ} (17)h←h+1 (18)endifendifendfor Output: str,Kh,Ch,circle 切片过程中有提到获取起始原子,终止原子的方法,也就是算法伪代码中的getPrevAtom(),getNextAtom()。本小节将对SMILES表达式中获取指定位置原子的过程做详细说明。获取SMILES表达式对应位置i的原子公式如下 步骤1 从起始位置对应的字符进行判断,将常用的双原子列表存入配置文件中。其中常见的双原子有Br,Cl。 步骤2 判断是否为双原子。位置i的字符设置为ch1,若ch1对应双原子第一个字符,且位置i+1的字符ch2对应双原子第二个字符,则起始原子=ch1+ch2。若不是,则起始原子=ch1。 步骤3 判断是否为括号。若为括号,当前位置加1,继续对新的字符判断。 步骤4 判断是否存在离子。若存在"["字符,则继续寻找直到遇到"]"字符。则"["与"]"之间组成的字符串即为起始原子,起始原子="["+ch1+...+chi+...+chn+"]"。 获取切片结果集后,需要根据切片结果集进行匹配检测。匹配方式如图1所示。将两个待匹配的SMILES表达式进行切片处理,待匹配的SMILES表达式切片结果记为A,被匹配的SMILES表达式结果记为B。将A跟B的结果进行比较: 步骤1 若A的环个数大于B的环个数,则匹配不成功,不具有子结构关系。 步骤2 若A种类大于B种类,则匹配不成功,不具有子结构关系。 步骤3 若A每种切片个数大于B对应切片个数,则匹配不成功,不具有子结构关系。 步骤4 不满足上述条件时,匹配成功,具有子结构关系。 图1 SMILES匹配流程 本文的实验数据是参照《合成香料技术手册》[10]提到的1001种合成香料,根据CAS[11]编号从相关化工站点中抓相关香料信息,其中包含SMILES表达式的香料有638种。本文从ChemNet网中抓取638条化工香料,存储到xml文档中,文档中数据共包含638种CAS号,每种CAS号对应着一种SMILES表达式,其中数据的来源分布见表1。 表1 化工香料数据的种类 实验分成两个过程:①检查香料切片的准确性;②将香料的子结构与香料结构进行对比,检测匹配的准确性。判断SMILES表达式之间结构关系的准确性满足式(1) AR=SR*MR (1) 其中,AR为子结构准确率,SR为切片的准确率,MR为匹配的准确率。 将抓取后的638种化工香料进行算法切片,存储每种香料的切片结果。同时将这些切片进行人工切片处理,将切片处理结果跟算法切片结果进行对比,若算法切片跟人工切片结果相同,则切片准确。如对香料檀香210进行切片处理,按照切片定义,最小切片种类有CC,C(C),C=C,circle。其中CC代表起始原子与终止原子均为碳原子,原子之间以单键相连;C(C)代表起始原子与终止原子为碳原子。原子之间以单链相连,并具有支链关系;C=C代表起始原子与终止原子为碳原子。原子之间以双链相连;circle代表此香料化学式拥有环状结构的个数。切片结果见表2。 根据切片结果可知,算法切片将该化合物SMILES切割成CC,C(C),C=C,C=O这4种片段,具有circle结构,个数分别为8,5,1,1,1。算法切片跟人工切片结果一致,故可判定对香料檀香210进行切片处理准确。经过实验,对638种香料进行切片处理,其提取出的信息总条数为638,提取出的正确信息条数为636,根据处理式(2)求出切片正确率=636/638=99.7% 表2 檀香210的切片结果 SR=CI/TI (2) 其中,CI为提取正确信息条数,TI为提取信息总条数。 再从638条香原料中随机抽取50条香料数据做匹配实验,通过对每一条香料可能存在的子结构与此香料结构进行匹配验证。匹配的正确性是通过将每一条香料子结构的SMILES与该香料结构的SMILES进行匹配检测,若匹配成功则具有子结构关系。如对3-蒈烯进行匹配实验,首先对该化合物进行切片处理,切片结果见表3;再对3-蒈烯可能存在的子结构进行切片处理,切片结果见表4。将子结构的切片结果与3-蒈烯的切片结果进行对比,通过匹配算法判断是否匹配,进而验证这个子结构与该化合物的匹配关系,实验数据见表4。 表3 3-蒈烯切片结果 表4 3-蒈烯子结构的匹配结果 根据匹配结果可知,3-蒈烯所有可能子结构的SMILES与该香料结构的SMILES进行匹配检测,3-蒈烯所包含的子结构按照图1匹配算法得出的结论是匹配的。经过实验,50种香料共计928种子结构。匹配正确的条数921条,错误条数7条。根据处理式(3)求出匹配准确率=921/928=99.2% MR=CI/TI (3) 根据切片实验跟匹配实验的结果可知,切片算法的准确率为99.7%,匹配算法的准确率为99.2%。根据处理式(1)得出SMILES表达式之间是否具有子结构关系的准确率为98.9%,具有很高的准确性。其中切片过程中0.3%错误率是因为香料存在一个CAS号对应多个SMILES表达式的情况,而本文目前只考虑的一种CAS号对应一种SMILES表达式;匹配过程0.8%的错误率是因为SMILES表达式中环位置标记数字是相同的,代表着环之间不交叉,如表达式C1CC1C1CC1。本算法中考虑的是SMILES表达式中每种环位置的标记数字是不同的。 本文的实验数据来自香料企业生产的1001种合成香料,以算法切片匹配做实验组,人工切片匹配作对照组。通过实验可知,该算法检测SMIELS表达式之间子结构关系的准确性很高。本算法通过将SMILES表达式切割成一个个不可再分的片段,再去比较各片段的数量来判断化学式之间是否具有子结构匹配关系,本算法可用于判断化合物之间的结构关系。该方法简单,匹配准确性高,可为下一步做基于SMILES结构检索数据库的工作做依据参考。 [1]Toropov A A,Toropova A P,Martyanov S E,et al.Comparison of SMILES and molecular graphs as the representation of the molecular structure for QSAR analysis for mutagenic potential of polyaromatic amines[J].Chemometrics and Intelligent Laboratory Systems,2011,109(1):94-100. [2]Suzuki M,Manago N,Ozeki H,et al.Sensitivity study of SMILES-2 for chemical species[C]//Sensors,Systems,and Next-Generation Satellites XIX,2015. [3]Brefo-Mensah E K,Palmer M.mol2chemfig,a tool for rendering chemical structures from molfile or SMILES format to LATE X code[J].Journal of Cheminformatics,2012,4(1):24. [4]Dalke A.Parsers for SMILES and SMARTS[J].Chemistry Central Journal,2008,2(1):1. [5]Bonnici V,Giugno R,Pulvirenti A,et al.A subgraph isomorphism algorithm and its application to biochemical data[J].BMC Bioinformatics,2013,14(7):1-13. [6]LI Yan,ZHOU Jiaju.Application of VF algorithm in chemical structure retrieval[J].Computer and Applied Chemistry,2002,19(5):575-576(in Chinese).[李琰,周家驹.VF算法在化学结构检索中的应用[J].计算机与应用化学,2002,19(5):575-576.] [7]Dallakian P,Haider N.FlaME:Flash molecular editor-a 2D structure input tool for the web[J].Journal of Cheminforma-tics,2011,3(1):6. [8]LI Chuangye.Network retrieval of compound structure[D].Tianjin:Hebei University of Technology,2007(in Chinese).[李创业.化合物结构的网络检索[D].天津:河北工业大学,2007.] [9]PAN Kai.The design and implementation of the retrieval method of chemical molecular substructure in ChemDataBase database[D].Lanzhou:Lanzhou University,2009(in Chinese).[潘凯.ChemDataBase数据库中化学分子子结构检索方法的设计与实现[D].兰州:兰州大学,2009.] [10]LIU Shuwen.Handbook of synthetic perfume technology[M].Beijing:China Light Industry Press,2000(in Chinese).[刘树文.合成香料技术手册[M].北京:中国轻工业出版社,2000.] [11]Makarova K S,Koonin E V.Evolution and classification of CRISPR-Cas systems and Cas protein families[M]//CRISPR-Cas Systems.Springer Berlin Heidelberg,2013:61-91.2.3 获取原子

2.4 匹配过程
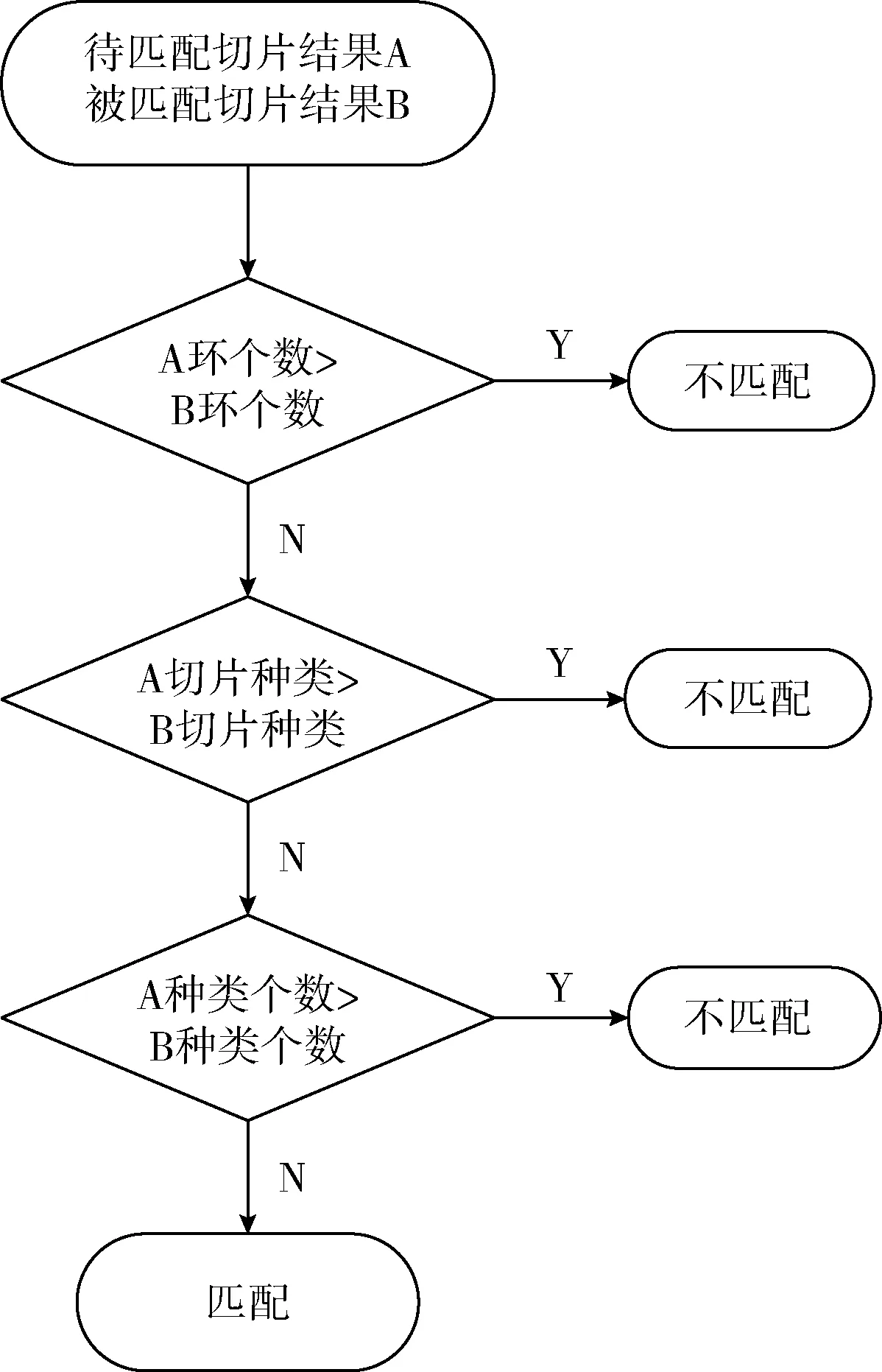
3 实验与评估

3.1 切片实验

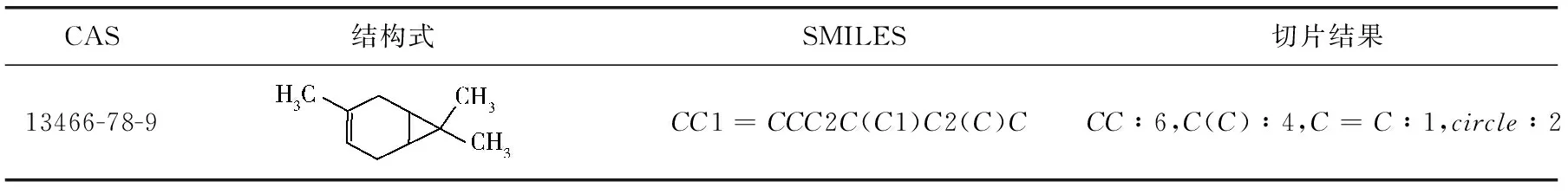
3.2 匹配实验

3.3 实验结论
4 结束语
——香草传

