牦牛KDM4A基因克隆、组织表达谱及其在卵母细胞和颗粒细胞中的表达
韩 杰,熊显荣,蔡雯祎,杨显英,阿果约达,张燕红,李 键
(西南民族大学生命科学与技术学院,成都 610041)
牦牛 (Bosgrunniens)生活在我国高原及其周围海拔3 000 m以上高山地区,能够很好地适应高寒、缺氧的生态环境,能充分利用高原地区天然草地为人们提供优质健康的食品。然而牦牛性成熟晚,繁殖和生长性能低下,严重阻碍了牦牛业和经济的发展。近些年,对牦牛育种的研究不断深入,并取得了一定的进展[1-2]。牦牛在动物基因遗传资源研究中也是一个极其珍贵的基因库[3],因此,牦牛的研究对提高牧区的生活水平和丰富中国遗传资源多样性具有重大意义。
组蛋白赖氨酸甲基化修饰是一个可逆过程,分别由赖氨酸甲基化转移酶(KTMs)和去甲基化酶(KDMs)两个家族催化完成,其中KDM家族成员均包含JmjC催化结构域蛋白,这些JmjC催化结构域属于cupin金属酶催化超家族[4-5]。这些酶在酵母、人、小鼠等物种间高度保守[6],并且可以使甲基化的组蛋白脱甲基化,改变其表观结构,通过调节染色质构造、损伤和修复及基因活性等作用,进而对基因组功能进行调控[7]。KDM4家族是由4个成员构成,包括:KDM4A、KDM4B、KDM4C和KDM4D,它们能够维持特异性催化H1.4K26me2/3、H3K9me2/3和H3K36me2/3的去甲基化[8]。KDM4组蛋白去甲基化酶家族除了包含JmjC催化结构域外,一侧还包括JmjN催化结构域,同样是催化组蛋白尾去甲基化所必须的[9-11]。组蛋白赖氨酸去甲基化酶4A(又称为KDM4A/JMJD2A/JHDM3A)可以催化H3K9me2/3和H3K36me2/3去甲基化[12]。对KDM4A基因的研究表明,其蛋白和mRNA广泛存在于生殖系统以及各个组织中,且KDM4A基因可通过影响周期蛋白和激素受体来间接调控生殖细胞的活动,进而参与生殖系统的调控[12-17]。
目前,关于KDM4A基因的研究主要集中在人和动物的生长发育和肿瘤等相关疾病上,而该基因在牦牛生殖细胞中的研究未见相关报道,为进一步研究KDM4A基因在牦牛卵母细胞及颗粒细胞中的作用,本研究克隆获得牦牛KDM4A基因,并对该基因序列进行生物信息学及蛋白功能结构预测分析;应用实时荧光定量PCR技术检测牦牛KDM4A基因在颗粒细胞和卵母细胞成熟过程中的表达情况,为进一步研究牦牛的KDM4A基因及其在牦牛生殖生理过程中所发挥的作用奠定基础。
1 材料与方法
1.1 主要试剂与仪器
Trizol Reagent购自Invitrogen(美国)公司,PrimeScriptTMRT Reagent Kit反转录试剂盒、Premix TaqTMDNA聚合酶、pMDTM19-T载体、SYBR®Premix Ex TaqTMⅡ试剂盒均购自TaKaRa(大连)公司,DNA胶回收试剂盒购自Axygen(北京)公司, DNA Marker、感受态细胞DH5α均购于天根生化科技(北京)有限公司,卵母细胞成熟液M199(添加雌二醇、FSH、LH、血清成分)购自Gibco公司,其他无特殊说明均为国产分析纯生化试剂。离心机、荧光定量PCR仪、琼脂糖凝胶成像系统、电泳仪、恒温培养箱均购自美国Biodine rad公司。
1.2 牦牛组织样品的采集
试验样品采自四川成都青白江区向阳镇屠宰场,屠宰后,立即无菌采集牦牛心、肝、脾、肺、肾、小肠、卵巢、子宫、胃、睾丸、大脑11个组织;所有样品放入已编号的冻存管中,迅速将其投入液氮中保存。
1.3 牦牛卵母细胞的采集和成熟培养
采集的卵巢,用含双抗的生理盐水(80 mg·L-1青霉素和100 mg·L-1链霉素)冲洗干净,放入25 ℃生理盐水(预先放入双抗)的保温瓶中,在2 h内送回实验室。用加有双抗的37 ℃生理盐水冲洗牦牛卵巢3遍后,使用一次性注射器(12号针头)抽取直径约为3~8 mm卵泡的卵泡液,将获得的卵泡液置于90 mm的平皿中,轻轻摇匀,在体视显微镜下,收集卵丘-卵母细胞复合体(Cumulus-oocyte complex,COCs)。将所取得的复合体放入卵母细胞成熟液中清洗3遍,移入50 μL的卵母细胞成熟液的微滴(每个微滴中放25个卵母细胞并由甘油覆盖)中,38.5 ℃,5% CO2,高湿度条件下培养[18],分别收集GV期、MI期和MII期卵母细胞及颗粒细胞,置于去RNA酶的离心管中。
1.4 总RNA的提取和反转录
按照Trizol法提取样本组织的总RNA。采用Cell-to-cDNATMII Kit试剂盒,对培养得到的不同时期的颗粒细胞和卵母细胞进行总RNA的提取。应用核酸分析仪测其纯度和浓度,选用OD值为1.8~2.0的RNA作为模板。根据反转录试剂盒PrimeScriptTMRT Reagent Kit说明书以Oligo(dT)为引物,对总RNA进行反转录合成cDNA第一链,分别编号后,-20 ℃保存。
1.5 引物设计及合成
根据NCBI中已报道的黄牛(Bostaurus)KDM4A(GenBank登录号:NM_001206316.1)和GAPDH(GenBank登录号:AC_00162.1)基因的mRNA序列,使用Primer5.0软件设计引物(表1),由成都擎科梓熙生物技术有限公司合成。
1.6 目的基因的扩增克隆
以牦牛卵巢cNDA为模板,利用PCR技术扩增牦牛KDM4A基因CDs区序列。PCR反应体系为25 μL,其中 cNDA 1.0 μL,Premix TaqTMDNA聚合酶12.5 μL,上、下游引物(10 μmol·L-1)各1.0 μL, ddH2O 9.5 μL;PCR扩增条件:94 ℃预变性4 min;94 ℃变性45 s,58 ℃退火45 s(前两段目的基因退火温度为58 ℃,第3段目的基因退火温度为56 ℃),72 ℃延伸1 min,35个循环;72 ℃ 7 min。并使用15 g·L-1琼脂糖凝胶电泳检测扩增结果,根据DNA胶回收试剂盒说明书切胶回收目的条带,然后将回收产物与pMDTM19-T载体在16 ℃连接过夜,第2天将DH5α感受态细胞均匀涂于已制备好的LB固体培养基(AMP+)平板上,37 ℃培养过夜后挑取白色单菌落,接种于LB液体培养基(AMP+)中摇床培养6 h,以菌液为模板进行PCR鉴定,将鉴定结果呈阳性的菌液,送成都擎科梓熙生物技术有限公司测序。
表1 引物信息
Table 1 Primer information
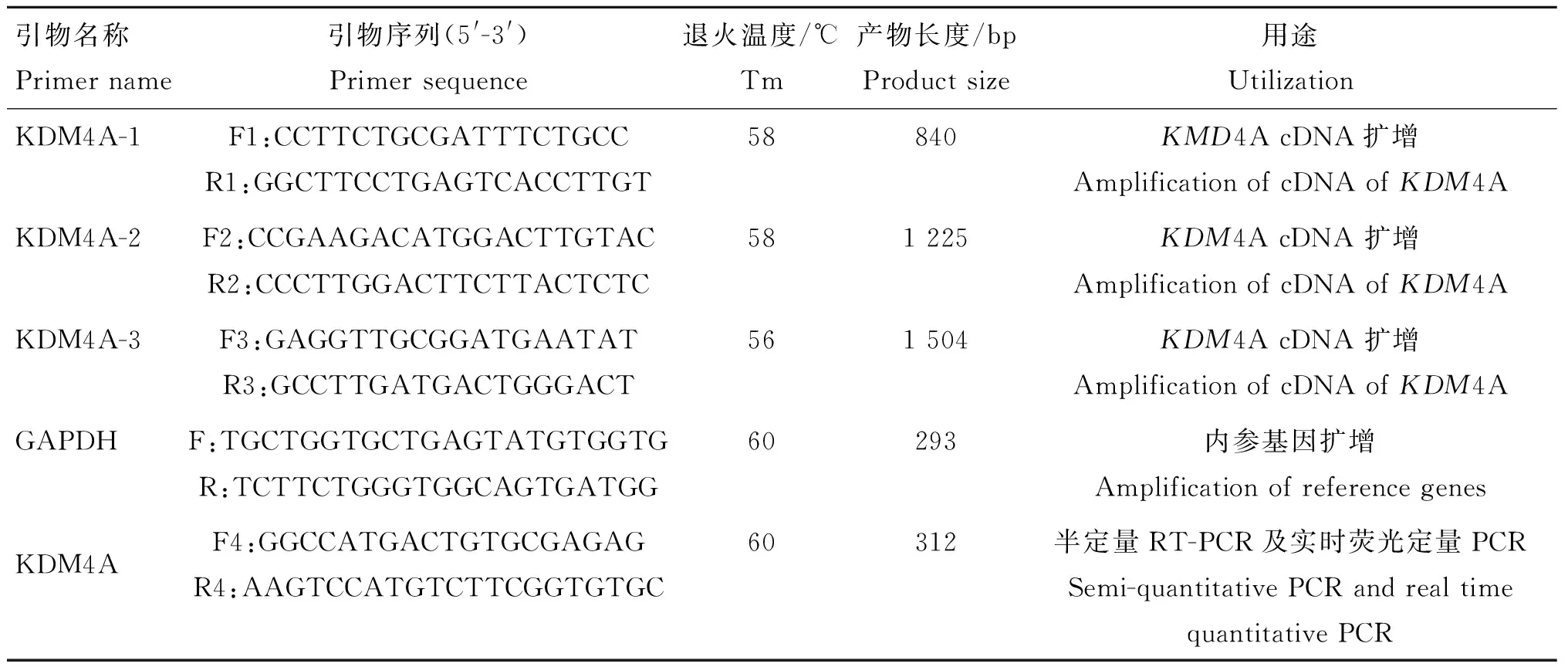
引物名称Primername引物序列(5'-3')Primersequence退火温度/℃Tm产物长度/bpProductsize用途UtilizationKDM4A-1F1:CCTTCTGCGATTTCTGCCR1:GGCTTCCTGAGTCACCTTGT58840KMD4AcDNA扩增AmplificationofcDNAofKDM4AKDM4A-2F2:CCGAAGACATGGACTTGTACR2:CCCTTGGACTTCTTACTCTC581225KDM4AcDNA扩增AmplificationofcDNAofKDM4AKDM4A-3F3:GAGGTTGCGGATGAATATR3:GCCTTGATGACTGGGACT561504KDM4AcDNA扩增AmplificationofcDNAofKDM4AGAPDHF:TGCTGGTGCTGAGTATGTGGTGR:TCTTCTGGGTGGCAGTGATGG60293内参基因扩增AmplificationofreferencegenesKDM4AF4:GGCCATGACTGTGCGAGAGR4:AAGTCCATGTCTTCGGTGTGC60312半定量RT-PCR及实时荧光定量PCRSemi-quantitativePCRandrealtimequantitativePCR
F.正向引物;R.反向引物
F.Forword primer; R.Reverse primer
1.7 牦牛KDM4A基因生物信息学分析
利用NCBI中ORF Finder对获得的KDM4A序列进行开放阅读框分析,并获得氨基酸序列,同时进行氨基酸相似性比对;利用在线软件ExPASY工具包中Protparam、ProtScale、SignalP、TMHMM、NetPhos、SOPMA、SWISS-MODEL及SMART分别对牦牛KDM4A蛋白的理化性质、疏水性、信号肽、跨膜区、磷酸化位点、二级结构、三级结构及结构域进行预测分析;在NCBI中找到需要建树的相关物种该基因序列,下载保存后,并利用MEGA 5.0软件进行比对和系统进化树构建。
1.8 牦牛KDM4A基因组织表达谱
采用RT-PCR检测KDM4A基因在牦牛心、胃、肝、肺、卵巢、脾、肾、小肠、子宫、睾丸、大脑组织中的表达,RT-PCR反应体系为25 μL: Premix TaqTMDNA聚合酶12.5 μL,上、下游引物(10 μmol·L-1)各1.0 μL,各组织cNDA模板1.0 μL, ddH2O 9.5 μL;PCR反应条件:94 ℃ 4 min;94 ℃ 45 s,60 ℃ 30 s, 72 ℃ 30 s,40个循环;72 ℃ 7 min。最后使用10 g·L-1琼脂糖凝胶电泳检测结果。
1.9 牦牛卵母细胞和颗粒细胞中KDM4A基因表达分析
采用实时荧光定量PCR(Quantitative real-time PCR, RT-qPCR)检测牦牛GV期、MI期及MII期的卵母细胞和颗粒细胞中KDM4A基因的表达。RT-qPCR反应体系为15 μL:其中SYBR®Premix Ex TaqTMⅡ 7.5 μL,上、下游引物(10 μmol·L-1)各0.5 μL,cNDA 1.0 μL, ddH2O 5.5 μL;PCR扩增条件:95 ℃ 3 min;95 ℃ 10 s,60 ℃ 30 s, 共40个循环。每个样本进行3次重复,用2-ΔΔCt法对定量结果进行分析。
1.10 数据分析
数据用“平均值±标准误(Mean±S)”表示,采用SPSS 18.0软件进行显著性分析,P<0.01为差异极显著,P<0.05为差异显著。
2 结 果
2.1 牦牛KDM4A基因CDS扩增及克隆测序
经RT-PCR方法扩增,琼脂糖凝胶电泳检测,获得和预期目的基因KDM4A片段大小一致的单一清晰条带(图1),进一步克隆测序验证获得牦牛KDM4A基因核苷酸序列为3 289 bp(图2),包括48 bp的5′端非编码区,3 201 bp的CDs区和40 bp的3′端非编码区。
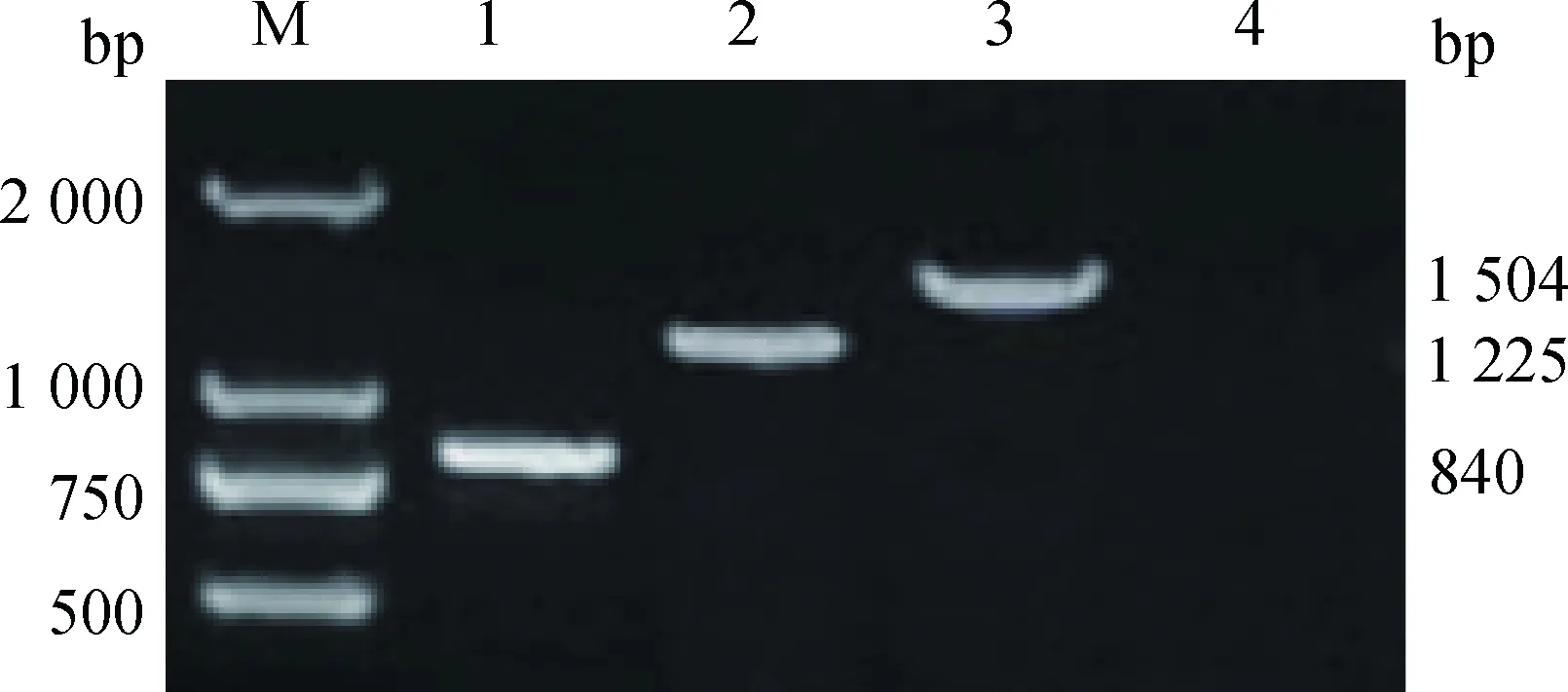
M.DL2000 DNA marker;1.KDM4A-1 PCR 产物;2.KDM4A-2 PCR产物;3.KDM4A-3 PCR产物;4.阴性对照(ddH2O)M.DL2000 DNA marker; 1. KDM4A-1 PCR product of yak KDM4A; 2. KDM4A-2 PCR product of yak KDM4A; 3. KDM4A-3 PCR product of yak KDM4A; 4.Negative control(ddH2O)图1 牦牛KDM4A基因PCR扩增结果Fig.1 Result for PCR amplification of yak KDM4A
2.2 物种间KDM4A基因同源性比较及进化树构建
利用DNAMAN软件将克隆测序所得的牦牛KDM4A基因CDs区与黄牛(NM_001206316.1)、山羊(XM_018043005.1)、马(XM_005607123.2)、猫(XM_019836867.1)、人(NM_014663.2)、犬(XM_005629109.2)、猕猴(XM_015136353.1)、小鼠(NM_001161823.1)、绵羊(XM_015091927.1)、野猪(XM_013992005.1)、原鸡(XM_015291306.1)、爪蟾(NM_001114026.1)的进行比对,同源性分别为99.6%、94.5%、90.6%、89.9%、89.4%、88.4%、88.1%、85.4%、84.2%、76.7%、73.2%、69.1%。使用MEGA 5.0软件对13个物种KDM4A基因编码区序列构建了系统进化树,结果表明,KDM4A基因CDs区核苷酸具有很高的保守性,牦牛与黄牛KDM4A基因的亲缘关系最近,其次是山羊等(图3)。
2.3 牦牛KDM4A蛋白结构和功能预测
采用ExPASY中Protparam在线工具对该蛋白理化性质进行分析,结果表明,其分子式为C5388H8382N1496O1656S58,相对分子质量122.48 ku,理论等电点5.71,总原子数16 980,带负电残基数(Asp+Glu) 161,带正电残基数(Arg+Lys) 138。出现频率较高的氨基酸残基有Glu(9.8%)、Ser(8.4%)、Leu(7.2%)、Ala(6.8%),且不包括吡咯赖氨酸(Pyl)和硒半胱氨酸(Sec)。总平均亲水性-0.567<0,脂肪系数67.81,估计哺乳动物网织红细胞(体外)半衰期30 h,不稳定指数为55.55。综上推测,KDM4A蛋白可能是亲水不稳定的酸性蛋白。
上行表示牦牛KDM4A基因核苷酸序列;下行表示推测的氨基酸序列。下划线表示起始密码子;*表示终止密码子The upper lines show the nucleotide sequence and the lower lines show the deduced amino acid sequence of KDM4A. The initial codon ATG is underlined; * indicates the terminal codon TAG图2 牦牛KDM4A基因核苷酸及其推测的氨基酸序列Fig.2 Nucleotide sequence and the deduced amino acid sequence of yak KDM4A
疏水性预测分析显示,KDM4A蛋白亲水性分值在第478位和第479位最小为-3.500,第880位最大为2.389,且较大多数氨基酸残基具有亲水性,推测该蛋白是亲水性蛋白,与所分析的蛋白理化性质结果一致。跨膜结构域和信号肽分析表明,牦牛KDM4A蛋白无跨膜结构和信号肽。蛋白修饰磷酸化位点预测发现,该蛋白有丝氨酸(Ser)位点68个,苏氨酸(Thr)位点31个和酪氨酸(Tyr)位点16个。KDM4A蛋白二级结构预测知,360个α-螺旋占33.33%,99个β-转角占9.17%,409个无规卷曲占37.87%,212个延伸链占19.63%(图4)。可推测α-螺旋和无规卷曲为该蛋白二级结构主要的组成元件,这与三级结构预测显示结果基本相符(图4)。对该蛋白结构域预测发现,该蛋白包括6个结构域。其中JmjN domain为第13~55位氨基酸;JmjC domain为第142~308位氨基酸;PHD domain有两个分别为第711~769和第831~887位氨基酸;TUDOR domain有两个分别为第899~956和第957~1 013位氨基酸(图5)。
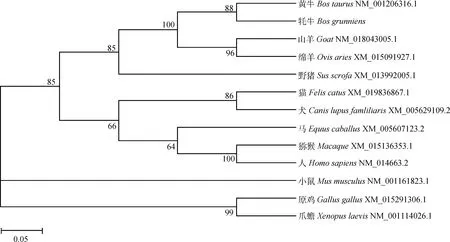
图3 不同物种间基于KDM4A的系统进化树Fig.3 Phylogenetic tree of KDM4A of various species
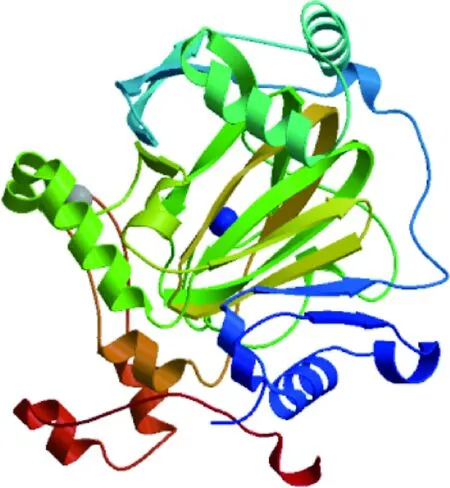
图4 牦牛KDM4A蛋白三级结构预测Fig.4 Tertiary structure prediction of KDM4A protein in yak

图5 牦牛KDM4A蛋白的结构域预测Fig.5 The domain prediction of KDM4A protein in yak
2.4 牦牛KDM4A基因的组织表达谱
采用普通PCR方法,以GAPDH为内参基因,对KDM4A基因在牦牛心、肝、脾、肺、肾、小肠、卵巢、子宫、胃、睾丸、大脑组织中表达情况进行研究,结果显示,在牦牛各组织中KDM4A基因普遍表达,但存在差异,其中该基因在睾丸、脾、卵巢中的表达相对较高,肝、肺、小肠、子宫、胃中的表达次之,在心、肾和大脑中的表达相对较低(图6)。
2.5 牦牛不同时期卵母细胞及颗粒细胞中KDM4A基因的表达
以内参基因GAPDH作为参照,利用实时荧光定量PCR,检测KDM4A基因在牦牛卵母细胞和颗粒细胞成熟过程中mRNA的表达情况(图7)。以GV期卵母细胞KDM4A基因的表达量作为参考,约为MI期和MII期卵母细胞中KDM4A基因表达量的1.8倍和2.6倍(P<0.05),MI期和MII期卵母细胞中KDM4A基因表达差异不显著(P>0.05)(图7A)。以GV期颗粒细胞中KDM4A的表达量作为参照,MII期颗粒细胞中的KDM4A的表达量约为GV期的1.7倍(P<0.05),与MI期颗粒细胞中KDM4A的表达量差异不显著(P>0.05)(图7B)。
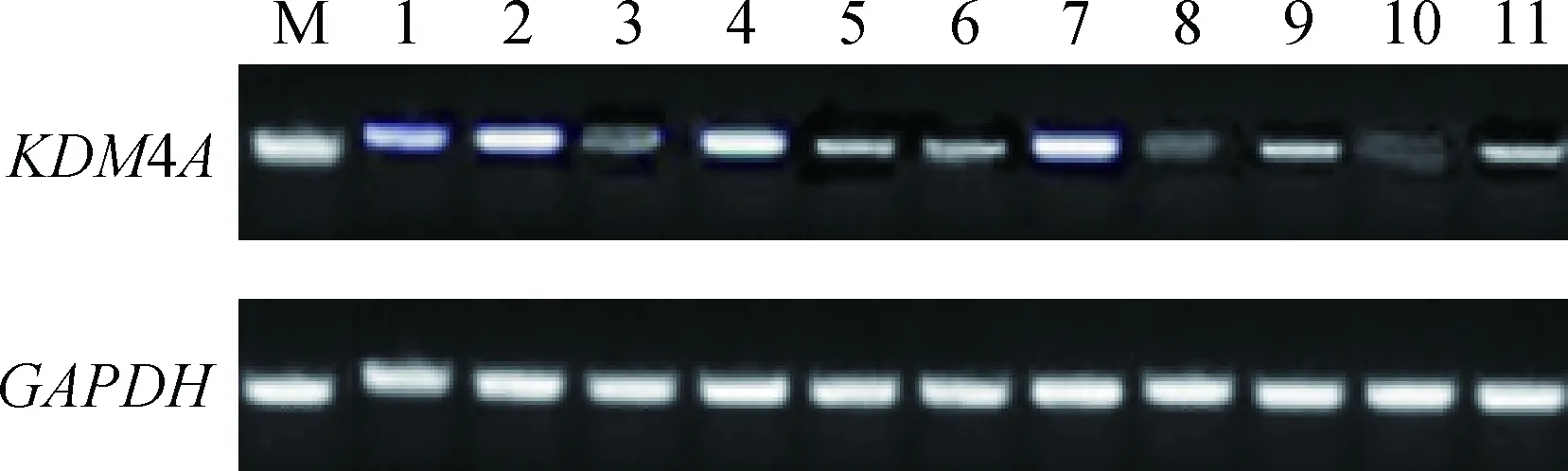
M.DNA相对分子质量标准DL2000; 1.肝;2.睾丸;3.心;4.脾;5.肺;6.小肠;7.卵巢;8.肾;9.子宫;10.大脑;11.胃M.DNA marker DL2000; 1.Liver; 2. Testis; 3. Heart; 4. Spleen; 5. Lung; 6. Intestine; 7. Ovary; 8. Kidney; 9. Uterus; 10. Cerebrum; 11.Stomach图6 牦牛KDM4A基因的组织表达Fig.6 Tissues expression of KDM4A gene in yak
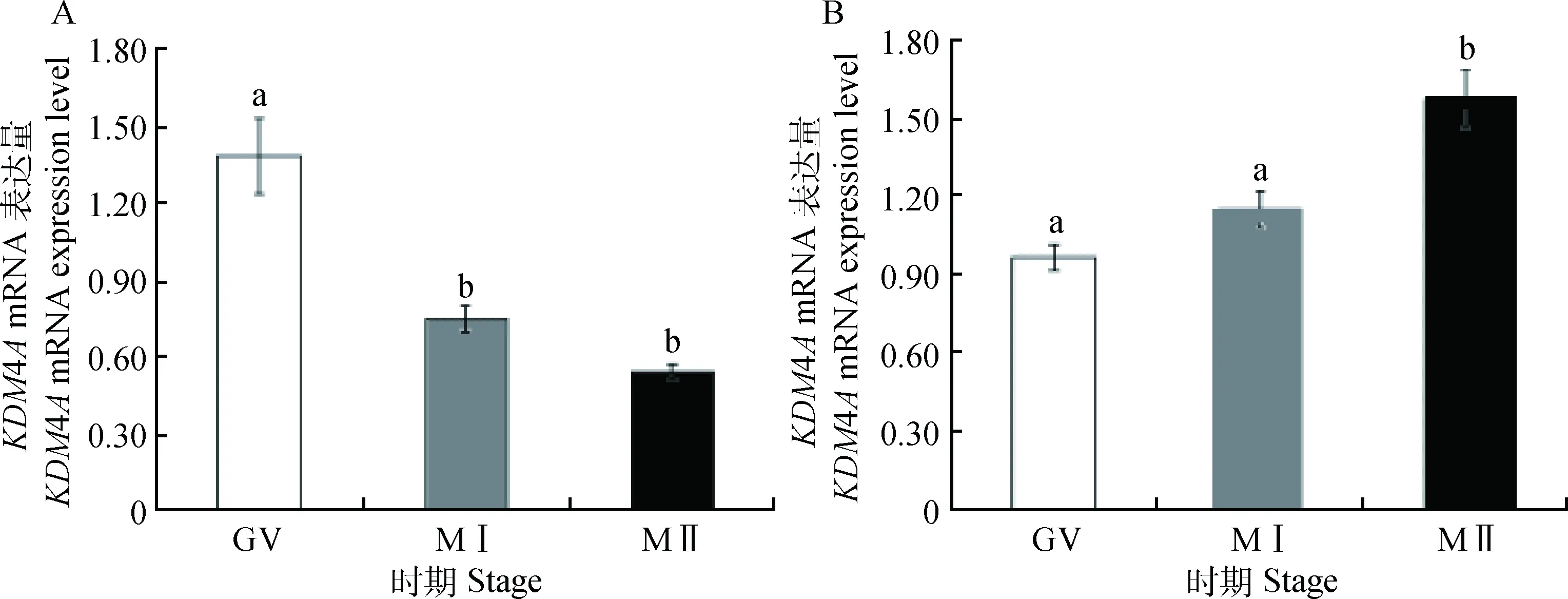
相同字母代表差异不显著(P>0.05);不同字母代表差异显著(P<0.05)The same letter shows no significant difference between stages(P>0.05); different letters show significant difference (P<0.05)图7 KDM4A基因在牦牛不同时期卵母细胞(A)和颗粒细胞(B)中的表达水平Fig.7 The expression of KDM4A in yak during different stages of oocyte (A) and granulosa cell (B)
3 讨 论
KDM4A是KDM4家族成员之一,该基因主要催化H3K9me2/3和H3K36me2/3去甲基化。KDM4A作为转录抑制因子能调节细胞周期及卵母细胞的发育。因此,KDM4A的研究对探索卵母细胞减数分裂调控机制有一定作用。
本研究成功克隆并获得了牦牛KDM4A基因包含完整CDs区的核苷酸序列,与黄牛对比,发现编码区存在10个碱基突变,使编码区第261位和第508位氨基酸,变化由T→P,E→K,但这种氨基酸的突变是否对该蛋白的功能有影响需更深入的研究。同源性分析发现,该基因与黄牛、山羊等的同源性较高,且与进化树分析的亲缘关系相符,暗示牦牛KDM4A基因在进化中具有较高的保守性[6]。
本试验检测了KDM4A基因在牦牛不同组织中的表达,并通过普通PCR对KDM4A基因的组织表达分析表明,该基因mRNA在牦牛的心、脾、肝、肺、卵巢、肾、子宫、睾丸、大脑、小肠、胃中均有表达,这与文献[19]的结果一致,且其表达量在睾丸、脾、卵巢中较高,在睾丸中表达量高的原因可能和KDM4A与结合了配体的雄激素受体进一步结合,形成复合物,大大提高其受体活性有关[20]。Y.Yaron等[21]发现,Tp53对卵巢功能有一定的调节作用,主要是与卵泡的选择闭锁和卵巢内分泌有关。同时,编码P53蛋白的基因Tp53的信使RNA表达水平受KDM4A信使RNA的表达水平影响,并呈正相关。因此,推测KDM4A基因可通过调控Tp53蛋白的表达,参与卵巢功能的调节,所以该基因在卵巢中表达量较高。还有研究发现该基因在多种肿瘤疾病中有超表达[19],从以上结果可推测,KDM4A基因除了参与哺乳动物生殖系统外,还参与免疫、消化、神经、骨骼肌及内分泌等系统的相关生物学调节。在生物体中KDM4A自身蛋白水平具有细胞周期依赖性,并且其自身蛋白含量通过蛋白-泛素酶体途径调控[12]。
卵丘细胞是在卵母细胞周围与之进行代谢联系的颗粒细胞群,其主要功能是维持卵母细胞减数分裂、诱导其发生减数分裂、以及在促进细胞质的成熟方面起一定作用。同时,卵母细胞在颗粒细胞成熟分化等方面也起着不可或缺的作用。有研究发现,KDM4A基因可通过调控颗粒细胞表面的激素受体来间接调控该细胞的增殖分化,同时,该基因也可以通过影响细胞周期蛋白的表达来影响卵母细胞减数分裂[17,22]。因此,研究该基因在卵母细胞和颗粒细胞中的表达对进一步探讨该基因对牦牛生殖机能的调控有一定意义。
本研究采用实时荧光定量PCR的方法检测了该基因在牦牛的卵母细胞及颗粒细胞成熟过程中的表达差异。结果指出,KDM4AmRNA在这两种细胞发育的不同时期均有表达,但其表达量存在差异,表明KDM4A基因在卵母细胞及颗粒细胞成熟过程中发挥着不可忽视的作用。并对KDM4A基因在不同时期的颗粒细胞中的表达量进行分析,结果显示,KDM4A基因在MII期表达量较其它时期高。有研究发现,KDM4A可以作为雌激素受体(ERα)、雄激素受体(AR)的辅激活子,激活它们各自靶基因,有助于雌激素与颗粒细胞上的ER结合,促进颗粒细胞分化[12,16];雄激素与颗粒细胞上的AR结合,促进颗粒细胞增殖[17]。B.X.Li等[20]发现KDM4A可调控P53的表达水平,并且呈正相关。Tp53(编码P53的基因)可促进排卵后的颗粒细胞转化为颗粒黄体细胞,并对颗粒黄体细胞分泌催产素、孕酮和前列腺素E有促进作用,从而对前列腺素E的分泌产生抑制作用[23],因此该基因在MII期表达量较高。随后,对KDM4A基因在不同时期的卵母细胞中表达量进行分析,发现该基因在GV期表达水平较其他时期高。研究表明,MPF在卵母细胞成熟过程中起一定的调控作用,并且MPF的活性受cyclinB量的影响[24]。J.J.Eppig等[22]研究发现,大量的cyclinB在GVBD后小鼠卵母细胞中合成以提高MPF水平,促进卵母细胞发育成熟至MII期。高扬[25]发现稳定敲低KDM4A会使cyclinB1的表达明显增高。同时,普遍认为处于高活性状态的MPF,可使卵母细胞维持在MII期不向后转变[26]。由此可以推测,KDM4A在卵母细胞不同时期的表达水平可能与其对cyclinB1的表达调控有关。综合两个图可以看出,KDM4A基因在两种细胞中表达变化趋势是不一致的,其原因可能是KDM4A基因对卵母细胞和颗粒细胞的调控机制及在其中发挥的生物学功能不同所致。综上所述,KDM4A基因参与牦牛卵母细胞和颗粒细胞的成熟过程。
4 结 论
本研究成功克隆获得了牦牛KDM4A基因序列。生物信息学分析表明,KDM4A基因编码区在生物进化中具有很高的保守性。本试验使用荧光定量PCR检测KDM4A基因在牦牛卵母细胞及颗粒细胞中的表达,且在不同时期mRNA表达量存在明显差异,表明,KDM4A基因参与了卵母细胞及颗粒细胞的成熟过程。为进一步研究KDM4A基因对牦牛生殖机能的调控奠定基础。
[1] 兰道亮, 熊显荣, 位艳丽, 等. 基于RNA-Seq高通量测序技术的牦牛卵巢转录组研究: 进一步完善牦牛基因结构及挖掘与繁殖相关新基因[J]. 中国科学: 生命科学, 2014, 57(9): 925-935. LAN D L, XIONG X R, WEI Y L, et al. RNA-Seq analysis of yak ovary: improving yak gene structure information and mining reproduction-related genes[J].ScienceChina:LifeSciences, 2014, 57(9): 925-935. (in Chinese)
[2] 兰道亮,熊显荣,柴志欣,等.牦牛发情期卵巢比较转录组学研究[J].畜牧兽医学报,2016,47(9): 1830-1839. LAN D L,XIONG X R,CHAI Z X, et al. Comparative transcriptome analysis between yak and cattle estrus ovary[J].ActaVeterinariaetZootechnicaSinica, 2016,47(9): 1830-1839. (in Chinese)
[3] QIU Q, ZHANG G J, MA T, et al. The yak genome and adaptation to life at high altitude[J].NatGenet, 2012, 44(8): 946-949.
[4] ALLIS C D, BERGER S L, COTE J, et al. New nomenclature for chromatin-modifying enzymes[J].Cell, 2007, 131(4): 633-636.
[5] TSUKADA Y I, FANG J, ERDJUMENT-BROMAGE H, et al. Histone demethylation by a family of JmjC domain-containing proteins[J].Nature, 2006, 439(7078): 811-816.
[6] GREER E L, SHI Y. Histone methylation: a dynamic mark in health, disease and inheritance[J].NatRevGenet, 2012, 13(5): 343-357.
[7] KLOSE R J, KALLIN E M, ZHANG Y. JmjC-domain-containing proteins, and histone demethylation[J].NatRevGenet, 2006, 7(9): 715-727.
[8] CHEN Z Z, ZANG J Y, WHETSTINE J, et al. Structural insights into histone demethylation by JMJD2 family members[J].Cell, 2006, 125(4): 691-702.
[9] TAN H D, WU S G, WANG J X, et al. The JMJD2 members of histone demethylase revisited[J].MolBiolRep, 2008, 35(4): 551-556.
[10] FODOR B D, KUBICEK S, YONEZAWA M, et al. Jmjd2b antagonizes H3K9 trimethylation at pericentric heterochromatin in mammalian cells[J].GenesDev, 2006, 20(12): 1557-1562.
[11] KLOSE R J, YAMANE K, BAE Y, et al. The transcriptional repressor JHDM3A demethylates trimethyl histone H3 lysine 9 and lysine 36[J].Nature, 2006, 442(7100): 312-316.
[12] TAN M K M, LIM H J, HARPER J W. SCF(FBXO22) regulates histone H3 lysine 9 and 36 methylation levels by targeting histone demethylase KDM4A for ubiquitin-mediated proteasomal degradation[J].MolCellBiol, 2011, 31(18): 3687-3699.
[13] LIU W, YIN J, ZHAO G, et al. Differential regulation of cyclin B1 degradation between the first and second meiotic divisions of bovine oocytes[J].Theriogenology, 2012, 78(6): 1171-1181.
[14] MA J Y, OUYANG Y C, LUO Y B, et al. Cyclin O regulates germinal vesicle breakdown in mouse oocytes[J].BiolReprod, 2013, 88(5): 110.
[15] POLI A, RAMAZZOTTI G, MATTEUCCI A, et al. A novel DAG-dependent mechanism links PKCɑ and Cyclin B1 regulating cell cycle progression[J].Oncotarget, 2014, 5(22): 11526-11540.
[16] BERRY W L, SHIN S, LIGHTFOOT S A, et al. Oncogenic features of the JMJD2A histone demethylase in breast cancer[J].IntJOncol, 2012, 41(5): 1701-1706.
[17] FINDLAY J K, DRUMMOND A E. Regulation of the FSH receptor in the ovary[J].TrendsEndocrinolMetab, 1999, 10(5): 183-188.
[18] GUO X, DING X Z, PEI J, et al. Efficiency ofinvitroembryo production of yak (Bosgrunniens) cultured in different maturation and culture conditions[J].JApplAnimRes, 2012, 40(4): 323-329.
[19] LABBÉ R M, HOLOWATYJ A, YANG Z Q. Histone lysine demethylase (KDM) subfamily 4: structures, functions and therapeutic potential[J].AmJTranslRes, 2014, 6(1): 1-15.
[20] LI B X, LI J, LUO C L, et al. Expression of JMJD2A in infiltrating duct carcinoma was markedly higher than fibroadenoma, and associated with expression of ARHI, p53 and ER in infiltrating duct carcinoma[J].IndianJExpBiol, 2013, 51(3): 208-217.
[21] YARON Y, SCHWARTZ D, EVANS M I, et al. P53 tumor suppressor gene expression in the mouse ovary during an artificially induced ovulatory cycle[J].JReprodMed, 1999, 44(2): 107-114.
[22] EPPIG J J, O’BRIEN M, WIGGLESWORTH K. Mammalian oocyte growth and developmentinvitro[J].MolReprodDev, 1996, 44(2): 260-273.
[24] KIRKPATRICK M, JENKINS C D. Genetic segregation and the maintenance of sexual reproduction[J].Nature, 1989, 339(6222): 300-301.
[25] 高 扬. JMJD2A生物学功能的研究和探索[D]. 厦门: 厦门大学, 2014: 35-36. GAO Y. The research and exploration of JMJD2A biological function[D]. Xiamen: Xiamen University, 2014: 35-36. (in Chinese)
[26] 朱必才,张子峰,高建国,等.哺乳动物卵母细胞减数分裂研究进展[J]. 动物学杂志,2002, 37(4): 79-83. ZHU B C, ZHANG Z F, GAO J G, et al. Recent progress in mammalian oocyte meiosis study[J].ChineseJournalofZoology, 2002, 37(4): 79-83. (in Chinese)

