HIV-1耐药突变的研究进展
, ,,,
1 简 介
随着抗逆转录病毒(ARV)治疗有效和覆盖面的增加,联合国已经制定了消除艾滋病流行的新策略。首先是从2016年始到2020年止,新检出艾滋病毒感染的人数需从2015年的2 000万例减少到2020年的50万例以下。从降低感染人数上实现(90-90-90)的目标和计划。即要求90%艾滋病病毒感染者应被确诊, 90%的HIV/AIDS病例应得到抗逆转录病毒的治疗(ART),而90%的患者经治疗有效[1-2]。为实现这些目标, WHO于2016年建议需要对所有感染艾滋病毒者给予治疗;并在高危地区和人群进行预防性服药(PrEP)[3],降低高危人群的感染风险。实施这个策略虽然可有效的降低感染者数,但也增加了耐药传播的风险[4]。
截止至目前,已有6类(核苷类逆转录酶抑制剂、非核苷类逆转录酶抑制剂、蛋白酶抑制剂、整合酶链转移抑制剂、CCR5拮抗剂和融合抑制剂)27种抗逆转录病毒药物和13种组合药已被美国FDA批准用于艾滋病治疗,但或是毒副作用大、或是效果不好,其中已有11种不再被推荐使用,此处仅论述5类(除T20融合抑制剂外)16种较常用的抗逆转录病毒药物(不包括组合药物在内)[5-7],如表1所示。
表1 常用的抗反转录病毒药物
Tab.1 Common antiretroviral drugs

药物种类Typeofdrugs药名Nameofdrugs缩写Abbreviation中低收入国家使用Useinmiddle⁃andlow⁃incomecountries高收入国家使用Useinhigh⁃incomecountries核苷类逆转录酶抑制剂(NR⁃TIs)拉米夫定(Lamivudine)3TC一线,二线一线,二线恩曲他滨(Emtricitabine)FTC一线,二线一线,二线替诺福韦(Tenofovir)TDF一线,二线一线,二线阿巴卡韦(Abacavir)ABC不常使用一线,二线需检测HLA抗原齐多夫定(Zidovudine)AZT一线、二线等备选挽救用药非核苷类逆转录酶抑制剂(NNRTIs)依非韦仑(Efavirenz)EFV一线一线备选利匹韦林(Rilpivirine)RPV不常使用一线备选依曲韦林(Etravirine)ETR不常使用二线,挽救用药奈韦拉平(Nevirapine)NVP一线备选不常使用蛋白酶抑制剂(PIs)阿扎那韦(Atazanavir)ATV二线一线备选地瑞纳韦(Darunavir)DRV二线备选一线,二线三线挽救用药洛匹那韦(Lopinavir)LPV二线不常使用整合酶链转移抑制剂(INS⁃TIs)埃替拉韦(Elvitegravir)EVG不常使用一线雷特格韦(Raltegravir)RAL二线备选一线三线多替拉韦(Dolutegravir)DTG一线备选一线,二线挽救用药CCR5拮抗剂马拉维若(Maraviroc)MVC不常使用挽救用药
所列三种蛋白酶抑制剂(PIs)在具体使用中均以利托那韦(RTV)为增强剂,用“/r”表示为增强型蛋白酶抑制剂。一线表示一线用药,二线表示二线用药,三线表示三线用药,多重耐药发生后才使用挽救用药。
2 HIV-1耐药突变的机制
HIV-1遗传变异是HIV-1逆转录酶(RT)高速率复制发生错误的结果。而重组病毒则是一个以上的病毒变异株同时感染同一个细胞时发生的,在病毒感染的过程中积累起前病毒变异株[8]。虽然大多数的HIV感染是由单一的病毒株启动的[9],但感染数周后就会出现与初始感染病毒相关的无数变异株(或称为准种)[1]。耐药变异株的选择产生取决于病毒复制过程处于不完全抑制治疗的程度,容易获得特定药物耐药突变(DRM),受药物敏感性和病毒复制系统的影响。虽然自然存在的耐药病毒每天都出现在未经治疗的患者中,但在没有选择性药物压力作用下,这些变异株很少会上升到可检测的水平,因此几乎所有临床治疗产生的耐药株,主要都是由于药物的选择压力而产生的。
对一些抗逆转录病毒来说,需要多种耐药突变才能降低其在治疗时的敏感性,而对另一些病毒来说,单一耐药突变也就足够了。多种耐药突变的数量要求和耐药突变的影响与病毒对某种药物抗性的基因屏障有关。耐药突变可分为主要和次要两类,主要耐药突变直接降低药物治疗的敏感性,而次要耐药突变使病毒对药物的敏感性进一步降低。抗逆转录病毒降低血浆HIV-1 RNA水平的程度称之为其抗病毒效力,一个抗逆转录病毒内在的抗病毒效力与其耐药基因屏障的结合影响其治疗效果见(图1)[11]。
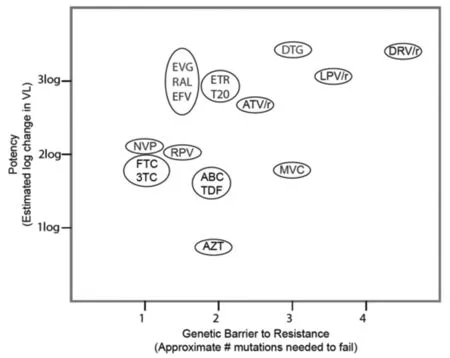
抗逆转录病毒药物一起出现在同一个椭圆都应考虑大致相当的效力和耐药基因屏障。If more than one antiretroviral drug is co-present in an oval, almost comparable potency and genetic barrier to drug resistance should be considered.图1 抗逆转录病毒药物的效力与耐药基因屏障Fig.1 Potency of antiretroviral drugs and genetic barrier to drug resistance
不同类药物之间基本上没有交叉耐药性,但同类药物的交叉耐药性很常见[12],这是因为多种耐药突变株对同类抗病毒药物均降低了敏感性而引起的,但也有对其他同类抗逆转录病毒药物均敏感的例外情况。因此,应当掌握抗逆转录病毒交叉耐药的一般知识,在使用一种以上的药物时应同时考虑同类药物的配伍。大多数一线联合治疗方案能够阻断HIV-1病毒的复制能力,此外HIV-1病毒自身耐药基因屏障也可保持长期的抑制病毒复制作用。抗病毒治疗失败和出现耐药在大多数情况下是因为抗病毒药物治疗的依从性差,这就使病人体内病毒的复制不受抑制,在药物的选择性压力下,形成HIV耐药。
3 HIV-1耐药突变的检测
HIV-1耐药检测可以进行表型或基因型检测。表型抗病毒药物敏感性检测系在细胞培养中进行,以抑制HIV-1复制的50%药物浓度(IC50)为单位(率)计算的,一个病人对药物的敏感与否,是通过与标准敏感株IC50倍增或倍减来确定的。几乎所有的表型检测都是使用野生型病毒为骨架,在骨架上插入从患者血液中扩增的病毒基因片段(蛋白酶/ 逆转录酶,整合酶,膜蛋白)形成重组病毒后进行敏感性分析[13];出于生物安全缘故,已开发出用其他非感染人的病毒膜蛋白代替HIV的env做为重组病毒的表型检测试剂盒。由于成本和检测时间长,表型检测一般用于药物开发、抗性研究或者复杂的临床病例检测。抗逆转录病毒的不同毒株对药物敏感性表型检测的变化较大,这与药物药代动力学和药理学的因素有关。如对NRTIs的检测必须是三磷酸化的活性形式,但体内发生的耐药速率不同于体外细胞检测的结果。 此外野生型株与NRTI耐药分离株之间IC50的倍数变化范围很大,TDF耐药株可低至5倍,而AZT、3TC,和FTC等基本相似,可高达200倍或更多,也就是说更容易耐药[14-15]。类似情况也存在于NNRTIs、PIs和INSTIs中,但敏感性差异没有NRTIs大。
基因型耐药检测(SGRT)就是对从病人血浆RNA中反转录为cDNA的PCR产物进行Sanger法的测序,检测靶点在于逆转录病毒是否发生蛋白酶,逆转录酶和整合酶耐药突变。因为每个病例中的病毒种群都不一致,因此很常见在核苷酸序列谱特定位置上,会出现不同的核苷酸,这也说明病例个体中的病毒是一个准种群[16];其次存在测序的背景“噪音”,这与大量的电泳混合物、不典型的突变和载脂蛋白家族突变等因素有关,表现在信号弱和难以定性的低峰型,这些问题都说明了要加强测序的质量内控[17]。
基因检测的下限一般是丰度值低于20%左右的耐药突变,用点突变(PMA)和二代测序(NGS)可测定低于该阈值下限的耐药突变株,但用这两种检测法所得结果差别较大。对低丰度的耐药突变的检测错误也有可能来自于PCR或测序。为了减少检测错误的概率,二代测序中使用了高度特异性的Primer IDs(具有标签引物的随机序列并连接待测序列特异性的引物模板)可以解决这个问题[18]。
HIV/AIDS患者起始治疗前和治疗失败时一般要用SGRT检测,在高收入国家这种检测是常规进行的;但在低中收入国家,因为成本的缘故,有相当多的病例不能进行SGRT的检测;此外SGRT的检测可以采用经批准的商用试剂盒,也可以按照WHO推荐的方案,用in-house的方法,采用的方法不同,对结果的解释也存在差异。基于这个原因,Dudley等[19]用48份标本混合并用Primer ID等序列识别技术进行NGS测序鉴定耐药突变,用这种测序法可进一步加大检测标本的混合数量,多的可检出几百个序列。采用这种NGS“宽序”测序的策略[20],其成本明显低于SGRT。但NGS测序需要专门的技术人才和昂贵的设备,还需要把样品集中在少数有条件的中心实验室处理,因此该法目前难以推广使用。近期Panpradist等[21]报告了低中收入国家根据一线治疗方案用亲和素、辣根过氧化物酶或荧光标记的点突变测定法准确性高、成本低、容易推广使用、并且可作即时分析等优点;但该法不能测定序列,因此也无法用于系统进化树分析或发现新的耐药突变。
4 耐药检测结果的解释
正确解释耐药检验结果特别对于临床医生来说难度大。各治疗方案产生的耐药模式不同,所造成的耐药水平也不同。一种耐药突变呈现对某一类药物的低水平抗性,但在其他药物仍有效的情况下并不能说该药绝对不能用。进一步复杂的基因型检测甚至高通量测序可能也无法检测到每一个病人体内所有的耐药突变,而对这些突变的推断部分则依靠对病人过去的治疗方案的分析,所以治疗失败要改变方案时,要具体问题具体分析。
第一,耐药突变可以根据其是否通过治疗产生的或是病毒复制过程自发产生的多态性来定性。治疗失败的主要原因是药物无法抑制病毒的复制,即所谓病毒对药物的敏感性下降,这种情况属于耐药突变;在不存在选择性药物压力的情况下而对使用药物产生所谓抗性,有两种情况:一是病毒自发产生多态性,二是对治疗产生不依从性,这两种情况都不能说是真正意义上的耐药突变。所以不管是对NRTIs、NNRTIs、PIs和INSTIs等药物来说,HIV的耐药突变在治疗前产生是很少见的。
第二,耐药突变检测的临床意义表现在对首次抗逆转录病毒治疗的病人效果的确定,如果效果不好,那么可以认为病人在起始治疗之前体内就已存在耐药突变病毒,只是没有经过基因型耐药检测就用药引起的,这种突变情况可以通过对预保存的冻干血液标本进行回顾性检测而得知[22-23]。
第三,耐药突变还可以通过影响体内和体外对治疗药物敏感性的反应来定性。评价病毒对某种药物的易感性可以用表型检定法,由特定耐药突变引起的药物敏感性的降低可以通过临床分离株来研究,这种研究还可以解释是否存在多重耐药突变[24-26]。
第四,斯坦福大学的HIV耐药数据库(HIVdb)提供了在线基因型检测耐药数据的分析,该程序接受用户提交的逆转录酶、蛋白酶或整合酶序列数据,并根据阈值返回耐药分析的结果,有效地帮助临床医生和实验室研究人员解释HIV-1基因型检测结果[27]。包括我国在内的部分国家使用了WHO推荐的in-house基因检测,并把数据提交给斯坦福大学的HIV耐药数据库进行分析,但这种分析法在病毒载量高时可用,对低丰度耐药株还无法测定其耐药种类。
5 四类常用药物的耐药突变(表2)
表2 HIV-1耐药基因检测靶点
Tab.2 Targets for detecting HIV-1 drug resistance

抗病毒药物Antiretroviraldrugs位点名称NameoflocusNRTIsM184V/I、K65R、K70E/G/Q、L74V/I,Y115F、Q151M、M41L、D67N、K70R、L210W、T215Y/F、K219Q/E、A62V、V75I、F77L、F116YNNRTIsL100I、K101E/P、K103N/S、V106A/M、Y181C/I/V、Y188C/H/L、G190A/S/E、M230L、E138K/G/Q/APIsV32I、G48V/M、I50V/L、V82A/T/L/F/S/C/M、I84V/A/C、M46I/L、I54V/M/L/T/S/A、L33F、L76V、N88S、V11I、I47V、T74P、L89VINSTIN155H±E92Q、Q148H/R/K±G140S/A、Y143C、E138±G140、T66I/K、S147G、R263K、G118R
5.1NRTI耐药突变 NRTI耐药突变有两个机制:1)识别性突变(discriminatory mutations),又称为结合阻断突变。逆转录酶在这类突变中有两种作用,第一是掺入NRTI,终止了病毒DNA链的延伸,病毒复制无法进行下去;其次逆转录酶作用于感染细胞本身,其作用是掺入正常的dNTPs进行细胞基因组的复制,这使的NRTI对逆转录酶不产生作用。M184V和Q151M是识别性突变的代表,尤以第一种方式为主; 2)引物解链突变(primer unblocking mutations)。其实质就是将NRTIs从被阻断的DNA合成链中切除下来,一旦NRTIs被切除,逆转录酶就可以继续合成DNA。逆转录酶切除NRTIs功能的突变称为胸苷类似物突变(Thymidine Analog Mutations,TAMs ),也称为剪切性突变[28]。
最常见的NRTI识别性耐药突变是M184V/I。M184V/I的突变是药物选择引起的,其对胞嘧啶核苷类似物如3TC 和FTC敏感性大为降低,可产生高于200倍的高水平耐药;M184V/I对阿巴卡韦(ABC)也有低水平的耐药。在含有3TC 或 FTC的联合药物中,抗病毒治疗失败时M184I产生在前,M184V随后出现,二者竞争的结果最后总是以M184V突变出现,大多数情况下,出现这类突变最好的做法是继续使用这些药物[23],因为对病毒来说,这类突变只是稍降低了病毒的适应性但却大大增强了AZT和TDF对病毒的敏感性,从治疗来说这是有利的。另外,3TC和FTC的耐受性良好,它们的副作用或毒性也低。
其次常见的识别性突变包括K65R、K70E/G/Q、L74V/I,Y115F和Q151M。K65R突变最初是由TDF引起的,该突变对ABC和d4T影响程度较低,但却大大增强了对AZT的敏感性。TDF虽只引起约两倍的敏感性降低,但在M184V突变产生后,含有TDF的联合用药常导致抗病毒治疗失败。
L74V/I和Y115F突变主要是由ABC的选择压力引起的。然而L74V/I和Y115F突变往往也由TDF选择压力引起且时间较长,特别是在中低收入国家(LMICs)检出的抗病毒治疗失败就有TDF引起的此类突变[29]。K70E/G/Q突变也由含TDF和ABC的方案选择引起的,体外检测认为病毒敏感性稍有下降与TDF和ABC用药有关[30]。Q151M突变只偶尔出现在重度治疗的失败病例中,通常只在与几种次要突变(A62V、 V75I、 F77L 和 F116Y)结合时出现,与AZT和 ABC的高抗性突变有关,但对TDF、 3TC 和 FTC只是中等抗性。
TAMs突变包括M41L、D67N、K70R、L210W、T215Y/F和K219Q/E M41L,虽然这类突变主要由AZT 和 d4T选择压力引发,但它们对TDF 和 ABC也有低水平的交叉耐药性。TAMs突变以型1 和型2两种模式出现,但这两种模式经常重叠发生[28]。型1 TAMs的突变包括M41L、L210W 和 T215Y,与型2 TAMs(D67N, K70R, T215F和d K219Q/E)比较,它们可引起更高水平的对TDF 和 ABC表型和临床的交叉抗性,所有3种型1 TAMs的突变在临床上可显著影响含有TDF和 ABC的联合用药[31-32]。
5.2NNRTI耐药突变 NNRTIs遗传突变屏障较低。用NVP治疗时只需1个耐药突变, EFV需 1至2个, ETR需2个[12]。
该类药物往往产生交叉耐药性,其主要机制有两种:1)大多数的的NNRTIs耐药突变可降低两个或两个以上NNRTIs对病毒的敏感性[12];2)由于NNRTIs耐药遗传屏障低,因此引起多个独立性的NNRTIs耐药谱系的出现,有些突变位点甚至用SGRT都难以检出[26]。最常见的NNRTI突变是L100I 、K101E/P、 K103N/S、 V106A/M、 Y181C/I/V、 Y188C/H/L、 G190A/S/E和 M230L,每一种这些突变都会引起对NVP中或高水平的表型耐药。 除Y181C/I/V 外,同样引发EFV耐药;虽然Y181C突变对 EFV敏感性只降低了2倍,但它与含有NVP药物组对抗病毒治疗失败有关,这原因也可能与极少数15608522突变参入其中有关。除K103N /S、V106A /M、Y188C/H和G190A/S,他们也引起ETR和RPV的表型耐药[19]。
此外,在患者接受RPV药物治疗中,以前无法识别的突变E138K/G/Q/A变为最常见的突变。如表型检测认为E138K突变对 RPV敏感性影响很小,但在含RPV一线治疗方案的患者中,该位点则是最常见抗病毒治疗失败的耐药突变,感染的病毒亚型中约有5%与有关的天然ARV抗药患者中E138A突变对RPV治疗的突变最为明显,因此(TDF/FTC / RPV)的这样的方案一般不列为耐药突变分析之列。
相对而言,ETR在NNRTIs类药物中的遗传突变屏障最高,因此它也是最常用的药物组合。在二线组合治疗药物中,如与增效型的DRV / r并用时,依据ETR耐药突变的数量和权重,可以预测抗病毒治疗是否有效[30]。
5.3PIs的耐药突变 HIV-1蛋白酶的功能主要是对前病毒多聚蛋白进行水解,以致水解后的各蛋白能装配使成熟的病毒释放,完成HIV的复制及生命循环。表1已说明蛋白酶抑制剂各单药须用利托那韦(RTV)为增效剂使其治疗效果倍增。
“凡有血肉的,不再被洪水灭绝,也不再有洪水毁坏地了”,“我把虹放在云彩中,这就可作我与地立约的记号了。我使云彩盖地的时候,必有虹现在云彩中。我使纪念我与你们和各样有血肉的活物所立的约,水就再不泛滥毁坏一切有血肉的物了。”(《创世纪·第九章》)
LPV /r又称克立芝、ATV/r和DRV /r是常用的三种增效型PIs, 其中则以ATV/r和DRV /r在高收入国家(HICs)中最常用 ,而 LPV / r在LMICs中则广泛被用于二线治疗。 LPV / r、 DRV /r和ATV/r都具有耐药突变的高基因屏障,需要多个基因耐药位点突变才能显示其抑制病毒复制, 换用LPV / r和DRV /r,即使在单药治疗(或称简化治疗),分别用这两种药物治疗时仍然有效[33];相对而言,ATV/r要比LPV / r和DRV / r的遗传屏障要低,主要原因有三方面:一是少数的耐药突变就可引起体外抗病毒敏感性的降低;二是即使在体外检测时,对从患者分离到的病毒其敏感性降低作用很小时,该药也会增加对患者的治疗失败率;其三ATV/r单药治疗效果比LPV / r和DRV /r差,这就很难以用于简化治疗[34]。尽管ATV/r有这样的问题,这三种PIs的耐药突变发生的频率仍比NRTI/NNRTI联合用药方案发生耐药突变的频率要低得多。使用了含PIs方案治疗,一般不发生耐药突变;虽然也有治疗失败发生,这种情况主要还是由治疗依从性问题引起的[35-36];另外还有可能是由蛋白酶以外的突变引起治疗失败,比较有争议的是gag基质蛋白(MA)及gp41细胞质表位(gp41-CD)的突变造成PI耐药突变的说法[37]。历史上在HICs使用未增强的蛋白酶抑制剂曾导致出现不少的耐药突变,有报道称有80多种,而绝大多数这些药物的突变是由选择压力引起的[38],这些突变包括了V32I、 G48V/M、 I50V/L、 V82A/T/L/F/S/C/M和 I84V/A/C;在蛋白酶的档板区(flap)包括了M46I/L 、I54V/M/L/T/S/A,在酶的核心区包括了 L33F、 L76V和 N88S,这些突变位点都很明显的降低了病毒对药物的敏感性。
对于增效型PIs来说,一般要产生次要突变位点和代偿性gag裂解突变位点[39],这种突变是PIs高遗传屏障作用的特征,正因为有这种特征,所以在治疗失败的情况下,需要换用DRV / r和LPV / r的方案继续治疗。在长期使用情况下,由这两种药物引起的耐药突变也是难免的,已知DRV/r耐药突变位点是下列中的3-5个:V11I、V32I、L33F、I47V、I50V、I54L /M、T74P、L76V,I84V和L89V。
5.4INSTIs的耐药突变 RAL是第一个经FDA批准的整合酶抑制剂,容易发生耐药突变,突变位点为: N155H ±E92Q,Q148H / R / K±G140S /A和Y143C / R,这些位点常出现在接受RAL晚期抢救治疗的患者中。
EVG有两个与RAL共同的突变位点:N155H±E92Q 和 Q148H/R/K±G140SA;E92Q则是最常由EVG引起的耐药突变[40];还有两个由EVG引起的耐药突变:T66I/K 和 S147G,这两个突变也与RAL有低水平的交叉耐药性。对这两种INSTIs来说,因为它们之间存在着广泛重叠的抗性遗传机制,所以在含其中一种INSTI治疗失败的情况下,不要换另一种INSTIs。
与RAL和EVG则相反, DTG需要多位点的耐药突变在临床上才有意义,因此它一般用于挽救治疗用药[41],其产生的耐药突变为Q148和 E138±G140, 当Q148突变出现时治疗失败较为常见。DTG也产生R263K和G118R突变,但这两个耐药突变并不常见。
6 HIV-1嗜性变化对药物的影响
马拉维若(Maraviroc)是一种CCR5受体拮抗剂,CCR5受体是HIV感染的辅助受体,其对HIV拮抗的机制主要是通过跨膜G蛋白偶联受体变构抑制gp120包膜的CCR5-嗜性株结合到R5受体上来完成的。简而言之,马拉维若正是结合到R5受体上而抑制了HIV利用这一受体的功能[42-43]。病人接受R5抑制剂最常见的治疗失败机制是原先存在的少数CXCR4(X4)病毒复制造成的,马拉维若的耐药突变也可能由另一方式产生,即HIV-1 gp120结合到另一种抑制剂结合的R5受体上,这种抗药性的报道多数是在体外试验中证实的,只出现在少量的临床分离株。80%以上的患者最初感染的病毒多是R5嗜性,而X4嗜性病毒多出现在晚期病人中,约50%的晚期病人携带X4嗜性病毒。R5嗜性病毒多时,X4嗜性病毒就很少。在使用马拉维若之前,可用表型或基因型检测病人是否感染了X4嗜性病毒。
嗜性基因型检测主要对Env膜V3环测序。gp120 V3环带正电荷的氨基酸残基的位点11-25一般鉴定为R5嗜性,但也不绝对,有些可能是X4嗜性株。约85%特异性和50%~70%敏感性由测序可检出X4嗜性的突变株,无法通过测序确定嗜性的部分原因在于一些嗜性的决定因素是在V3环外[44]。因此可用对病毒cDNA多个独立部分测序或NGS测序可以提高基因型嗜性检测的敏感性。
7 HIV-1亚型的耐药影响
在美国,B型病毒占HIV-1毒株的95%以上,欧洲占60%,其他国家大约只占约10%。因此在早期抗病毒治疗研发以及突变研究主要都是对B亚型病毒。但现有的抗逆转录病毒药物治疗对B亚型和非B亚型同样有效,此外,在B亚型病毒引起的突变同样也存在于非B亚型中。
然而,有几个耐药突变好发在某些的HIV-1亚型上,大多数这种亚型特异性的耐药突变主要是由于单一密码子的突变引起的。NNRTI V106M的耐药突变更多发生在用含NVP 或EFV治疗的C亚型病毒感染的病例,这是因为在C亚型病毒中V106M突变只需要单一碱基对的变化GTG(V)= > ATG(M),但在其他亚型中则需要两个碱基对的改变GTA(V)= > ATG(M)。而CRF01_AE亚型在NRTIs治疗中容易发生V75M 的耐药突变, G亚型则容易发生V82M的PIs 耐药突变,A亚型(来自前苏联国家) 在NNRTI治疗中则优先发生G190S耐药突变,可见亚型的耐药突变的发生与亚型型别关系不大,但其耐药机制则基本相同[45]。
8 暴露前预防与耐药性
截至2015年9月,WHO为男同性性接触者(MSM)、高风险异性性接触者及静脉注射吸毒者推荐含TDF 的暴露前PrEP。尽管HIV-1感染的风险在使用PrEP后明显降低,但那些服用后仍被感染的病例发展为耐药突变可能性大。在系统回顾和用Meta分析PrEP使用 风险,发现仅约0.1%(11/9222)使用者发展为FTC 或 TDF耐药突变,进一步分析发现这11人中有7个在开始使用PrEP时,即已处于急性HIV-1感染阶段,从这个意义上来说发生耐药突变的感染率甚至更低[46]。每一个PrEP使用者发生HIV感染而又产生FTC耐药突变都是因为没有意识到本身是处在急性HIV感染的环境中才去使用PrEP, 由此估计在这种场合下使用PrEP预防感染率可达到8%~50%。 PrEP最常见的耐药突变是与FTC的M184V/I突变有关,其次是由TDF引起的 K65R突变[47]。PrEP相关的获得性耐药突变可以通过例行的实验室检测来确定偶发感染并启动对感染者的治疗。虽然PrEP引发的耐药突变很低,但随着更广泛地实施这种干预,耐药突变可能增加,特别是那些在感染场所暴露太久的高危人群。
9 耐药突变的管理控制
耐药突变是用药后引起体内病毒相应位点的变异,而导致对原治疗药物抗性,使治疗方案失效。如简介中所述,耐药突变分为治疗前、可传播和获得性耐药突变三类。
9.1治疗前耐药突变(PDR) 没有用药物治疗,一般不会产生突变。所以真正意义上的PDR很少, WHO把这类突变主要归因于LMICs因经济社会发展及药物资源不足,使患者因药物不敷而退出治疗;其次是孕期妇女因担心药物对胎儿的不良影响而退出治疗;第三则是患者本身的行为造成。这三类人中断了原来的治疗,在进入第二次治疗前由之前治疗所引起的耐药突变统称为PDR。对PDR进行监测很重要,通过监测可以对LMICs的HIV耐药突变情况有所了解。现已知安哥拉PDR占16%、古巴22%、墨西哥12%,巴布亚新几内亚岛16%、博茨瓦纳10%、南非14%[48]。在国家层面上进行PDR的监测可以预测一线治疗和用药的合理组成。如PDR发生水平低,则预示病人参与WHO推荐的EFV TDF/3TC或FTC的一线治疗方案效果好;如PDR发生水平高,有两种情况需要注意区别对待,一种是一线治疗方案可用于所有起始治疗者,另一种仅针对那些之前曾有治疗经历的病例(如一线治疗失败者、女性暴露于母婴阻断,还包括那些曾经的药物预防者)启用另外的一线治疗方案。虽然WHO强烈推荐各LMICs要通过监测收集这些数据,但这些国家政策执行力低,也未意识到HIV耐药突变是对公共卫生的严重威胁,药物资源有限、国家间竞争激烈以及捐赠者的意愿等阻碍了对PDR的监测,这就需要WHO大力推进药物资源和耐药突变管理的全球行动计划。
9.2TDR突变 这类突变一是从治疗失败的患者感染获得;二是从未治疗的患者中感染获得。SGRT检测表明在美国新发感染者中已感染耐药毒株的构成比约是12%~24%,在欧洲约是10%[49]。大多数中低收入国家如撒哈拉以南非洲、南亚、东南亚等国在5%以下, 拉丁美洲、东欧和亚洲的高收入国家介于5%~10%之间。实际上可传播耐药突变是获得性耐药突变传播流行的结果,所以关键问题在于要加强对患者的治疗管理。但也应当认识到,TDR病毒存在回复突变的能力,虽然耐药的结果使病毒的适应性有所降低,但经过几年后,多种耐药病毒逐渐转换回到野生型。病毒适应性降低大的TDR比那些降低小的转换回到野生型反而更快;此外TDR很弱时其转换回到野生型的周期也很长[50]。在没有选择性药物压力的情况下,PIs和NNRTIs的TDR株逐渐恢复为野生型株时间平均约为3年;对于NRTIs类,回复突变存在更多的变数。M184V通常在一年内消失;T215FY 也消失很快;但T215回复慢,一般要10年以上[51]。
9.3ADR突变 70%~80%以上的治疗失败归因于ADR的产生[52]。ADR突变的模式与治疗失败时根据所用的方案和患者对药物的依从性而有所不同,依从性差也会酿成治疗失败。
在HICs,治疗失败且病毒载量> 1000拷贝/毫升时一定要做SGRT检测去确定耐药突变的原因[53]。4周内间断性地出现治疗失败的病例中,SGRT检出ADR突变的可能性是很大的。基因型检测可知治疗失败还是依从性问题,对于前者来说检测结果也可以用于帮助设计二线治疗方案,而非ADR治疗失败的病人可从依从性方面来改善治疗[54];原则上,不要只添加一种药物至已失败的方案中,否则将造成新选择压力引起的ADR的产生。经历了多次治疗失败并有多种ADR的患者需要进行挽救治疗,这种治疗需要多种抗逆转录病毒药物。
在大多数低收入国家,一般靠经验判断是否发生了获得性耐药突变,而二线药的治疗原则上采用WHO推荐的方案。患者间隔3个月病毒载量检测均在1 000个拷贝/毫升以上,可以肯定该患者已获得耐药性突变,继后所选的二线药物包括增效蛋白酶抑制剂,即ATV/r 或 LPV/r以及两种 NRTIs类药品;也可以选择DRV /r再加上两个NRTIs。二线治疗方案中的两种NRTI的选用一般不能选用一线治疗已失败的药品。
10 总结和结论
在过去的10多年里,全球对艾滋病进行了前所未有的扩大规模的治疗,2015年就有 1 700万人接受了抗逆转录病毒治疗。然而,随着治疗增加,HIV的感染率、发病率和死亡率都在进一步地降低,但不可否认耐药突变也随着增多,这是不可避免的结果。关注耐药突变,积极进行耐药监测,采取改变方案、改善依从性等方法去应对,这应当成为医防人员的共识。
我国目前用于艾滋病免费治疗的药品包括用于二线治疗的蛋白酶在内也才7种,未引进PrEP药品或采用PrEP对高危人群进行预防,MSM高危人群中感染HIV者疫情上升快,局部异性性接触者感染率也有增加的趋势,无论是对MSM、还是对异性性接触感染者的干预收效有限,疫情持续成上升趋势;此外与西方发达国家相比,我国新药研制技术相对落后,而人群流动性却不断增长,使TDR和ADR的病例不断增加,防治形势不容乐观。为解决艾滋病毒耐药性需要所有国家和合作伙伴共同行动起来,以预防、监测和应对艾滋病毒耐药性,为实现到2030年终结艾滋病流行这一可持续发展目标保驾护行,使其不断取得进展。
[1] UNAIDS. Fast-Track: Ending the AIDS Epidemic by 2030[EB/OL][2017-10-27]. 2014. Available at:http://www.unaids.org/ sites/ default/files/media_asset/JC2686_WAD2014report_en.pdf, Geneva, Switzerland.
[2] UNAIDS. Fact Sheet 2016[EB/OL][2017-10-27]. Available at: http://www.unaids.org/en/resources/fact-sheet.,Geneva, Switzerland.
[3] World Health Organization. Consolidated guidelines on the use of antiretroviral drugs for treating and preventing HIV infection: recommendations for a public health approach-Second edition[EB/OL][2017-10-27]. Available at: http://www.who.int/hiv/pub/arv/arv-2016/en/. Geneva, Switzerland.
[4] Phillips AN, Cambiano V, Miners A, et al. Effectiveness and cost-effectiveness of potential responses to future high levels of transmitted HIV drug resistance in antiretroviral drug-naive populations beginning treatment: modelling study and economic analysis[J]. Lancet HIV, 2014, 1(2): e85-93. DOI: 10.1016/S2352-3018 (14) 70021-9
[5] Gallant JE, Koenig E, Andrade-Villanueva JF, et al. Brief report: Cobicistat compared with ritonavir as a pharmaco enhancer for atazanavir in combination with emtricitabine/tenofovir disoproxil fumarate: week 144 results[J]. J Acquir Immune Defic Syndr, 2015, 69(3): 338-340. DOI: 10.1097/QAI.0000000000000598
[6] Margot NA, Johnson A, Miller MD, et al. Characterization of HIV-1 resistance to tenofovir alafenamide in vitro[J]. Antimicrob Agents Chemother, 2015, 59(10): 5917-5924. DOI: 10.1128/AAC.01151-15
[7] Waning B, Diedrichsen E, Moon S. A lifeline to treatment: the role of Indian generic manufacturers in supplying antiretroviral medicines to developing countries[J]. J Int AIDS Soc, 2010, 13: 35. DOI: 10.1186/1758-2652-13-35
[8] Abram ME, Ferris AL, Shao W, et al. Nature, position, and frequency of mutations made in a single cycle of HIV-1 replication[J]. J Virol, 2010, 84(19): 9864-9878. DOI: 10.1128/JVI.00915-10
[9] Keele BF, Giorgi EE, Salazar-Gonzalez JF, et al. Identification and characterization of transmitted and early founder virus envelopes in primary HIV-1 infection[J]. Proc Natl Acad Sci U S A, 2008, 105(21): 7552-7557. DOI: 10.1073/pnas.0802203105
[10] Coffin JM. HIV population dynamicsinvivo: implications for genetic variation, pathogenesis, and therapy[J]. Science, 1995, 267(5197): 483-489. DOI: 10.1126/science.7824947
[11] Clutter DS, Jordan MR, Bertagnolio S, et al. HIV-1 drug resistance and resistance testing[J]. Infect Genet Evol, 2016, 46: 292-307. DOI: 10.1016/j.meegid.2016.08.031
[12] Melikian GL, Rhee SY, Varghese V, et al. Non-nucleoside reverse transcriptase inhibitor (NNRTI) cross-resistance: implications for preclinical evaluation of novel NNRTIs and clinical genotypic resistance testing[J]. J Antimicrob Chemother, 2014, 69(1): 12-20. DOI: 10.1093/jac/dkt316
[13] Petropoulos CJ, Parkin NT, Limoli KL, et al. A novel phenotypic drug susceptibility assay for human immunodeficiency virus type 1[J]. Antimicrob Agents Chemother, 2000, 44(4): 920-928. DOI: 10.1128/AAC.44.4.920-928.2000
[14] Melikian GL, Rhee SY, Varghese V, et al. Non-nucleoside reverse transcriptase inhibitor (NNRTI) cross-resistance: implications for preclinical evaluation of novel NNRTIs and clinical genotypic resistance testing[J]. J Antimicrob Chemother, 2014, 69(1): 12-20. DOI: 10.1093/jac/dkt316
[15] Whitcomb JM, Huang W, Limoli K, et al. Hypersusceptibility to non-nucleoside reverse transcriptase inhibitors in HIV-1: clinical, phenotypic and genotypic correlates[J]. AIDS, 2002, 16(15): F41-47.
[16] Woods CK, Brumme CJ, Liu TF, et al. Automating HIV drug resistance genotyping with RECall, a freely accessible sequence analysis tool[J]. J Clin Microbiol, 2012, 50(6): 1936-1942. DOI: 10.1128/JCM.06689-11
[17] Rhee SY, Sankaran K, Varghese V, et al. HIV-1 protease, reverse transcriptase, and integrase variation[J]. J Virol, 2016, 90(13): 6058-6070. DOI: 10.1128/JVI.00495-16
[18] Keys JR, Zhou S, Anderson JA, et al. Primer ID informs next-generation sequencing platforms and reveals preexisting drug resistance mutations in the HIV-1 reverse transcriptase coding domain[J]. AIDS Res Hum Retroviruses, 2015, 31(6): 658-668. DOI: 10.1089/AID.2014.0031
[19] Dudley DM, Chin EN, Bimber BN, et al. Low-cost ultra-wide genotyping using Roche/454 pyrosequencing for surveillance of HIV drug resistance[J]. PLoS One, 2012, 7(5): e36494. DOI: 10.1371/ journal.pone.0036494
[20] Lapointe HR, Dong W, Lee GQ, et al. HIV drug resistance testing by high-multiplex "wide" sequencing on the MiSeq instrument[J]. Antimicrob Agents Chemother, 2015, 59(11): 6824-33. DOI: 10.1128/AAC.01490-15
[21] Panpradist N, Beck IA, Chung MH, et al. Simplified paper format for detecting HIV drug resistance in clinical specimens by oligonucleotide ligation[J]. PLoS One, 2016, 11(1): e0145962. DOI: 10.1371 /journal. pone.0145962
[22] Chung MH, Beck IA, Dross S, et al. Oligonucleotide ligation assay detects HIV drug resistance associated with virologic failure among antiretroviral-naive adults in Kenya[J]. J Acquir Immune Defic Syndr, 2014, 67(3): 246-253. DOI: 10.1097/QAI.0000000000000312
[23] Lee GQ, Bangsberg DR, Muzoora C, et al. Prevalence and virologic consequences of transmitted HIV-1 drug resistance in Uganda[J]. AIDS Res Hum Retroviruses, 2014, 30(9): 896-906. DOI: 10.1089/AID. 2014. 0043
[24] Melikian GL, Rhee SY, Taylor J, et al. Standardized comparison of the relative impacts of HIV-1 reverse transcriptase (RT) mutations on nucleoside RT inhibitor susceptibility[J]. Antimicrob Agents Chemother, 2012, 56(5): 2305-2313. DOI: 10.1128/AAC.05487-11
[25] Melikian GL, Rhee SY, Varghese V, et al. Non-nucleoside reverse transcriptase inhibitor (NNRTI) cross-resistance: implications for preclinical evaluation of novel NNRTIs and clinical genotypic resistance testing[J]. J Antimicrob Chemother, 2014, 69(1): 12-20. DOI: 10.1093/jac/dkt316
[26] Rhee SY, Blanco JL, Jordan MR, et al. Geographic and temporal trends in the molecular epidemiology and genetic mechanisms of transmitted HIV-1 drug resistance: an individual-patient- and sequence-level meta-analysis[J]. PLoS Med, 2015, 12(6): e1001845. DOI: 10.1371/journal.pmed.1001845
[27] Wensing AM, Calvez V, Gunthard HF, et al. Update of the drug resistance mutations in HIV-1[J]. Top Antivir Med, 2014, 22(3): 642-650.
[28] Tang MW, Shafer RW. HIV-1 antiretroviral resistance: scientific principles and clinical applications[J]. Drugs, 2012, 72(9): e1-25. DOI: 10.2165/11633630-000000000-00000
[29] Skhosana L, Steegen K, Bronze M, et al. High prevalence of the K65R mutation in HIV-1 subtype C infected patients failing tenofovir-based first-line regimens in South Africa[J]. PLoS One, 2015, 10(2): e0118145. DOI: 10.1371/journal.pone.0118145
[30] TenoRes Study Group. Global epidemiology of drug resistance after failure of WHO recommended first-line regimens for adult HIV-1 infection: a multicentre retrospective cohort study[J]. Lancet Infect Dis, 2016, 16, 565-575. DOI: 10.1016/S1473-3099 (15)00536-8
[31] Lanier ER, Ait-Khaled M, Scott J, et al. Antiviral efficacy of abacavir in antiretroviral therapy-experienced adults harbouring HIV-1 with specific patterns of resistance to nucleoside reverse transcriptase inhibitors[J]. Antiviral Therapy, 2004, 9: 37-45.
[32] Miller MD, Margot N, Lu B, et al. Genotypic and phenotypic predictors of the magnitude of response to tenofovir disoproxil fumarate treatment in antiretroviral-experienced patients[J]. J Infect Dis, 2004, 189: 837-846. DOI: 10.1086/381784
[33] Collins SE, Grant PM, Shafer RW. Modifying antiretroviral therapy in virologically suppressed HIV-1-infected patients[J]. Drugs, 2016, 76: 75-98. DOI: 10.1007/s40265-015-0515-6
[34] Castagna A, Maggiolo F, Penco G, et al. Dolutegravir in antiretroviral-experienced patients with raltegravir- and/or elvitegravir-resistant HIV-1: 24-week results of the phase III VIKING-3 study[J]. J Infect Dis, 2014, 210: 354-362. DOI: 10.1093/infdis/jiu051
[35] El Bouzidi K, White E, Mbisa JL, et al. Protease mutations emerging on darunavir in protease inhibitor-naive and experienced patients in the UK[J]. J Intl AIDS Society, 2014, 17: 19739. DOI: 10.7448/IAS.17.4.19739
[36] Lopez-Cortes LF, Castano MA, Lopez-Ruz MA, et al. Effectiveness of litonavir-boosted protease inhibitor monotherapy in clinical practice even with previous virological failures to protease inhibitor-based regimens[J]. PLoS One, 2016, 11: e0148924. DOI: 10.1371/journal.pone.0148924
[37] Sutherland KA, Mbisa JL, Cane PA, et al. Contribution of Gag and protease to variation in susceptibility to protease inhibitors between different strains of subtype B human immunodeficiency virus type 1[J]. J General Virol, 2014, 95: 190-200. DOI: 10.1099/vir.0.055624-0
[38] Rhee SY, Sankaran K, Varghese V, et al. HIV-1 protease, reverse transcriptase, and integrase variation[J]. J Virol, 2016, 90: 6058-6070. DOI: 10.1128/JVI.00495-16
[39] Rhee SY, Taylor J, Fessel WJ, et al. HIV-1 protease mutations and protease inhibitor cross-resistance[J]. Antimicrob Agents Chemother, 2010, 54: 4253-4261. DOI: 10.1128/AAC.00574-10
[40] White KL, Kulkarni R, McColl DJ, et al. Week 144 resistance analysis of elvitegravir/cobicistat/ emtricitabine/t enofovir DF versus efavirenz/emtricitabine/tenofovir DF in antiretroviral-naive patients[J]. Antivir Ther, 2015, 20: 317-327. DOI: 10.3851/IMP2885
[41] Castagna A, Spagnuolo V, Galli L, et al. Simplification to atazanavir/ritonavir monotherapy for HIV-1 treated individuals on virological suppression: 48-week efficacy and safety results[J]. AIDS, 2014, 28: 2269-2279. DOI: 10.1097/QAD.0000000000000407
[42] Tan Q, Zhu Y, Li J, et al. Structure of the CCR5 chemokine receptor-HIV entry inhibitor maraviroc complex[J]. Science, 2013, 341: 1387-1390. DOI: 10.1126/science.1241475
[43] Jiang X, Feyertag F, Meehan CJ, et al. Characterizing the diverse mutational pathways associated with R5-tropic maraviroc resistance: HIV-1 that uses the drug-bound CCR5 coreceptor[J]. J Virol, 2015, 89: 11457-11472. DOI: 10.1128/JVI.01384-15
[44] Archer J, Weber J, Henry K, et al. Use of four next-generation sequencing platforms to determine HIV-1 coreceptor tropism[J]. PLoS One, 2012, 7: e49602. DOI: 10.1371/journal.pone.0049602
[45] Kolomeets AN, Varghese V, Lemey P, et al. A uniquely prevalent nonnucleoside reverse transcriptase inhibitor resistance mutation in Russian subtype A HIV-1 viruses[J]. AIDS, 2014, 28: 1-8. DOI: 10.1097/QAD.0000000000000485
[46] Liegler T, Abdel-Mohsen M, Bentley LG, et al. HIV-1 drug resistance in the iPrEx preexposure prophylaxis trial[J]. J Infect Dis, 2014, 210: 1217-1227. DOI: 10.1093/infdis/jiu233
[47] Baeten, Donnell D, Ndase P, et al. Antiretroviral prophylaxis for HIV prevention in heterosexual men and women[J]. N Engl J Med, 2012, 367: 399-410. DOI: 10.1056/NEJMoa1108524
[48] Avila-Rios S, Garcia-Morales C, Tapia-Trejo D, et al. HIV pre-treatment drug resistance in Mexico: A nationally representative WHO durvey. Abstract 64, 2016, XXV International HIV Drug Resistance Workshop, Boston, USA.
[49] Li JF, Linley L, Kline R, et al. Sensitive sentinel mutation screening reveals differential underestimation of transmitted HIV drug resistance among demographic groups[J]. AIDS, 2016, 30: 1439-1445. DOI: 10.1097/ QAD.0000000000001099
[50] Mourad R, Chevennet F, Dunn DT, et al. A phylotype-based analysis highlights the role of drug-naive HIV-positive individuals in the transmission of antiretroviral resistance in the UK[J]. AIDS, 2015, 29: 1917-1925. DOI: 10.1097/QAD.0000000000000768
[51] Castro H, Pillay D, Cane P, et al. Persistence of HIV-1 transmitted drug resistance mutations[J]. J Infect Dis, 2013, 208: 1459-1463. DOI: 10.1093/infdis/jit345
[52] Schultze A, Phillips AN, Paredes R, et al. HIV resistance testing and detected drug resistance in Europe[J]. AIDS, 2015, 29: 1379-1389. DOI: 10.1097/QAD.0000000000000708
[53] Gonzalez-Serna A, Min JE, Woods C, et al. Performance of HIV-1 drug resistance testing at low-level viremia and its ability to predict future virologic outcomes and viral evolution in treatment-naive individuals[J]. Clin Infect Dis, 2014, 58: 1165-1173. DOI: 10.1093/cid/ciu019
[54 ] Bonner K, Mezochow A, Roberts T, et al. Viral load monitoring as a tool to reinforce adherence: a systematic review[J]. J Acquired Immune Deficiency Syndromes, 2013, 64: 74-78. DOI: 10.1097/QAI.0b013e31829f05ac
[55] Paton N, Kityo C, Hoppe A, et al. Assessment of second-line antiretroviral regimens for HIV therapy in Africa[J]. N Engl J Med, 2014, 371: 234-247. DOI: 10.1056/NEJMoa1311274

