新型流感病毒快速实验室鉴定研究进展
王蔚+卢洪洲
2017年入冬以来,我国南北方省份流感活动水平上升较快,当前处于冬季流感流行高峰水平。全国流感监测结果显示,流感样病例就诊百分比和流感病毒检测阳性率均显著高于过去三年同期水平,流感活动水平仍呈现上升态势,本次冬季流感活动强度要强于往年。2018年1月,为有效应对流感疫情,国家卫生计生委和国家中医药管理局联合印发了《关于做好2018年流感防治工作的通知》和《流行性感冒诊疗方案(2018年版)》。
2018年1月8日,由浙江大学李兰娟院士领衔、上海市公共卫生临床中心卢洪洲教授等专家共同参与完成的“以防控人感染H7N9禽流感为代表的新发传染病防治体系重大创新和技术突破”项目获“2017年度国家科技进步奖特等奖”。这是该奖项设立以来,我国医药卫生行业、教育行业“零”的突破。该项目在发现新病原、确认传染源、明确发病机制、有效临床救治、研发新型疫苗和诊断技术等方面取得了重大创新和技术突破。
流感防控的诸多环节中,流感病毒的快速鉴定至关重要,但却一直是个技术难点。为此,本刊特向上海市公共卫生临床中心相关专家约稿,希望广大医药工作者,特别是临床一线门急诊医护人员对流感疫情引起高度重视,并对相关检测鉴定技术有所了解。
摘 要 流感病毒是新发突发传染病防控重点,而临床标本中微量未知新型流感病毒快速鉴定一直是个难点。本文介绍最新高通量分子诊断技术研究进展,通过组合应用荧光多重PCR、consensus PCR、一代测序和二代测序技术等不同技术,不经过细胞培养分离,快速鉴定临床标本中新型甲型流感病毒的技术流程。
关键词 新型甲型流感病毒 高通量分子诊断技术 病原体快速鉴定体系
中图分类号:R511.7; R446 文献标识码:A 文章编号:1006-1533(2018)03-0001-04
Research progress on rapid laboratory identification of novel influenza A virus
WANG Wei*, LU Hongzhou**
(Shanghai Public Health Clinical Center, Fudan University, Shanghai 201508, China)
ABSTRACT Influenza virus is a key pathogen for the prevention and control of the emerging infectious diseases, and the rapid identification of a new novel genotype of influenza virus in clinical specimens has been a difficult problem. This paper introduces the research progress of new high-throughput molecular diagnostic techniques, and the procedure of rapid identification of novel influenza A virus from clinical samples without cell culture by using in combination of fluorescence multiple PCR, consensus PCR and next-generation sequencing technology.
KEY WORDS novel influenza A virus; high throughput molecular diagnosis technology; rapid identification system of pathogens
流感病毒是一種重要的呼吸道传染病原体。每年流行季可导致数十万人死亡。不仅如此,人类历史上多次发生流感大流行,造成百万甚至千万人死亡。进入2000年以来,新型甲型流感病毒如pamH1N1病毒在2009年造成全球大流行,新发现H5N1、H7N9、H5N6和H10N8等禽流感病毒在全球或局部地区不断出现人感染病例[1-5]。因此,流感全球大暴发的潜在威胁一直存在,是新发突发传染病防控的重点。
流感病毒属于正黏病毒科,根据基因组和抗原特性分为甲乙丙三个属。甲型流感病毒为有包膜负链RNA病毒,基因组分成8个节段,分别编码11个病毒蛋白(PB2、PB1、PBl-F2、PA、HA、NP、NA、M1、M2、NS1、NEP)。其中,血凝素HA蛋白和神经酰胺酶NA蛋白是病毒的表面抗原蛋白,根据各自抗原特性和序列特征细分成不同亚型,已知HA蛋白常见亚型有18种,NA蛋白有11种。甲型流感病毒亚型命名是根据不同HA和NA蛋白的组合。水禽类是流感病毒的贮存宿主,携带了几乎所有18种HA和11种NA亚型病毒,人感染的流感病毒亚型主要为H1、H2、H3和N1、N2。H1N1和H3N2流感病毒是人季节性流感。当人流感病毒与某个禽流感病毒发生重组产生新病毒,或者某个禽流感病毒通过自身点突变的积累而获得感染人体细胞的能力,而人类对这些新病毒并没有免疫能力,很可能会再次发生流感大流行。
新型流感病毒疫情出现时,若实验室在第一时间确诊病原体,对于患者的及时救治、有效防控措施的制定等都是至关重要的。目前全新分子生物学技术在此领域的应用使新病原诊断的周期不断缩短,灵敏度和准确性也不断提高。
2013年3月初,上海市公共卫生临床中心在会诊上海市第五人民医院一位不明原因重症肺炎患者时,规范地留取临床标本,按照未知呼吸道病原体鉴定流程,采用荧光多重PCR、consensus PCR、一代测序和二代测序技术,从临床标本中直接确定病原体为一种新型甲型H7N9禽流感病毒,并获得临床毒株全长基因组序列,对毒株的耐药位点进行耐药分析[6]。通常新病毒确诊需要通过鸡胚或细胞接种分离得到毒株然后测序得到毒株的全基因组序列[2]。由于病毒鸡胚/细胞分离株不可避免地受到培养过程中环境选择的压力,导致毒株发生适应性突变以及优势毒株优先得到扩增,因此不能充分反映临床标本中病毒群落的生物学特性。为了进一步研究病毒从禽到人跨种传播和毒力变化的原因、评估抗病毒药物的疗效以及预测病毒人际传播的潜在可能,从患者临床标本中直接得到病毒全基因组序列和遗传多态性信息已经成为被广泛接受的最新研究策略[4-5]。endprint
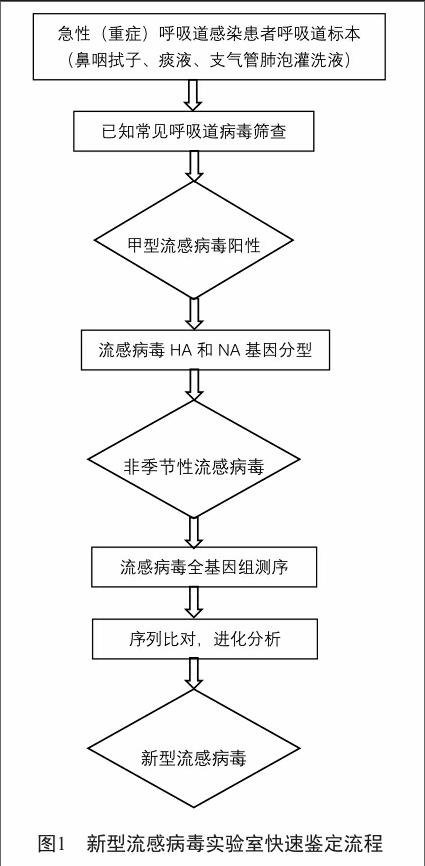
本文重点介绍采用组合应用不同高通量分子诊断技术的策略,直接从临床标本入手,快速鉴定新型甲型流感病毒的技术流程。该流程包括三个步骤:流感病毒病毒诊断、甲型流感病毒分型和新型流感病毒鉴定(图1)。
1 甲型流感病毒检测
1.1 核酸检测
流感病毒与鼻病毒、呼吸道合胞病毒和腺病毒等10多种病原体引发的呼吸道感染临床症状极为相似且流行季节重叠。因此,对急性呼吸道感染患者的临床标本(鼻咽擦拭子、痰液、支气管肺泡灌洗液)需要进行流感病毒和其他已知常见呼吸道病原体初步筛查。筛查采用分子检测方法,以不同病毒基因各自高度保守序列为靶序列,设计一系列引物和探针,各个单重PCR扩增产物通过片段大小或荧光信号筛查阳性标本[7-8]。
以Luminex和Perkin Elmer公司分别研发的“液相悬浮芯片”、BioFire公司研发的FilmArray等为代表的高通量病原体核酸检测技术,采用多重PCR或多重实时荧光PCR扩增病毒核酸,再结合下游不同杂交技术的策略[9-10]。与传统单个病原体特异性PCR相比,这些技术一次可同时检测10多种甚至几十种病原,为及时快速地筛查鉴定病原体提供了有效的手段,但是检测成本较高和配套设备复杂昂贵等因素限制了这些技术在临床应用上的推广。
1.2 血清学诊断
患者急性期(发病5 d内)血清和恢复期血清(病程2~4周)双份血清,在相同条件下做血凝抑制(hemaglutination inhibition, HI)试验可作为流感病毒感染辅助诊断,但是诊断的灵敏性和特异性较差不适用于流感病毒快速诊断。
1.3 病毒分离与鉴定
流感病毒确诊的金标准是毒株分离培养。流感病毒分离培养采用鸡胚或猴胚肾细胞(MDCK细胞)接种临床呼吸道标本,3~4 d后取鸡胚囊液或细胞上清液做血凝实验或实时荧光定量PCR检测确定阳性结果。该方法操作要求高且耗时久,不适用于流感病毒的快速诊断。
2 甲型流感病毒分型
流感病毒是高突变病毒,其基因组的复制需要病毒PA、PB1和PB2蛋白组成的病毒RNA聚合酶复合物,复制后的基因节段与新合成的病毒蛋白装配成子代病毒。由于病毒聚合酶缺乏5à3端核酸校正功能,因此在RNA复制过程中易出现错配,导致子代病毒基因组和病毒蛋白氨基酸突变。突变累积会造成病毒蛋白抗原性改变,产生病毒对人体免疫逃逸,是人季节性流感流行的原因。同时,当两种以上不同亚型的流感病毒感染同一宿主细胞时,各自基因节段在子代病毒装配过程中发生重配,从而产生新型重组病毒。因此,流感病毒分型对病毒的流行监测具有重要意义。
流感病毒基因组有8个节段,其中M和NP基因高度保守,一般作为甲型流感病毒的型别鉴定。HA和NA基因则用于人季节性甲型流感病毒(H1N1和H3N2)分型。
HA和NA基因分型可采用传统PCR或荧光定量PCR方法,利用已知基因序列信息进行多序列的比对(alignment),找出基因组中合适的区域段作为靶点,设计H1~H18和N1~N11不同亚型特异性引物和探针[11-12]。由于流感病毒具有高突变率,引物选择和数据分析较为复杂。随着病毒变异不断积累导致靶序列的较大变化,需要相应地更新或更换检测引物。
3 新型流感病毒鉴定
由于新型甲型流感病毒是不同亚型甲型流感病毒的8个基因节段重组而来,因此新型甲型流感病毒的鉴定需要确定8个片段基因序列,通过序列比对和进化分析确定病毒亚型。
为获得病毒8个基因节段序列,研究人员建立了基于保守序列的PCR扩增(consensus PCR)。首先,通过序列比对找到各个相对保守的序列区域设计PCR通用引物。用consensus PCR扩增所获片段,通过直接测序得到基因序列[13]。该方法特异性强,但是要求临床标本中病毒有较高滴度,而且实验周期较长。
近年发展起来的高通量测序技术(high-throughput sequencing, HTS)能夠更简单更全面检测病毒全基因组序列,它通过导入特异性接头序列,对每个输入(input)的临床标本中的DNA分子进行大规模平行扩增和合成测序,然后通过生物信息分析,将所得每条序列与数据库序列进行比对、拼接和组装,从而完成病毒鉴定[14]。它的最大检测优势在于不需要经过细胞培养,直接对临床标本中的核酸池(pool)进行测序;而它的弱点在于检测特异性较低,主要原因是建库过程中无法完全去除宿主基因组和环境微生物基因组的污染。另外,它必需有专业生物信息平台的支持,才能对产生的海量测序数据进行后期处理。因此,高通量测序技术较适用于无法体外培养分离的病毒、高度变异病毒、重组病毒以及新病毒的鉴定和研究[14]。
目前高通量技术平台有3家,分别由Roche、ABI和Illumina公司开发。相对于一代Sanger测序法,这些技术被统称为二代测序技术。三家研发了不同的化学扩增体系、测序信号检测系统和分析软件,其各自具体原理本文不再赘述[14]。三家平台各有所长,根据其通量、平均读长、准确性和使用成本的差异,适合于不同研究应用。对于新病原鉴定,一个HTS平台最重要技术指标是:①模板DNA最低起始量;②测序时间和成本;③测序通量;④测序读长。相较于商业化测序服务公司配备大型HTS平台,临床诊断实验室多选择小型HTS平台包括Miseq和Ion Torrent PGM,直接从单个临床标本中获得病毒全基因组序列。
值得一提的是,尽管高灵敏度的检测方法可以帮助发现标本中的病毒核酸,但如何确认被检测到的微生物是导致疾病或疫情暴发的病原体,始终是病原体鉴定中的核心问题[15]。新病原体与导致疾病之间因果关系的论证需要遵从科赫(Koch)法则。endprint
4 結语
临床标本中微量已知病毒的鉴定通常根据其形态学、特异性抗原和特定基因序列,经过或不经过细胞培养分离,采用血清学和分子学检测方法来实现。但是,未知新病毒的检测一直是个难点。新型流感病毒的及时鉴定对于临床救治和传染病疫情控制至关重要,新的快速、高通量、高敏感性和高特异性的检测鉴定技术正在不断地被研发和应用。正在研发和验证之中的还包括纳米金颗粒技术、流式细胞技术、三代测序技术与现有的核酸诊断技术的联用等。未来,随着这些技术的不断成熟、标准化和低成本化,有望在临床诊断和疫情防控中被大规模应用。
参考文献
[1] Tran TH, Nguyen TL, Nguyen TD, et al. Avian influenza A(H5N1) in 10 patients in Vietnam[J]. N Engl J Med, 2004, 350(12): 1179-1188.
[2] Gao R, Cao B, Hu Y, et al. Human infection with a novel avian-origin influenza A (H7N9) virus[J]. N Engl J Med, 2013, 368(20): 1888-1897.
[3] Gao HN, Lu HZ, Cao B, et al. Clinical findings in 111 cases of influenza A (H7N9) virus infection[J]. N Engl J Med, 2013, 368(24): 2277-2285.
[4] Yang ZF, Mok CK, Peiris JS, et al. Human infection with a novel avian influenza A (H5N6) virus[J]. N Engl J Med, 2015, 373(5): 487-489.
[5] Chen H, Yuan H, Gao R, et al. Clinical and epidemiological characteristics of a fatal case of avian influenza A H10N8 virus infection: a descriptive study[J]. Lancet, 2014, 383(9918): 714-721.
[6] Hu Y, Lu S, Song Z, et al. Association between adverse clinical outcome in human disease caused by novel influenza A H7N9 virus and sustained viral shedding and emergence of antiviral resistance[J]. Lancet, 2013, 381(9885): 2273-2279.
[7] Wang W, Cavailler P, Ren P, et al. Molecular monitoring of causative viruses in child acute respiratory infection in endemo-epidemic situations in Shanghai[J]. J Clin Virol, 2010, 49(3): 211-218.
[8] Wang W, Ren P, Sheng J, et al. Simultaneous detection of respiratory viruses in children with acute respiratory infection using two different multiplex reverse transcription-PCR assays[J]. J Virol Methods, 2009, 162(1-2): 40-45.
[9] Luminex. Targeted and syndromic molecular diagnostics testing[EB/OL]. [2018-01-25]. http://www.luminexcorp.com/ clinical/infectious-disease.
[10] Biofire. Syndromic testing: the right test, the first time[EB/ OL]. [2018-01-25]. http://www.biofiredx.com.
[11] WHO. Diagnostics for influenza publications, 2010–2011[EB/ OL]. [2018-01-25]. http://www.who.int/influenza/resources/ research/RA_Progress_Report_App11.pdf?ua=1.
[12] Wang W, Ren P, Mardi S, et al. Design of multiplexed detection assays for identification of avian influenza A virus subtypes pathogenic to humans by SmartCycler real-time reverse transcription-PCR[J]. J Clin Microbiol, 2009, 47(1): 86-92.
[13] Hoffmann E, Stech J, Guan Y, et al. Universal primer set for the full-length amplification of all influenza A viruses[J]. Arch Virol, 2001, 146(12): 2275-2289.
[14] Qui?ones-Mateu ME, Avila S, Reyes-Teran G, et al. Deep sequencing: becoming a critical tool in clinical virology[J]. J Clin Virol, 2014, 61(1): 9-19.
[15] Firth C, Lipkin WI. The genomics of emerging pathogens[J]. Annu Rev Genomics Hum Genet, 2013, 14: 281-300.endprint

