槐叶决明SSR标记开发与应用
刘玉洋,叶生月,闵 会,王慧中*,卢江杰*
(1.杭州师范大学 生命与环境科学学院,浙江 杭州 310036; 2.浙江省药用植物种质改良与质量控制技术重点实验室,浙江 杭州 310036; 3.桐庐县农业产业化办公室,浙江 桐庐 311500; 4.浙江康恩贝制药股份有限公司,浙江 杭州 310052)
决明属植物全世界约590种,我国原产约10种,中部、东南部、南部及西南部各省区均有分布[1]。入药用的约19种,具清热明目、润肠通便的功效,常用来治疗便秘、眼病、皮癣、水肿、高血压和头痛等病症。常见以种子入药的有小决明(CassiatoraL.)、望江南(C.occidentalisL.)和槐叶决明(C.sopheraL.)等[2]。小决明的种子即最常用的中药“决明子”,目前广泛用于治疗高血脂症、高血压,由于泻下作用缓和又可降脂,成为减肥用品中的最常用组分之一。望江南在医药上常用作缓泻剂,种子炒后治疟疾;根有利尿功效;鲜叶捣碎治毒蛇毒虫咬伤。但有微毒,牲畜误食过量可以致死,近来关于望江南中毒病例的报道逐渐增多[3]。槐叶决明也称“茳芒决明”,据《中药大辞典》中记载:“茳芒决明,性平,无毒,味甘滑”。现代医学研究还表明槐叶决明叶的醇提物有松弛支气管平滑肌等作用[4-6]。
民间历来有望江南代替决明使用的情况,而望江南和槐叶决明由于形态相似,也常混用[2]。所以如何区分决明属不同种是中药质量研究的热点之一。宋贤丽等[7]采用高效毛细管电泳对决明属植物种子中的水溶性成分进行分析,可以较好地区分6种决明属植物。此外,蒽醌类、黄酮类和萜类等化学成分的化学指纹图谱被广泛应用于决明属植株的研究[8-9]。近年来,DNA分子标记也开始在决明属植物中得到应用。如王静等[10]建立了SRAP分子标记体系,可用于决明属植物的遗传多样性和种质鉴定等研究。但是可用的SRAP分子标记数量有限,一定程度上限制了决明属植物大规模研究工作的开展。随着高通量测序技术被越来越广泛地应用于转录组分析,通过转录组序列开发全基因组SSR分子标记,用于遗传多样性分析、遗传图谱构建、目标基因定位以及指纹图谱绘制等研究成为可能[11]。Jain等[12]在沙棘转录组中发现7.69%的序列含有SSR位点,而且平均每6.704 kb就有一个SSR位点。Li等[13]通过同色兜兰3.77 Gb的转录组数据,从3 975条含有SSR位点的序列中获得4 989对SSR引物。Zeng等[14]从箭叶淫羊藿转录组序列中随机选择32个SSR分子标记进行验证,其中18对引物可以扩增出条带。目前决明属的SSR标记开发尚未见报道,本研究通过分析槐叶决明转录组数据,利用生物信息学方法搜寻SSR位点,并设计SSR标记的引物,进而利用SSR标记分析小决明、望江南和槐叶决明等决明属材料,以期利用开发的决明属SSR分子标记进行该属植物的种质鉴定和遗传多样性分析等相关研究。
1 材料与方法
1.1 材料
实验材料为不同地区收集的1份槐叶决明种子、3份望江南种子和5份小决明种子,具体信息见表1。种子发芽后,温室内正常培养,待植株长到20 cm高后,采集新鲜叶片,液氮冷冻处理后,超低温保存,用于后续提取DNA。
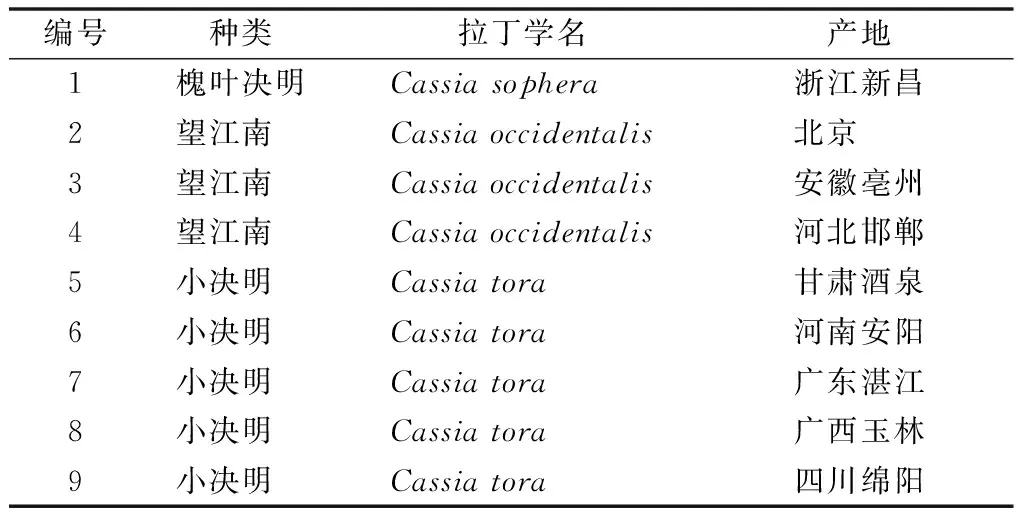
表1 材料编号名称与产地
1.2 方法
1.2.1 基因组DNA的提取与检测
DNA提取参考Doyle等[15]的CTAB法,利用超微量紫外分光光度计(NanoUV-3000)进行DNA浓度测定。并利用琼脂糖凝胶电泳检测DNA纯度。
1.2.2 转录组数据分析和SSR位点引物设计
利用MISA软件对浙江省药用植物种质改良和质量控制技术重点实验室拥有的槐叶决明转录组数据进行生物信息学分析,按照软件默认参数鉴定单碱基重复SSR、双碱基重复SSR、三碱基重复SSR、四碱基重复SSR、五碱基重复SSR和六碱基重复SSR。
使用软件Primer Premier 5.0进行引物设计。设计时设置的主要参数为:EST序列长度在150 bp以上;引物长度控制在18~25 bp;避免产生二级结构如dimer,hairpin,falseprimer等;CG含量在40%~70%;退火温度处于45.0~65.0 ℃;PCR扩增产物的长度在100~300 bp。引物由上海生工公司合成。
1.2.3 SSR分子标记PCR扩增和引物筛选
PCR反应体系(10 μL)如下:10×PCR Buffer 1 μL,10 mmol·L-1dNTPs 0.3 μL,10 μmol·L-1的正向和反向引物各0.5 μL,2 U·L-1Taq酶0.5 μL,20 ng·μL-1DNA模板1 μL,ddH2O 6.2 μL。PCR反应程序如下:94 ℃预变性3 min,32个循环(94 ℃变性30 s,各引物退火温度下复性40 s,72 ℃延伸90 s),最后72 ℃延伸10 min。选择2个材料的基因组DNA筛选合成的SSR引物,选出条带清晰且多态性好的引物用于后续所有材料的PCR扩增。
1.2.4 聚丙烯酰胺凝胶电泳和数据分析
将8%丙烯酰胺40 mL,10%过硫酸铵280 μL,TEMED 26 μL,混合均匀后水平灌胶,插上68孔梳子,水平放置直至胶凝固(30 min左右)。在电泳槽中加入适量1×TBE缓冲液,上样,在200 V电压下电泳分离2 h。电泳结束后,用蒸馏水将凝胶漂洗2 min后,置入800 mL 0.1%的AgNO3溶液中染色25 min(期间使用摇床摇动);之后在蒸馏水中漂洗2次,每次10 s。将漂洗之后的凝胶放入含有800 mL 1%的氢氧化钠溶液中(含2 mL甲醛)显影,摇床摇动直至凝胶上出现清晰的条带。用蒸馏水漂洗10 s。
使用BIO-RAD visadoc 3.0(Bio-RAD,USA)成像系统进行凝胶扫描后,通过Quantity One软件依据DNA Marker计算每个条带的分子量大小,并依据SSR标记的预计大小和重复单元进行矫正,确定等位变异大小(bp)和数量,由软件PowerMarker version 3.25[16]进行遗传丰富度(A)和多态信息量(PIC)等多样性指数计算。
2 结果与分析
2.1 槐叶决明转录组SSR位点分布特点
利用MISA软件对拼接得到的103 305条槐叶决明unigene进行SSR位点搜索,共检测到15 753个SSR位点,可用于标记开发。如表2所示,单碱基重复数SSR位点数占39.55%,其中以A/T占比最大(38.51%);双碱基重复数占27.40%,其中AG/CT占9.36%;三碱基重复数占30.84%,其中AAG/CTT占9.36%;四碱基重复数占1.44%,其中AAAG/CTTT占0.37%;五碱基重复数占0.23%,其中AAAAG/CTTTT占0.03%;六碱基重复数占0.53%,其中AAGAGG/CCTCTT占0.01%。
2.2 SSR分子标记开发和验证
对15 753个SSR位点利用Primer Premier 5.0进行引物设计,最终有10 110个SSR位点可以设
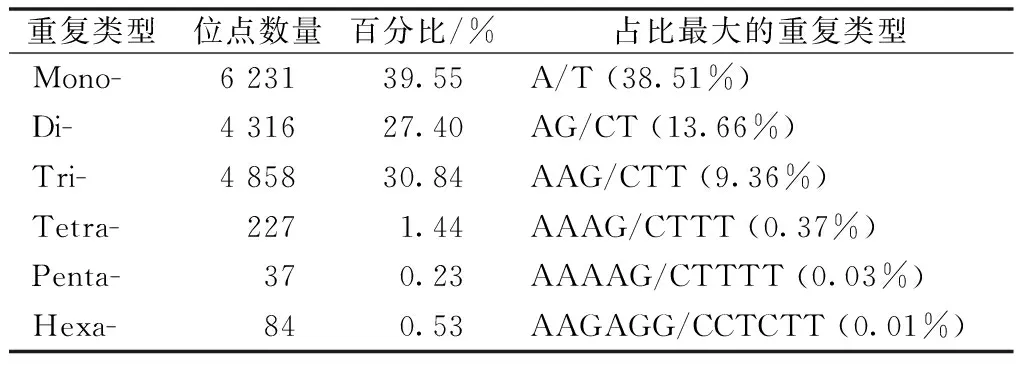
表2 槐叶决明转录组SSR位点各类型的情况
计SSR引物。随机选择50对SSR引物由上海生工合成,通过PCR扩增,聚丙烯酰胺凝胶凝胶电泳,银染,拍照,经初步筛选得到25对条带清晰、有差异的SSR引物可用于后续不同产地槐叶决明遗传多样性分析。这些标记的等位基因数量从2到7不等,平均有2.72个等位基因;遗传丰富度从0.20到0.73不等,平均为0.45;PIC从0.18到0.70不等,平均为0.39(表3)。图1为SSR引物CPtSSR008在不同试验材料中的电泳结果。
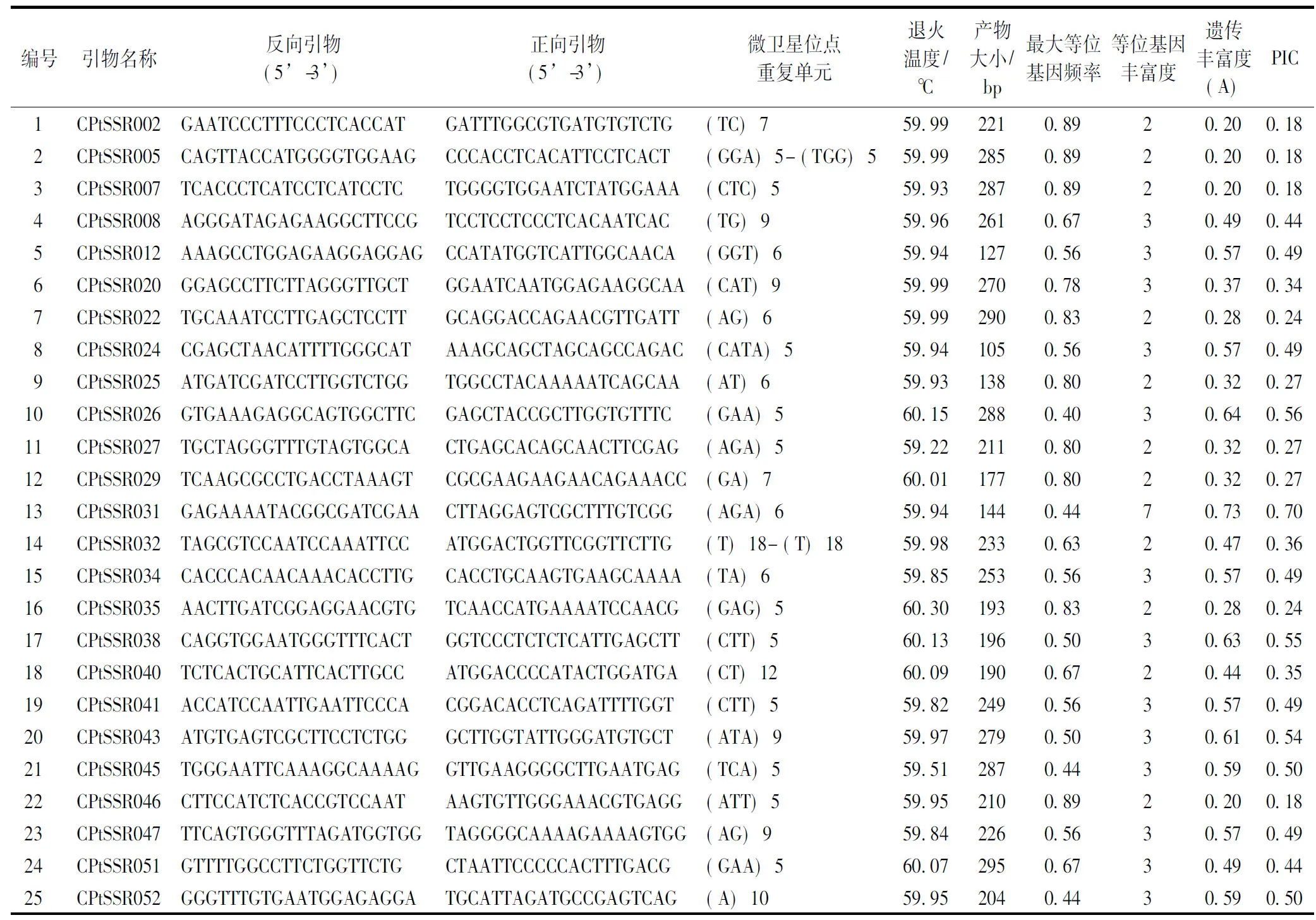
表3 本研究中用于遗传多样性分析的25对SSR分子标记信息
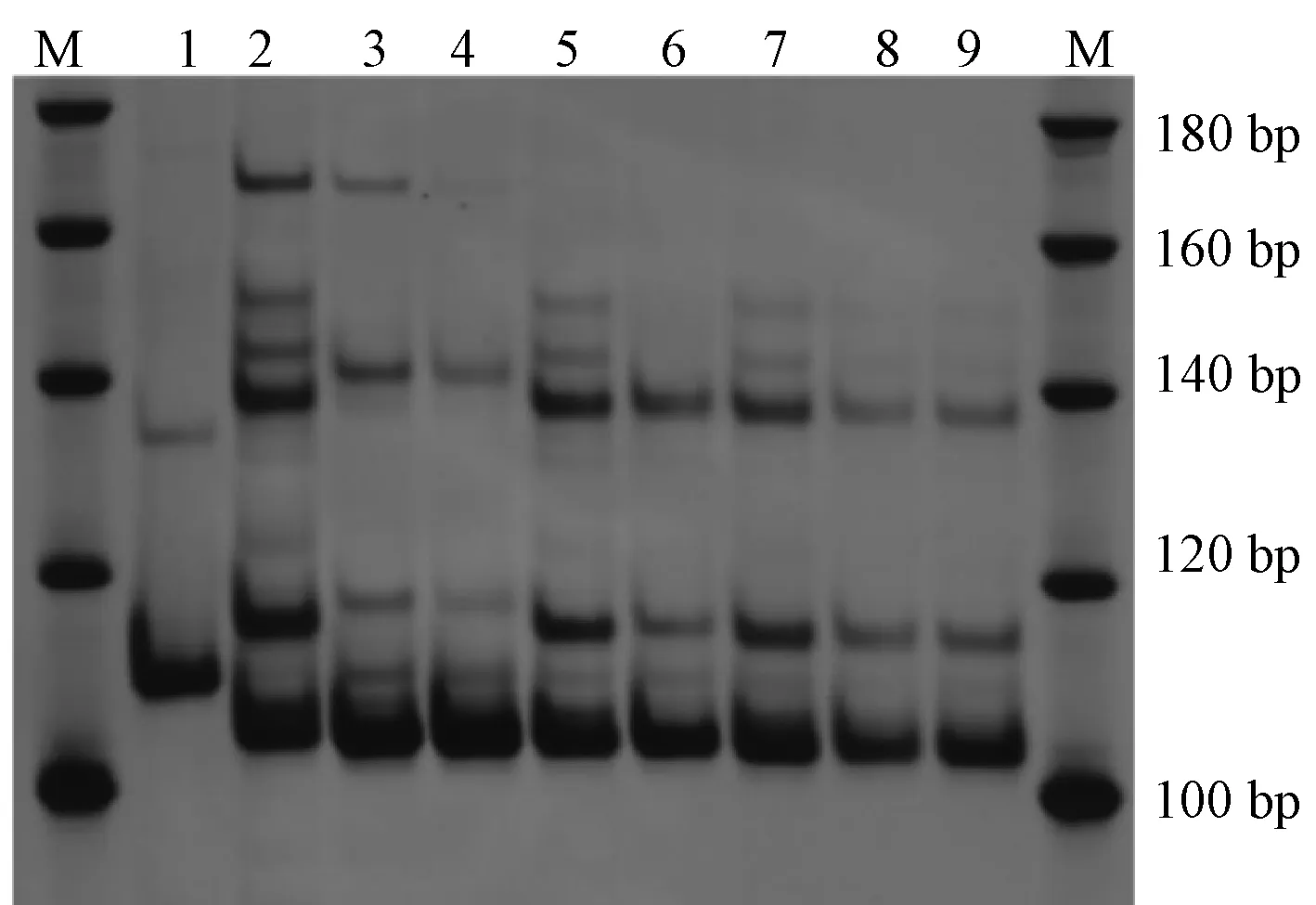
1~9分别表示本研究中使用的决明属种质资源材料,与表1相同。M为DNA标准分子量图1 引物CPtSSR008的电泳结果
2.3 不同决明属材料的遗传多样性分析
根据25对SSR引物电泳结果,通过聚类距离比较分析发现:槐叶决明和不同产地决明子、不同产地决明子之间遗传距离在0.111~0.963,其中槐叶决明、小决明和望江南各种之间,以及小决明和望江南不同地理来源之间均存在较大遗传差异(表4)。
进一步通过遗传聚类分析,我们发现槐叶决明、小决明和望江南可分为三类:槐叶决明为第一类;河北、安徽、北京产地的望江南为第二类;四川、广西、广东、甘肃、河南产地的小决明为第三
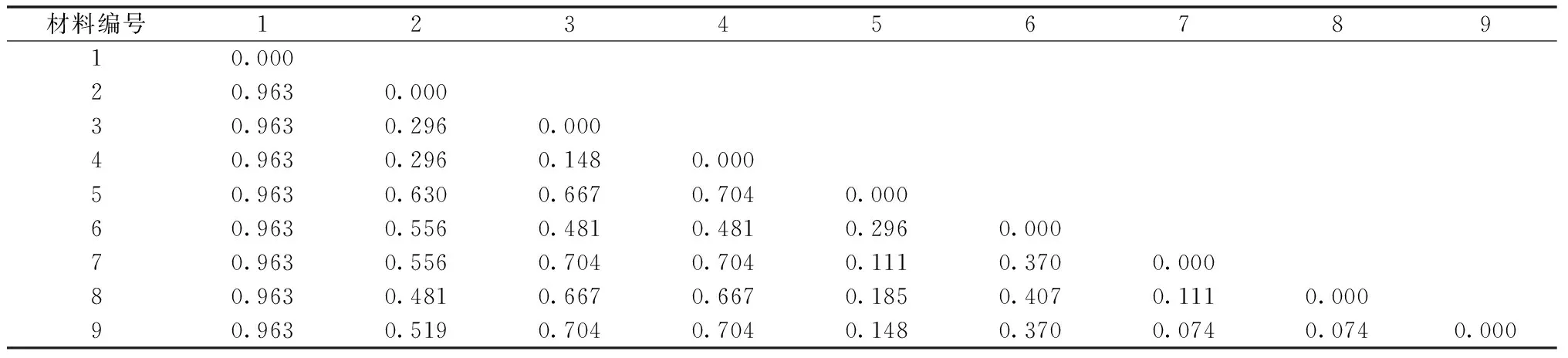
表4 各材料的遗传距离
类(图2)。从种子外观上看,槐叶决明种子与决明差异较大,这与利用SSR分子标记鉴定的结果一致,而不同产地的槐叶决明和望江南材料无论种子还是植株根茎叶差异都不大,很难从外观上鉴定出不同,利用SSR分子标记技术,我们鉴定出了槐叶决明和不同产地望江南的差异,从而为槐叶决明和望江南的种质资源分析鉴定提供依据。
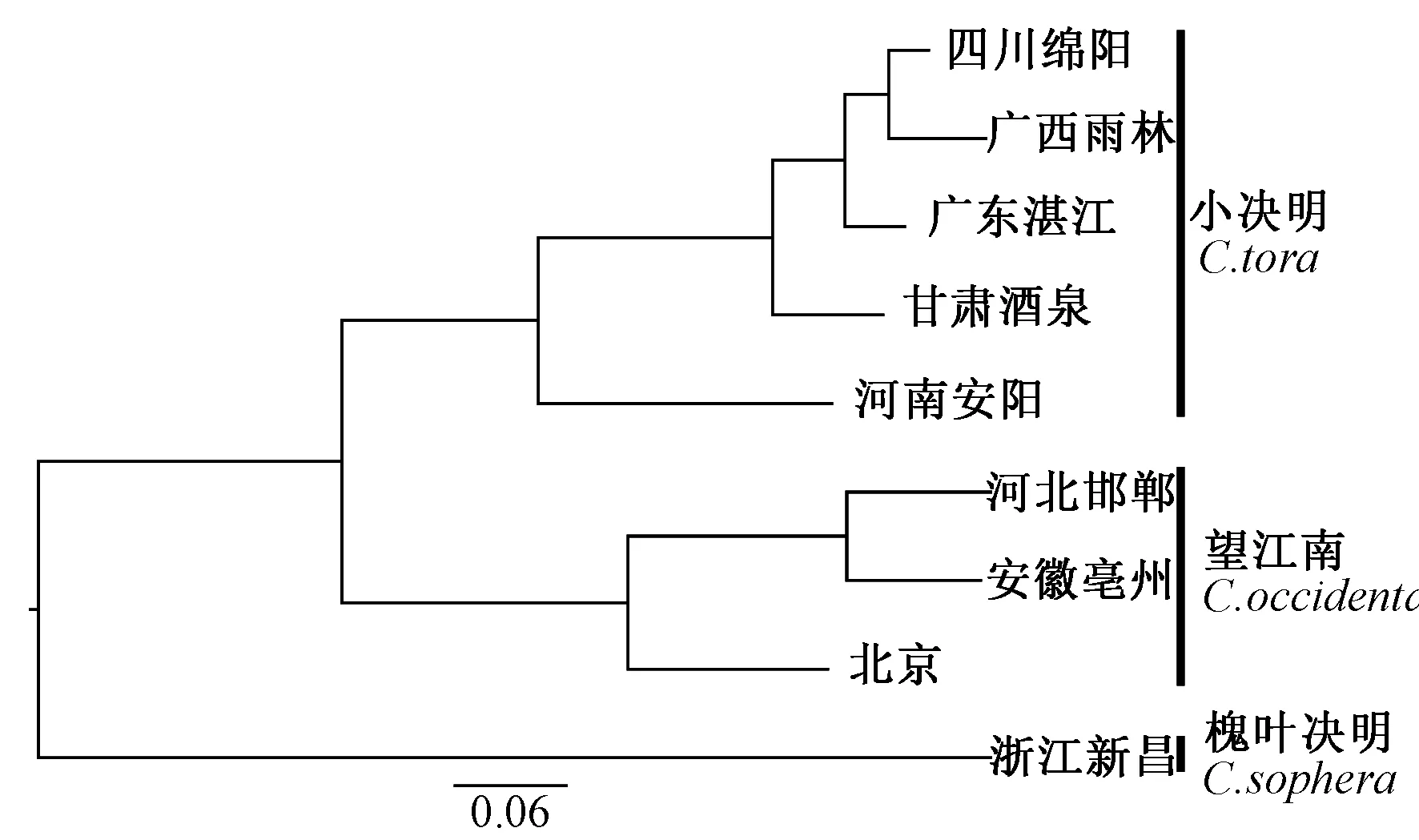
图2 基于SSR分子标记的决明属遗传聚类
3 小结与讨论
通过分析槐叶决明转录组数据,本研究共发掘了15 753个SSR位点,并成功设计出10 110对SSR引物。随机选取其中的50对SSR引物中,有电泳条带清晰和多样性高的SSR引物25对,可用于本实验室收集的决明属材料遗传多样性分析以及种质资源鉴定。因此,我们预计本研究共开发决明属SSR标记5 000个左右。而且这些标记可为槐叶决明、小决明和望江南3个种为代表的决明属材料相关研究提供分子标记数据库。
[1] 中国科学院中国植物志编辑委员会.中国植物志[M]. 北京:科学出版社, 1988: 125.
[2] 连文琰. 中国决明属药用植物简报[J]. 中草药, 1986, 17(70): 27.
[3] 韩文雯, 李天祥. 望江南子的生药学研究[J]. 天津药学, 2011, 23(3): 49-51.
[4] SINGH R, HUSSAIN S, VERMA R, et al. Anti-mycobacterial screening of five Indian medicinal plants and partial purification of active extracts ofCassiasopheraandUrticadioica[J]. Asian Pacific Journal of Tropical Medicine, 2013,6(5):366-371.
[5] TIWARI R D, MISRA G. Chemical examination of the flowers ofCassiasophera[J]. Planta Medica, 1975, 28(2): 182-185.
[6] ZHAO Y, LIU J P, LU D, et al. A novel cycloartane triterpene glycoside from the seeds ofCassiasopheraL. [J]. Natural Product Research, 2007, 21(6): 494-499.
[7] 宋贤丽, 郭宝林, 刘克武, 等. 6种决明属药用植物种子毛细管电泳法鉴别[J]. 中国中药杂志, 2003,28(6): 14-19.
[8] 李婷, 冯占民, 杨巡纭, 等. 决明属植物的化学成分及药理作用研究进展[J]. 林产化学与工业, 2012, 32(6): 107-118.
[9] 胡勇, 陈丽琼, 朱霞, 等. 决明属化学成分与药理作用研究进展[J]. 现代中药研究与实践, 2013, 27(5): 69-71.
[10] 王静, 张跃进, 梁宗锁, 等. 决明DNA提取、SRAP-PCR优化及引物筛选[J]. 西北农业学报, 2014, 23(2): 137-142.
[11] KALIA R K, RAI M K, KALIA S, et al. Microsatellite markers: an overview of the recent progress in plants [J]. Euphytica, 2011, 177(3): 309-334.
[12] JAIN A, CHAUDHARY S, SHARMA P C. Mining of microsatellites using next generation sequencing of seabuckthorn (HippophaerhamnoidesL.) transcriptome [J]. Physiology and Molecular Biology of Plants: An International Journal of Functional Plant Biology, 2014, 20(1): 115-123.
[13] LI D M, ZHAO C Y, LIU X R, et al. De novo assembly and characterization of the root transcriptome and development of simple sequence repeat markers inPaphiopedilumconcolor[J]. Genetics and Molecular Research, 2015, 14(2): 6189-6201.
[14] ZENG S, XIAO G, GUO J, et al. Development of a EST dataset and characterization of EST-SSRs in a traditional Chinese medicinal plant,Epimediumsagittatum(Sieb. Et Zucc.) Maxim [J]. BMC Genomics, 2010, 11:94.
[15] DOYLE J J, DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochemical Bulletin, 1987, 19(1): 11-15.
[16] LIU K, MUSE S V. PowerMarker: an integrated analysis environment for genetic marker analysis [J]. Bioinformatics, 2005, 21(9): 2128-2129.

