一种快速提取土壤微生物DNA的方法
陶兴玲
(长江大学生命科学学院,湖北 荆州 434025)
雷琼
(湖北省荆州文物保护中心,湖北 荆州 434020)
马立安
(长江大学生命科学学院,湖北 荆州 434025)
土壤微生物资源丰富,每克土壤含有大约107个原核生物细胞,但用传统的技术培养的微生物仅占微生物总数的0.01%~10%[1],而大部分的微生物处于不可培养的状态,相当多的菌种由于无法培养使之不能被充分地开发和利用,因此从自然环境中提取微生物的DNA 对不可培养细菌的检测、某些目标菌株的跟踪或重组基因在自然条件下的行为有重要的意义[2~5]。
自20世纪70年代开始,研究者就开始关注土壤微生物DNA的提取方法。目前土壤微生物DNA的提取方法主要有直接法[6]和间接法[7,8]。采用间接法提取的DNA纯度比较高,但提取量很少;采用直接法获得的DNA量大并且能代表大部分的土著微生物种群,但是纯度较低。纯化是一件繁琐的事情,目前纯化的方法有氯化铯密度梯度超速离心法、PVPP(聚乙烯基吡咯烷酮)法、色谱法、电泳法、试剂盒、透析和过滤法等[9],但这些纯化方法往往造成核酸的大量损失。陈旭玉等[10]介绍的方法不仅可以获得大片段,并且不需要纯化即可直接用于PCR扩增和DNA酶切等后续操作,每克土壤DNA提取量约为2.1~4.6μg,但是此方法耗费时间长、较繁琐。本研究在陈旭玉等[10]的方法基础上,对该方法的SDS裂解细胞温育时间、PEG沉淀DNA时间、CTAB溶解DNA沉淀温育时间、异丙醇沉淀DNA放置时间、SDS裂解细胞离心条件以及异硫氰酸胍试剂的加入与否等实验条件进行了优化,旨在建立一种经济、快速、简单的土壤微生物DNA提取方法。
1 材料与方法
1.1 土壤样品
土壤样品分为2种:一种采自潜江某油田作业油井及废弃老井附近,共采集7个土样,分别记为:1#、2#、3#、4#、5#、6#、7#;另一种是采自长江大学西校区农学院实验基地:麦田、草地、大豆田、玉米田、水稻田、树林、生活淤泥。土样采集深度为地表20~25cm,所有样品无菌密闭保存于4℃冰箱中。
1.2 试剂
主要试剂有十二烷基硫酸钠(SDS)、乙二胺四乙酸(EDTA)、十六烷基三甲基溴化铵(CTAB)、异硫氰酸胍,均为分析纯级。
1.3 土壤微生物DNA提取方法
原方法:参照陈旭玉等[10]报道的方法。
优化后的方法:(1)SDS裂解土壤微生物细胞:称取10g土壤于50mL离心管,加18.5mL SDS裂解液(0.25mol/L NaCl,0.1mol/L EDTA,4% SDS),涡旋1.5min,68℃温育20min,每10min轻轻摇动,7000r/min离心10min,将上清液移入50mL干净的离心管。(2)PET沉淀DNA去除杂质:加0.125倍体积的5mol/L醋酸钾和0.42倍40% PEG-8000,6000r/min离心15min。(3)CTAB溶解DNA沉淀:加18mL 2×CTAB(2% CTAB,1.4mol/L NaCl,0.1mol/L MEDTA)中溶解沉淀,68℃温育5min。(4)去除蛋白质:加等体积的苯酚/氯仿/异戊醇(25∶24∶1),轻柔混匀,6000r/min离心10min。(5)异丙醇沉淀DNA:在DNA上清液中加0.6~1.0倍体积的异丙醇,13000r/min离心20min。(6)70%的酒精洗涤DNA沉淀:用70%的酒精洗2~3次,自然晾干,加500μL TE缓冲液或无菌水。
1.4 提取方法的优化
优化其中一个条件的时候,其他实验条件参照陈旭玉等[10]的方法,逐条优化。实验过程中,在进行各项条件优化以及后续稳定性和可操作性实验时,均用麦田土壤。
细胞裂解优化内容为:SDS裂解细胞温育时间分别设置15、20、25、30、45、60min 6个不同时间段;SDS裂解细胞离心条件分别设置7000r/min 10min、8000r/min 10min、9000r/min 10min、6000r/min 15min 4种不同条件;异硫氰酸胍的加入与否设置加入与不加入2种条件。
DNA提取优化内容为:PEG沉淀DNA时间分别设置0、5、10、15min 4个不同时间段;CTAB溶解DNA沉淀温育时间分别设置5、8、11、14min 4个不同时间段;异丙醇沉淀DNA放置时间分别设置0、15、30、45、60、180min 6个不同时间段。
1.5 优化提取方法的稳定性研究
采取2种重复方式进行实验,一种是取同一种样品在3个不同时间操作,每次3个重复。另一种是取同一样品进行5个重复操作。
1.6 优化提取方法的应用性研究
采取不同土质的土壤样品分别进行实验,一种采自潜江某油田作业油井及废弃老井附近的7个土壤样品,另一种是采自长江大学西校区农学院实验基地的7个土壤样品,每个样品2次重复。
1.7 优化提取方法的可操作性研究
3人同时操作,每人重复3个样品。

1:15min;2:20min;3:25min;4:30min;5:45min;6:60min图1 不同SDS裂解细胞温育时间下的DNA琼脂糖凝胶电泳图
2 结果与分析
2.1 土壤微生物DNA提取方法的优化
2.1.1 细胞裂解优化结果
采用6个不同时间段进行SDS裂解细胞温育时间优化,结果如图1所示。由图1可知,采用温育20min 即可达到所需要求,故可将SDS裂解细胞温育时间从1h缩短至20min。
采用4种不同条件进行SDS裂解细胞离心条件优化,结果如图2所示。由图2可知,采用7000r/min 10min、8000r/min 10min 均可达到所需要求,最终选择7000r/min 10min,以通过提高转速缩短离心时间,从而缩短试验时间。
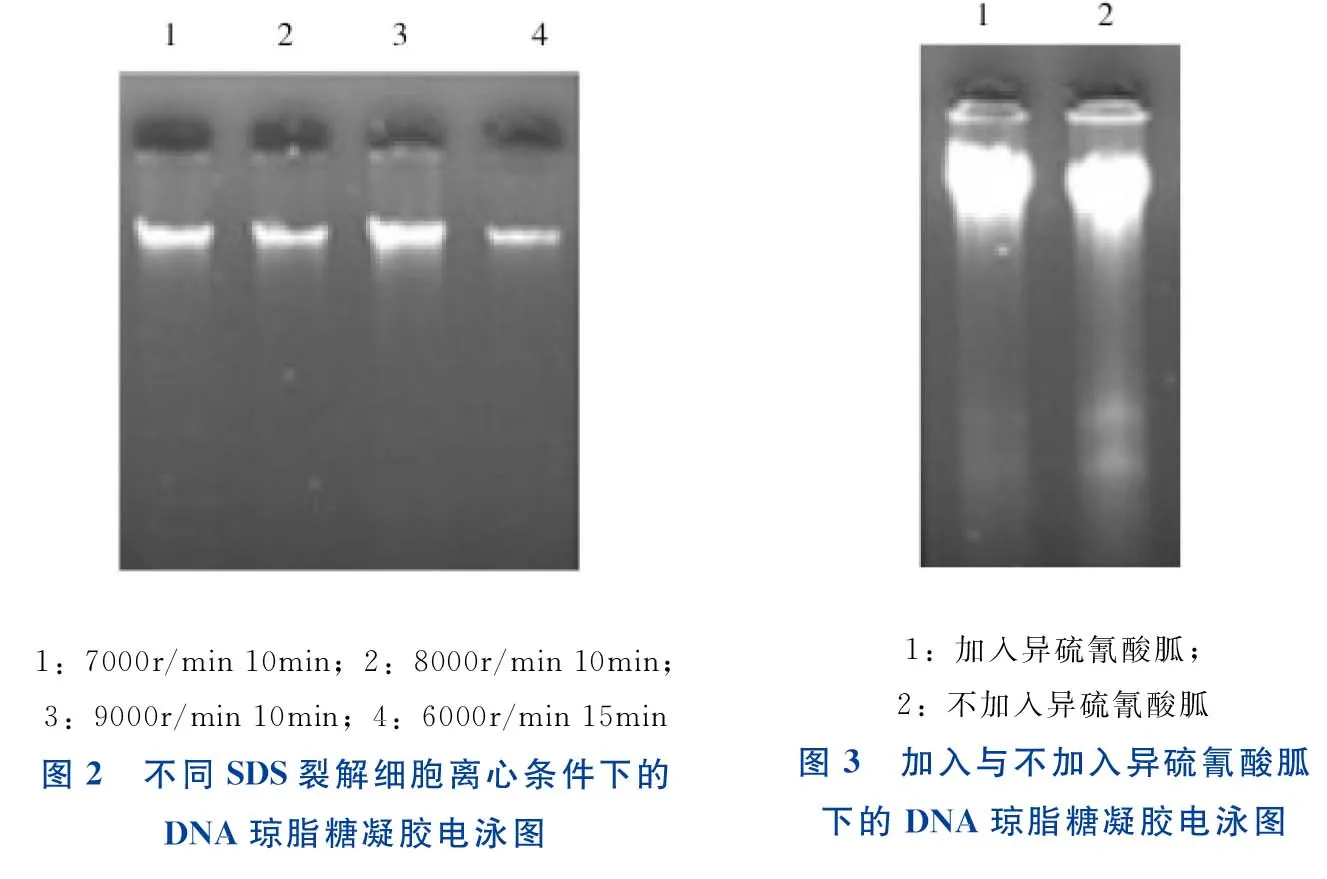
1:7000r/min10min;2:8000r/min10min;3:9000r/min10min;4:6000r/min15min图2 不同SDS裂解细胞离心条件下的DNA琼脂糖凝胶电泳图1:加入异硫氰酸胍;2:不加入异硫氰酸胍图3 加入与不加入异硫氰酸胍下的DNA琼脂糖凝胶电泳图
采用加入与不加入异硫氰酸胍2种条件进行比较,结果如图3所示。由图3可知,不加入异硫氰酸胍试剂对提取结果影响不明显,因此优化的实验方法不加入异硫氰酸胍,可使该实验方法更经济。
2.1.2 DNA提取优化结果
采用4个不同时间段进行PEG沉淀DNA时间优化,结果如图4所示。由图4可知,采用0 min 即可达到所需要求,故可将PEG沉淀DNA这一步骤省去,以缩短试验时间。
采用4个不同时间段进行CTAB溶解DNA沉淀温育时间优化,结果如图5所示。由图5可知,采用5min 即可达到所需要求,故可将CTAB溶解DNA沉淀温育时间从15min 缩短至5min。
采用6个不同时间段进行异丙醇沉淀DNA放置时间优化,结果如图6所示。由图6可知,采用放置0 min 对提取结果影响不明显,故可将异丙醇沉淀DNA这一步骤省去,以缩短试验时间。
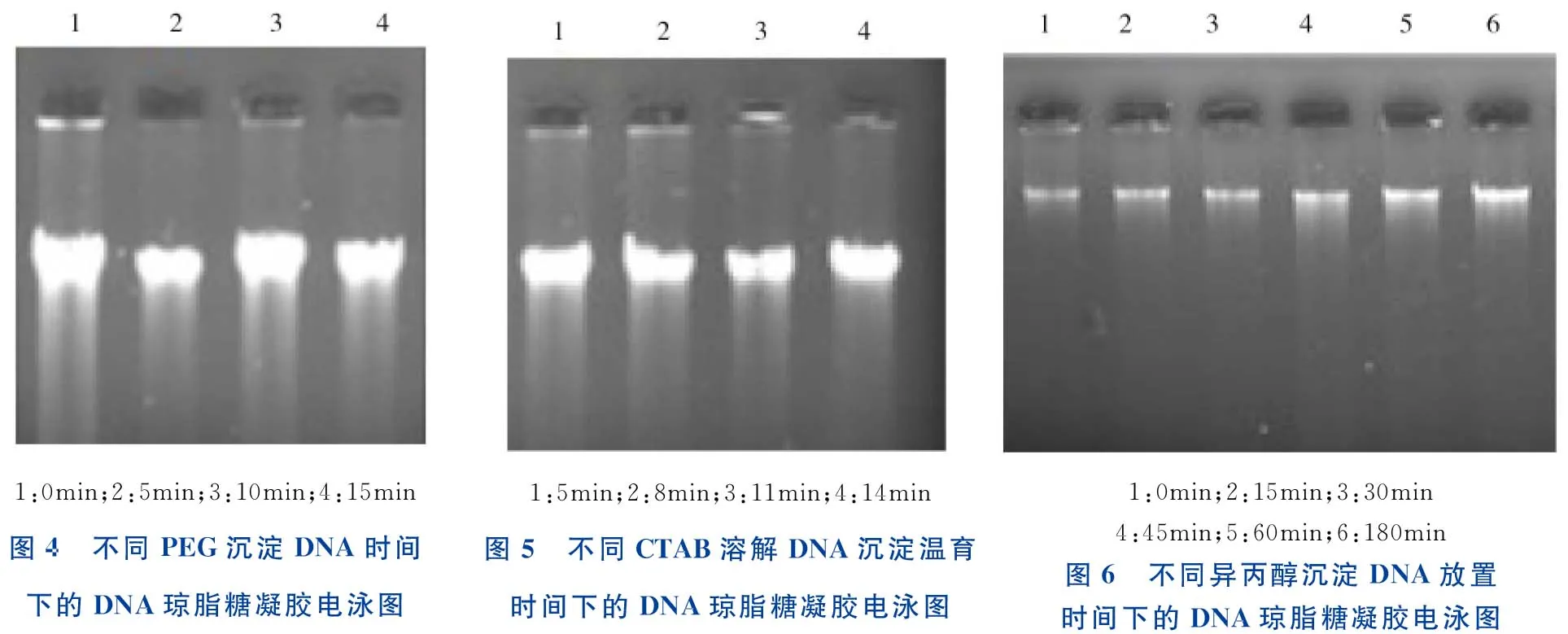
1:0min;2:5min;3:10min;4:15min图4 不同PEG沉淀DNA时间下的DNA琼脂糖凝胶电泳图1:5min;2:8min;3:11min;4:14min图5 不同CTAB溶解DNA沉淀温育时间下的DNA琼脂糖凝胶电泳图1:0min;2:15min;3:30min4:45min;5:60min;6:180min图6 不同异丙醇沉淀DNA放置时间下的DNA琼脂糖凝胶电泳图
2.2 优化提取方法的稳定性
采取2种重复方式进行研究,结果如图7所示。由图7可知,同一样品3个不同时间的3个重复中,条带8的提取量较差;同一样品的5个重复中,条带11的提取效果不佳,其余条带提取效果均较好,由此可知该方法具有一定稳定性。
2.3 优化提取方法的应用性
采取不同土质的土壤样品分别进行实验,结果如图8所示。由图8a可知,5#和6#土样的提取效果较好,其他编号的土样提取效果一般,但都有提取量,可能由于不同土壤样品中微生物含量有差异,从而导致提取量不同。由图8b可知,草地、玉米田、水稻田和生活淤泥的提取效果更好,小麦田、树林等都有提取量。根据不同土壤样品的实验结果可知,优化的实验方案适用于不同地质土壤样品DNA的提取。
2.4 优化提取方法的可操作性
优化提取方法的可操作性研究结果如图9所示。由图9可知,3人提取的3个样品中,均有2个样品提取效果较好,可见该实验方案对于不同的人员均具有一定的可操作性。
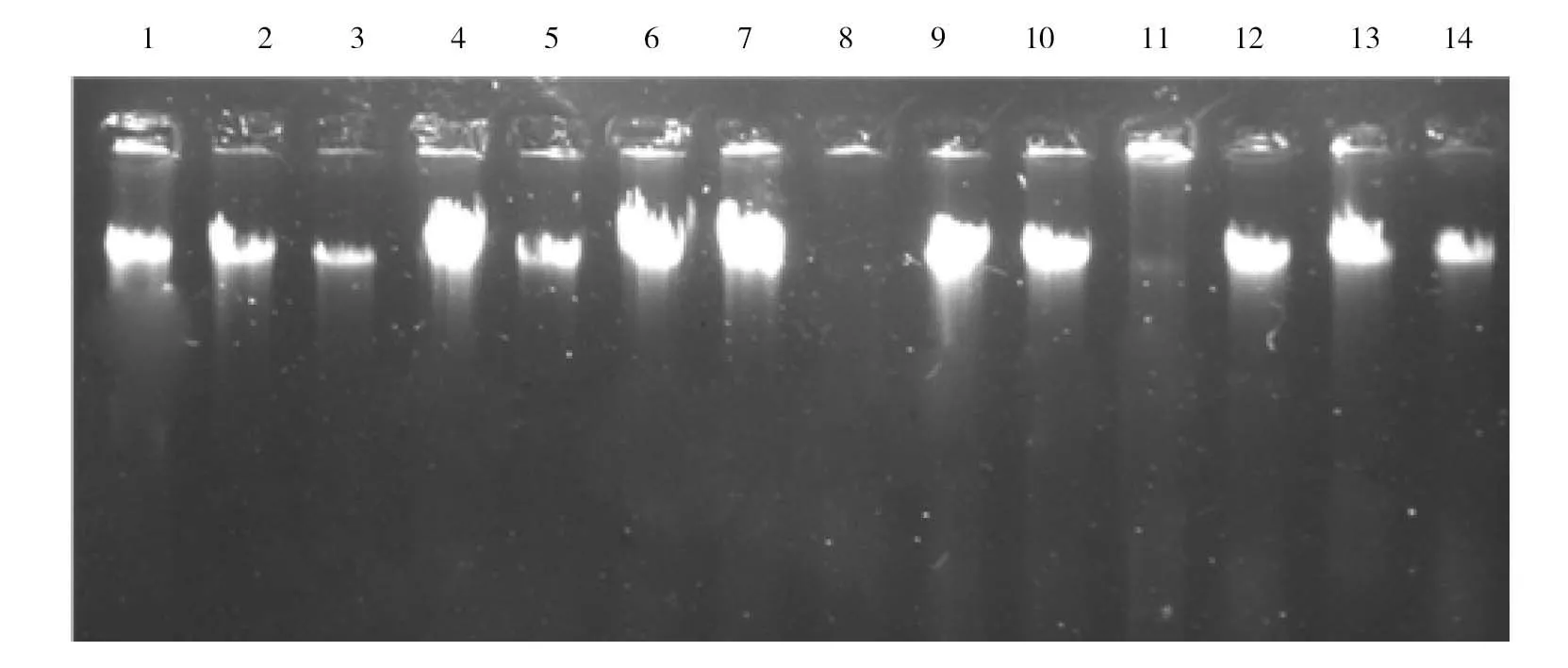
1~3:第1次3的个重复;4~6:第2次的3个重复;7~9:第3次的3个重复;10~14:同一次的5个重复。图7 稳定性DNA琼脂糖凝胶电泳图

a.油井附近土壤样品1~2:1#;3~4:2#;5~6:3#;7~8:4#;9~10:5#;11~12:6#;13~14:7#

b.实验基地土壤样品1~2:麦田;3~4:草地;5~6:大豆田;7~8:玉米田;9~10:水稻田;11~12:树林;13~14:生活淤泥图8 不同土质的土壤样品的DNA琼脂凝胶电泳图

1~3:分别为同一人;4~6:分别为同一人;7~9:分别为同一人。图9 不同人员操作提取的DNA琼脂糖凝胶电泳图
2.5 2种土壤微生物DNA提取方法的比较
通过对陈旭玉等[10]报道的方法进行单因素实验,得出了优化后的实验方法。由表1可知,优化后方法和原方法相比,实验时间缩短了250 min,省去了异硫氰酸胍试剂。
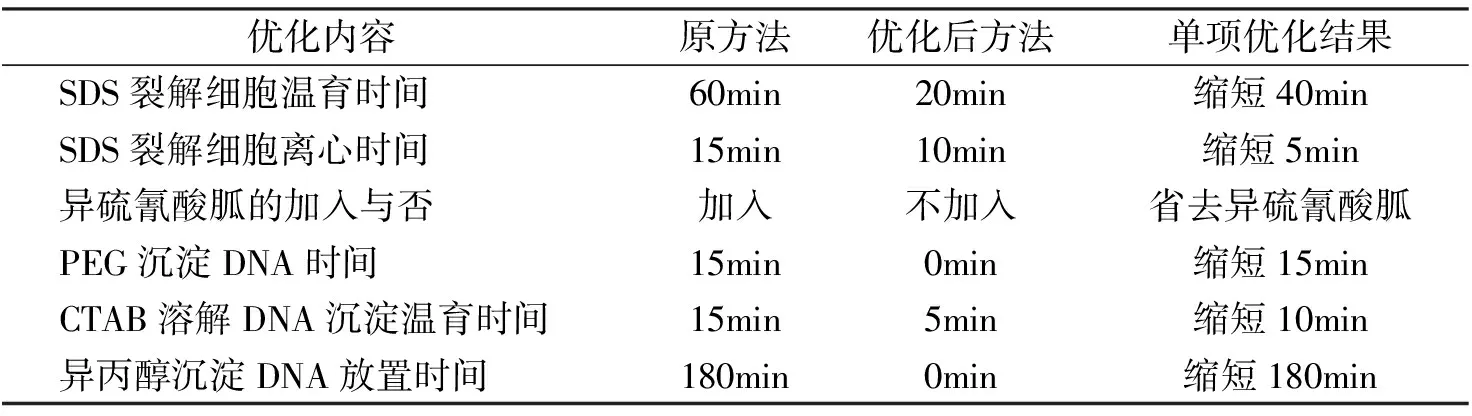
表1 2种方法优化内容与结果比较
3 讨论与结论
本研究优化了陈旭玉等[10]的方法,优化后的土壤微生物DNA提取方法,其稳定性、适用性和可操作性均达到满意的结果。但是判断该方法的优化结果时,单一地从DNA条带的亮暗来评判,只是定性研究并没有定量试验。后续研究中,如果能够从DNA的提取量、纯度等方面进行试验验证,通过定量研究,优化的结果将得到更细致的描述。
优化后的方法与原方法相比,实验时间缩短了250min,省去了异硫氰酸胍试剂。因此,优化后方法是一种经济、快速、简单的土壤微生物DNA提取方法。
[1]沈菊培,张丽梅,郑袁明.土壤宏基因组学技术及其应用[J].应用生态学报,2007,18(1):212~218.
[2]张于光,李迪强,王慧敏,等.用于分子生态学研究的土壤微生物DNA提取方法[J].应用生态学报,2005,16(5):956~960.
[3]张秀敏,徐秀娥,王海祥.从土壤样品中提取和纯化微生物DNA方法研究进展[J].保定师范专科学校学报,2005,18(2):34~37.
[4]Smalla K,Cresswell N,Mendonca-Hagler L C,etal.Rapid DNA extraction protocol from soil for polymerase chain reaction-mediated amplification[J].Journal of Applied Bacteriology,1993,74:78~85.
[5]Zhou J,Bruns M A,Tiedje J M.DNA recovery from soils of diverse composition[J].Applied and Environmental Microbiology,1996,62:316~322.
[6]Kuske C R,Barns S M,Busch J D.Diverse uncultivated bacterial groups from soils of the arid southwestern United States that are present in many geographic regions[J].Applied and Environmental Microbiology,1997,63:3614~3621.
[7]Ogram A,Sayler G.S,Barkay T.The extraction and purification of microbial DNA from sediments [J].J Microbiol Methods,1987,7:57~66.
[8]Faegri A,Torsvik V.L,Goksoyr J.Bacterial and fungal activities in soil:separation of bacteria and fungi by a rapid fractionated centrifugation technique[J].Soil Biol.Biochem,1977,9:105~112.
[9]Torsvik V.L,Goksoyr J.Determination of bacterial DNA in soil[J].Soil Biol Biochem,1978,10:7~12.
[10]陈旭玉,周亚奎,余贤美,等.一种直接用于PCR的土壤微生物DNA提取方法[J].中国农学通报,2008,24(4):33~36.
[编辑] 余文斌

