代谢工程技术方法研究进展
杨祖明,李炳志
(1. 天津大学 系统生物工程教育部重点实验室,天津 300072;2. 天津大学 化工学院 天津化学化工协同创新中心,天津 300072)
代谢工程是发酵菌株改造的重要技术平台,通过引入外源途径或者改造优化代谢网络,从而将微生物改造成天然的“细胞工厂”。随着世界能源和经济结构的调整以及发展面临环境问题的日益突出,微生物发酵作为一种绿色环保的生产技术,在能源、医药、环境等行业被广泛应用,在工业生产领域也取得了丰硕的成果,如:青蒿素[1]、番茄红素[2]和1,4-丁二醇[3]等。
过去,代谢工程主要集中在通过实验分析,鉴定宿主细胞内代谢途径的限速步骤,确定关键节点,然后进行基因敲除和过表达,消除限速步骤。然而,尽管这些努力在实验室中取得了很多成功例子,但是真正能用于大规模工业生产的例子非常有限,主要集中在乙醇、丁醇、甘油、有机酸、氨基酸和抗生素等代谢物。近些年,合成生物学、分子生物学以及计算机科学的发展,为突破这些问题的限制提供了可能,在代谢网络分析、提高菌株性能、途径酶的共定位表达等方面提供了新的技术和策略支撑。
1 代谢网络分析
代谢工程是基于细胞代谢网络的系统研究。在对代谢途径进行合理地改造后,还需要对细胞整体的生理变化、代谢网络的结构和通量进行分析,然后提出合理可行的遗传操作方案。目前,分析这些代谢网络的方法主要有代谢通量分析( metabolic flux analysis,MFA)、代谢控制分析(metabolic control analysis,MCA)和通量平衡分析(flux balance analysis,FBA)(图1)。
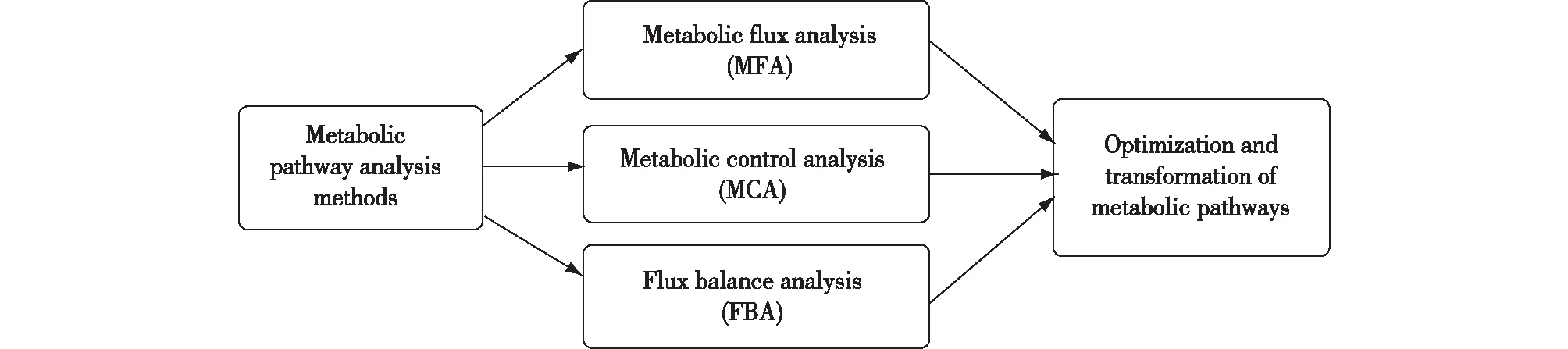
图1 代谢网络分析Fig.1 Analysis of metabolic pathways
1.1 代谢通量分析
在MFA中,使用稳定的13C同位素标记技术,在被标记的碳源环境中培养细胞,当代谢网络中同位素分布达到稳态时,通过气相色谱-质谱技术(GC-MS)或13C-核磁共振(NMR)测量代谢物中同位素的分布,然后基于测量结果,通过细胞内主要反应的化学计量模型和细胞内代谢物的质量守恒来计算细胞内通量,运用底物的摄取速率和产物的分泌速率计算细胞外通量。通量计算的结果最终以代谢通量路径图的形式展现出来,其中包括主要的生化反应以及每个反应的稳态通量。这些信息将帮助代谢工程进行下一步的分析,确定通路中的关键节点,发现宿主细胞内不寻常的通路,并估计产品合成的最大理论产量以及复杂网络途径中的多个辅因子和中间体[4]。根据分析结果,从而可以选定遗传操作的目标,改变细胞的代谢网络分布,使代谢通量更多地流向目标产品。如,Ghosh等[5]使用稳定的13C同位素标记技术测定了酿酒酵母(S.cerevisiae)中脂肪酸(fatty acid)合成路径的碳流量分布,经过通量分析,发现了其中限制脂肪酸生产的两个重要因素:①由甘油-3-磷酸脱氢酶(glycerol-3-phosphate dehydrogenase,由GPD1编码)催化二羟丙酮磷酸(dihydroxyacetone phosphate)合成甘油-3-磷酸(glycerol-3-phosphate)的反应和脂肪酸的合成路径存在竞争关系。②脂肪酸合成的关键前体乙酰辅酶A(acetyl-CoA)会被苹果酸合成酶(malate synthase,由MLS1编码)催化,流向苹果酸(malate)的合成。通过下调MLS1的表达和敲除GPD1等遗传修饰,最终使脂肪酸的产量提高了70%。
1.2 代谢控制分析
由于细胞内代谢网络中存在许多平行反应、代谢循环和双向反应,MFA并不能提供足够准确的测量, 20世纪90年代后期开发出了很多复杂的算法,使得对细胞内通量实现了更为精准的测量[6-7]。这也对代谢控制分析(MCA)的发展起到了积极的作用。MCA作为传统控制理论的逻辑延伸,依赖实验来准确测定代谢途径的通量及其对系统扰动的响应变化,可以计算通路中每种酶的通量控制系数(flux control coefficient,FCC)表征酶对细胞内代谢物通量的控制程度;浓度控制系数(concentration control coefficient,CCC)表征酶对给定代谢物浓度的控制程度;弹性系数(elasticity coefficient,EC)表征酶响应于扰动(例如底物浓度、抑制剂浓度)的能力。 FCC和CCC是整个网络的特性,而EC是特定酶的函数。综合而言,这些信息可用于梳理代谢网络的控制结构,阐明酶活性的相对变化如何影响通路流量,可以帮助指导分子生物学技术的合理应用,将更多的通量转移到目标产品。例如,在线性代谢途径中,如果一种酶的FCC值较高,那么它就可以被认定是速率限制的主要因素。因此,将其过表达可以使该途径的通量增加。Cintolesi等[8]将MCA方法用于对大肠杆菌(E.coli)的甘油发酵进行代谢定量分析,利用计算的通量控制系数(FCC)预测甘油发酵过程,结果发现糖酵解通量是由甘油脱氢酶(glycerol dehydrogenase,由gldA编码)和二羟基丙酮激酶(dihydroxyacetone kinase,由dhaKLM编码)控制。通过遗传操作,使gldA和dhaKLM过表达,结果与MCA分析一致,甘油利用率和乙醇产量显著增加[9]。
1.3 通量平衡分析
微生物全基因组序列的测定以及基因功能注释工具的开发极大地促进了基因组水平代谢网络模型的构建,提高了代谢网络结构的分析能力。通量平衡分析(FBA)是一种通过构建基因组水平代谢网络模型(genome-scale metabolic model,GEM),从系统水平上认识微生物的复杂代谢网络,模拟预测环境扰动或遗传改组后细胞的代谢响应,筛选出基因敲除和过表达的最佳组合,以增加生物体生产目标产品的能力[10]。随着新组学数据的开发和基因组注释方法的改进,还可以在代谢网络模型中引入相关的反应监管调控机制[11]对模型进行不断改进。近些年,研究人员针对大肠杆菌开发了一种非常全面的GEM模型,包括2 077个反应和1 039个代谢物[12]。这些模型在实际中具有相当多的用途,例如,研究人员在该模型的基础上,运用通量平衡分析的方法确定了一株高产番茄红素菌株的基因敲除目标,最终使其产量相对于亲本菌株增加了37%[13]。与此同时,研究人员也开发了很多计算程序,被用来自动计算基因优化策略[14]。例如,Optknock程序[14-15]通过解决双层优化问题,内部优化生物量和外部优化目标产品产量,利用二元理论搜索多个基因敲除候选者;OptGene可用于优化非线性目标函数[16],其算法可以迅速鉴定出菌株的基因缺失组合;RobustKnock[17]用来识别与主要代谢路径存在竞争关系的途径。
2 逆向代谢工程策略
大多数代谢工程最初的努力主要集中于以上几种经典的代谢工程分析方法,通过通量测定和代谢途径分析,确定动力学瓶颈,然后理性分析设计,合理地操纵新陈代谢将通量转向目标产品。但是通常菌株的表现型和基因的联系非常复杂,通过理性分析设计的方法很难获得工业生产的菌株表型。这种情况下,可以使用逆向代谢工程策略,与理性代谢工程不同,它不一定依赖于代谢途径分析的先验知识。而是运用各种突变技术对菌株的基因组进行随机突变,再高通量筛选突变体,以获得优良的表现型,然后利用高通量基因组测序技术对野生型菌株和突变菌株的基因组序列进行测序比对,获得突变信息,再通过转录组学、蛋白质组学和代谢组学等相关组学技术,鉴定其中发生的有利突变,最后,将有利突变引入野生菌株中,剔除不利突变,经过多次循环操作,最终获得只含有利突变的菌株[18](图2)。逆向代谢工程成功的关键在于突变文库的遗传多样性和突变的质量水平,下面回顾最近的一些组合技术方法在逆向代谢工程中的发展应用[19]。
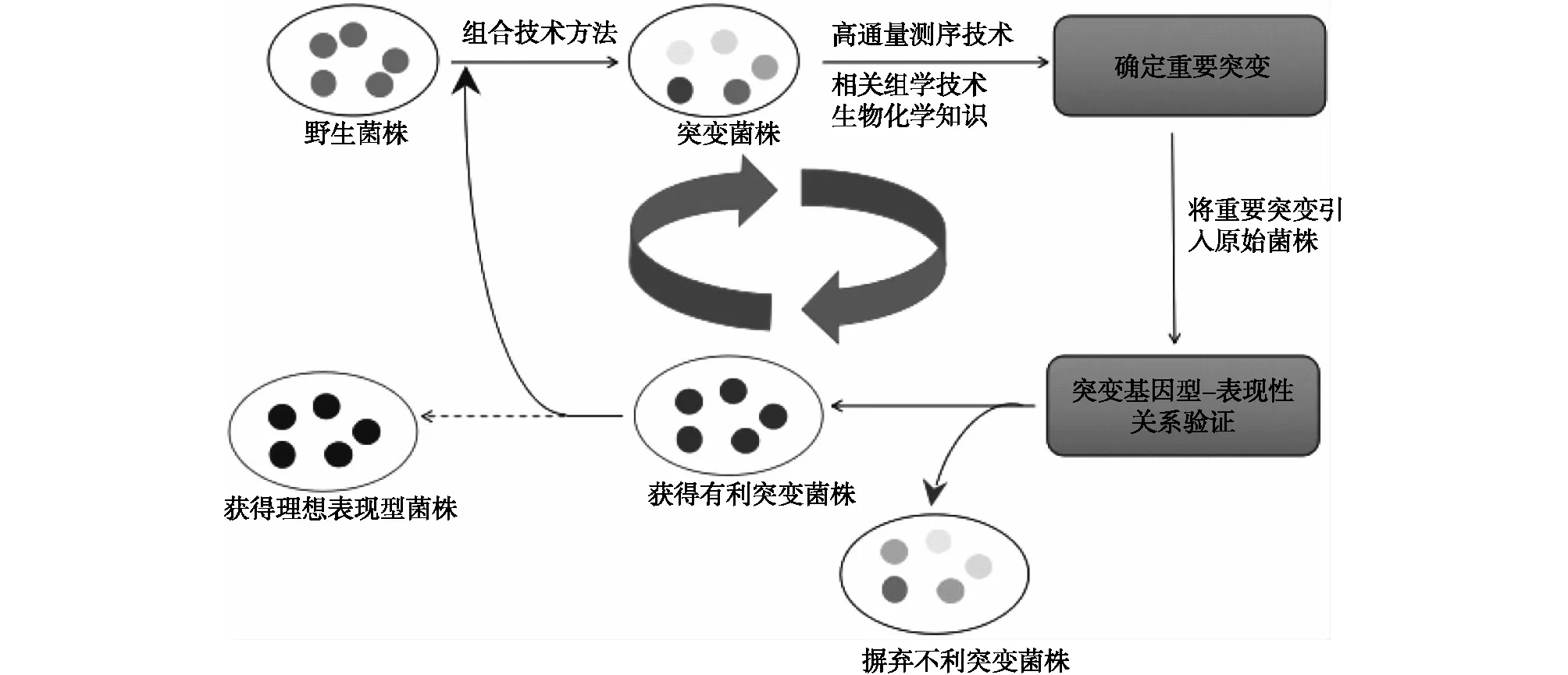
图2 逆向代谢工程策略Fig.2 Inverse metabolic engineering
2.1 定向驯化
在特定的生长条件下,使菌株自发地发生突变以提高对环境的适应能力是经典的定向驯化方法。这种方法在工业生产和科学研究中被广泛运用,产生了许多成功的例子,包括增加菌株对乙醇和异丁醇的耐受性[20-21]、提高酿酒酵母对木糖和半乳糖的利用[22-23]以及利用大肠杆菌生产D-乳酸(D-lactate)[24]等。
2.2 转座子诱变
这种类型的诱变可以使转座元件在整个基因组中随机插入,同时使插入的基因序列发生功能性破坏。转座子诱变技术在天蓝色链霉菌(Streptomycescoelicolor)中十一烷基多糖苷的生产[25]、枯草芽孢杆菌(Bacillussubtilis)中核黄素的生产[26]以及酿酒酵母中类异戊二烯[27]的生产中都起到了积极的促进作用。
2.3 细胞全局转录调控机制工程(global transcription machinery engineering,gTME)
细胞代谢网络具有高度的复杂性,菌株的某些特定表型往往由多个基因共同控制,过去主要通过敲除和过表达对其中一个或几个基因进行修饰,但是往往得不到理想的适用于工业生产菌株。随着对转录过程认识的深入,人们开始直接操控转录组来控制整个细胞内各种基因的转录。RNA聚合酶能够对转录行使全局调控功能,因此突变RNA聚合酶可以使多种基因控制的菌株表型得到优化。在RNA聚合酶参与转录的过程中,σ等各种转录因子负责转录的识别、激活和控制[28],运用全局转录调控技术对这些转录因子进行适当地修饰,能够使整个基因组的转录发生全局性改变。Alper 等[29]利用全局转录调控技术,将大肠杆菌中编码σ70因子的rpoD基因用易错PCR进行突变,获得了一个rpoD基因突变文库,并将突变后的rpoD基因引入野生型大肠杆菌,使得大肠杆菌对乙醇的耐受能力大幅度提高。
2.4 锌指结构突变
锌指结构是锌指蛋白中高度特异的DNA 结合域,能识别3个碱基序列,在细胞内的多种调节蛋白里都发现了锌指结构[30-31]。 Park等[32]对多个锌指结构进行突变,构建了人工的锌指文库,并引入酵母,得到了对渗透胁迫作用和真菌药物咪康唑(ketoconazole) 抗性提高的酵母突变体。此外,由于锌指蛋白能够特异性结合DNA序列,研究人员构建生成了结合转录激活或抑制功能区的人工转录因子文库,并在提高动物细胞和酿酒酵母生产重组蛋白效率的研究中取得了成功的应用[33]。
2.5 核糖体工程
将微生物细胞暴露在抑制核糖体功能的致死浓度抗生素里,例如氯霉素、利福平、链霉素或卡那霉素,并且筛选对这些抗生素在不同水平上有抗性的突变体。通常对这些抗生素的抗性来自核糖体组成部分的突变,比如核糖体蛋白和RNA。这些变化可导致异常的蛋白质合成和提高特殊蛋白的生产。Tanaka及其同事[34]的研究表明,在编码核糖体蛋白S12的rpsL基因上引起突变,导致丁酸梭菌(Clostridiumsaccharoperbutylacetonicum)对链霉素(streptomycin)的抗性提高,此外还突变了与丁醇合成相关的基因,最终使丁醇产量增加了1.6倍,达到16.5 g/L,结果分析显示,在抗性增加的突变体中,参与丁醇合成的丁醛脱氢酶(AdhE)和丁醇脱氢酶(BdhAB)的活性都显著增加。
3 代谢途径酶的共定位表达
近年来,随着分子生物学的发展,代谢工程不再过度集中于通过过表达或敲除特定基因以调节代谢通量的走向,一些更加精细的调节方法正在开发中。其中,优化多步代谢途径中关键酶的空间分布的方法被广泛应用,这些方法包括:构建融合蛋白、蛋白质支架组装、亚细胞结构定位(图3)。通过优化酶的空间分布,可以提高蛋白和底物之间的相互作用,减少中间体流入竞争路径,减少细胞内噪声干扰并提供适宜的催化环境。
3.1 构建融合蛋白
构建融合蛋白是目前最简单并且被广泛运用的共定位方法,通过将两个或多个基因与接头序列融合产生一个具有多功能的融合蛋白。接头区使各个蛋白在空间上相互靠近,同时又能使结构域分离,减少了蛋白之间的相互折叠干扰,使各个蛋白保持其天然活性[35-36]。Zhang等[37]将合成路径中的4-香豆酰基-乙酰连接酶(4-coumaroyl-CoA ligase)和二苯乙烯合酶(stilbene synthase)用一个三氨基酸序列连接起来,使白藜芦醇的产量比原来提高了15倍。随后的动力学和结构研究证实,产量的提高是由于酶活性位点的接近而不是来自任何酶的催化活性的提高[38],这也说明了融合蛋白克服了中间体扩散的限制,进而提高了产物浓度和产量。
3.2 蛋白质支架组装
将路径酶连接到支架的方法在代谢工程领域也产生了非常好的效果,虽然这个方法比较复杂,但是相对于融合蛋白更具优点,如可以根据需要调节支架结构和改变结合酶数量。支架的空间架构是路径优化的关键,因为支架的构型和比例对于平衡反应通量以提高目标产品产率是至关重要的。蛋白质支架在甲羟戊酸途径中取得了非常好的效果,在最佳的支架结构下,甲羟戊酸(mevalonate)途径通量提高了77倍[39]。麻省理工学院的Prather实验室也运用同样的策略共定位葡糖二酸途径的3个酶,尽管从相当高的质量浓度(约0.5 g/L)开始,但是最后通过蛋白质支架组装技术将葡糖二酸的浓度提高了5倍[39-40]。与融合蛋白一样,蛋白质支架不仅能催化连续的途径步骤,而且还能够增加蛋白质之间的相互作用的速率[41]。另外,支架蛋白也可以被工程化以产生新的信号传导途径以产生新的表型,如工程化MAP激酶(MAP kinase)途径的研究[42]。
3.3 亚细胞结构定位
将代谢途径酶共定位到亚细胞结构是最为复杂的空间定位方法。这些亚细胞结构的空间大于支架蛋白,能够容纳大量蛋白质,并且能够更好地限制物质跨越边界进行传递。因此,这些亚细胞结构可以整合中间体以防止对细胞的毒性作用,并且能够限制竞争路径以及增加底物、酶和辅因子的局部浓度以提高产率。
在真核宿主中,亚细胞结构具有不同于细胞质的pH、氧化还原电位、辅因子和代谢物组成,这些条件可能对酶促反应热力学更有利,帮助宿主实现更高的催化效率。Avalos等[43]将酵母中生产异丁醇的下游路径由细胞质全部定位到线粒体,由于中间体利用度和酶浓度增加,并且限制了竞争路径,使得异丁醇的产量提高了260%。目前,涉及亚细胞结构定位主要使用N末端靶向序列进行蛋白质靶向的方法,通常被靶向的亚细胞结构有过氧化物酶体、液泡、内质网和线粒体等。表1列举了近年来在亚细胞结构定位方面取得的一些成果。

图3 酶的共定位表达技术Fig.3 Enzyme co-localization technology

表1 亚细胞结构定位研究汇总
4 代谢工程与合成生物学
合成生物学,即生物学的工程化,通过采用工程化的设计思路,借助计算机科学、化学和生物学等学科的相关理论,构建标准的基因元件和模块,然后根据设计的需要,采用“自下而上”的策略,组装合成新的基因电路、生物模块、细胞乃至生命体。随着合成生物学的核心技术人工合成基因组的进步以及人工模块库的不断丰富,合成生物学为代谢工程提供了许多新的方法策略(图4),相对于传统技术方法,合成生物学的优势主要体现在:①模块的标准化和计算方法的发展进步,不仅节省了时间、人力,还提高了代谢路径组装设计的可预测性和可靠性。②合成生物学拓展了人工改造的尺度,可以利用不同物种来源的基因元件和生物模块设计及组合来合成非天然分子。③优化改造宿主,改变内部环境,提高路径和宿主的适配性。④基因电路的发展为响应发酵环境变化,实现更精准的调控提供了可能。
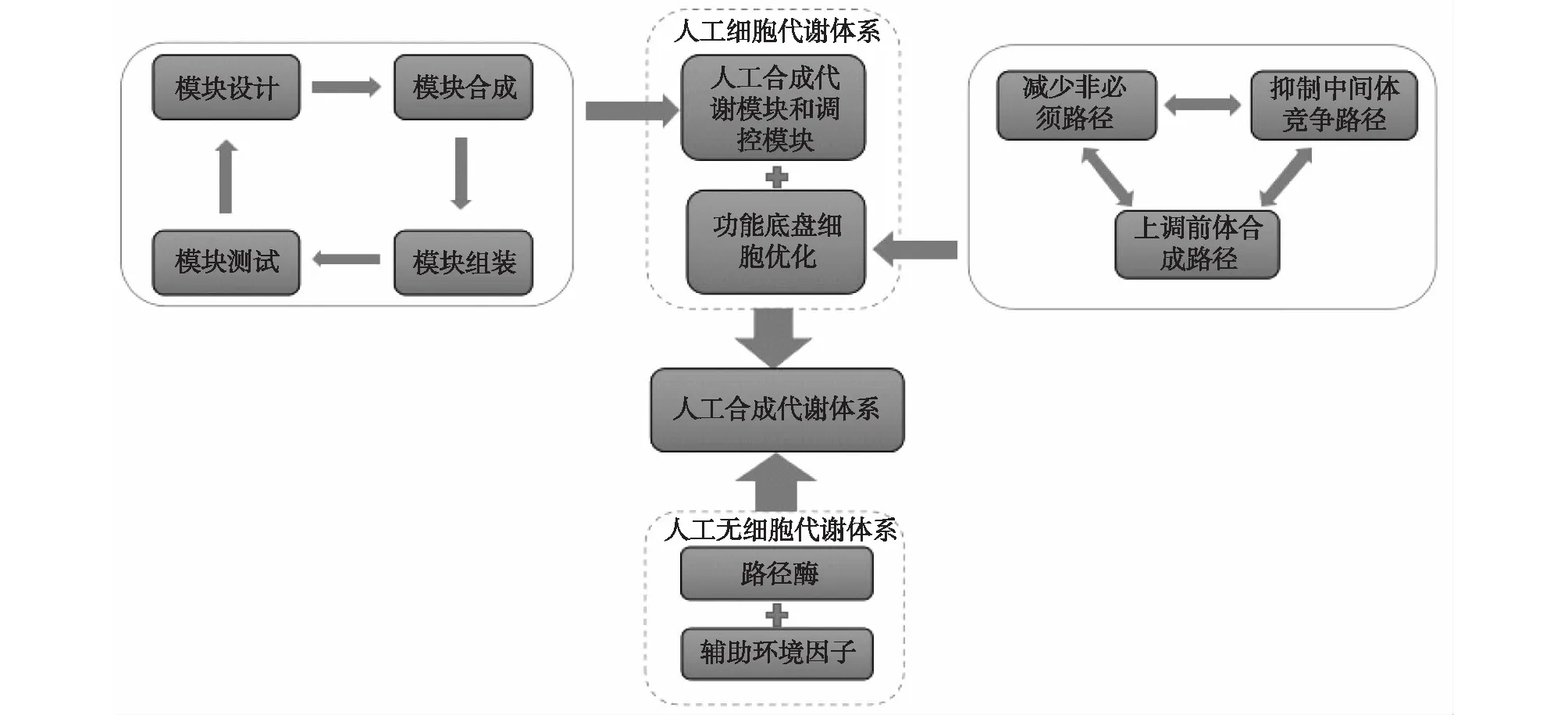
图4 代谢工程与合成生物学Fig.4 Metabolic engineering and synthetic biology
4.1 宿主细胞的改造和开发
宿主细胞是代谢途径正常发挥生物学功能的基础,不仅能够保留复制生物装置的遗传信息,而且能够为生物装置的运行提供能量、还原力、各种因子和基础物质。过去代谢工程的研究主要集中在实验室标准模式生物——酿酒酵母或者大肠杆菌中,主要是由于它们的生理学、遗传学背景已经了解得非常透彻,全基因组已经完成测序而且开发了很多成熟的分子生物学工具。然而,将许多非常有应用前景的异源生物代谢途径组装到酿酒酵母或者大肠杆菌中,运行的结果并不理想,而且由于未来工业化生产条件的限制,如高温、苛刻的酸性或碱性等条件,需要对目前的宿主底盘细胞进行改造,使其和代谢模块适配。目前,利用合成生物学的方法改造宿主细胞主要有两种策略:人工合成最小基因组(minimal genome)和人工全基因组合成[54]。最小基因组既能够为宿主细胞的生长和繁殖提供必要的分子机制和物质基础,又降低了细胞内不必要的代谢途径、反馈调控路径的影响,简化了宿主细胞的遗传背景,提高代谢效率。Pósfai等[55]通过基因序列比对,删除了大肠杆菌基因组中引起 DNA结构不稳定的 DNA 片段和非必需功能的基因,将E.coliMG1655菌株的基因组减少了15.27%,最后得到了在丰富培养基上的生长速度没有明显降低的菌株,这一研究成果充分说明了在宿主细胞中构建最小基因组的可行性。近些年,由于DNA合成技术的发展和物种基因组信息不断丰富,大大促进了从全基因组水平上对宿主细胞遗传改造的进程。最近中美英等国科学家联合参与的人工合成酿酒酵母基因组Sc2.0计划顺利完成,标志着人类实现了从读取生命信息到编码生命信息的里程碑式的跨越,也意味着人类向化学物质合成生命迈出了一大步。对酿酒酵母基因组的合成并不只是简单地复制,在保证菌株在各种环境条件下正常生长的同时,加入了大量的特异性重组位点和特异性识别标签,使基因组具有更好的灵活性和可操作性[56],为后续开展遗传操作改良菌株提供了基础。
4.2 非天然化学品的生物合成
有别于传统代谢工程,利用合成生物学技术合成非天然化学品有着很多独特的优势,主要体现在以下几个方面:①合成生物学技术可以将不同物种来源的基因元件和生物模块进行设计组装,构建非天然分子合成路径。②利用合成生物学的基因组装技术可以快速将大片段 DNA 组装成完整的代谢通路。③可以根据天然酶反应和化学逆合成分析方法从头设计构建自然界中不存在的代谢途径。Hanai 等[57]通过跨种属组合表达来自丙酮丁醇梭菌(Clostridiumacetobutylicum)乙酰乙酸脱羧酶基因adc和乙酰辅酶A乙酰基转移酶基因thl、来自拜氏羧菌(Clostridiumbeijerinckii)的乙醇脱氢酶基因adh和来自大肠杆菌的乙酰乙酰辅酶A转移酶基因atoAD,在大肠杆菌内首次构建了合成异丙醇的代谢途径,通过实验室发酵培养30.5 h,异丙醇产量达到了81.6 mmol/L。
如果目标产物的代谢途径未知,就需要采用从头合成代谢途径(de novo biosynthetic pathways)的方法。Yim等[58]使用这种方法在大肠杆菌体内构建了1,4-丁二醇的代谢路径。首先他们通过检查功能基团的转化,用计算机搜索了超过10 000条合成路径,然后建立诸如热力学可行性、最大理论产量、路径长度等标准排查筛选,最终建立了由5个异源酶催化合成目标产物的生物合成路径,然后经过后期改造优化,最终1,4-丁二醇的产量达到了18 g/L。
4.3 代谢工程的精准调控
迄今为止,在代谢工程的大多数应用中,异源途径的表达通常使用静态调节策略。在计算和实验工具的帮助下,通过使用基因敲除、启动子替换和异源基因的引入在细胞中改变静态通量分布方面非常成功。然而,在实际发酵过程中,随着细胞周围生长环境的变化,如底物浓度、溶氧量、细胞密度等,细胞的生长速率会明显降低,导致生产目标化合物的能力受限于细胞生物量的减少。因此,为了实现目标化合物的高产,需要在宿主代谢过程中引入动态调节机制,根据细胞生长环境的变化,重新平衡代谢通量的分布。Zhang等[59]设计了一种动态响应调控系统(dynamic sensor-regulator system,DSRS)来调控大肠杆菌中脂肪酸乙酯(fatty acid ethyl ester,FAEE)的生产。在该代谢通路中,平衡脂肪酸乙酯合成路径中关键中间体脂肪酰基辅酶A(fatty acyl-CoA)和乙醇对于提高脂肪酸乙酯的生产是至关重要的,因为脂肪酰基辅酶A的过高积累不仅会和细胞内生物膜的合成路径争夺脂肪酸(fatty acid),还需要消耗过多的乙酰辅酶A(acetyl-CoA),同时乙醇水平过高还会对细胞的生长和代谢产生不利的影响,见图5。由图5可知:该系统的调控作用主要是基于一种转录调控因子FadR,它能够感应大肠杆菌中脂肪酸乙酯代谢通路中脂肪酸的水平,当细胞内脂肪酸浓度较低时,FadR会对P1、P2和P3启动子起抑制作用,进而降低细胞内脂肪酰基辅酶A和乙醇的浓度,抑制脂肪酸乙酯的合成。当细胞内脂肪酸浓度较高时,首先脂肪酸会在酰基辅酶A合成酶(acyl-CoA synthase,ACS)的作用下转化成脂肪酰基辅酶A,然后脂肪酰基辅酶A会和FadR结合形成FadR-FA复合体,解除FadR对P1 、P2和P3启动子的抑制,提高了细胞内脂肪酰基辅酶A和乙醇的浓度,促进了脂肪酸乙酯的合成。最终在DSRS系统的调控下,脂肪酸乙酯的产量达到了1.5 g/L,产率提高了3倍。
生物传感器用于动态控制的一些例子已经成功实现。然而,到目前为止,其中许多需要耗费大量时间筛选适当的生物传感器和通过启动子工程平衡基因表达。合成和系统生物学新工具的快速发展将有助于扩大动态代谢工程领域,并简化实施所需的过程。通过组合双稳态开关[60]、生物振荡器[61]、生物放大器[62]以及能够执行逻辑功能的AND、OR和NOR门等基因元件,可以构建传导复杂生物信号的基因线路[63],来响应细胞发酵环境的变化,进而更精准地调控细胞内通量的流向。
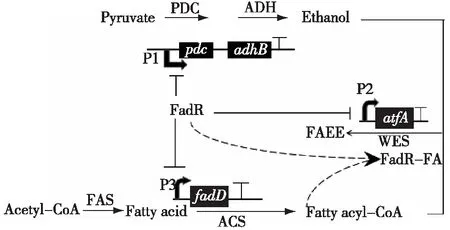
PDC—pyruvate decarboxylase(edcoded by pdc);ADH—alcohol dehydrogenase (encoded by adhB );ACS—acyl-CoA synthase(edcoded by fadD );WES—wax-ester synthase(encoded by atfA);FAS—fatty acid synthase 图5 脂肪酸乙酯合成路径动态响应调控系统Fig.5 Dynamic sensor-regulator system of fatty acid ethyl ester synthesis path
4.4 模块化工程
优化和平衡多基因调控的代谢途径通常是低效的,往往一个限制性步骤刚解决,新的代谢瓶颈又会出现,需要经过多轮的设计、优化,非常耗时。为了解决上述问题,模块化代谢路径已经发展成为一种有希望的策略[64],可以将代谢途径划分成若干个模块,然后利用调控元件组装不同活性模块,最后通过调整模块间的相对活性,得到代谢模块相对活性平衡的最佳重组菌株[65]。根据模块化工程采用策略的差异,可将其分为三种不同的类型:①基于代谢网络中生化反应的模块化。根据生化反应产物的浓度、毒性大小等生化特性划分模块,通过平衡不同模块的相对强度,减少了中间代谢物的积累对菌体产生的毒害作用和其他竞争路径的干扰[66]。②基于代谢网络分支点的模块化。以代谢网络分支点为基础,将分支点处的中心代谢路径和其他分支路径模块化,通过微调中心代谢路径和分支路径的通量比,实现了菌株生长和目标产品合成的协同[67]。③基于酶促反应效率的模块化[68]。根据酶促反应效率的不同,将代谢网络中的酶促反应划分为不同的模块,通过优化不同模块的催化效率,提高中间体的催化效率和转运效率。 Liu等[69]利用模块化工程技术优化了枯草芽孢杆菌中的N-乙酰氨基葡萄糖(GlcNAc)的路径,将枯草芽孢杆菌胞内代谢途径分为 3 个模块,最后将活性强度不同的 3 个模块进行拼装组合,筛选出活性相对平衡的最佳重组菌,成功地将N-乙酰氨基葡萄糖的产量提高了4.3 倍,达到 2.0 g/g。
4.5 无细胞代谢工程
虽然通过代谢工程途径异源表达化学物质成功的案例正在不断增长,但是真正可以用来实现工业生产的化学品却非常有限。这主要是由于细胞本身所固有的局限性,主要体现在:①细胞代谢网络错综复杂,使得合理设计难以预测,平衡网络中的代谢通量需要大量的分析和实验,但是产量提升效果很有限。②目标产物或中间体的积累对细胞产生毒性,而且存在很多竞争途径和抑制调控网络,也大大限制了代谢产量。③细胞内物质在不同细胞器之间的跨膜运输障碍降低了目标产品的合成效率。④工业化大规模生产对发酵条件的控制提出了严苛的要求,往往很难达到和实验室小规模生物合成相同的条件,而且代谢产生了很多副产物,不利于下游分离。近些年,无细胞代谢工程(cell-free metabolic engineering,CFME)的发展正在克服这些限制,提供替代的解决方案,扩大了传统代谢工程的应用范围。通过体外组合由纯化酶或细胞粗裂解物制备的催化蛋白质来生产目标产物。相对于体内系统,无细胞代谢工程能够通过分子“设计—建造—测试”平台而非重新设计构建生命装置元件,实现合成路径的快速调试和优化,其前所未有的控制和设计自由度推动了这一领域的快速发展。Korman等[70]设计了一个由27个酶组成的无细胞合成系统,通过改变萜烯合酶,能够将葡萄糖转化为不同的单萜,一次添加葡萄糖能使系统连续稳定工作至少5 d,通过优化反应条件,使葡萄的转化率大于95%,单萜产量超过了15 g/L,比传统微生物发酵的产量高出了一个数量级,凸显了CFME系统合成天然产品的巨大潜力。
5 结语与展望
代谢工程发展20多年来,国内外针对代谢工程的研究主要集中在代谢网络的分析评估和优化控制两个领域。在分析评估方面,研究人员基于不同的策略并运用计算机辅助技术开发了很多分析评估方法,为代谢网络的优化控制提供了基础。代谢网络的优化控制是提高代谢效率和产量的关键。该技术的发展主要集中在以下三个方面:
1)逆向代谢工程。随着高通量基因组测序技术的快速发展,能够低成本地对野生型菌株和突变菌株的基因组序列进行快速比对,获得突变信息,再通过转录组学、蛋白质组学、代谢组学等相关组学技术鉴定其中的有利突变并将其引入菌株。但是运用组合技术方法突变会产生巨大的突变文库,给筛选带来了压力,需要针对特定菌株和产品开发出适合的高通量筛选技术。
2)酶工程策略。过去酶工程策略主要集中在改变酶的活性和特异性等方面,最近三维超高分辨率显微镜技术的进步推动了途径酶共定位表达技术的快速发展,通过优化酶在细胞内的空间分布,减少了中间体流失,提高了底物和酶浓度,使代谢网络运行效率大幅提高。
3)合成生物学与代谢工程。合成生物学技术为代谢工程的模块化、精准化提供了有力支撑,能够通过不同模块间的适配和生物传感器的响应,实现代谢网络中的通量平衡和实时调控。
另外,合成生物学技术在改造底盘细胞,减少细胞噪声干扰,提高代谢路径和底盘细胞的适配性方面也发挥了积极的作用。但是由于细胞自身代谢网络的高度复杂性,未来研究人员不仅需要在基础领域进一步加深对生命的认识,还可以通过无细胞合成技术来打破细胞本身所固有的局限性,实现更多大宗化学品的生物合成。随着全球对可持续发展的呼声越来越高,未来,在系统生物学、生物信息学的知识背景下,结合合成生物学、计算机辅助设计及基因编辑技术提供的策略和技术支持,代谢工程的应用范围将不断地扩大。
[1] PADDON C J,KEASLING J D.Semi-synthetic artemisinin:a model for the use of synthetic biology in pharmaceutical development[J].Nat Rev Microbiol,2014,12(5):355-367.
[2] CHEN Y,XIAO W,WANG Y,et al.Lycopene overproduction inSaccharomycescerevisiaethrough combining pathway engineering with host engineering[J].Microb Cell Fact,2016,15(1):113.
[3] BARTON N R,BURGARD A P,BURK M J,et al.An integrated biotechnology platform for developing sustainable chemical processes[J].J Ind Microbiol Biotechnol,2015,42(3):349-360.
[4] WOOLSTON B M,EDGAR S,STEPHANOPOULOS G.Metabolic engineering:past and future[J].Annu Rev Chem Biomol Eng,2013,4:259-288.
[5] GHOSH A,ANDO D,GIN J,et al.13C metabolic flux analysis for systematic metabolic engineering ofS.cerevisiaefor overproduction of fatty acids[J].Front Bioeng Biotechnol,2016,4:76.
[6] SCHMIDT K,NIELSEN J,VILLADSEN J.Quantitative analysis of metabolic fluxes inEscherichiacoli,using two-dimensional NMR spectroscopy and complete isotopomer models[J].J Biotechnol,1999,71(1):175-189.
[7] ANTONIEWICZ M R,KELLEHER J K,STEPHANOPOULOS G.Elementary metabolite units (EMU):a novel framework for modeling isotopic distributions[J].Metab Eng,2007,9(1):68-86.
[8] CINTOLESI A,CLOMBURG J M,RIGOU V,et al.Quantitative analysis of the fermentative metabolism of glycerol inEscherichiacoli[J].Biotechnol Bioeng,2012,109(1):187-198.
[10] PRICE N D,PAPIN J A,SCHILLING C H,et al.Genome-scale microbial in silico models:the constraints-based approach[J].Trends Biotechnol,2003,21(4):162-169.
[11] LEE S Y,PARK J M,KIM T Y.4 Application of metabolic flux analysis in metabolic engineering[J].Methods Enzymol,2011,498:67.
[12] FEIST A M,HENRY C S,REED J L,et al.A genome-scale metabolic reconstruction forEscherichiacoliK-12 MG1655 that accounts for 1260 ORFs and thermodynamic information[J].Mol Syst Biol,2007,3(1):121.
[13] ALPER H,JIN Y S,MOXLEY J,et al.Identifying gene targets for the metabolic engineering of lycopene biosynthesis inEscherichiacoli[J].Metab Eng,2005,7(3):155-164.
[14] SIMEONIDIS E,PRICE N D.Genome-scale modeling for metabolic engineering[J].J Ind Microbiol Biotechnol,2015,42(3):327-338.
[15] BURGARD A P,PHARKYA P,MARANAS C D.Optknock:a bilevel programming framework for identifying gene knockout strategies for microbial strain optimization[J].Biotechnol Bioeng,2003,84(6):647-657.
[16] PATIL K R,ROCHA I,F RSTER J,et al.Evolutionary programming as a platform for in silico metabolic engineering[J].BMC Bioinf,2005,6(1):308.
[17] TEPPER N,SHLOMI T.Predicting metabolic engineering knockout strategies for chemical production:accounting for competing pathways[J].Bioinformatics,2010,26(4):536-543.
[18] 李桂莹,张新波,王智文,等.逆向代谢工程的最新研究进展[J].生物工程学报,2014,30(8):1151-1163.
[19] SKRETAS G,KOLISIS F N.Combinatorial approaches for inverse metabolic engineering applications[J].Comput Struct Biotechnol J,2012,3:e201210021.
[20] YOMANO L,YORK S,INGRAM L.Isolation and characterization of ethanol-tolerant mutants ofEscherichiacoliKO11 for fuel ethanol production[J].J Ind Microbiol Biotechnol,1998,20(2):132-138.
[21] ATSUMI S,WU T Y,MACHADO I M,et al.Evolution,genomic analysis,and reconstruction of isobutanol tolerance inEscherichiacoli[J].Mol Syst Biol,2010,6(1):449.
[22] HONG K K,VONGSANGNAK W,VEMURI G N,et al.Unravelling evolutionary strategies of yeast for improving galactose utilization through integrated systems level analysis[J].Proc Natl Acad Sci USA,2011,108(29):12179-12184.
[23] SCALCINATI G,OTERO J M,VAN VLEET J R,et al.Evolutionary engineering ofSaccharomycescerevisiaefor efficient aerobic xylose consumption[J].FEMS Yeast Res,2012,12(5):582-597.
[24] UTRILLA J,LICONA-CASSANI C,MARCELLIN E,et al.Engineering and adaptive evolution ofEscherichiacolifor D-lactate fermentation reveals GatC as a xylose transporter[J].Metab Eng,2012,14(5):469-476.
[25] XU Z,WANG Y,CHATER K F,et al.Large-scale transposition mutagenesis ofStreptomycescoelicoloridentifies hundreds of genes influencing antibiotic biosynthesis[J].Appl Environ Microbiol,2017,83(6):e02889-16.
[28] 乔志新,于群.全局转录调控及其在代谢工程中的应用[J].生物技术通讯,2009,20(5):689-691.
[29] ALPER H,STEPHANOPOULOS G.Global transcription machinery engineering:a new approach for improving cellular phenotype[J].Metab Eng,2007,9(3):258-267.
[30] BROWN R S.Zinc finger proteins:getting a grip on RNA[J].Curr Opin Struct Biol,2005,15(1):94-98.
[31] 赵心清,姜如娇,白凤武.启动子和细胞全局转录机制的定向进化在微生物代谢工程中的应用[J].生物工程学报,2009,25(9):1312-1315.
[32] PARK K S,DONG-KI L,LEE H,et al.Phenotypic alteration of eukaryotic cells using randomized libraries of artificial transcription factors[J].Nat Biotechnol,2003,21(10):1208-1214.
[33] PARK K S,SEOL W,YANG H Y,et al.Identification and use of zinc finger transcription factors that increase production of recombinant proteins in yeast and mammalian cells[J].Biotechnol Prog,2005,21(3):664-670.
[34] TANAKA Y,KASAHARA K,HIROSE Y,et al.Enhancement of butanol production by sequential introduction of mutations conferring butanol tolerance and streptomycin resistance[J].J Biosci Bioeng,2017,124(4):400-407.
[35] LI J,NAYAK S,MRKSICH M.Rate enhancement of an interfacial biochemical reaction through localization of substrate and enzyme by an adaptor domain[J].J Phys Chem B,2010,114(46):15113-15118.
[36] WRIGGERS W,CHAKRAVARTY S,JENNINGS P A.Control of protein functional dynamics by peptide linkers[J].Peptide Sci,2005,80(6):736-746.
[37] ZHANG Y,LI S Z,LI J,et al.Using unnatural protein fusions to engineer resveratrol biosynthesis in yeast and mammalian cells[J].J Am Chem Soc,2006,128(40):13030-13031.
[38] WANG Y,YI H,WANG M,et al.Structural and kinetic analysis of the unnatural fusion protein 4-coumaroyl-CoA ligase::stilbene synthase[J].J Am Chem Soc,2011,133(51):20684-20687.
[39] DUEBER J E,WU G C,MALMIRCHEGINI G R,et al.Synthetic protein scaffolds provide modular control over metabolic flux[J].Nat Biotechnol,2009,27(8):753-759.
[40] MOON T S,DUEBER J E,SHIUE E,et al.Use of modular,synthetic scaffolds for improved production of glucaric acid in engineeredE.coli[J].Metab Eng,2010,12(3):298-305.
[41] HIRAKAWA H,NAGAMUNE T.Molecular assembly of P450 with ferredoxin and ferredoxin reductase by fusion to PCNA[J].ChemBioChem,2010,11(11):1517-1520.
[42] PARK S H,ZARRINPAR A,LIM W A.Rewiring MAP kinase pathways using alternative scaffold assembly mechanisms[J].Science,2003,299:1061-1064.
[43] AVALOS J L,FINK G R,STEPHANOPOULOS G.Compartmentalization of metabolic pathways in yeast mitochondria improves the production of branched-chain alcohols[J].Nat Biotechnol,2013,31(4):335-341.
[44] SZCZEBARA F M,CHANDELIER C,VILLERET C,et al.Total biosynthesis of hydrocortisone from a simple carbon source in yeast[J].Nat Biotechnol,2003,21(2):143-149.
[45] FARHI M,MARHEVKA E,MASCI T,et al.Harnessing yeast subcellular compartments for the production of plant terpenoids[J].Metab Eng,2011,13(5):474-481.
[46] LV X,WANG F,ZHOU P,et al.Dual regulation of cytoplasmic and mitochondrial acetyl-CoA utilization for improved isoprene production inSaccharomycescerevisiae[J].Nat Commun,2016,7:12851.
[47] CHEN X,DONG X,WANG Y,et al.Mitochondrial engineering of the TCA cycle for fumarate production[J].Metab Eng,2015,31:62-73.
[48] GIDIJALA L,KIEL J A,DOUMA R D,et al.An engineered yeast efficiently secreting penicillin[J].PLoS ONE,2009,4(12):e8317.
[49] LEE P C,GEOL YOON Y,SCHMIDT-DANNERT C.Investigation of cellular targeting of carotenoid pathway enzymes inPichiapastoris[J].J Biotechnol,2009,140(3):227-233.
[50] CHEN L,ZHANG J,CHEN W N.Engineering theSaccharomycescerevisiaeβ-oxidation pathway to increase medium chain fatty acid production as potential biofuel[J].PLoS ONE,2014,9(1):e84853.
[51] SHENG J,STEVENS J,FENG X.Pathway compartmentalization in peroxisome ofSaccharomycescerevisiaeto produce versatile medium chain fatty alcohols[J].Sci Rep,2016,6:26884.
[52] BAYER T S,WIDMAIER D M,TEMME K,et al.Synthesis of methyl halides from biomass using engineered microbes[J].J Am Chem Soc,2009,131(18):6508-6515.
[53] THODEY K,GALANIE S,SMOLKE C D.A microbial biomanufacturing platform for natural and semisynthetic opioids[J].Nat Chem Biol,2014,10(10):837-844.
[54] 王俊姝,祁庆生.合成生物学与代谢工程[J].生物工程学报,2009,25(9):1296-1302.
[56] WU Y,LI B Z,ZHAO M,et al.Bug mapping and fitness testing of chemically synthesized chromosome X[J].Science,2017,355:eaaf4706.
[57] HANAI T,ATSUMI S,LIAO J.Engineered synthetic pathway for isopropanol production inEscherichiacoli[J].Appl Environ Microbiol,2007,73(24):7814-7818.
[58] YIM H,HASELBECK R,NIU W,et al.Metabolic engineering ofEscherichiacolifor direct production of 1,4-butanediol[J].Nat Chem Biol,2011,7(7):445-452.
[59] ZHANG F,CAROTHERS J M,KEASLING J D.Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids[J].Nat Biotechnol,2012,30(4):354-359.
[60] CHAKRAVARTY S,BARIK D.Steady state statistical correlations predict bistability in reaction motifs[J].Mol Biosyst,2017,13(4):775-784.
[61] LI Z,LIU S,YANG Q.Incoherent inputs enhance the robustness of biological oscillators[J].Cell Syst,2017,5(1):72-81.e4.
[62] NISTALA G J,WU K,RAO C V,et al.A modular positive feedback-based gene amplifier[J].J Biol Eng,2010,4(1):4.
[63] DANIEL R,RUBENS J R,SARPESHKAR R,et al.Synthetic analog computation in living cells[J].Nature,2013,497:619-623.
[64] CHEN X,GAO C,GUO L,et al.DCEO biotechnology:tools to design,construct,evaluate,and optimize the metabolic pathway for biosynthesis of chemicals[J].Chem Rev,2017,doi:10.1021/acs.chemrev.6b00804.
[65] NA D,YOO S M,CHUNG H,et al.Metabolic engineering ofEscherichiacoliusing synthetic small regulatory RNAs[J].Nat Biotechnol,2013,31(2):170-174.
[66] AJIKUMAR P K,XIAO W H,TYO K E,et al.Isoprenoid pathway optimization for taxol precursor overproduction inEscherichiacoli[J].Science,2010,330:70-74.
[67] LO T M,TEO W S,LING H,et al.Microbial engineering strategies to improve cell viability for biochemical production[J].Biotechnol Adv,2013,31(6):903-914.
[68] YADAV V G,DE MEY M,LIM C G,et al.The future of metabolic engineering and synthetic biology:towards a systematic practice[J].Metab Eng,2012,14(3):233-241.
[69] LIU Y,ZHU Y,LI J,et al.Modular pathway engineering ofBacillussubtilisfor improvedN-acetylglucosamine production[J].Metab Eng,2014,2(3):42-52.
[70] KORMAN T P,OPGENORTH P H,BOWIE J U.A synthetic biochemistry platform for cell free production of monoterpenes from glucose[J].Nat Commun,2017,8:15526.

