胃癌患者癌组织与癌旁组织菌群比较分析
宫大伟,王莉莉,周建华,于新娟,孟新颖,周长虹,董全江
青岛大学医学院附属青岛市市立医院 1.中心实验室; 2.消化科; 3.口腔医学中心,山东 青岛 266000
肠道微生物群落(菌群)能够影响人体众多的生理功能,如能量代谢、免疫系统成熟及抵抗感染[1-2]。菌群失调参与到糖尿病、风湿性疾病和过敏等疾病的发病机制中[3-4]。人体肠道菌群多样性和结构的改变与炎症性肠病发生密切相关。越来越多的证据表明,菌群能够促进肿瘤的发生、发展[5]。
胃内菌群主要由变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、梭杆菌门(Fusobacteria)和放线菌门(Actinobacteria)构成。INS-GAS-小鼠模型中菌群缺乏能够延迟胃癌的发生,表明胃内菌群能够促进癌症发生[6]。与慢性胃炎和肠化生相比,胃癌患者胃内菌群的丰度和均匀度均增高[7-8],这与此前的研究一样,都表明更加多样化的胃内菌群与胃癌发生有关[9]。胃内菌群的结构似乎在慢性胃炎、肠上皮化生、胃癌的癌症发生过程中逐渐改变[7,10]。因此,胃癌组织的菌群结构与慢性胃炎明显不同。胃癌患者胃内菌群的组成也发生明显改变[7-8,10-11],但不同的研究中发现的癌症相关细菌并不一致。
在肿瘤环境中从正常黏膜向恶性病变过渡时菌群特征变化情况尚不清楚。以往的结直肠癌研究发现,癌性病变组织有着由众多物种组成的肠道菌群[12]。与癌旁正常黏膜相比,大肠癌病变组织中一些细菌丰度增加[13]。Fusobacterium和大肠埃希氏菌被发现具有促癌活性,且在癌症病变组织中过度增加[14-15]。目前,关于胃癌患者恶性病变部位菌群特征的研究相对缺乏。为了进一步阐明胃黏膜癌变过程中微生物的作用,本研究通过与胃癌患者癌旁正常胃黏膜比较,探讨胃癌癌变组织中菌群生物多样性、结构、群落类型及组成的变化。
1 资料与方法
1.1患者样本采集本研究共纳入2015年3月至2016年8月在青岛市市立医院进行手术治疗的88例晚期胃癌患者,年龄(62.3±0.5)岁,男58例,女30例。每例患者签署知情同意书,该研究方案得到青岛市市立医院医学研究伦理委员会同意批准。为了最大程度减少对胃内菌群的影响,所有入选患者在标本收集前8周内没有接受抗生素及质子泵抑制剂治疗。癌组织取自癌症病变处,癌旁组织取自手术切除组织边缘的正常黏膜。一块组织进行常规组织学检查,另一块贮存于-80 ℃冰箱中用于后续DNA提取。
1.2荧光原位杂交分析组织固定于甲醛溶液中过夜并用石蜡包埋,4 mm切片。按照之前报道[16]的方法进行荧光原位杂交检测。用5′标记有Cy3的探针(EUB 338,GCTGCCTCCCGTAGGAGT)来检测胃黏膜细菌[17]。在Olympus IX71显微镜上对切片进行成像。
1.3DNA提取通过对DNA提取进行胃内菌群分析。首先对胃黏膜标本进行研磨,然后用1 U的DNase降解外源细菌DNA。然后将样品在终浓度为50 g/L溶菌酶中处理。用QIAGEN血液和组织试剂盒提取基因组DNA。
1.4定量PCR反应为了定量胃黏膜的细菌数量,根据HARMS等[18]报道的方法对细菌16S rRNA基因进行实时荧光定量多聚核苷酸链式反应(real-time quantitative PCR,qPCR)。引物和探针序列如下:正向引物(5′-ATGGCTGTCGTCAGCT-3′),反向引物(5′-ACGGGCGGTGTGTAC-3′)及荧光探针(5′-CAACGAGCGCAACCC-3′)。通过对含有16S rRNA基因全长的质粒进行一系列稀释,并构建标准曲线。计算每μg DNA 16S rRNA基因拷贝数作为细菌数量。
为了定量胃黏膜Fusobacterium菌属丰度,对其16S rRNA基因进行扩增,其中引物和探针序列遵照之前研究方法[19]。前列腺素转运蛋白(PGT)作为内参基因[20]。在胃癌的癌组织和癌旁组织中对Fusobacterium丰度计算倍差(2-ΔΔCt)。ΔCt为目的基因与内参基因阈值循环数之差。
1.5焦磷酸测序和数据分析为了分析胃黏膜菌群,我们对细菌16S rRNA的V1~V3区进行PCR扩增,其引物为8F/533R。通过Q5高保真DNA聚合酶进行25个循环的PCR扩增。运用454 GS FLX平台对扩增产物进行测序。去除所得序列的引物、标签及接头序列并对序列按照以下标准进行过滤:长度>200 nt,同聚物<9,模糊碱基<0和平均质量>25。12个样本共产生138 143条序列,运用 UCHIME软件进行嵌合体去除。序列相似度达到97%划分为一个可操作单元。使用核糖体数据库项目Naïve Bayes Classifier对其分类学分析[21-22]。使用QIIME软件进行稀疏曲线、α多样性和β多样性分析[23]。胃黏膜菌群的丰度通过Chao1指数进行评估,其能够反映微生物群落中理论上的物种数量。Shannon指数包含物种的丰度和物种的均匀度,其常被用来评估胃黏膜菌群的生物多样性[23]。依赖于Bray-Curitis指数的非度量多维尺度分析(non-metric multi-dimensional scaling,NMDS)用于分析不同微生物群落间的相似度[24]。Dirichlet多项混合模型(DMM模型)用于进行群落类型分析[25]。本研究短序列已放于美国国家生物技术信息中心的网站中(研究号:SRP060550、SRP082742)。
1.6统计学分析采用SPSS 18.0软件进行统计分析及运用Prism软件画图。通过t检验或配对秩和检验对数据进行统计分析。P<0.05为差异有统计学意义。
2 结果
2.1胃内细菌分布癌旁组织中,荧光原位杂交发现细菌主要位于黏膜表面。它们分布相对分散,但发现在黏膜表面脱落的黏液中出现聚集,可以偶尔在胃腺中发现其存在(见图1A)。在癌组织中存在相似细菌分布模式(见图1B)。但在癌组织深处未发现细菌存在。
2.2胃癌中总细菌数量对88例胃癌患者的癌组织与癌旁组织细菌运用qPCR进行细菌定量。在胃癌的癌组织中细菌数量(log)为7.89±0.88,轻微低于癌旁组织细菌数量(8.13±0.89),但差异无统计学意义(P>0.05)。
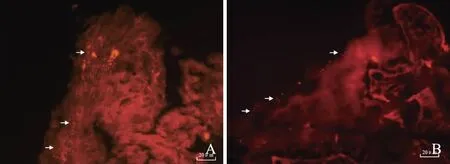
注:细菌红标(箭头所示)。
2.3癌组织与癌旁组织中胃内菌群分析为了进一步研究胃癌患者胃内菌群,对6例患者的细菌16S rRNA基因扩增子进行高通量测序用于比较癌组织与癌旁组织之间的菌群特征。癌组织中的Chao1指数为880±192,轻微低于癌旁组织(985±242),但其差异无统计学意义(P>0.05)。癌组织中Shannon多样性指数为2.31±0.73,而癌旁组织为1.93±0.52(P>0.05)。表明胃癌患者癌组织与癌旁组织相比,菌群多样性未发生改变。
通过主坐标分析(principal coordinate analysis,PCoA)及NMDS分析对菌群结构进行相似度研究,结果发现癌组织与癌旁组织样本呈现一个分散的分布模式(见图2)。癌组织样本未发生明显分离,表明在胃癌患者中菌群结构同样未发生明显改变。
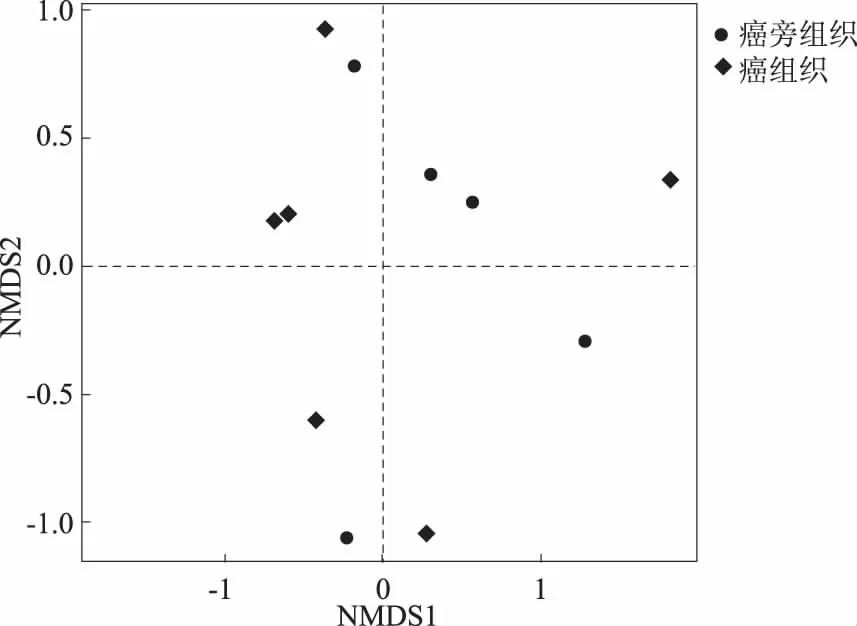
注:较小的点间距离对应着较小的群落构成差异,反之亦然。
运用DMM分型方法,将12个样本在菌群属水平的相对丰度拟合到Dirichlet中用于增加Dirichlet混合成分K数量,通过使用拉普拉斯近似的负对数模型证据支持在胃内菌群中存在a、b、c三种群落类型(见图3),发现在胃癌患者中主要群落类型为c型(66.7%)。同一个体的癌组织和癌旁组织的群落类型基本一致。
组成分析发现,胃内菌群的优势菌门为Proteobacteria(见图4)。在菌门水平,癌组织与癌旁组织比较发现其Firmicutes、Bacteroidetes、Fusobacteria及Actinobacteria相对丰度轻微增加,但差异无统计学意义(P>0.05)。在菌属水平上,Fusobacterium、Moryella、Porphyromonas菌属的相对丰度在癌组织中明显增加(癌组织vs癌旁组织:0.43%vs0.13%;1.32%vs0.04%;0.45%vs0.05%)。配对秩和检验表明这些细菌的丰度差异有统计学意义(P=0.048、0.036、0.021)。

图3 Dirichlet混杂拟合模型Fig 3 Model fit for mixture of Dirichlets
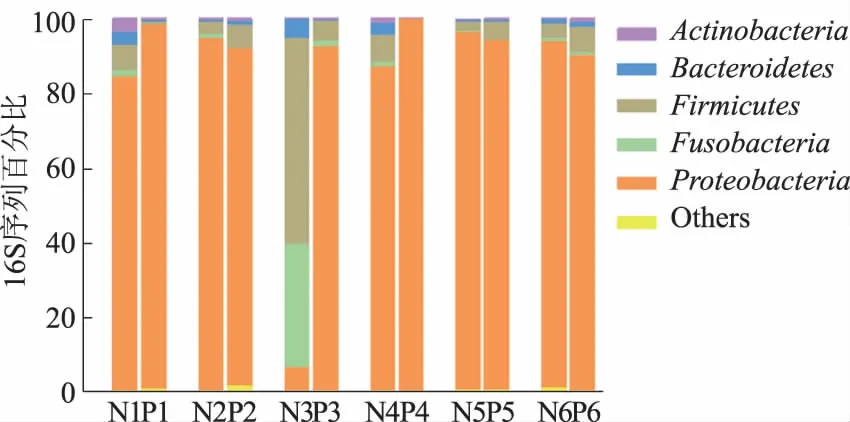
注: N:癌组织;P:癌旁组织。
2.4梭杆菌属在癌组织中数量增加Fusobacterium菌属是结直肠癌发生过程中一种潜在的致癌菌[20]。通过在88例患者成对的胃黏膜标本中对该菌属进行qPCR,定量Fusobacterium菌属细菌数量。结果表明癌组织中Fusobacterium菌属平均总丰度是癌旁组织的3倍(P=0.043)。
3 讨论
本研究发现,细菌存在于癌组织表面。癌性病变处的细菌数量与毗邻的正常胃黏膜相比无明显改变。这些发现表明,癌性病变处的微环境对细菌的生长和分布影响较小。进一步分析发现,癌组织中菌群的物种丰度、均匀度、群落结构和菌群类型均未发生变化。癌组织与癌旁组织菌群的高度相似同样表明癌性病变处的微环境对菌群特征影响较小。这与结直肠癌的研究结果一致,结直肠癌的癌变部位与毗邻的黏膜组织相比,其菌群α-多样性变化差异无统计学意义[13]。结肠癌癌变组织的菌群结构与癌旁组织相比未发生明显变化。综上所述,虽然癌变组织的坏死和异常分泌可能会引起微环境的改变,但结果发现,癌性病变本身并不会显著改变微生物群落。
菌群组成分析表明,Fusobacterium、Moryella、Porphyromonas菌属在癌组织中相对丰度升高,qPCR同样证明Fusobacterium菌属出现过度生长。这些研究结果表明,这些菌属与胃癌的发生可能相关。Fusobacterium、Moryella、Porphyromona菌属定植于胃肠道黏膜,如口腔、食管、胃及结肠黏膜中均发现它们的存在[26-28]。且在相关研究中发现,结肠腺瘤和结肠癌中Fusobacterium菌属丰度增加[12-13,28]。Fusobacterium菌属和Porphyromonas菌属也被发现与口腔鳞状细胞癌相关[29]。也有研究[30]报道,Porphyromonas菌属参与胰腺癌的发生。此外也有研究[27]发现,Moryella菌属与食管鳞癌存在明显相关性。这些细菌与癌症的密切联系表明,它们可能参与了胃癌的发生。最近研究[31-32]发现,F.nucleatum能够通过炎症NF-κB信号通路引起肿瘤形成。F.nucleatum表达黏附分子FadA,FadA能够结合上皮细胞E-钙黏蛋白,从而抑制其肿瘤抑制活性[33]。此外,Fusobacterium、Moryella和Porphyromonas菌属能够参与到炎症性疾病的发生,进而又可以促进癌症的发生和发展[34-35]。目前的数据虽然表明这些细菌与胃癌之间存在潜在联系,但也不能够排除上述菌属在癌症发生、发展中仅仅作为旁观者存在的可能。它们在胃癌发生中的作用仍需进一步研究。
在胃肠道癌症中发现,菌群组成发生改变,癌症中增加的物种可以用作癌症发现、风险评估和预后的生物标记物[36-38]。初步研究表明,F.nucleatum可能是疾病进展和肿瘤严重程度的一个潜在指标[20,39]。在我们的研究中,定量分析发现,胃癌组织中Fusobacterium菌属平均总丰度3倍于配对的癌旁组织,是否可以运用Fusobacterium菌属作为发现胃癌的生物标记物仍有待进一步研究。
本研究表明,在胃癌中,癌性病变部位的菌群特征未发生明显改变。同时也发现了具有潜在促癌特性的细菌,它们在胃癌中的作用仍有待进一步研究。
[1] PARFREY L W, KNIGHT R. Spatial and temporal variability of the human microbiota [J]. Clin Microbiol Infect, 2012, 18 Suppl 4: 8-11. DOI: 10.1111/j.1469-0691.2012.03861.x.
[2] LOZUPONE C A, STOMBAUGH J I, GORDON J I, et al. Diversity, stability and resilience of the human gut microbiota [J]. Nature, 2012, 489(7415): 220-230. DOI: 10.1038/nature11550.
[3] NICHOLSON J K, HOLMES E, KINROSS J, et al. Host-gut microbiota metabolic interactions [J]. Science, 2012, 336(6086): 1262-1267. DOI: 10.1126/science.1223813.
[4] MILLION M, LAGIER J C, YAHAV D, et al. Gut bacterial microbiota and obesity [J]. Clin Microbiol Infect, 2013, 19(4): 305-313. DOI: 10.1111/1469-0691.12172.
[5] WANG L L, YU X J, ZHAN S H, et al. Participation of microbiota in the development of gastric cancer [J]. World J Gastroenterol, 2014, 20(17): 4948-4952. DOI: 10.3748/wjg.v20.i17.4948.
[6] LOFGREN J L, WHARY M T, GE Z, et al. Lack of commensal flora in helicobacter pylori-infected INS-GAS mice reduces gastritis and delays intraepithelial neoplasia [J]. Gastroenterology, 2011, 140(1): 210-220. DOI: 10.1053/j.gastro.2010.09.048.
[7] EUN C S, KIM B K, HAN D S, et al. Differences in gastric mucosal microbiota profiling in patients with chronic gastritis, intestinal metaplasia, and gastric cancer using pyrosequencing methods [J]. Helicobacter, 2014, 19(6): 407-416. DOI: 10.1111/hel.12145.
[8] WANG L, ZHOU J, XIN Y, et al. Bacterial overgrowth and diversification of microbiota in gastric cancer [J]. Eur J Gastroenterol Hepatol, 2016, 28(3): 261-266. DOI: 10.1097/MEG.0000000000000542.
[9] CORREA P. A human model of gastric carcinogenesis [J]. Cancer Res, 1988, 48(13): 3554-3560.
[10] AVILES-JIMENEZ F, VAZQUEZ-JIMENEZ F, MEDRANO-GUZMAN R, et al. Stomach microbiota composition varies between patients with non-atrophic gastritis and patients with intestinal type of gastric cancer [J]. Sci Rep, 2014, 4: 4202. DOI: 10.1038/srep04202.
[11] DICKSVED J, LINDBERG M, ROSENQUIST M, et al. Molecular characterization of the stomach microbiota in patients with gastric cancer and in controls [J]. J Med Microbiol, 2009, 58(Pt4): 509-516. DOI: 10.1099/jmm.0.007302-0.
[12] NAKATSU G, LI X, ZHOU H, et al. Gut mucosal microbiome across stages of colorectal carcinogenesis [J]. Nat Commun, 2015, 6: 8727. DOI: 10.1038/ncomms9727.
[13] ALLALI I, DELGADO S, MARRON P I, et al. Gut microbiome compositional and functional differences between tumor and non-tumor adjacent tissues from cohorts from the US and Spain [J]. Gut Microbes, 2015, 6(3): 161-172. DOI: 10.1080/19490976.2015.1039223.
[14] TAHARA T, YAMAMOTO E, SUZUKI H, et al. Fusobacterium in colonic flora and molecular features of colorectal carcinoma [J]. Cancer Res, 2014, 74(5): 1311-1318. DOI: 10.1158/0008-5472.CAN-13-1865.
[15] ARTHUR J C, PEREZ-CHANONA E, MUHLBAUER M, et al. Intestinal inflammation targets cancer-inducing activity of the microbiota [J]. Science, 2012, 338(6103): 120-123. DOI: 10.1126/science.1224820.
[16] SHEN X J, RAWLS J F, RANDALL T, et al. Molecular characterization of mucosal adherent bacteria and associations with colorectal adenomas [J]. Gut Microbes, 2010, 1(3): 138-147. DOI: 10.4161/gmic.1.3.12360.
[17] DAIMS H, BRÜHL A, AMANN R, et al. The domain-specific probe eub338 is insufficient for the detection of all bacteria: development and evaluation of a more comprehensive probe set [J]. Syst Appl Microbiol, 1999, 22(3): 434-444. DOI: 10.1016/S0723-2020(99)80053-8.
[18] HARMS G, LAYTON A C, DIONISI H M, et al. Real-time pcr quantification of nitrifying bacteria in a municipal wastewater treatment plant [J]. Environ Sci Technol, 2003, 37(2): 343-351.
[19] BOUTAGA K, VAN WINKELHOFF A J, VANDENBROUCKE-GRAULS C M, et al. Periodontal pathogens: a quantitative comparison of anaerobic culture and real-time PCR [J]. FEMS Immunol Med Microbiol, 2005, 45(2): 191-199. DOI: 10.1016/j.femsim.2005.03.011.
[20] CASTELLARIN M, WARREN R L, FREEMAN J D, et al. Fusobacterium nucleatum infection is prevalent in human colorectal carcinoma [J]. Genome Res, 2012, 22(2): 299-306. DOI: 10.1101/gr.126516.111.
[21] WANG Q, GARRITY G M, TIEDJE J M, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy [J]. Appl Environ Microbiol, 2007, 73(16): 5261-5267. DOI: 10.1128/AEM.00062-07.
[22] LOZUPONE C A, KNIGHT R. Species divergence and the measurement of microbial diversity [J]. FEMS Microbiol Rev, 2008, 32(4): 557-578. DOI: 10.1111/j.1574-6976.2008.00111.x.
[23] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data [J]. Nat Methods, 2010, 7(5): 335-336. DOI: 10.1038/nmeth.f.303.
[24] LOOFT T, JOHNSON T A, ALLEN H K, et al. In-feed antibiotic effects on the swine intestinal microbiome [J]. Proc Natl Acad Sci U S A, 2012, 109(5): 1691-1696. DOI: 10.1073/pnas.1120238109.
[25] HOLMES I, HARRIS K, QUINCE C. Dirichlet multinomial mixtures: generative models for microbial metagenomics [J]. PLoS One, 2012, 7(2): e30126. DOI: 10.1371/journal.pone.0030126.
[26] CARLIER J P, K'OUAS G, HAN X Y. Moryella indoligenes gen. Nov., sp. Nov., an anaerobic bacterium isolated from clinical specimens [J]. Int J Syst Evol Microbiol, 2007, 57(Pt 4): 725-729. DOI: 10.1099/ijs.0.64705-0.
[27] CHEN X, WINCKLER B, LU M, et al. Oral microbiota and risk for esophageal squamous cell carcinoma in a high-risk area of China [J]. PLoS One, 2015, 10(12): e0143603. DOI: 10.1371/journal.pone.0143603.
[28] SALAZAR C R, SUN J, LI Y, et al. Association between selected oral pathogens and gastric precancerous lesions [J]. PLoS One, 2013, 8(1): e51604. DOI: 10.1371/journal.pone.0051604.
[29] PUSHALKAR S, JI X, LI Y, et al. Comparison of oral microbiota in tumor and non-tumor tissues of patients with oral squamous cell carcinoma [J]. BMC Microbiol, 2012, 12: 144. DOI: 10.1186/1471-2180-12-144.
[30] MICHAUD D S. Role of bacterial infections in pancreatic cancer [J]. Carcinogenesis, 2013, 34(10): 2193-2197. DOI: 10.1093/carcin/bgt249.
[31] KOSTIC A D, CHUN E, ROBERTSON L, et al. Fusobacterium nucleatum potentiates intestinal tumorigenesis and modulates the tumor-immune microenvironment [J]. Cell Host Microbe, 2013, 14(2): 207-215. DOI: 10.1016/j.chom.2013.07.007.
[32] DIDONATO J A, MERCURIO F, KARIN M. NF-κB and the link between inflammation and cancer [J]. Immunol Rev, 2012, 246(1): 379-400. DOI: 10.1111/j.1600-065X.2012.01099.x.
[33] RUBINSTEIN M R, WANG X, LIU W, et al. Fusobacterium nucleatum promotes colorectal carcinogenesis by modulating E-cadherin/β-catenin signaling via its FadA adhesin [J]. Cell Host Microbe, 2013, 14(2): 195-206. DOI: 10.1016/j.chom.2013.07.012.
[34] BASHIR A, MISKEEN A Y, HAZARI Y M, et al. Fusobacterium nucleatum, inflammation, and immunity: the fire within human gut [J]. Tumour Biol, 2016, 37(3): 2805-2810. DOI: 10.1007/s13277-015-4724-0.
[35] LENZO J C, O’BRIEN-SIMPSON N M, ORTH R K, et al. Porphyromonas gulae has virulence and immunological characteristics similar to those of the human periodontal pathogen porphyromonas gingivalis [J]. Infect Immun, 2016, 84(9): 2575-2585. DOI: 10.1128/IAI.01500-15.
[36] YU J, FENG Q, WONG S H, et al. Metagenomic analysis of faecal microbiome as a tool towards targeted non-invasive biomarkers for colorectal cancer [J]. Gut, 2017, 66(1): 70-78. DOI: 10.1136/gutjnl-2015-309800.
[37] MARCHESI J R, DUTILH B E, HALL N, et al. Towards the human colorectal cancer microbiome [J]. PLoS One, 2011, 6(5): e20447. DOI: 10.1371/journal.pone.0020447.
[38] BAXTER N T, RUFFIN M T 4TH, ROGERS M A, et al. Microbiota-based model improves the sensitivity of fecal immunochemical test for detecting colonic lesions [J]. Genome Med, 2016, 8(1): 37. DOI: 10.1186/s13073-016-0290-3.
[39] FLANAGAN L, SCHMID J, EBERT M, et al. Fusobacterium nucleatum associates with stages of colorectal neoplasia development, colorectal cancer and disease outcome [J]. Eur J Clin Microbiol Infect Dis, 2014, 33(8): 1381-1390. DOI: 10.1007/s10096-014-2081-3.

