激肽释放酶KLK7小分子抑制剂的筛选及活性初步研究
王芳宇,戴俊斌,谭 潇,何丽芳,王德平,李玉林
(1.衡阳师范学院 生命科学与环境学院,功能金属有机材料湖南省重点实验室,湖南 衡阳 421008;2.南华大学 药学与生物科学学院,湖南 衡阳 421001;3.三峡大学 医学院,湖北 宜昌 443002)
激肽释放酶(Kallikreins,KLKs)是具有胰蛋白酶或胰凝乳蛋白酶特异性的丝氨酸蛋白酶,其编码基因定位于19q13.4,编码15个分泌型丝氨酸蛋白酶KLK 1-KLK 15[1]。已经证明,KLKs存在于正常人的皮肤角质层细胞中,目前发现,至少有8种KLK(KLK 5,KLK 6,KLK 7,KLK 8,KLK 10,KLK 11,KLK 13和KLK 14)存在于人的角质层和汗液中,其中KLK7占总量的40 %。KLK7可能是皮肤损伤的一个关键酶,而且在特应性皮炎(Atopic Dermatitis,AD)病人中它可能参与一个系统性炎症反应,在AD的角质层KLK7升高最为突出[2],这提示KLK7在AD的发病机制上可能是至关重要的,因此,抑制KLK7的活性对于AD的治疗显得最为重要。
本研究以KLK7作为靶分子,根据KLK7的晶体结构,采用美国ChemDiv化合物数据库进行计算机模拟筛选,挑选能与KLK7催化域匹配结合的化合物(3 000个分子),然后对3 000个分子进行人工挑选,挑选时综合考虑化合物与靶蛋白的结合模式、预测的亲合性数据以及成药性指标,进一步得到300个分子。分析这300个分子对KLK7的抑制作用,以期得到具有较好抑制活性的小分子,作为AD治疗的药物先导化合物。
1 材料与方法
1.1 材料
有机化合物数据库:ChemDiv 公司提供的有机小分子化合物筛选库,约含 105万个有机小分子化合物。
小分子化合物:美国TOPSCIENCE公司;
激肽释放酶7(Recombinant Human Kallikrein 7 :R&D; MCa-R-P-K-P-V-E-Nval-W-R-K(Dnp)-NH2:R&D;
多功能荧光酶标仪:Bio-tek公司(美国)。
1.2 方法
1.2.1 计算机虚拟筛选
以KLK7作为靶分子,根据KLK7的晶体结构,采用美国ChemDiv化合物数据库 (1 044 623个分子)进行计算机模拟筛选。分两步进行,第一步基于药效团模型进行筛选,共得到342 366个化合物;第二步,对获得的化合物进行分子对接筛选,挑选与KLK7催化域匹配结合效果好的化合物(3 000个分子)(虚拟筛选委托上海陶素生化技术有限公司完成)。
1.2.2 人工挑选
对虚拟筛选中排名靠前的3 000个化合物进行人工挑选,挑选时综合考虑化合物与靶蛋白的结合模式、预测的亲合性数据以及成药性指标。最终挑选匹配效果最好的前300个化合物分子(Top300)进行后续的研究。
1.2.3 Top300小分子化合物对KLK7的抑制效果分析
KLK7分别与小分子化合物在37 ℃下作用15分钟,然后加入KLK7的荧光底物MCa-R-P-K-P-V-E-Nval-W-R-K(Dnp)-NH2,15分钟(37 ℃)后测酶活性。不含小分子化合物的为阴性对照,计算抑制率=(1-a/b)×100 %,a为待测样品的酶活,b为阴性对照的酶活。
1.2.4 化合物IC50的测定
选取抑制率高的化合物设置成6.5、12.5、25、50、100和200 μM等6个浓度梯度,分别与KLK7 在37 ℃下作用15分钟,然后加入KLK7的荧光底物MCa-R-P-K-P-V-E-Nval-W-R-K(Dnp)-NH2,15分钟(37 ℃)后测酶活性。
2 结 果
2.1 Top300小分子化合物的获得
重新dock Top3000分子,得到Score和RMSD值。Score代表对docking的打分,Score值越小越好。RMSD代表dock后分子构象与起始构象的差别。对Score值根据分子量作修正,得到revised Score值。以revised Score为X轴,RMSD值为Y轴作图,如图1。在图1作辅助线,以辅助线为轴,越靠近图表左下方的分子,它的Score值与RMSD值越小,分子预测效果越好。反之,越靠近右上方的分子它的Score值与RMSD值越大,分子预测效果越差。将所有分子的坐标投影到辅助线上得到projection value,然后排序,挑选Top300个分子 (表1)。

图1 revised Score与RMSD值之间呈微弱正相关

表1 挑选的Top300分子

续表
续表
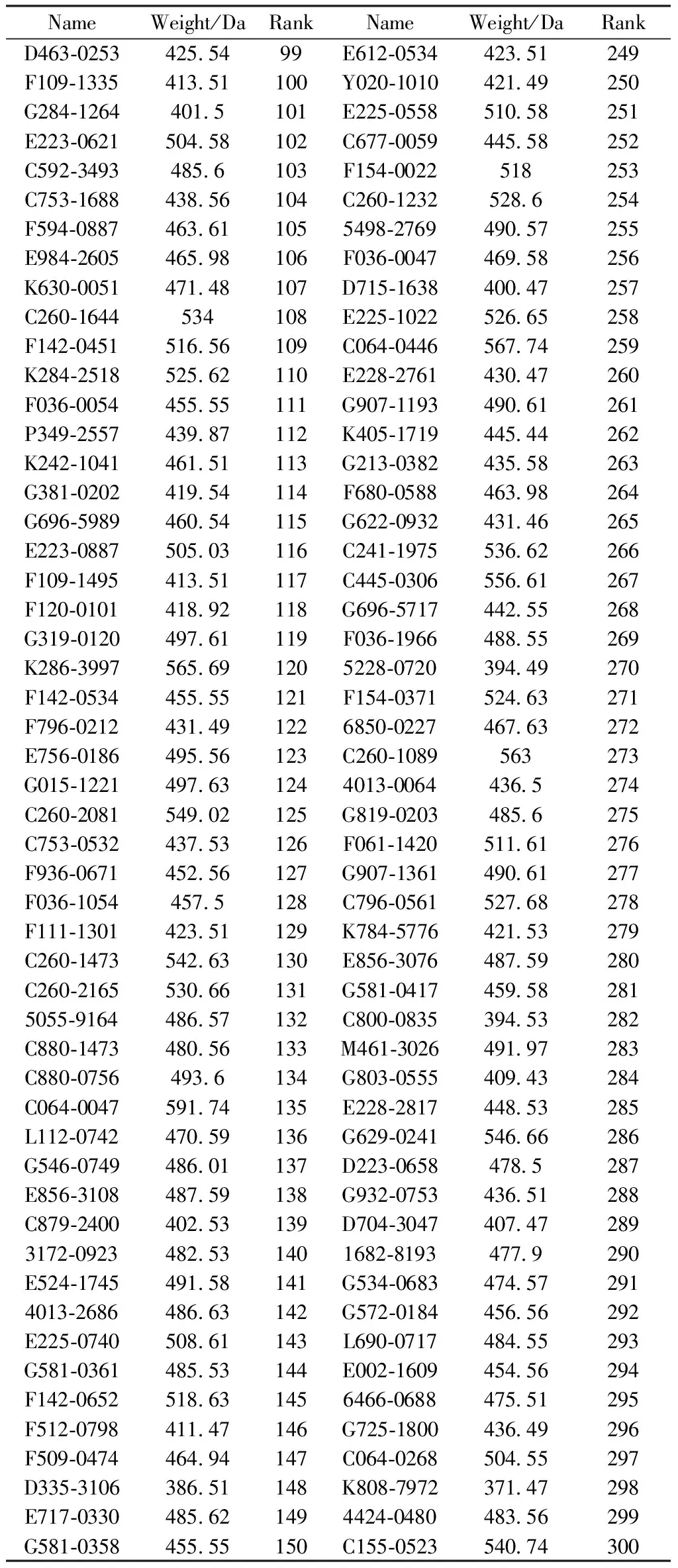
NameWeight/DaRankNameWeight/DaRankD463⁃0253425 5499E612⁃0534423 51249F109⁃1335413 51100Y020⁃1010421 49250G284⁃1264401 5101E225⁃0558510 58251E223⁃0621504 58102C677⁃0059445 58252C592⁃3493485 6103F154⁃0022518253C753⁃1688438 56104C260⁃1232528 6254F594⁃0887463 611055498⁃2769490 57255E984⁃2605465 98106F036⁃0047469 58256K630⁃0051471 48107D715⁃1638400 47257C260⁃1644534108E225⁃1022526 65258F142⁃0451516 56109C064⁃0446567 74259K284⁃2518525 62110E228⁃2761430 47260F036⁃0054455 55111G907⁃1193490 61261P349⁃2557439 87112K405⁃1719445 44262K242⁃1041461 51113G213⁃0382435 58263G381⁃0202419 54114F680⁃0588463 98264G696⁃5989460 54115G622⁃0932431 46265E223⁃0887505 03116C241⁃1975536 62266F109⁃1495413 51117C445⁃0306556 61267F120⁃0101418 92118G696⁃5717442 55268G319⁃0120497 61119F036⁃1966488 55269K286⁃3997565 691205228⁃0720394 49270F142⁃0534455 55121F154⁃0371524 63271F796⁃0212431 491226850⁃0227467 63272E756⁃0186495 56123C260⁃1089563273G015⁃1221497 631244013⁃0064436 5274C260⁃2081549 02125G819⁃0203485 6275C753⁃0532437 53126F061⁃1420511 61276F936⁃0671452 56127G907⁃1361490 61277F036⁃1054457 5128C796⁃0561527 68278F111⁃1301423 51129K784⁃5776421 53279C260⁃1473542 63130E856⁃3076487 59280C260⁃2165530 66131G581⁃0417459 582815055⁃9164486 57132C800⁃0835394 53282C880⁃1473480 56133M461⁃3026491 97283C880⁃0756493 6134G803⁃0555409 43284C064⁃0047591 74135E228⁃2817448 53285L112⁃0742470 59136G629⁃0241546 66286G546⁃0749486 01137D223⁃0658478 5287E856⁃3108487 59138G932⁃0753436 51288C879⁃2400402 53139D704⁃3047407 472893172⁃0923482 531401682⁃8193477 9290E524⁃1745491 58141G534⁃0683474 572914013⁃2686486 63142G572⁃0184456 56292E225⁃0740508 61143L690⁃0717484 55293G581⁃0361485 53144E002⁃1609454 56294F142⁃0652518 631456466⁃0688475 51295F512⁃0798411 47146G725⁃1800436 49296F509⁃0474464 94147C064⁃0268504 55297D335⁃3106386 51148K808⁃7972371 47298E717⁃0330485 621494424⁃0480483 56299G581⁃0358455 55150C155⁃0523540 74300
2.2 Top300小分子化合物对KLK7的抑制效果
1) 200 μM浓度下筛选得到13个对KLK7抑制效率为100 %的化合物,编号分别是26,44,47,52,54,63,73,125,138,165,166,251,260;
2)将上步筛选到的13个化合物的反应浓度降低至50 μM,检测它们在50 μM浓度时对KLK7的抑制效率,发现44号化合物在50 μM时对KLK7没有抑制作用,其余12个化合物依然有抑制作用但抑制效率下降,其中%inhibition在50 %以上的化合物有5个,编号分别为47,54,125,166,260。

表 2 50 M浓度下13个化合物对KLK7的抑制率
注:*表示%Inhibition>50
2.3 抑制率高的五个化合物对KLK7的IC50
进一步研究50 μM浓度下对KLK7的抑制率>50 %的五个化合物对KLK7的抑制效率,根据测得的F.U/h计算出%inhibition,再根据% inhibition和抑制剂浓度用KaleidaGraph软件进行曲线拟合,计算各个抑制剂对KLK7的IC50(其中54号化合物抑制效果检测不理想,结果没有列出)。结果显示,47号、125号、166号和260号化合物对KLK7的IC50依次是:37.166 μM,25.32 μM,28.955 μM,36.078 μM。

表3 47号化合物检测数据
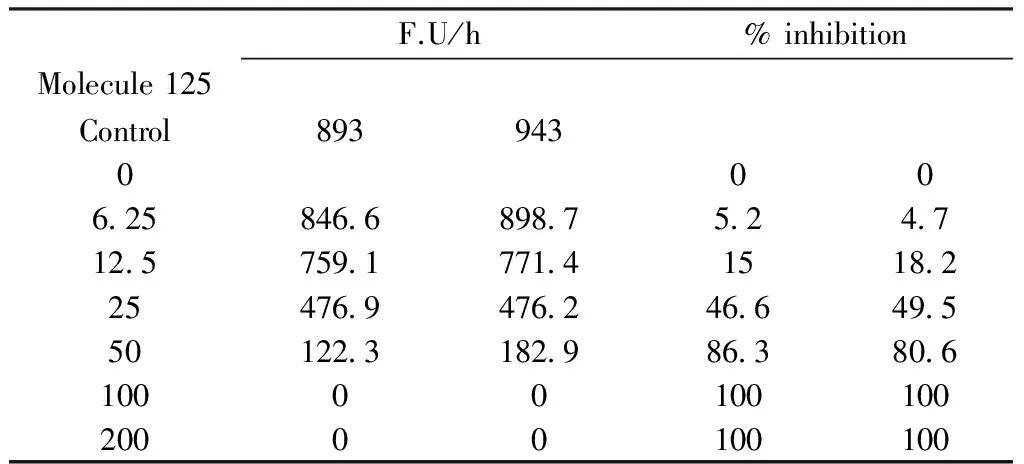
表4 125号化合物检测数据

表5 166号化合物检测数据

表6 260号化合物检测数据
3 讨 论
近年来,使用信息工具进行药物设计和虚拟筛选在短时间内来获得药物先导化合物越来越受到重视。计算机工具发现新药先导化合物的应用可帮助研究人员迅速设计和评估新的药物。计算机资源减少了获得药物的时间和相关经济成本,使得在低成本下有靶标性地预测潜在药物成为可能[3]。
虚拟筛选依赖分子模拟技术,从大量的有机化合物中遴选出可能有效的候选化合物,避免了对化合物的盲目筛选,从而大大降低了发现活性先导化合物的成本。近些年来,国内外针对一些重大疾病药物靶标均有许多成功使用虚拟筛选的实例[4]。从原理上来讲,虚拟筛选可以分为两类,即基于分子对接的虚拟筛选和基于药效团的虚拟筛选。基于分子对接的虚拟筛选从靶蛋白的三维结构出发,研究靶蛋白结合位点的特征性质以及它与小分子化合物之间的相互作用模式,根据与结合能相关的亲合性打分函数对蛋白和小分子化合物的结合能力进行评价,最终从大量的化合物分子中挑选出结合模式比较合理的、预测得分较高的化合物,用于后续的生物活性测试。基于药效团的虚拟筛选一般是利用一系列已知活性的小分子化合物产生一个与药物作用对应的药效团模型,然后根据这个模型在化合物数据库中搜索能够与它匹配的化学分子结构,最后对这些挑选出来的化合物进行实验筛选研究。
本实验选用的有机化合物数据库ChemDiv 数据库,是由 ChemDiv 公司提供的有机小分子化合物筛选库,是目前世界上提供数量最多,结构具有丰富多样性的化合物库。采用的药物分子设计软件GOLD (GOLD 软件套装5.2版,由英国Cambridge Crystal Data Center发行)是由 Sheffield 大学,GlaxoSmithKline 公司和 Cambridge Crystallographic Data Center(CCDC)共同开发的对接程序[5]。它采用遗传算法产生小分子配体的结合构象,分子对接时配体是完全柔性的,蛋白分子可以考虑部分柔性。GOLD 程序自带多种打分函数,包括GoldScore[6]、ChemScore[7]、ASP[8]。新版中整合了打分函数ChemScore_Kinase 和ChemScore_PLP。
本实验通过筛选得到的300个小分子化合物,进一步检测抑制效率较高的化合物并测定其对KLK7的IC50。其中47号、125号、166号和260号化合物对KLK7的IC50依次是:37.166 μM,25.32 μM,28.955 μM,36.078 μM,说明这四个化合物对KLK7有较好的抑制作用,可以进一步研究。
[1] YOUSEF G M,DIAMANDIS E P.The new human tissue kallikrein gene family:structure,function,and association to disease[J].Endocrine reviews,2001,22(2): 184-204.
[2] KOMATUS N,SAIJOH K,KUK C,et al.Human tissue kallikrein expression in the stratum corneum and serum of atopic dermatitis patients[J].Experimental Dermatology,2007,16(6):513-519.
[3] CUMMING J G,DAVIS A M,MURESAN S,et al.Chemical predictive modelling to improve compound quality[J].Nat Rev Drug Discov,2013,12(12):948-962.
[4] GHOSH S,NIE A H,AN J,et al.Structure-based virtual screening of chemical libraries for drug discovery[J].Curr Opin Chem Biol,2006,10:194-202.
[5] JONES G,WILLETT P,GLEN R C.Molecular recognition of receptor sites using an algorithm with a description of desolvation[J].J Mol Biol,1995,245:43-53.
[6] JONES G,WILLETT P,GLEN R.C,et al.Development and validation of a genetic algorithm for flexible docking[J].J Mol Biol,1997,267(3):727-748.
[7] ELDRIDGE M D,MURRAY C W,AUTON T R,et al.Empirical scoring functions: I.the development of a fast empirical scoring function to estimate the binding affinity of ligands in receptor complexes[J].J Comput Aided Mol Des,1997,11(5):425-445.
[8] MOOIJ W T,VERDONK M L.General and targeted statistical potentials for protein-ligand interactions[J].Proteins,2005,61(2):272-287.
(编校 陈志阳)

