雨生红球藻PHOT的基因克隆和生物信息学分析
崔红利,陈 军,王林萍,胡章立,秦 松
(1.大连大学生命科学与技术学院,辽宁 大连 116622;2.中国科学院烟台海岸带研究所海岸带生物学与生物资源利用重点实验室,山东 烟台 264003;3.中国科学院大学,北京 100049;4.深圳大学生命与海洋科学学院,广东 深圳 518060)
雨生红球藻PHOT的基因克隆和生物信息学分析
崔红利1,陈 军2,3,王林萍1,胡章立4,秦 松2
(1.大连大学生命科学与技术学院,辽宁 大连 116622;2.中国科学院烟台海岸带研究所海岸带生物学与生物资源利用重点实验室,山东 烟台 264003;3.中国科学院大学,北京 100049;4.深圳大学生命与海洋科学学院,广东 深圳 518060)
【目的】本文旨在获得雨生红球藻中蓝光受体向光素(Phototropin,PHOT)的全长序列并进行生物信息分析。【方法】采用同源克隆和RACE结合的方法获得雨生红球藻中编码HaePHOT的cDNA序列全长,并对HaePHOT进行生物信息分析。【结果】HaePHOT的开放阅读框全长为2139 bp,编码712个氨基酸,预测的等电点8.66,理论分子量78.73 kD。经BLASTP程序分析,与拟南芥来源PHOT的相似性达到58 %,与莱茵衣藻来源PHOT的相似性达到68 %。通过序列比对和结构域分析,PHOT蛋白的典型结构域存在于HaePHOT中,包括发色团结合和激酶结构域。系统进化分析表明,高等植物和真核绿藻来源PHOTs来自共同祖先。【结论】首次从雨生红球藻中获得编码PHOT的基因序列,为下一步雨生红球藻中PHOT的表达和功能研究奠定基础,同时为解析蓝光调控雨生红球藻虾青素合成的分子机制提供线索。
向光素;cDNA全长;结构域;系统进化;雨生红球藻
【研究意义】向光素(phototropin, PHOT)是植物和绿藻特有的蓝光受体[1-2]。PHOT由N端光感知(Light-oxygen-voltage, LOV)结构域、Jα螺旋区和C端丝氨酸/苏氨酸激酶(Serine/threonine kinase, STK)结构域组成[2-5]。其中N端光感知结构域依次含有两个序列极为相似的光-氧-电LOV结构域(LOV1和LOV2),在感知蓝光信号、结合黄素单核苷酸FMN(Flavin mononucleotide)发色团及光依赖自磷酸化过程中发挥重要作用[6];Jα螺旋区位于LOV2和C端激酶之间,大约20个保守氨基酸,可介导激酶(STK)与光感知(LOV)结构域的耦合,是蓝光诱导PHOT发生自磷酸化所必需的条件[7];C端STK结构域与其他蛋白互作,在蓝光信号转导方面发挥重要作用[7-9]。磷酸化、构象改变及蛋白互作是PHOT介导蓝光信号转导的重要机制,已有大量的磷酸化位点和互作蛋白被鉴定[10-13],磷酸化的PHOT通过构象改变激活STK激酶的活性,进而与其他蛋白互作,共同调控多种生理反应[14-18]。拟南芥包括两个向光素(AtPHOT1和AtPHOT2),在功能上冗余而又不完全相同,例如AtPHOT1和AtPHOT2共同调节光形态建成、气孔开度、叶片伸展、叶片运动及叶绿体积聚运动[19-21]。有趣的是AtPHOT1 介导叶绿体的积聚运动,与蓝光信号强弱无关;而AtPHOT2 在低强度蓝光照射时介导叶绿体的积聚运动,在高强度蓝光照射时介导叶绿体的逃避运动[19-22]。尽管高等植物中蓝光受体PHOT的基因克隆、功能鉴定及信号通路方面有一些研究报道,但在绿藻中的报道很少。【前人研究进展】与高等植物相比,绿藻中PHOT的功能和信号通路研究还很不完善,莱茵衣藻(Chlamydomonasreinhardtii)来源CrePHOT已经完成分子克隆、序列分析及功能研究。RNA干扰实验验证CrePHOT参与细胞有性生活周期[23],并能影响大量基因在转录水平的表达,涉及捕光复合物形成、叶绿素和类胡萝卜素的生物合成[24]。通过突变技术,表明CrePHOT调控眼点大小和细胞周期[24-26]。除此之外,通过拟南芥AtPHOT1和AtPHOT2双突变株的恢复实验,CrePHOT部分功能也得到了验证,上述结果暗示绿藻和高等植物来源PHOTs的功能与作用机制较为保守[19]。团藻(Volvoxcarteri)中VcaPHOT的相关研究表明其表达与细胞的发育相关[27]。最近,海洋生境绿藻(Ostreococcustauri)中OtaPHOT相关研究表明其作用机制和功能与拟南芥存在差异[11]。目前,雨生红球藻中未见PHOT的相关报道。雨生红球藻,也称红球藻(Haematococcuspluvialis)是一种淡水单细胞绿藻,是目前已知虾青素含量最高的物种,在胁迫条件(高温、高盐、强光和营养缺乏等)下虾青素含量可达细胞干重的4 %,是极为理想的天然虾青素来源[28]。但红球藻中虾青素的合成是一个诱导的过程,即合成与积累总是发生在不适合红球藻细胞生物量积累的胁迫条件下,两者之间的矛盾已成为利用红球藻高效生产天然虾青素的限制因素[29]。大量研究表明,光是影响虾青素合成的重要因子[30-33]。并且在同等光强条件下,与自然光(白光)相比,蓝光更有效诱导红球藻中类胡萝卜素羟化酶基因的表达和虾青素的积累[34-35]。但相关基因如何被激活,作用机制不清楚。高等植物研究表明光调控类胡萝卜素合成存在新机制:光受体介导的信号通路可调控类胡萝卜素代谢相关功能基因(psy、lcyb、bch、zep和vde)在转录水平上的表达[36-38]。因此,可推测类似信号通路也存在于红球藻中。开展红球藻中PHOT功能的研究是探讨上述新调控机制的理想途径,但获得基因序列和生物信息学分析是前提条件。【本研究切入点】本文以雨生红球藻为研究对象,利用同源克隆与RACE结合的方法克隆HaePHOT基因的cDNA 全长,对其理化性质、结构及系统进化进行了分析,进一步对功能进行了预测。【拟解决的关键问题】为该基因的功能鉴定和信号通路的研究奠定了基础。
1 材料与方法
1.1 供试材料
1.1.1 实验材料 本实验所用Haematococcuspluvialisstrain Flotow 1844购自藻类和原生生物培养中心(Dunstaffnage Marine Laboratory),由本实验室保存。采用EG:JM有机培养基和BBM无机培养基,培养条件为25 μmol m-2s-1的光照强度,于(22±1) ℃,12 h/12 h的光/暗周期,静置培养每天摇动2~3次。大肠杆菌(E.coil)采用Top 10为本实验室保存。LB培养基,包括10 g/L胰蛋白胨,5 g/L酵母萃取物,10 g/L NaCl,用5 M NaOH调pH至7.0~7.2,定容,高压灭菌15~20 min。固体培养基加入1 %的琼脂。
1.1.2 实验试剂 提取RNA的试剂盒(RNAiso plus)、反转录试剂盒(PrimeScriptTMRT reagent Kit with gDNA Eraser)、RACE试剂盒(SMARTerTMRACE cDNA Amplication Kit)、PCR试剂盒(TaKaRa LATaq®)、DNA片段回收试剂盒(MiniBEST Agarose Gel DNA Extraction Kit)及测序载体(pMD-18T)等均购买于宝生物公司。无水乙醇、氯仿、异丙醇等购自国药集团化学试剂有限公司,琼脂糖购自Biowest 公司。
1.1.3 实验仪器 超净工作台(上海博迅)、光照培养箱 GTOP(浙江托普仪器有限公司)、公司的电泳设备(GE)、VeriFlexTM96 PCR仪(Well Applied Biosystems)、凝胶成像仪 GelDox XR Bio(伯乐)、小型低温高速离心机(Thermo Fisher)、Nano Drop 2000 分光光度计(赛默飞世尔科技公司)。

表1 文中用到的引物信息
1.2 试验方法
1.2.1 总RNA的提取 采用Takara公司的RNAiso plus试剂盒。具体过程参考产品说明书。RNA 提取过程涉及到的所有耗材包括枪头,离心管采用RNA提取专用的耗材,购自Axygen公司。其它的玻璃和瓷器包括研钵等经过180 ℃干热处理8 h 以上。实验过程中保持工作台面干净,并用75 %酒精擦拭,操作人员穿戴实验室,戴口罩,并及时更换1次性手套,整个操作过程中尽量减少人员流动。具体操作过程如下:①离心收集藻株,采用PBS缓冲溶液悬浮洗藻体3~5次,并及时存放于液氮中保存;②取适量的藻体,在液氮条件下迅速研磨成粉末状,快速加入1 mL RNAiso plus,涡旋仪上震荡均匀,室温孵育3 min;③加入氯仿200 μl,管盖,震荡混匀,室温孵育3 min;④在4 ℃条件下,12 000 r/min 离心15 min,转移上清到一新的离心管中;⑤加等体积的异丙醇,混合均匀, -20 ℃静置30 min;⑥在4 ℃条件下,12 000 r/min 离心15 min;⑦倒掉上清,重新离心,用移液器将残液吸出,防止 RNA 沉淀丢失;⑧用 1 mL 70 % 乙醇洗沉淀1次,4 ℃下,12 000 r/min 离心5 min;⑨室温下晾干,大概3~5 min,加入适量DEPC 处理水,溶解;⑩采用超微量分光光度计进行定量分析,同时采用凝胶电泳仪进行电泳检测。提取后的RNA于-80 ℃保存。
1.2.2 模板的制备 包括普通cDNA模板的制备和RACE模板制备两大类。普通模板的制备参照TaKaRa 公司的PrimeScriptTMRT reagent Kit with gDNA Eraser试剂盒说明书进行。包括基因组DNA 处理和反转录两步。RACE 模板的制备参照Clontech 公司的 SMARTerTMRACE cDNA Amplication Kit说明书。
1.2.3 引物设计 在比较基因组学分析的基础上,根据与雨生红球藻物种相近5株绿藻物种(C.reinhardtii,C. sp. NC64A,V.carteri,C.vulgaris, 和C. sp. C-169) 向光素PHOT基因序列比对的结果,找出高度保守的氨基酸序列,用CODEHOP软件设计兼并引物。根据同源克隆片段设计RACE引物,获得全长的基础上设计表达框引物(表1),引物合成由上海生工生物工程公司完成。
1.2.4 同源片段的获得 以1.2.2中制备的普通cDNA为模板,结合1.2.3中设计的同源克隆引物,采用TaKaRa公司的TaKaRa LATaq®扩增体系进行PCR。反应体系的配置按照试剂盒说明书的要求,PCR反应条件如下:94 ℃ 5 min,1 个循环;94 ℃ 30 s,55 ℃(根据引物的退火温度)30 s,72 ℃ 1 Kb/min,30个循环;72 ℃ 7 min,4 ℃ 保温。循环结束后,取3 μl PCR 产物,用 1.2 %的琼脂糖胶电泳检测,根据引物位置确定目的条带的大小,将可能的目的条带进行胶回收,进行后续的连接、转化及测序。
1.2.5 全长的获得 在获得同源克隆片段的基础上,设计两端RACE引物(表1),以1.2.2中制备的RACE cDNA为模板,根据试剂盒说明书进行两端片段的PCR。产物进行琼脂糖凝胶电泳检测、连接、转化及挑选阳性克隆测序。
1.2.6 生物信息分析 HaePHOT基因cDNA全长序列通过软件SeqMan software of DNAStar 7.1 (DNASTAR Inc., USA)拼接得到;序列翻译和ORF的查找借助在线软件(http://www.bio-soft.net/sms/)。序列鉴定通过在线BLASTP和tBLASTN程序完成,利用ClustalW软件进行多序列比对分析。目的基因翻译氨基酸的分子量(Mw)和等电点(pI)采用ExPASy Compute pI/Mw tool软件预测,跨膜结构与亚细胞定位分别采用Transmembrane Prediction Server和ChloroP 及TargetP Server分别预测。通过在线SMART和Pfam软件进行结构域分析,利用PhyML中的ML (Maximum likelihood) 法构建系统进化树(模型选择Leand Gascuel 进化模型)。

M: Marker DL2000图1 雨生红球藻总RNA的提取和Haephot基因克隆Fig.1 Total RNA of H. pluvialis and amplification of Haephot
2 结果与分析
2.1 雨生红球藻总RNA的提取
如图1所示,出现3条带,从上往下依次是28S rRNA,18S rRNA 和5S rRNA ,前2条带比较清晰,第3条带模糊,说明完整性较好,没发生明显的降解。RNA的浓度和纯度采用NanoDorp 2000微量分光光度计检测,A260/A280在1.8~2.0,A260/A230在1.9~2.0,所得到的RNA浓度为850 ng/μl,总体积为50 μl。说明RNA质量比较好,可以满足后续试验的要求。
2.2 雨生红球藻Haephot基因cDNA序列的获得
过同源克隆和RACE相结合方法,获得了雨生红球藻中编码向光素基因的cDNA 序列(HaePHOT, NCBI 注册号:KT354897)的全长(图2,封三)。全长为2692 bp,编码区2139 bp(137~2275),编码712个氨基酸,5′-非翻译区(5’-untranslated region, 5’-UTR)长度为136 bp,3′-非翻译区(3’-UTR)长度为417 bp,并带有poly (A) 尾巴。通过BLASTP程序分析,与拟南芥来源PHOT的相似性达到58 %,与莱茵衣藻来源PHOT的相似性达到68 %。
2.3 雨生红球藻HaePHOT的理化性质分析
利用ProtParam 软件分析HaePHOT基本理化性质。表明该蛋白质分子式为(C3510H5579N971O1027S24),开放阅读框翻译的氨基酸预测分子量为78.73 kD,预测的等电点为8.66。由20 种基本氨基酸组成,含量较高的氨基酸残基是亮氨酸(Leu, 10.4 %)和缬氨酸(Val, 9.0 %),含量较低的氨基酸残基是色氨酸(Trp, 0.8 %)和半胱氨酸(Cys, 1.3 %)。带正电荷氨基酸残基85个(Arg+Lys),带负电荷氨基酸残基81个(Asp+Glu)。预测该蛋白为稳定蛋白(不稳定系数为32.75)。信号肽预测(SignalP 4)结果显示不存在信号肽序列。DAS-TMfilte和TMHMM软件预测目的蛋白不存在跨膜区,是可溶性蛋白。细胞定位显示该蛋白定位细胞质。
2.4 雨生红球藻HaePHOT的二级结构分析
蛋白质的二级结构包括α-螺旋(H: α-helix)、β-折叠(T: β-turn)、延伸链(E: Extended strand)和无规则卷曲(C: Random coil)。基于软件SOPMA 二级结构预测结果表明,无规则卷曲占的比例最高(35.39 %),其次为由α-螺旋(27.95 %),延伸链和β-折叠分别占25.98 % 和10.67 %。综上所述,无规则卷曲和α-螺旋是HaePHOT 蛋白二级结构的主要组成,属于混合型蛋白。
2.5 雨生红球藻HaePHOT序列比对、保守结构域和磷酸化位点分析
利用SMART和CDD分析了HaePHOT中的保守结构域(图3A)。HaePHOT编码蛋白中存在N 端的2个重复PAS亚家族结构域(24-125和215-304),C端的丝氨酸/苏氨酸激酶结构域(387-702),上述两大结构域是向光素家族的典型特征。为了进一步研究PHOT蛋白中的保守结构域,对不同来源五种真核绿藻(PHOT-1,PHOT-2,PHOT-3,PHOT-4和PHOT-5)和高等植物拟南芥来源的PHOTs蛋白进行序列比对,发现在LOV1和LOV2中都存在结合发色团FMN保守结构域(GXNCRFLQG),其中C是结合FMN的活性位点(图3B)。蛋白质磷酸化是一种常见的翻译后修饰,可影响蛋白质的功能,因此磷酸化分析对研究蛋白质的功能具有重要意义。基于Scansite 3.0 软件分析了HaePHOT蛋白中可能的磷酸化位点(图3C)。预测到HaePHOT 中存在12个可能的磷酸化位点,其中第1个PAS结构域中存在4个(T23、V87、T106和T110),第2个PAS结构域中存在2个(P211和T293),激酶结构域中存在4个(T419、Y475、T543和P620)及C末端2个(P695和Y696)。
2.6 雨生红球藻HaePHOT高级结构预测
为进一步研究Hae PHOT蛋白的高级结构,采用同源建模方法对其高级结构进行了预测。因为PHOT蛋白包括3个保守结构域(LOV1、LOV2和STK)因此对3个保守结构域分别进行了高级结构研究(图4)。结果显示,LOV1和LOV2结构域具有几乎一样的高级结构,包括4个β-折叠和2个α-螺旋。其中结合发色团的保守半胱氨酸残基存在于整个高级结构的中心位置,图4A和图4B中的红色圆圈标记。LOV2与STK激酶之间通过一个大约20个氨基酸残基的Jα螺旋区域连接,其中Jα螺旋区域是图4C中的红色箭头所示。STK激酶结构域的高级结构与其他蛋白中STK激酶高级结构极为相似(图4D),暗示phot基因的形成可能是通过不同基因的不同结构域重新组合形成。

图3 雨生红球藻HaePHOT的结构域分析、序列比对及磷酸化位点预测Fig.3 Structural domain (A), amino acid sequence alignment (B) and phosphorylation sites (C) analysis of HaePHOT
2.7 雨生红球藻HaePHOT的起源和系统进化分析
在高等植物和真核绿藻中先后发现了PHOT蛋白,为了进一步研究不同物种来源PHOTs的起源和系统进化,采用PHYML软件进行系统进化树的构建(图5)。系统进化树表明雨生红球藻来源HaePHOT与其他真核绿藻(莱茵衣藻和团藻)聚为一类,并且与海洋生境的真核绿藻形成姐妹群,进而与高等植物来源的PHOTs形成单系群,并且PHOT的基因进化树与物种之间的进化树吻合。这些研究结果进一步暗示高等植物和真核绿藻来源PHOTs具有共同祖先,形成于真核绿藻和高等植物分枝之前。
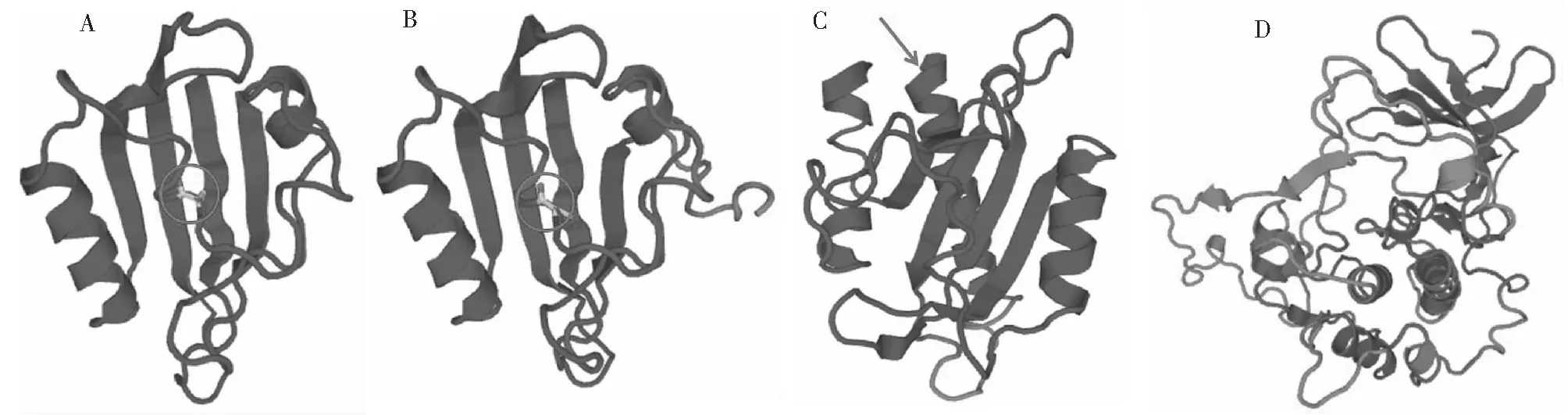
A为LOV1结构域部分的高级结构;B为LOV2结构域部分的高级结构;C为LOV2结构域部分(含有Jα螺旋区域)的高级结构;D为STK激酶结构域部分的高级结构图4 雨生红球藻HaePHOT的LOV1、LOV2及STK_c结构域的高级结构预测Fig.4 Model of structure of LOV1, LOV2 and STK_c from HaePHOT
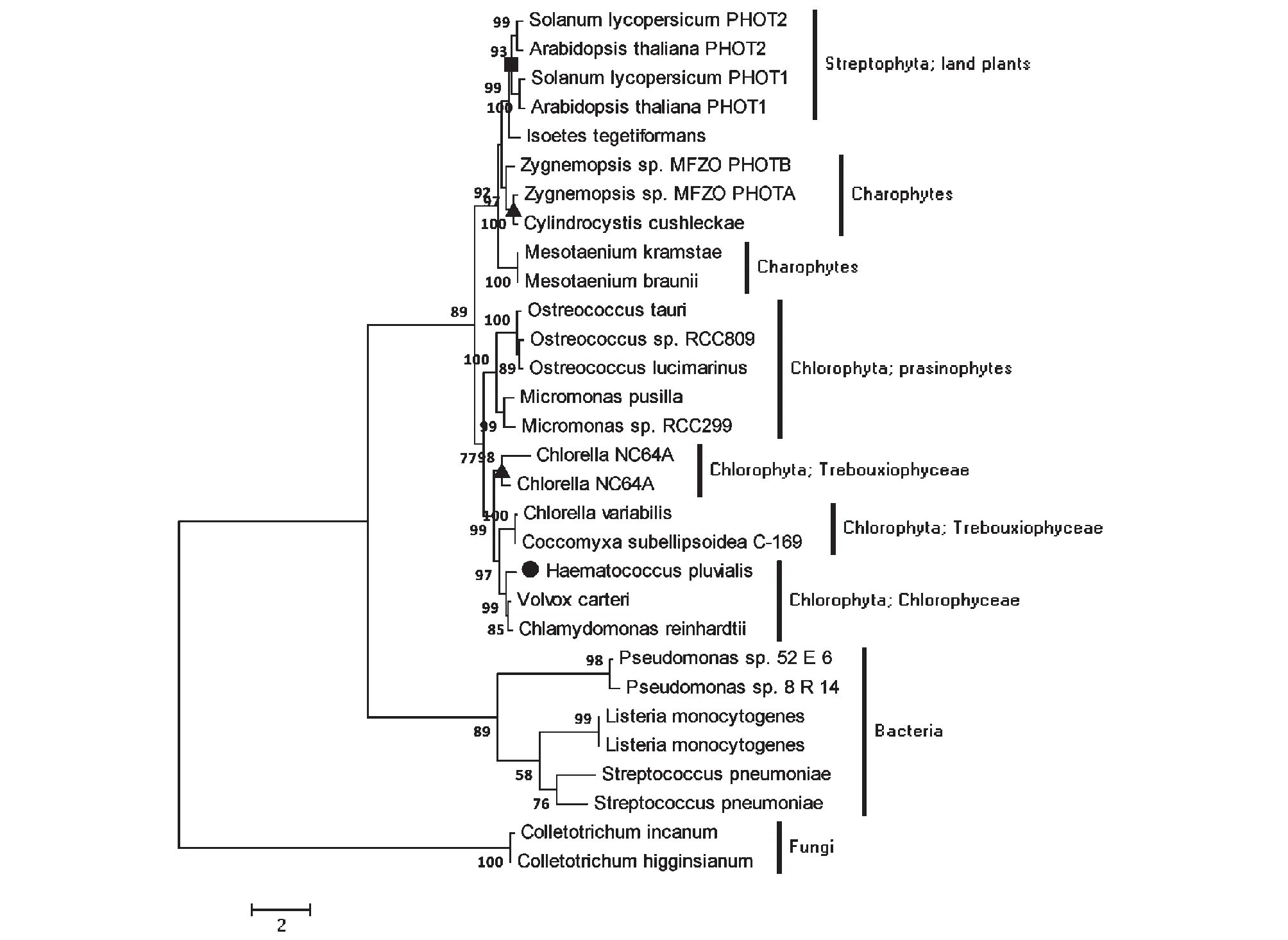
其中HaePHOT为黑色圆点标记,黑色三角形代表物种特异性的种内基因复制现象(基因复制发生在物种形成之后),黑色方框代表类群特异性的基因复制现象(基因复制发生在物种形成之前)。不同种类物种来源的PHOTs被分类标记(Solanum lycopersicum PHOT1和 PHOT2:NP_001234214和XP_010323569; Zygnemopsis sp. MFZO PHOTA 和PHOTB: AHZ63920和AHZ63918; Arabidopsis thaliana PHOT1和 PHOT2: PHOT1_ARATH和PHOT2_ARATH; Mesotaenium braunii PHOT: AHZ63922; Mesotaenium kramstage PHOT: AHZ63457)图5 不同物种来源PHOTs的系统进化分析Fig.5 Maximum likelihood tree of PHOTs from bacteria, fungi, higher plants and green alga
3 讨 论
光是环境中重要的信号因子之一,不仅为真核微藻提供能量,而且还为它们提供信息,调节其生长发育过程[8]。为适应光强、光质及光周期等光信息,真核微藻在漫长的进化过程中形成了一套精细的光信号接收和转导系统。光受体在光信号通路中发挥重要的作用,起着接收和转化的桥梁作用。高等植物拟南芥中PHOT的研究表明,PHOT可介导蓝光调控多种生理过程的信号通路,为真核微藻中研究PHOT的功能提供了线索[3,16,19]。
通过同源克隆和RACE结合方法获得了雨生红球藻中编码PHOT的基因cDNA序列全长。HaePHOT与拟南芥来源PHOT的相似性达到58 %,与莱茵衣藻来源PHOT的相似性达到68 %,暗示该基因可能是雨生红球藻中可能的编码PHOT的基因。向光素PHOT蛋白由N端与发色团FMN结合的并具有蓝光感知功能的光氧电结构域(LOV1和LOV2)和C端具有激酶活性的STK_c的结构域组成[4,7,9,13]。保守结构域分析表明,在HaePHOT中同样存在上述3个保守结构域,进一步暗示这个基因编码PHOT蛋白。
光感知结构域LOV在蓝光激发下,通过发色团FMN和胱氨酸Cys3发生可逆光循环,形成共价结合物,进而诱导PHOT蛋白的构象变化和激活激酶活性,最终磷酸化互作蛋白,LOV1和 LOV2对光照、氧气及电压差敏感[8,39-40]。GXNCRFLQG及其保守的结构域是LOV的典型特征,其中C是发色团FMN的结合位点[40],在HaePHOT中也发现了该保守区域。C 末端含有Ser/Thr蛋白激酶区域,属于蛋白激酶家族。蛋白质磷酸化是一种常见的翻译后修饰,影响蛋白的活性与功能,因此磷酸化分析对研究蛋白质的功能具有重要意义[41]。拟南芥中PHOT的研究表明,磷酸化在PHOT蛋白的光信号转导过程中发挥重要的作用,已经在At PHOT1 的 C末端鉴定出了多个磷酸化位点[42]。通过磷酸化位点预测软件,在HaePHOT蛋白中发现了12个可能的磷酸化位点,在任何一个保守结构域中都有分布,这与上述研究结果一致,并为后续磷酸化机制功能研究提供了重要线索。Jα螺旋区域的功能是连接LOV2和STK激酶,起着桥梁作用,在激活激酶活性的过程中发挥重要的作用[8],通过同源建模方法,在HaePHOT中发现了同样的Jα螺旋区域,该区域与LOV2区域紧密相邻。研究表明PHOT蛋白N端的两个LOV结构域不仅在氨基酸序列水平极为相似,同时具有几乎一样的高级结构[19]。同源建模结果表明HaePHOT蛋白的LOV1和LOV2的高级结构几乎一致,与上述研究结果吻合[19]。
含有LOV结构域蛋白介导的光信号转导机制广泛分布于几乎所有的生物中[43-44],但目前研究表明,PHOTs蛋白仅存在于高等植物和真核绿藻中,在其他类型真核微藻中并没有发现。同理,含有STK激酶保守结构域的蛋白也是广泛存在于几乎所有的生物中[45],上述研究结果对编码PHOT的基因起源和系统进化提出了挑战。通过构建系统进化树分析表明,当真菌来源的LOV结构域蛋白作为进化树的根时,高等植物和真核绿藻来源PHOTs形成一个单系群,暗示他们具有共同的祖先,这与前人的研究结果相符[1]。暗示phot基因的出现发生在高等植物和真核绿藻分开之前,在进化过程中的不同选择压力条件下,出现了基因复制现象并在功能上出现了分工。基因复制现象的发生存在2种形式,一种基因复制发生在物种形成之后,另一种是物种形成之前。并且基因的出现是通过不同基因的LOV结构域和STK结构域的重新组合形成,但这种假说还需要进一步数据支持。
总而言之,上述研究结果为雨生红球藻中HaePHOT的基因表达研究和确定其功能奠定了基础。同时保守结构域的分析和高级结构预测为蓝光信号感知和转导机制提供了依据,并将有助于研究PHOT在蓝光信号通路中相关调控因子的作用机制。例如,雨生红球藻是天然虾青素的理想来源,也是目前制备虾青素的首选藻种。多种研究表明高蓝光可有效诱导其虾青素的合成与积累,调控主要发生在转录水平上,但具体分子机制并不清楚。红球藻中PHOT基因的表达模式如何?是否具有功能?在蓝光诱导下,体内的PHOT如何感知和转导蓝光信号,并如何与其他已知的蛋白(COP1和CIBI等)互作?互作之后通过何种途径激活下游转录因子的表达从而启动红球藻中虾青素合成相关基因的表达,最终合成并积累虾青素,这些问题都有待进行研究。目前,由于有关PHOT的研究主要以高等植物拟南芥为主,PHOT介导的光信号转导路径中的许多疑问没有解决,更多参与PHOT 响应蓝光信号的转录因子有待进一步发现。雨生红球藻中PHOT 基因的同源克隆和生物信息学分析对于今后更深入系统地研究PHOT 蛋白响应蓝光的分子机理有一定的参考价值。
4 结 论
获得了雨生红球藻中蓝光受体向光素(Phototropin,PHOT)的cDNA全长序列并进行了生物信息分析。HaePHOT的开放阅读框全长为2139 bp,编码712个氨基酸,预测的等电点8.66,理论分子量78.73 kDa。与拟南芥来源PHOT的相似性达到58 %,与莱茵衣藻来源PHOT的相似性达到68 %。PHOT蛋白的典型结构域存在于HaePHOT中,包括发色团结合和激酶结构域。高等植物和真核绿藻来源PHOTs属于共同祖先。首次从雨生红球藻中获得编码PHOT的基因序列,存在与高等植物PHOT相似的结构域,暗示其作用机制与高等植物类似,为下一步雨生红球藻中PHOT的表达和功能研究奠定基础,同时为解析蓝光调控雨生红球藻虾青素合成的分子机制提供线索。
[1]Li F W, Rothfels C J, Melkonian M, et al. The origin and evolution of phototropins[J]. Frontiers in Plant Science, 2014(6): 637.
[2]崔红利, 陈 军, 侯义龙,等. 真核微藻蓝光受体及其功能研究[J]. 生物技术通报, 2017,33(4): 51-62.
[3]乔新荣, 段鸿斌, 叶兆伟. 植物向光素受体与信号转导机制研究进展[J]. 生物技术通报, 2014(8): 1-7.
[4]Happer S M, Neil L C, Gardner K H. Structural basis of a phototropin light switch[J]. Science, 2003, 301(5639): 1541-1544.
[5]Kaiserli E, Sullivan S, Jones M A, et al. Domain swapping to assess the mechanistic basis ofArabidopsisphototropin 1 receptor kinase activation and endocytosis by blue light[J]. Plant Cell, 2009, 21(10): 3226-3244.
[6]Hefti M H, Kees-Jan F, De V S C, et al. The PAS fold[J]. European Journal of Biochemistry, 2004, 271(6): 1198-1208.
[7]Nakabone Y, Kawaguchi Y, Kong S G, et al. Photoinduced oligomerization ofArabidopsisthalianaphototropin 2 LOV1[J]. Journal of Physical Chemistry B, 2014, 118(49): 14314-14325.
[8]Salomon M, Christie J M, Knieb E, et al. Photochemical and mutational analysis of the FMN-binding domains of the plant blue light receptor, phototropin[J]. Biochemistry, 2000, 39(31): 9401-9410.
[9]Okajima K. Molecular mechanism of phototropin light signaling[J]. Journal of Plant Research, 2016, 129(2): 1-9.
[10]Takayama Y, Nakasako M, Okajima K, et al. Light-induced movement of the LOV2 domain in an Asp720Asn mutant LOV2-kinase fragment ofArabidopsisphototropin2[J]. Biochemistry, 2011, 50(7): 1174-1183.
[11]Sullivan S, Petersen J, Blackwood L, et al. Functional characterization ofOstreococcustauriphototropin[J]. New Phytologist, 2016, 209(2): 612-623.
[12]Sullivan S, Thomson C E, Lamont D J, et al. InVivophosphorylation site mapping and functional characterization ofArabidopsisPhototropin 1[J]. Molecular Plant, 2008, 1(1): 178-194.
[13]Inoue S I, Shimazaki K I. Blue light-induced autophosphorylation of phototropin is a primary step for signaling[J]. Proceedings of the National Academy of Sciences, 2008, 105(14): 5626-5631.
[14]Christie J M, Yang H, Richter G L, et al. phot1 inhibition of ABCB19 primes lateral auxin fluxes in the shoot apex required for phototropism[J]. Plos Biology, 2011, 9(6): e1001076.
[15]Takemiya A, Sugiyama N, Fujimoto H, et al. Phosphorylation of BLUS1 kinase by phototropins is a primary step in stomatal opening[J]. Nature Communications, 2012, 4(3): 131-140.
[16]Takemiya A, Shimazaki K I.Arabidopsisphot1 and phot2 phosphorylate BLUS1 kinase with different efficiencies in stomatal opening[J]. Journal of Plant Research, 2016, 129(2): 1-8.
[17]D R, UV P, J M, et al. Modulation of phototropic responsiveness inArabidopsisthrough ubiquitination of phototropin 1 by the CUL3-Ring E3 ubiquitin ligase CRL3(NPH3)[J]. Plant Cell, 2011, 23(10): 3627-3640.
[18]乔新荣,陈 琼. 植物向光素信号通路中NPH3蛋白的研究进展[J]. 植物生理学报, 2015(6): 829-834.
[19]Christie J M. Phototropin blue-light receptors[J]. Plant Biology, 2007, 58(58): 21-45.
[20]乔新荣. 烟草PHOT2 基因的克隆及生物信息学分析[J]. 西南农业学报, 2014,27(6): 2635-2638.
[21]Sakai T, Kagawa T, Kasahara M, et al.Arabidopsisnph1 and npl1: Blue light receptors that mediate both phototropism and chloroplast relocation[J]. Proceedings of the National Academy of Sciences, 2001, 98(98): 6969-6974.
[22]Kagawa T, Sakai T, Suetsugu N, et al.ArabidopsisNPL1: a phototropin homolog controlling the chloroplast high-light avoidance response[J]. Science, 2001, 291(5511): 2138-2141.
[23]Huang K, Beck C F. Phototropin is the blue-light receptor that controls multiple steps in the sexual life cycle of the green algaChlamydomonasreinhardtii[J]. Proceedings of the National Academy of Sciences, 2003, 100(100): 6269-6274.
[24]Im C S, Eberhard S, Huang K, et al. Phototropin involvement in the expression of genes encoding chlorophyll and carotenoid biosynthesis enzymes and LHC apoproteins inChlamydomonasreinhardtii[J]. Plant Journal, 2006, 48(1): 1-16.
[25]Zorin B, Lu Y, Sizova I, et al. Nuclear gene targeting inChlamydomonasas exemplified by disruption of the PHOT gene[J]. Gene, 2009, 432(1-2): 91-96.
[26]Trippens J, Greiner A, Schellwat J, et al. Phototropin influence on eyespot development and regulation of phototactic behavior inChlamydomonasreinhardtii[J]. Plant Cell, 2012, 24(11): 4687-4702.
[27]Kianianmomeni A, Hallmann A. Transcriptional analysis ofVolvoxphotoreceptors suggests the existence of different cell-type specific light-signaling pathways[J]. Current Genetics, 2015, 61(1): 3-18.
[28]Kamath B S, Vidhyavathi R, Sarada R, et al. Enhancement of carotenoids by mutation and stress induced carotenogenic genes inHaematococcuspluvialismutants[J]. Bioresource Technology, 2008, 99(18): 8667-8673.
[29]Sandmann G. Carotenoid biosynthesis and biotechnological application[J]. Archives of Biochemistry & Biophysics, 2001, 385(1): 4-12.
[30]Wwiting F. Enhanced astaxanthin production from microalga,Haematococcuspluvialisby two-stage perfusion culture with stepwise light irradiation[J]. Bioprocess and Biosystems Engineering, 2014, 37(10): 1-9.
[31]Han D, Li Y, Hu Q. Astaxanthin in microalgae: pathways, functions and biotechnological implications[J]. Algae, 2013, 28(2): 131-147.
[32]顾洪玲,管 斌,孔 青,等. LED灯的光照对雨生红球藻细胞生长及虾青素积累的影响[J]. 海洋湖沼通报,2014(2):45-51.
[33]韦 韬,顾文辉,李 健,等. 雨生红球藻的光周期效应[J]. 植物学报,2013,48(2):168-173.
[34]Cui H, Yu X, Wang Y, et al. Gene cloning and expression profile of a novel carotenoid hydroxylase (CYP97C) from the green algaHaematococcuspluvialis[J]. Journal of Applied Phycology, 2014, 26(1): 91-103.
[35]Cui H, Yu X, Wang Y, et al. Evolutionary origins, molecular cloning and expression of carotenoid hydroxylases in eukaryotic photosynthetic algae[J]. BMC Genomics, 2013, 14(1): 135-143.
[36]Beel B, Prager K, Spexard M, et al. A flavin binding cryptochrome photoreceptor responds to both blue and red light inChlamydomonasreinhardtii[J]. Plant Cell, 2012, 24(7): 2992-3008.
[37]Toledoortiz G, Huq E, Rodr Guezconcepci N M. Direct regulation of phytoene synthase gene expression and carotenoid biosynthesis by phytochrome-interacting factors[J]. Proceedings of the National Academy of Sciences, 2010, 107(107): 11626-11631.
[38]Nisar N, Li L, Lu S, et al. Carotenoid metabolism in plants[J]. Molecular Plant, 2015, 8(1): 68-82.
[39]Jm C, Swartz T E, Bogomolni R A, et al. Phototropin LOV domains exhibit distinct roles in regulating photoreceptor function[J]. Plant Journal, 2002, 32(32): 205-219.
[40]Glantz S T, Carpenter E J, Melkonian M, et al. Functional and topological diversity of LOV domain photoreceptors[J]. Proceedings of the National Academy of Sciences, 2016, 113: e1442-e1451.
[41]姜 铮,王 芳, 何 湘,等. 蛋白质磷酸化修饰的研究进展[J]. 生物技术通讯,2009,20(2): 233-237.
[42]Suetsugu N, Wada M. Evolution of three LOV blue light receptor families in green plants and photosynthetic stramenopiles: phototropin, ZTL/FKF1/LKP2 and aureochrome[J]. Plant and Cell Physiology, 2013, 54(1): 8-23.
[43]Krauss U, Minh B Q, Losi A, et al. Distribution and phylogeny of light-oxygen-voltage-blue-light-signaling proteins in the three kingdoms of life[J]. Journal of Bacteriology, 2009, 191(23): 7234-7242.
[44]Losi A, Mandalari C, Rtner W. The Evolution and functional role of flavin-based prokaryotic photoreceptors[J]. Photochemistry and Photobiology, 2015, 91(5): 1021-1031.
[45]Masaaki M, Takuji S, Hiroshi T, et al. Serine/threonine protein kinase[M]. Springer Netherlands, 2008.
GeneCloningandBioinformaticsAnalysisofNovelBluePhotoreceptorPhototropinfromGreenAlgaHaematococcuspluvialis
CUI Hong-li1, CHEN Jun2,3, WANG Lin-ping1, HU Zhang-li4, QIN Song2
(1.School of Life Science and Technology, Dalian University, Liaoning Dalian 116622,China; 2.Key Laboratory of Coastal Biology and Biological Resources Utilization, Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Shandong Yantai 264003,China; 3.University of Chinese Academy of Sciences, Beijing 100049, China; 4.College of Life and Marine Science,Shenzhen Key Laboratory of Marine Bioresources and Ecology, Shenzhen University, Guangdong Shenzhen 518060, China)
【Objective】In this study, a full-length complementary DNA (cDNA) sequence of a novel blue photoreceptor phototropin (PHOT), designatedHaephot, was cloned, and the bioinformatics from the green algaHaematococcuspluvialiswas analyzed. 【Method】The homology cloning and rapid-amplification of cDNA ends (RACE) strategies were applied to obtain the sequence ofHaephot, and its sequence was analyzed. 【Result】The cloned cDNA ofHaephotcontained an open reading frame (ORF) of 2139 bp encoding a protein of 712 amino acids, and the deduced protein had a calculated molecular mass of 78.73 kD with an estimated isoelectric point (pI) of 8.66. Multiple sequence alignment analysis revealed that HaePHOT was homologs to known PHOTs, e.g.,Arabidopsisthaliana(58 %) andChlamydomonasreinhardtii(68 %). Conserved domains analysis showed that the typical catalytic motifs of the PHOT were highly conserved in the protein sequences of HaePHOT. The phylogenetic analysis of the PHOTs implied that HaePHOT had higher similarity to the PHOTs from high plants and green algae, which indicated that they share a common ancestor origin. 【Conclusion】The first identified algalphotgene from green algaHaematococcuspluvialiswas obtained, which facilitated the expression and functional investigations of PHOT-mediated signaling pathways involved in astaxanthin biosynthesis regulation.
Phototropin; Full-length of cDNA; Structure domain; Phylogeny and evolution;Haematococcuspluvialis
1001-4829(2017)12-2639-09
10.16213/j.cnki.scjas.2017.12.005
2017-01-04
国家级大学生创新创业训练计划项目(2016112580 38);国家自然基金项目(41376139)
崔红利(1987-),男,山西临汾人,博士,研究方向:微藻功能基因挖掘及利用,E-mail: cuihongli@dlu.edu.cn。
S154.38+4
A
(责任编辑陈 虹)

