PCR鉴定沙门氏菌血清分型方法的建立与应用
赵建梅,李月华,宋传周,赵 格,曲志娜
(1.中国动物卫生与流行病学中心,山东青岛 266032;2.平原县畜牧兽医局王庙镇动检分所,山东德州 253100)
PCR鉴定沙门氏菌血清分型方法的建立与应用
赵建梅1,李月华1,宋传周2,赵 格1,曲志娜1
(1.中国动物卫生与流行病学中心,山东青岛 266032;2.平原县畜牧兽医局王庙镇动检分所,山东德州 253100)
为建立快速有效、经济适用的沙门氏菌血清型鉴定方法,利用PrimerPlex软件设计畜禽中常见沙门氏菌的菌体(O)抗原和鞭毛抗原(H)的引物,优化引物比例和退火温度等反应条件,建立沙门氏菌血清型鉴定的PCR法,并对46株沙门氏菌分离株进行血清型鉴定,同时与传统玻片凝集法和美国CDC的液相芯片(Luminex-xMAP)法进行比较。优化后获得了O抗原中B、C1、D和E1群的多重PCR引物,以及鞭毛抗原H1相抗原基因(fliC)和H2相抗原基因(fljB)的2对引物;建立了一套基于沙门氏菌O和H抗原的多重PCR血清型鉴定法;PCR法和Luminex-xMAP法鉴定出7种沙门氏菌血清型,传统玻片凝集法鉴定出6种血清型;PCR法与传统玻片凝集法和Luminex-xMAP鉴定结果的符合率均为95.65%。本研究建立的PCR沙门氏菌血清型鉴定法简便、快速,预期可在沙门氏菌的血清分型研究中发挥作用。
沙门氏菌;血清型;PCR;鉴定;符合率
沙门氏菌是一种可以引起人和动物腹泻的食源菌,有重要的公共卫生意义。血清型是沙门氏菌研究中重要的分类依据[1-2],是研究沙门氏菌流行情况和公共健康监测的重要参数。及时了解和掌握养殖动物中血清型的分布状况,对研究沙门氏菌的流行态势及其追踪溯源具有重要价值。
菌体抗原(O)、鞭毛抗原(H)和荚膜抗原(Vi)决定了沙门氏菌的血清型[2]。沙门氏菌O和H抗原构成沙门氏菌属绝大部分血清型鉴定的物质基础。rfb基因编码合成多聚糖菌体(O)抗原决定簇,rfb基因群有12个开放阅读框。这些阅读框是编码O抗原的合成和装配的基因。基因组编码序列差异决定了O抗原的多样性。沙门氏菌的fliC和fljB基因分别是编码H1相和H2相鞭毛抗原基因。大约20 000个鞭毛蛋白聚合形成细丝状鞭毛,鞭毛蛋白的末端保守且形成发夹结构,中间部分多样性形成了鞭毛抗原多样性[3]。
本研究参照我国报道的沙门氏菌主要血清型[4-11],设计了菌体抗原中B、C1、D和E1群的编码基因的多重PCR引物,根据鞭毛抗原编码基因末端保守的特点,设计了fliC和fljB基因的PCR引物,建立了一套基于沙门氏菌O和H抗原的多重PCR血清型鉴定方法,并与传统玻片凝集法和美国疾病控制与预防中心(CDC)的液相芯片(Luminex-xMAP)法进行了比较。
1 试验材料和方法
1.1 材料
1.1.1菌株来源 肠炎沙门氏菌(CVCC3276)、猪霍乱沙门氏菌(CVCC3756)、鼠伤寒沙门氏菌(CVCC2222)、鸡伤寒沙门氏菌(CVCC3759)、鸡白痢沙门氏菌(CVCC1790)、大肠杆菌(CVCC25922)、金黄色葡萄球菌(CVCC25923)和空肠弯曲杆菌(ATCC33560):购自中国兽医药品监察所;鸭沙门氏菌(CMCC50774):购自中国生物制品所;小肠结肠耶尔森氏菌(CMCC52207):购自广东省微生物研究所;46株沙门氏菌分离株:由本实验室保存。
1.1.2主要仪器与试剂 PCR扩增仪:购自美国Bio-Rad;Luminex 200检测系统、离心机、涡旋混匀仪、恒温培养箱、xMAP Salmonella serotyping assay kit:购自美国Luminex;沙门氏菌抗血清:购自泰国S&A;沙门氏菌显色培养基:购自郑州博赛科马嘉;2× Go Taq Green mix:购自Promga;Cary-Blair运输培养基、缓冲蛋白胨水(BPW)、亚硒酸盐胱氨酸(SC)增菌液、胰蛋白大豆胨培养基和细菌琼脂粉:购自北京陆桥技术有限公司。
1.2 方法
1.2.1引物设计 从GenBank中下载沙门氏菌代表菌株O抗原B、C1、D和E1群编码基因序列,以及fliC和fljB基因序列。在PrimerPlex软件中设置产物大小的差异、引物和产物的解链温度、5′和3′端最大△G值,以及GC含量等参数,以找出最优引物。
1.2.2DNA模板提取 参照邵碧英等[12]的方法,用煮沸法提取标准菌株和沙门氏菌分离株的细菌DNA用作PCR反应模板。
1.2.3条件优化 O抗原基因的每条引物均配制成20 pmol/μL母液,等比例混合,根据扩增效果对引物的浓度进行调整优化。将2× Go Taq Green mix、多重引物混合物、DNA模板和去离子水配制25 μL的反应体系;扩增的退火温度设置5个梯度,分别为55、52、50、48和45 ℃,按照95 ℃变性、72 ℃延伸的条件进行扩增。fliC和fljB基因扩增:用2× Go Taq Green mix,fliC或fljB基因的上、下游引物混合物,DNA模板,以及去离子水,配制25 μL的PCR反应体系;以95 ℃变性、56 ℃退火和72 ℃延伸为条件,进行PCR扩增。
1.2.4沙门氏菌血清型判定 PCR扩增反应结束后,取5 μL PCR产物进行琼脂糖凝胶电泳,对照DNA Marker判定O抗原相应的群,将H1相和H2相产物送生物公司进行测序。将测序结果输入美国国立生物技术信息中心(NCBI)网站的nucleotide BLAST进行序列比对,根据得分判定相应H抗原。得到O和H抗原后,按照考夫曼-怀特沙门氏菌属抗原表解判定待检沙门氏菌血清型。
1.2.5传统玻片凝集法 参照GB/T 4789.4—2016。
1.2.6Luminex-xMAP法 按 xMAP salmonella serotyping assay kit说明书操作。
1.2.73种鉴定方法的比较 利用建立的PCR鉴定法与传统玻片凝集法和Luminex-xMAP法,分别对46株沙门氏菌分离株血清型进行鉴定,对结果进行比较分析。
2 结果
2.1 引物优化
经PrimerPlex软件中产物大小、解链温度、△G值和GC含量等参数的优化设置,引物序列与扩增片段大小见表1。
2.2 PCR反应条件优化

表1 沙门氏菌血清型O和H抗原PCR引物
经试验确定,O抗原多重PCR扩增最佳退火温度为50 ℃,B、C1、D和E1群引物的最佳比例为1:1:1:2。在25 μL的反应体系中加入2.5 μL多重O抗原基因引物混合物,DNA模板2 μL;扩增条件为95 ℃变性 5 min,35个循环(95 ℃变性30 s,50 ℃退火 30 s,72 ℃延伸 60 s),72 ℃延伸 10 min。
fliC和fljB基因25 μL扩增体系中加入引物混合物1 μL,DNA模板2 μL;扩增反应条件为:95 ℃变性 5 min,95 ℃变性 30 s,56 ℃退火 30 s,72 ℃延伸60 s,循环30次,72 ℃延伸10 min。
O抗原基因扩增出的条带大小如下:鼠伤寒沙门氏菌(B)在216 bp 左右,猪霍乱沙门氏菌(C1)在760 bp 左右,肠炎沙门氏菌、鸡白痢沙门氏菌和鸡伤寒沙门氏菌(D)在385 bp 左右,鸭沙门氏菌(E1)在1 323 bp左右(图1),与引物设计时的预计结果相同。而大肠杆菌、金黄色葡萄球菌、空肠弯曲杆菌和小肠结肠耶尔森氏菌无条带扩增。

图1 沙门氏菌标准株O抗原PCR结果
标准沙门氏菌株的fliC和fljB基因扩增片段送擎科生物科技有限公司测序后进行序列比对。结果分别是鼠伤寒沙门氏菌、猪霍乱沙门氏菌、肠炎沙门氏菌、鸡伤寒沙门氏菌、鸡白痢沙门氏菌和鸭沙门氏菌,与预测结果相同(表2)。

表2 6株沙门氏菌标准株PCR法鉴定沙门氏菌血清型结果
2.3 46株沙门氏菌分离株鉴定
2.3.1PCR法鉴定结果 25株鼠伤寒沙门氏菌、10株肠炎沙门氏菌、4株印第安纳沙门氏菌、4株德尔卑沙门氏菌,以及罗森沙门氏菌、火鸡沙门氏菌和胥伐成格隆沙门氏菌各1株(表3)。
2.3.2传统玻片凝集法 鉴定结果为26株鼠伤寒沙门氏菌、10株肠炎沙门氏菌、4株印第安纳沙门氏菌、4株德尔卑沙门氏菌,以及罗森沙门氏菌和雷根特沙门氏菌各1株。
2.3.3Luminex-xMAP法 将Luminex 200检测的数据整理后,用xMAP Salmonella Serotyping assay软件分析判定46株沙门氏菌分离株血清型。鉴定结果除2株未鉴定出外,与PCR法鉴定结果相同(表3)。
2.3.43种鉴定方法结果比较 PCR法、LuminexxMAP法和传统玻片凝集法鉴定沙门氏菌分离株中O抗原的结果一致;Luminex-xMAP法鉴定S1和S2沙门氏菌分离株的H抗原读数与阴性对照比例不显著,无法判定血清型;S9菌株H1相抗原PCR法和Luminex-xMAP法鉴定为d,而传统玻片凝集法鉴定为i;S46菌株H2相抗原PCR法和LuminexxMAP法鉴定为e,h,而传统玻片凝集法鉴定为f,g。对3种沙门氏菌血清型鉴定法的实验结果进行比对和统计,PCR法和Luminex-xMAP法与传统玻片凝集法结果的符合率均为95.65 %,(表4)。

表3 3种血清型鉴定法的实验结果
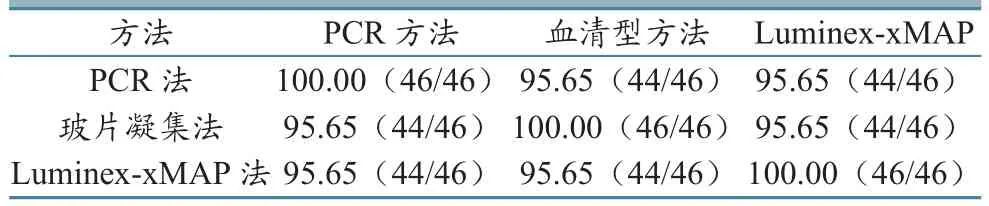
表4 3种沙门氏菌血清型鉴定方法结果符合率单位:%
3 讨论
本研究利用6株沙门氏菌标准株和4株非沙门氏菌标准株对所建立的PCR沙门氏菌血清型鉴定法进行了验证,证明本方法具有良好的特异性和准确性。对46株沙门氏菌分离株进行血清分型鉴定,同时用Luminex-xMAP法和传统玻片凝集法进行鉴定,发现3种鉴定方法之间的符合率均为95.65 %。
由于沙门氏菌O抗原编码基因的多态性和相似性,增加了设计多重引物的难度。在本研究O抗原多重引物设计过程中,就遇到了A和D血清型不能区分的问题。Luk等[2]在沙门氏菌研究中也遇到这个问题。结合畜禽中沙门氏菌血清型A群分布少的实际,本研究就在O抗原基因引物设计中主要参考D群抗原基因进行了多重PCR引物的设计。
试验中,传统玻片凝集法鉴定结果为阳性的菌株,PCR方法均有清晰的条带。从鉴定结果可以看出46株沙门氏菌分离株的O抗原3种鉴定结果相同,PCR法的H相序列Blast结果清晰明确。Luminex-xMAP法中S1和S2号菌株H相读数与阴性对照读数比率不明显,不能判定结果。分析3种鉴定结果存在差异的原因主要有以下几点:一是血清凝集实验结果判读时会因个人的判读标准不同而产生差异;二是与等位基因的分布相关,有研究报道沙门氏菌H相抗原基因,主要是fljB基因的等位基因在相变机制中发生突变而不表达,而这些基因仍在基因组中[13],导致PCR结果假阳性,血清分型不准确;三是在Luminex-xMAP法中H:t等位基因与H:t基因探针不结合而与H:G-complex探针出现反应[14]。基于以上原因,可以将传统玻片凝集法和PCR法相结合,以提高沙门氏菌血清分型的效率和准确率。
已知的沙门氏菌血清型有2 587种[14],传统的血清学鉴定方法操作简便,但血清价格昂贵,鉴定过程步骤多,耗费时间长,需保证250种分型血清的质量,且要鉴定2 587种的沙门血清型,要求实验人员有丰富的经验,即便如此,操作人员的主观判断仍有可能造成误差。鉴于沙门氏菌血清型在流行病学研究中的重要性和传统血清型鉴定方法的不足,研究人员开展了多种方法的研究以弥补传统血清鉴定方法的不足。高通量鉴定方法(如通用探针沙门氏菌血清分型法和PremiTest沙门氏菌分型法)的探针设计较复杂,使用的设备特殊,不易推广。环介导等温扩增(LAMP)方法只适合于单一血清组或血清型的检测[2]。石秀清等[15]以差异位点表为基础建立的血清分型方法鉴定的血清型数量较少,仅为14种。Haim L[16]建立了3个多重PCR体系鉴定伤寒、甲型副伤寒和乙型副伤寒沙门氏菌,Miguel D O等[17]建立了1个6轮realtime PCR反应方法可同时检测6种血清型,Sharon M T等[18]建立了针对肠炎等4种血清型的多重PCR检测系统[16]。方婷子等[13]采用2个三重PCR和3个单重PCR对沙门氏菌进行血清型鉴定,发现结果准确度达到98.70%,证明了PCR法在鉴定沙门氏菌血清型的有效性和准确率。本研究建立的PCR法成本在75元左右,与传统玻片凝集法(成本为35~185美元[14],折合人民币236~1 250元)、MLST(成本少于140元)和PFGE(成本约120元)相比成本低廉,操作人员的主观判断对结果影响小,分离到沙门氏菌即可对其进行血清型分型。这对沙门氏菌血清分型研究,尤其是对广大基层实验室及其工作人员而言,是一种非常适用的工具。
4 结论
本研究是基于动物源沙门氏菌血清型鉴定而建立的PCR法,与其他研究者建立的方法相比,不仅具有反应快速、高效、灵敏和成本低的优点,且理论上可以鉴定589种沙门氏菌血清型;只要有普通PCR仪的实验室就可以开展沙门氏菌血清型的分型鉴定,能满足一般实验室的要求。
[1] 陆承平. 兽医微生物学[M]. 4版,北京:中国农业出版社,2007:107-113.
[2] 李可,方莹,张晓峰,等. 沙门氏菌的血清分型及分子鉴定研究进展[J]. 食品安全质量检测学报,2016,7(10):3947-3951.
[3] CHLOE K B M,TANSY M P,SAHEER E G,et al. Towards the development of a DNA–sequence based approach to serotyping of Salmonella enteric[J].Biomed central microbiology,2004(4):31.DOI:10.1186/1471-2180-4-31.
[4] 赵建梅,王娟,王君玮,等. 山东地区生猪和家禽屠宰环节沙门氏菌血清型分布[J]. 中国食品卫生杂志,2015,27(5):503-508.
[5] 杨保伟,张秀丽,曲东,等. 2007—2008陕西部分零售畜禽肉沙门氏菌血清型和基因型[J]. 微生物学,2010,50(5):654-660.
[6] 杨怀珍,罗薇,孔雪英,等. 大雁、斑头雁和天鹅源沙门氏菌血清型及脉冲场凝胶电泳分型研究[J]. 中国畜牧兽医,2016,43(11):2873-2879.
[7] 梁德媚,许学斌,王伟芳,等. 上海市肉鸽沙门氏菌的分离鉴定及流行血清型和耐药性分析[J]. 中国畜牧兽医,2016(12):3322-3328
[8] 赵志伟,FRANCESCO C,韦平,等. 2009—2010年广西南宁畜禽食品及病禽中沙门氏菌的血清型调查[J].食品科学,2011,32(21):198-200.
[9] 邹明,魏蕊蕊,张纯平,等. 鸡源沙门氏菌的血清型、耐药性和耐药机制调查[J]. 农业生物技术学报,2013(7):855-862.
[10] 黄裕,阚式绂,汪清,等. 深圳市畜禽产品中沙门氏菌血清型与耐药性研究[J]. 食品安全质量检测学报,2016(6):2252-2257.
[11] 刘鲜鲜,王君玮,王娟,等. 山东部分生猪屠宰场屠宰环节沙门氏菌污染状况分析[J].中国动物检疫,2015,32(5):82-86.
[12] 邵碧英、陈彬、汤敏英,等. 沙门氏菌DNA提取及PCR反应条件的优化[J]. 食品科学,2007,28(7):331-334.
[13] 方婷子,史贤明,施春雷. 沙门氏菌血清型快速PCR鉴定方法的建立[J]. 中国食品学报,2017,17(2):212-218.
[14] MCQUISTON J R,WATERS R J,DINSMORE B A,et al. Molecular determination of H antigens of Salmonella by use of a microsphere-based liquid array[J]. Journal of clinical microbiology,2011,49(2):565-573.
[15] 石秀清,周秀娟,施春雷,等. 沙门氏菌属特异性检测靶点碱基差异位点的识别及其血清型决定力的分析[J]. 食品科学,2017(6):1-9.
[16] LEVY H,DIALLO S,TENNANT S M,et al. PCR method to identify Salmonella enterica serovars Typhi,paratyphi A,and paratyphi B among Salmonella isolates from the blood of patients with clinical enteric fever[J].Journal of clinical microbiology,2008,46(5):1861-1866.
[17] MUNOZ N,DIAZOSORIO M,MORCON J,et al.Development and evaluation of a multiplex real-time polymerase chain reaction procedure to clinically type prevalent Salmonella enterica serovars[J]. Journal of molecular diagnostics,2010,12(2):220-225.
[18] TENNANT S M,DIALLO S,LEVY H,et al.Identification by PCR of Non-typhoidal Salmonella enterica serovars associated with invasive infections among febrile patients in Mali[J]. Plos neglected tropical diseases,2010,4(3):e621.
Establishment and Application of PCR Assay for Serotype Identification of Salmonella
Zhao Jianmei1,Li Yuehua1,Song Chuanzhou2,Zhao Ge1,Qu Zhina1
(1. China Animal Health and Epidemiology Center,Qingdao,Shandong 266032,China;2. Wangmiao Animal Inspection Branch,Pingyuan Animal Husbandry and Veterinary Bureau,Dezhou,Shandong 253100,China)
In order to develop a rapid,effective,economic and applicable method for identifying Salmonella serotypes,the primers for O antigen and flagellum antigen(H)of common Salmonella bacteria in livestock and poultry were designed,and PCR method was established by optimizing the concentration of primers and the anneal temperature. Forty-six Salmonella isolates were tested by the PCR method,and the results of which were compared to the traditional agglutination method and the Luminex-xMAP method. After optimization,multi-primers of group B,C1,D,E1 in antigen O were obtained,as well as the antigens H1 and H2 encoded by genes of fliC and fljB. Then a set of multiple PCR method was established on the basis of Salmonella O and H antigens. Results showed 7 serotypes were identified by this method and Luminex-xMAP method,and 6 serotypes were identified by traditional agglutination test. The coincidence rate between traditional agglutination test and Luminex-xMAP and the PCR method was 96.56 %.The PCR assay was simple and rapid,and it would play important role in Salmonella serotype evaluation.
Salmonella;serotype;PCR;identification;coincidence rate
S852.61
B
1005-944X(2018)01-0021-05
10.3969/j.issn.1005-944X.2018.01.021
国家“十二五”科技基础性工作专项(2012FY111000)
同等贡献作者:赵建梅、李月华
曲志娜
杜宪)

