大竹蛏高通量转录组测序数据组装和分析
聂鸿涛, 姜力文, 郑梦鸽, 李东东, 闫喜武
大竹蛏高通量转录组测序数据组装和分析
聂鸿涛1,2, 姜力文1, 郑梦鸽1, 李东东1, 闫喜武1,2
(1.大连海洋大学水产与生命学院,辽宁大连116023;2.辽宁省贝类良种繁育工程技术研究中心,辽宁大连116023)
为进一步开发大竹蛏Solen grandis的基因资源,采用2代Illumina Hi-seq测序技术对大竹蛏的鳃组织进行了转录组测序,构建了转录组数据库,获得338 483 476条Clean Reads数据;拼接组装后获得190 856条Unigene数据,平均长度为1147 bp;与NR、NT、KO、SwissProt、PFAM、GO、KOG等数据库进行Blast信息比对 (E-value为10-5),共获得63 337个注释基因;与NR数据库比对发现,大竹蛏转录组基因序列与长牡蛎Crassostrea gigas具有较高的同源性,为53.3%;将大竹蛏转录组的Unigene的功能通过与KOG数据库进行注释比对划分为25类;GO数据库注释可分为三类,即细胞组分、生物过程和分子功能,共包括65个分支;KEGG分析发现,大竹蛏转录组数据中按照代谢通路可分为92类,利用Blast蛋白库比对和Estscan软件进行ORF预测,获得长度大于300 nt的ORF共50 681个;通过SSR分析,共获得73 089个SSR标记。本研究中获得的转录组信息可为今后进行大竹蛏分子标记的开发和关键基因的克隆及功能分析等研究提供基础数据。
大竹蛏;转录组;高通量测序;生物信息学分析
大竹蛏Solen grandis在分类上隶属于软体动物门、双壳纲、真瓣鳃目、竹蛏科、竹蛏属,分布较为广泛,是中国重要的经济贝类[1]。大竹蛏市场前景较好,经济价值高,因其肉味鲜美,深受消费者喜爱。大竹蛏的出肉率为45%,蛋白质含量高达82.7%[2],必需氨基酸种类齐全,具有较高的营养价值[3]。目前,有关大竹蛏的研究主要集中在人工育苗技术[4-8]、 繁殖生物学[9-11]、 养殖生态学[12-14]和分子生物学[15-17]等方面, 而关于大竹蛏组学方面的研究较少。近年来,高通量转录组测序技术的广泛应用[18],为开展大竹蛏重要经济性状的分子基础研究提供了重要的基因资源。
转录组测序能够在没有基因资源的情况下,通过对某物种的RNA测序分析其生长与代谢规律,揭示基因与生物学特性之间的关系,同时能够有效地开发大量基因资源[18]。该法特别适合基因信息匮乏的物种,并已在长牡蛎、扇贝、菲律宾蛤仔等多个海洋贝类中得到广泛应用。但关于大竹蛏转录组测序的研究还鲜有报道。为了进一步开发大竹蛏的基因资源,本研究中以大竹蛏野生群体为材料,利用Illumina HiSeq 2500技术对大竹蛏进行转录组测序和分析,获得了大竹蛏转录组数据库,得到了与大竹蛏生长、繁殖、代谢、免疫等相关基因资源,并发现了大量的微卫星分子标记 (SSR),旨在为大竹蛏分子标记开发、基因克隆和组学研究提供基础数据,同时也为今后大竹蛏功能基因的研究奠定基础。
1 材料与方法
1.1 大竹蛏的暂养
试验用大竹蛏购于大连市新长兴市场,大竹蛏个体健康、活动性强,壳长为 (9.4±0.39)cm,壳宽为 (1.58±0.45)cm,壳高为 (1.61±0.56)cm,湿质量为 (43.68±0.73)g。试验用海水为黑石礁海区经沉淀、沙滤后的海水,储存备用。试验开始前,先将大竹蛏暂养3 d,暂养期间不投喂,水温为 (12.0±0.3)℃,每天换水1次。用水质分析仪测定海水的盐度和pH,海水pH值为8.0±0.1, 盐度为 32.0±0.2。
1.2 方法
1.2.1 大竹蛏总RNA的提取 采用TRIzol方法提取大竹蛏鳃组织的总RNA,测定RNA浓度和质量。如果OD260nm/OD280nm的范围为1.8~2.2,28S/18S>2, OD260nm/OD230nm>1.8, RNA 完 整 值(RIN)>8.5,则认为提取的RNA合格,符合建库测序标注。
1.2.2 大竹蛏建库测序和拼接组装 大竹蛏转录组的建库测序和拼接组装委托北京诺禾致源生物信息科技有限公司完成,利用Trinity软件[19]进行拼接组装。
1.2.3 基因功能注释 进行基因功能注释的数据库包括 NR、 NT、 PFAM、 KOG/COG、 SwissProt、GO、KEGG[20]。拼接得到的Unigene通过Blastx与基因和蛋白数据库进行比对,得到注释信息,利用WEGO和BlastGO进行GO注释和功能分类统计,分别用COG和KEGG数据库进行Unigene的功能注释和代谢通路的定位。
1.2.4 ORF预测与SSR位点搜索 利用Blast比对到NR和SwissProt蛋白库提取ORF序列,没比对到的用Estscan软件预测已注释Unigene的ORF(Open Reading Frame)。微卫星标记的搜索使用MISA软件,对Unigenes进行SSR重复序列搜索(http://pgrc.ipk-gatersleben.de/misa/misa.htmh/)。
2 结果与分析
2.1 大竹蛏测序数据质量评价及序列组装分析
经HiSeq2500高通量测序获得338 483 476条读序 (clean data),数据量为50.77 G,测序数据已上传至SRA数据库 (PRJNA385244),GC含量为39.60%,Q30值为91.89%。利用Trinity软件对上述获得的clean reads进行组装,得到305 485条transcripts,平均长823 bp,N50为1536 bp;进一步去冗组装获得190 856条Unigenes,平均长度为1147 bp,N50为1875 bp。拼接得到的Unigene的N50为1875 bp。Unigene长度分布见图1。
2.2 Unigene的功能注释
将组装得到的190 856条Unigene与已知的NR、NT、 KO、 SwissProt、 PFAM、 GO、 KOG 这 7个数据库进行功能注释。如表1所示,全部Unigene在7个不同数据库中的同源比对数目不同,分别为 46 657、 7857、 19 044、 34 627、 50 377、50 481和25 168条。至少在一个数据库中获得注释信息的Unigene数量为63 337条,占总Unigene的33.18%;在7个数据库中都得到注释的基因数量为4744条 (表1)。
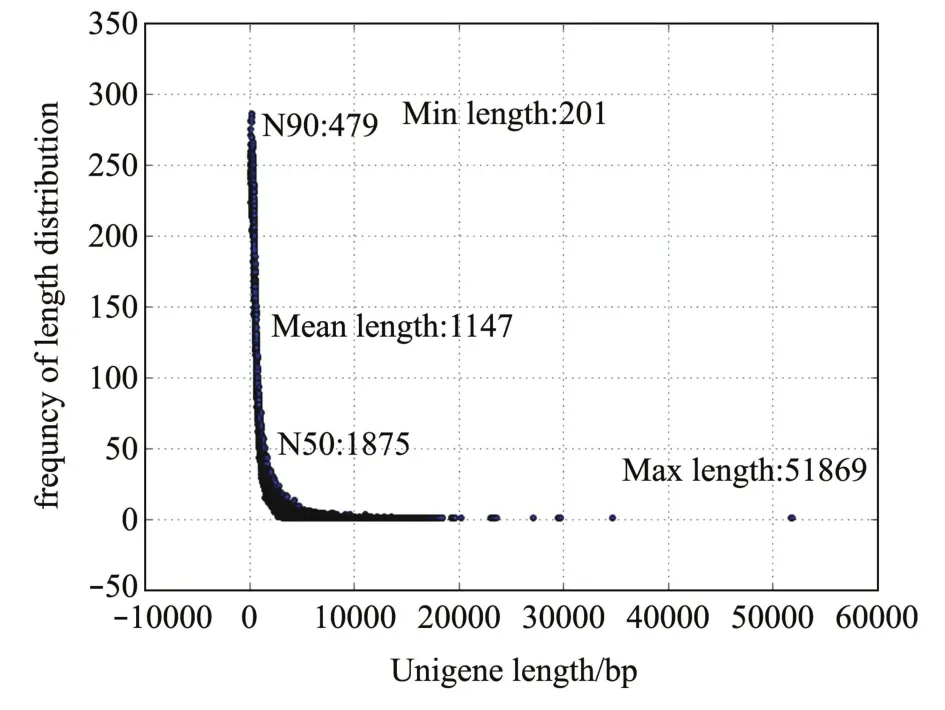
图1 大竹蛏Unigene长度分布Fig.1 Sequence length distribution of unigenes assembled from Illumina reads of the gill transcriptome in grand jacknife clam Solen grandis
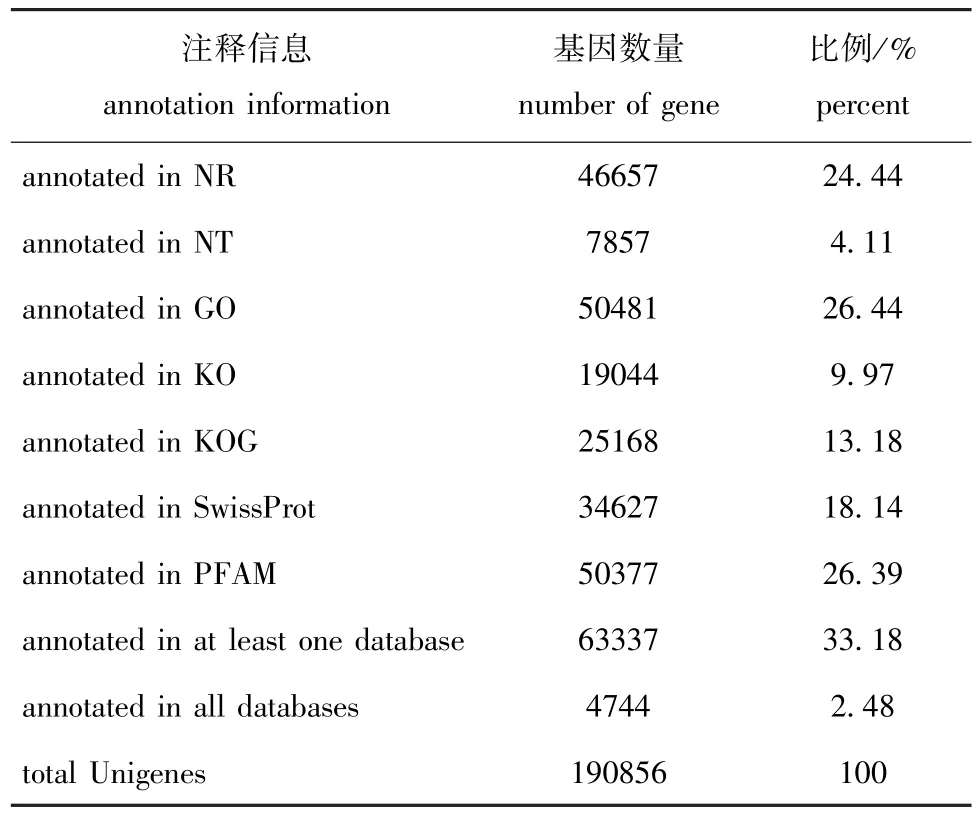
表1 大竹蛏基因注释成功率统计Tab.1 Number of genes annotated in different database in grand jacknife clam Solen grandis
从图2-A可以看出,比对到NR数据库中的46 657条 Unigenes,有 16.4%的 E值分布在 le-15~1e-5, 14.4%分布在 le-30~1e-15, 9.7%分布在 le-45~1e-30, 7.9%分布在 le-60~1e-45,15.4%分布在 le-100~1e-60, 17.8%分布在0~1e-100。从图2-B可以看出,从匹配的物种来源分析,大竹蛏Unigene注释到长牡蛎Crassostrea gigas的占 53.3%,注释到帽贝Lottia gigantea的占11.7%,注释到加州海兔Aplysia californica的占8.0%,在紫海胆Strongylocentrotus purpura和文昌鱼Branchiostoma floridae中注释到的Unigene均占1.9%,其余23.2%注释到其他物种。
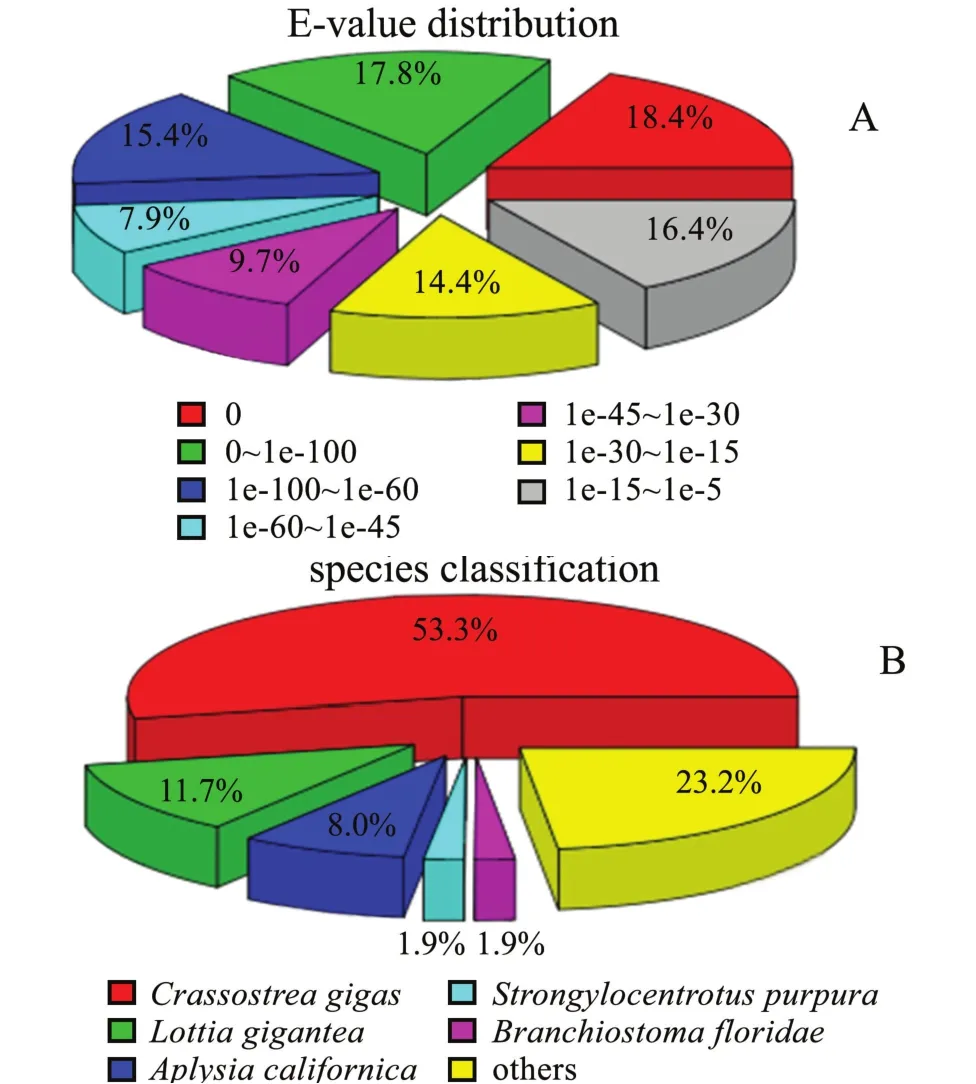
图2 NR注释的E-value分布和物种分布Fig.2 E-value distribution and species classification of sequences matched to the NR database
2.3 Unigene的功能分类
在基因的GO注释中,有50 481条Unigene(26.44%)获得GO数据库注释 (图3)。注释到的3大类包括:细胞组分分为20个亚类,生物过程分为25个亚类,分子功能分为10个亚类。从图3可见:生物过程获得的注释信息最多,为146 264条Unigene,占全部注释信息的76.64%,生物过程里涉及代谢过程、细胞过程和对刺激的应激效应的Unigene比较多,分别有23 801、29 427、9526条;其次是细胞组分,分为20个亚类,共有87 560条Unigene,占全部注释信息的45.88%,涉及到细胞部分、膜、细胞和细胞器的Unigene比较多,分别为15 763、11 013、15 763和10 280条;注释信息相对最少的是分子功能,共有58 699条Unigene(30.76%),排在前 2位的是结合与催化活性,Unigene数量分别为 27 847、19 094条。另外,KOG数据库功能注释结果显示,KOG分类中得到25种,包括25 168条Unigene,其中,信号转导机制包含Unigene最多 (5884条),其次是一般功能预测 (5569条),转录后修饰、蛋白折叠和分子伴侣 (2502条),胞内运输、分泌和囊泡运输 (1252条),转录 (1232条),细胞骨架 (1232条)。
2.4 Unigene代谢途径分析
在KEGG数据库中分析了190 856条Unigene,得到注释的Unigene有30 914条 (16.2%),KEGG代谢途径有230种。其中,细胞黏附的Unigene数量最多 (568条),其次是 Rap1信号通路 (512条)、涉及内吞作用 (482条)、PI3K-Akt信号通路 (472条)、催产素信号通路 (417条)、Ras信号通路 (403条)、cAMP信号通路 (402条)、嘌呤代谢 (293条)等代谢通路。
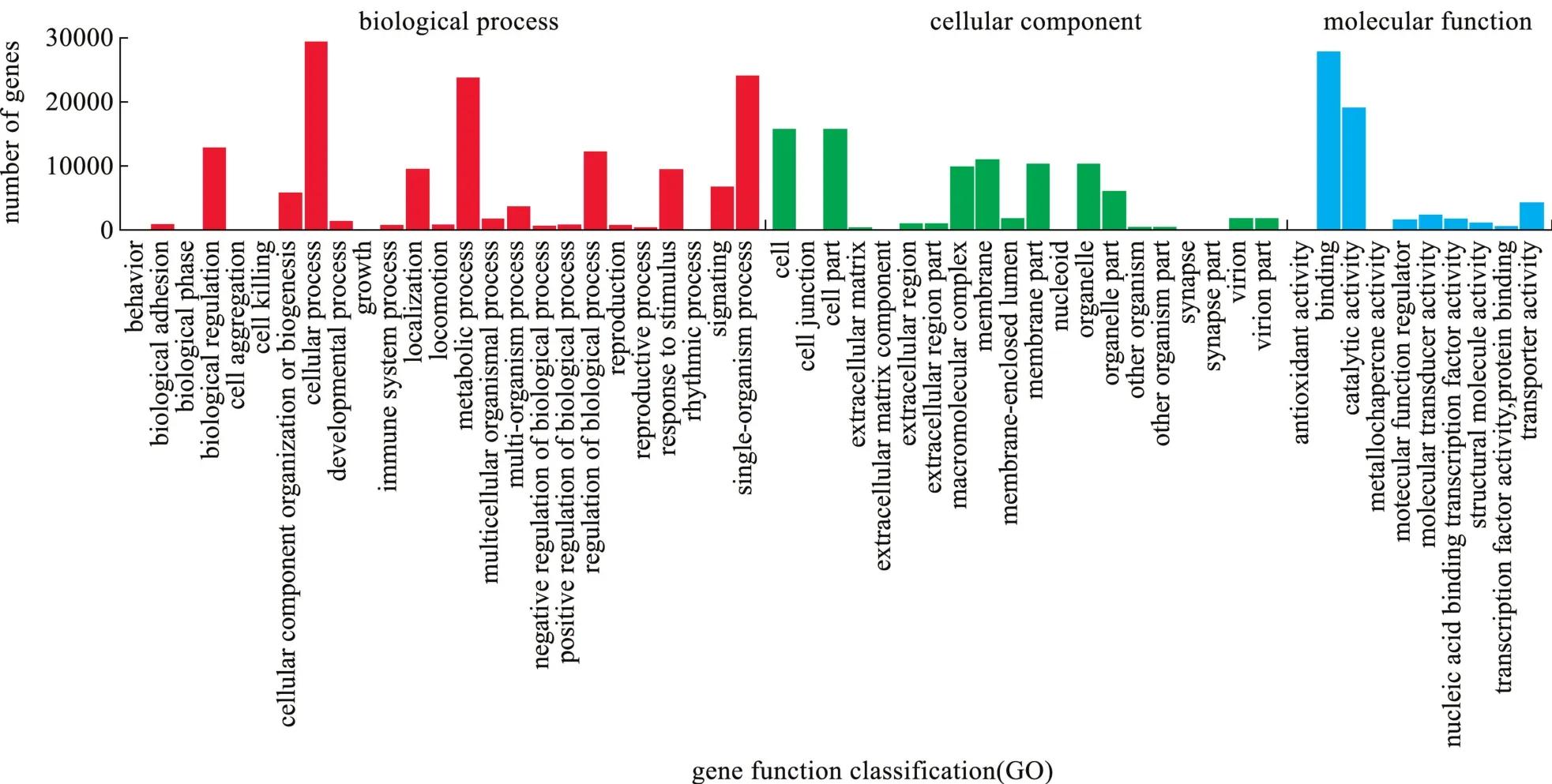
图3 大竹蛏转录组Unigene GO功能分类Fig.3 GO(Gene Ontology)categorization(biological process, cellular component and molecular function)of the unigenes in the gill transcriptome of grand jacknife clam Solen grandis
2.5 ORF预测与SSR位点分析
利用Blast比对到NR和SwissProt蛋白库提取ORF序列,没比对到的用Estscan预测Unigene,得到ORF共86 152个,其中,长度在300 nt以上的Unigene数量为50 681条,占58.83%(图4)。同时,利用MISA软件搜索到微卫星标记73 089个,其中单碱基微卫星最多,单碱基型 (50 676个,占69.33%),其他类型依次为二碱基型重复(15 449个,占21.14%)、三碱基型 (5692个,占7.79%)、四碱基型 (1250个,占1.71%),五碱基型和六碱基型最少,分别为20、2个,各占0.027%和0.003%(表2)。对大竹蛏微卫星位点的分析,可为大竹蛏分子标记的开发与应用研究提供重要资源。
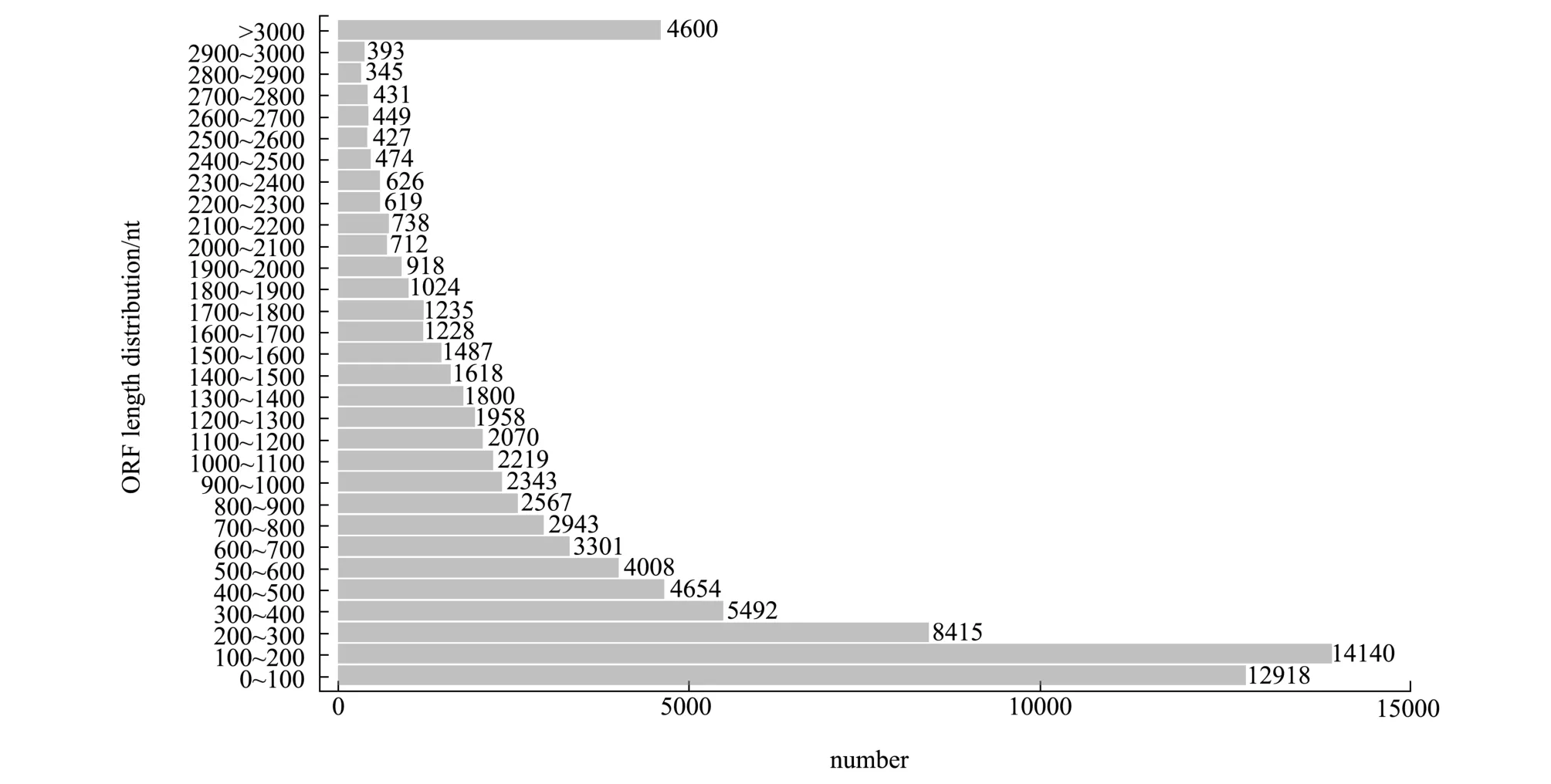
图4 大竹蛏ORF长度分布Fig.4 ORF length distribution in the gill transcriptome of grand jacknife clam Solen grandis
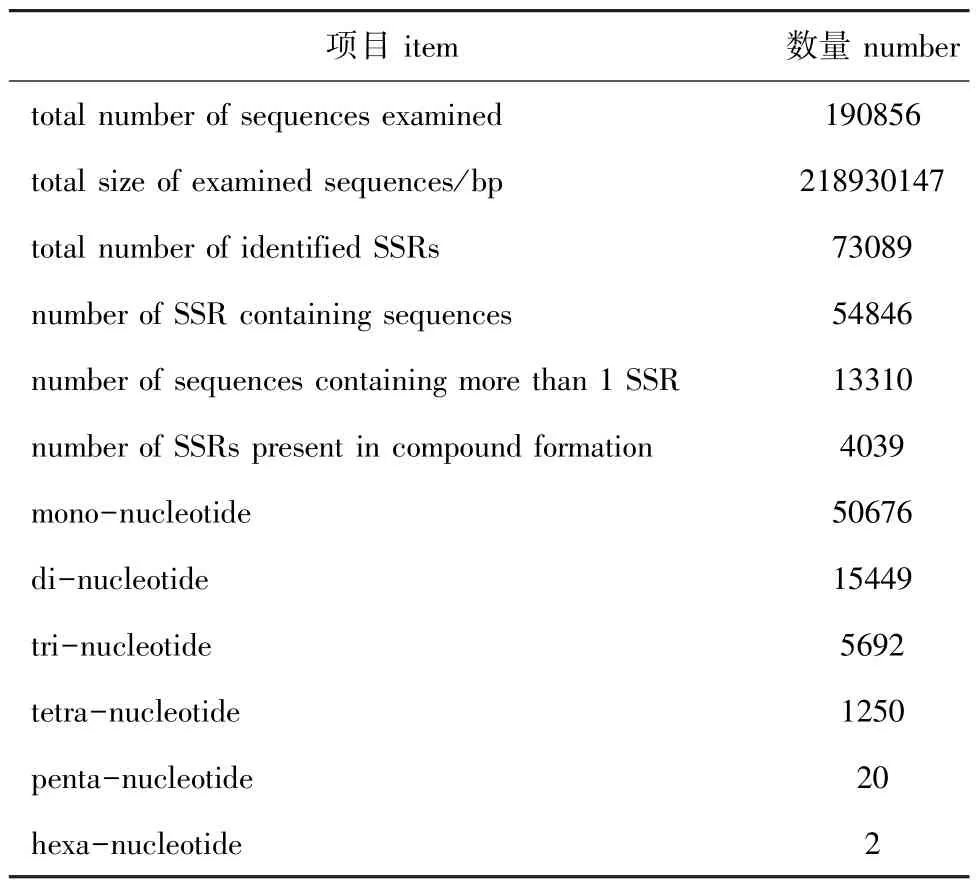
表2 大竹蛏SSR分析统计结果Tab.2 Number of SSR marker in the gill transcriptome of grand jacknife clam Solen grandis
3 讨论
3.1 大竹蛏鳃组织转录组组装与注释
近年来,高通量测序技术已在许多水产动物的转录组研究中得到广泛应用,包括鱼类[21-23]、贝类[24-26]和虾蟹类等[27-29]。 本研究中, 采用高通量测序技术对大竹蛏的鳃组织进行转录组测序,拼接组装后获得190 856条Unigene,平均长度为1147 bp,其中有65 683条Unigenes的长度在1 kb以上。所有的Unigene有63 337条 (33.18%)成功获得功能注释。但仍有127 519条 (66.82%)序列定位不清楚,原因可能是基因数据库中大竹蛏基因资源少,导致得到功能注释基因偏少。对拼接组装获得的190 856条Unigenes进行了代谢途径分析和功能分类。KOG功能注释包括25 168条Unigenes,分为25个KOG亚类。本文中获得了大竹蛏转录组数据库,得到了与大竹蛏生长繁殖、合成代谢与免疫抗逆等相关基因资源,完善了大竹蛏的基因数据库信息,为今后大竹蛏功能基因的研究奠定了基础。
3.2 大竹蛏鳃组织转录组基因资源
近年来,有越来越多的水产经济动物转录组和基因组测序结果的发表,基因数据库中的资源得到不断地丰富和完善。在基因资源非常充足的情况下,很多未知功能的基因将被注释到具体的功能,因此,可以利用这个途径来挖掘一些新的功能基因。鳃是贝类重要的呼吸和滤食器官,研究表明,鳃在贝类的免疫方面也发挥重要作用[30-31]。转录组研究可以进行不同组织测序数据的比较分析,但本研究中选取了大竹蛏的鳃组织进行建库测序,通过基因功能注释发现了一些重要的免疫基因,如凋亡抑制因子 (Inhibitors of apoptosis,IAP)、肿瘤坏死因子 (Tumor necrosis factors,TNF)和补体 C3(complement component C3)等。本研究结果不仅为今后的基因克隆和功能研究提供了重要的基础数据,同时为研究大竹蛏生长、免疫、繁殖、代谢等途径中功能基因的分子机制奠定基础。
3.3 大竹蛏转录组微卫星分子标记
与传统测序方法相比,高通量测序技术具有操作简单、通量大、效率高等优点。能够快速地获得成千上万条基因序列,并开发出大量的微卫星分子标记资源[32]。
本研究中,利用MISA微卫星搜索软件对大竹蛏转录组基因序列进行搜索,共发现微卫星分子标记73 089个。在得到的6种微卫星重复类型中,单碱基型重复数量最多,为50 676个,占所有微卫星标记的69.33%,这与对栉孔扇贝的研究结果类似[33]。目前,关于大竹蛏微卫星标记开发的报道较少,在大竹蛏中仅有少量的微卫星标记可以利用。因此,有必要进一步丰富大竹蛏的微卫星标记资源。
本研究中进行了大竹蛏转录组的初步探究,大竹蛏的基因数据库资源得到了补充和完善,为今后开展大竹蛏分子遗传学和组学方面的相关研究奠定了重要基础。本研究中获得了大量的大竹蛏微卫星候选标记,可为今后大竹蛏微卫星分子标记开发、遗传连锁图谱构建、家系分析和系谱鉴定、物种鉴定以及群体遗传学等研究提供重要资源。
[1] 齐钟彦.中国经济软体动物[M].北京:中国农业出版社,1998:233-234.
[2] 戴聪杰.大竹蛏软体部分营养成分分析及其评价[J].集美大学学报:自然科学版,2002,7(4):304-308.
[3] 戴聪杰.大竹蛏软体部分的氨基酸组成分析[J].莆田学院学报,2002,9(3):32-35.
[4] 陈爱华,姚国兴,张志伟.大竹蛏生产性人工繁育试验[J].海洋渔业,2009,31(1):66-72.
[5] 宋贤亭,于瑞海,马培振,等.大竹蛏室内人工育苗技术研究[J].海洋湖沼通报,2015(4):56-60.
[6] 王雪梅,路宜华,丰爱秀,等.大竹蛏健康苗种培育新模式的研究[J].水产养殖,2012,33(8):14-16.
[7] 侯和要,牟乃海,宋全山,等.大竹蛏人工繁殖技术研究[J].齐鲁渔业,2004,21(6):32-35.
[8] 杨辉.大竹蛏人工育苗技术研究[J].河北渔业,2015(5):41-69.
[9] 肖国强,柴雪良,邵艳卿,等.大竹蛏的繁殖生物学[J].海洋科学,2009,33(10):21-25.
[10] 吴杨平,陈爱华,姚国兴,等.大竹蛏胚胎发生及稚贝发育基本特征[J].动物学杂志,2012,47(4):74-81.
[11] 李碧全,陈东辉,郑杰民,等.大竹蛏性腺观测与人工催产技术的研究[J].集美大学学报:自然科学版,2011,16(2):97-102.
[12] 侯和要,王君霞,彭作波,等.不同盐度对大竹蛏存活的影响[J].齐鲁渔业,2004,21(5):5-6,4.
[13] 陈爱华,姚国兴,张志伟,等.温度、盐度和底质对大竹蛏稚贝生长及存活的影响[J].热带海洋学报,2010,29(5):94-97.
[14] 陈爱华,吴杨平,姚国兴,等.底质和温度对大竹蛏苗种生长存活的复合影响[J].江苏农业科学,2012,40(5):208-210.
[15] 张滔,刘相全,孙国华,等.大竹蛏AFLP分子标记反应体系的建立与优化[J].安徽农业科学,2011,39(29):17923-17926.
[16] 杨顶珑,韦秀梅,杨建敏,等.大竹蛏(Solen grandis)铁蛋白基因的克隆及其在转录水平上对微生物多糖的应答[J].海洋与湖沼,2013,44(3):664-669.
[17] Yuan Y,Li Q,Kong L F,et al.The complete mitochondrial genome of the grand jackknife clam,Solen grandis(Bivalvia:Solenidae):a novel gene order and unusual non-coding region[J].Molecular Biology Report,2012,39(2):1287-1292.
[18] Sánchez C C,Weber G M,Gao G T,et al.Generation of a reference transcriptome for evaluating rainbow trout responses to various stressors[J].BMC Genomics,2011,12:626.
[19] Grabherr M G,Haas B J,Yassour M,et al.Full-length transcriptome assembly from RNA-Seq data without a reference genome[J].Nature Biotechnology,2011,29(7):644-652.
[20] Kanehisa M,Goto S.KEGG:Kyoto encyclopedia of genes and genomes[J].Nucleic Acids Research,2000,28(1):27-30.
[21] Ju Z,Dunham R A,Liu Z.Differential gene expression in the brain of channel catfish(Ictalurus punctatus)in response to cold acclimation[J].Molecular Genetics and Genomics, 2002, 268(1):87-95.
[22] Long Y,Li L C,Li Q,et al.Transcriptomic characterization of temperature stress responses in larval zebrafish[J].PLoS One,2012,7(5):e37209.
[23] Mininni A N,Milan M,Ferraresso S,et al.Liver transcriptome a-nalysis in gilthead sea bream upon exposure to low temperature[J].BMC Genomics,2014,15:765.
[24] Zhang L L,Li L,Zhu Y B,et al.Transcriptome analysis reveals a rich gene set related to innate immunity in the eastern oyster(Crassostrea virginica)[J].Marine Biotechnology,2014,16(1):17-33.
[25] Milan M,Coppe A,Reinhardt R,et al.Transcriptome sequencing and microarray development for the Manila clam,Ruditapes philippinarum:genomic tools for environmental monitoring[J].BMC Genomics,2011,12:234.
[26] Shi M J,Lin Y,Xu G R,et al.Characterization of the Zhikong scallop(Chlamys farreri)mantle transcriptome and identification of biomineralization- related genes[J].Marine Biotechnology,2013,15(6):706-715.
[27] Zhao Q,Pan L Q,Ren Q,et al.Identification of genes differentially expressed in swimming crab Portunus trituberculatus response to low temperature[J].Aquaculture,2015,442:21-28.
[28] Shekhar M S,Kiruthika J,Ponniah A G.Identification and expression analysis of differentially expressed genes from shrimp(Penaeus monodon)in response to low salinity stress[J].Fish&Shellfish Immunology,2013,35(6):1957-1968.
[29] Chen K,Li E C,Xu T Y,et al.Transcriptome and molecular pathway analysis of the hepatopancreas in the Pacific white shrimp Litopenaeus vannamei under chronic low-salinity stress[J].PLoS One,2015,10(7):e0131503.
[30] Wei X M,Yang J M,Liu X Q,et al.Identification and transcriptional analysis of two types of lectins(SgCTL-1 and SgGal-1)from mollusk Solen grandis[J].Fish & Shellfish Immunology,2012,33(2):204-212.
[31] Nie H T,Liu L H,Huo Z M,et al.The HSP70 gene expression responses to thermal and salinity stress in wild and cultivated Manila clam Ruditapes philippinarum[J].Aquaculture,2017,470:149-156.
[32] Li H J,Liu M,Ye S,et al.De novo assembly,gene annotation,and molecular marker development using Illumina paired-end transcriptome sequencing in the clam Saxidomus purpuratus[J].Genes & Genomics,2017,39(6):675-685.
[33] 赵柏淞.栉孔扇贝(Chlamys farreri)BAC文库的构建及其基因组特征分析[D].青岛:中国海洋大学,2013.
Transcriptome data assembly and analysis of grand jackknife clam Solen grandis through high-throughput sequencing
NIE Hong-tao1,2, JIANG Li-wen1, ZHENG Meng-ge1, LI Dong-dong1, YAN Xi-wu1,2
(1.College of Fisheries and Life Science, Dalian Ocean University, Dalian 116023, China; 2.Engineering Research Center of Shellfish Culture and Breeding in Liaoning Province, Dalian 116023, China)
The Illumina Hi-seq sequencing technology was used to sequence the transcriptome in gills of grand jackknife clam Solen grandis and the data were analyzed by bioinformatics method to study the transcriptome of the grand jackknife clam.The transcriptome library of grand jackknife clam was found to contain 338 483 476 reads,and 190 856 Unigenes were obtained by assembling the Scaffolds in the transcriptome library,with an average length of 1147 bp.A total of 63 337 annotated genes were obtained compared with NR, KO, SwissProt, PFAM,GO, KOG and other databases for Blast(E-value 10-5).Comparing with the NR database, we found that the gene sequence in grand jackknife clam had a high homology(53.3%)with the Pacific oyster(Crassostrea gigas).Unigenes in the transcriptome of grand jackknife clam was divided into 25 classes according to the function by comparing Unigene with the KOG database.GO annotations was divided into 3 major categories:biological processes,cellular components and molecular functions including a total of 65 branches.The KEGG database revealed that the transcriptome data of grand jackknife clam was divided into 92 types according to the metabolic pathway.The ORF were predicted by Blast with protein database and estscan, 50 681 ORF with the length of more than 300 nt, and total of 73 089 SSR markers was found through repeat sequence motifs search and analysis.The transcriptome information in this study can provide the gene resources for cloning and functional analysis and molecular markers development of grand jackknife clam in the future.
Solen grandis; transcriptome; high-throughput sequencing; bioinformatics analysis
Q142;S968.3
A
10.16535/j.cnki.dlhyxb.2017.06.004
2095-1388(2017)06-0658-06
2017-03-30
现代农业产业技术体系建设专项 (CARS-48);辽宁省农业领域青年科技创新人才项目 (2014004);大连市科技计划项目(2014B11NC092,2016RQ065)
聂鸿涛 (1984—),男,博士,副研究员。E-mail:htnie@dlou.edu.cn
闫喜武 (1962—),男,博士,教授。E-mail:yanxiwu@dlou.edu.cn

