青海巨型住肉孢子虫线粒体cox1基因的克隆及进化分析
巨金玲 , 张洪波 , 陈 刚 , 康 明
(1.青海大学农牧学院 , 青海 西宁 810016 ; 2.青海省畜牧兽医科学院 , 青海 西宁 810016)
青海巨型住肉孢子虫线粒体cox1基因的克隆及进化分析
巨金玲1, 张洪波2, 陈 刚1, 康 明1
(1.青海大学农牧学院 , 青海 西宁 810016 ; 2.青海省畜牧兽医科学院 , 青海 西宁 810016)
为了研究青海省巨型住肉孢子虫线粒体细胞色素C氧化酶第一亚基(cox1)基因部分序列(pcox1)的遗传变异情况,并用pcox1序列构建巨型住肉孢子虫与其他住肉孢子虫的种群遗传关系。利用PCR技术扩增巨型住肉孢子虫的pcox1,将测序所获序列进行比对,并进行同源性分析和系统发育树的构建。结果显示,所获得的pcox1序列长度均为968 bp,与GenBank已公布的巨型住肉孢子虫相似度为99.1% ~ 99.8%。系统发育树表明,所有分离株与已知巨型住肉孢子虫位于同一分支。巨型住肉孢子虫pcox1序列种内相对保守(0.2%~0.7%),种间差异较大(6.8%~20.8%),可用作种间遗传变异研究的标记。
巨型住肉孢子虫 ; 藏羊 ; 线粒体DNA ;cox1基因 ; 种系发育关系
住肉孢子虫是一类细胞内的寄生性原虫,广泛发现于爬行类、鸟类和包括猴、鲸、各种家畜在内的哺乳动物,能引起家畜发热、贫血、消瘦、共济失调、母畜流产、严重者甚至死亡[1]。本属虫体的最后一代裂殖体进入横纹肌或心肌发育为肉孢子虫包囊[2],肌肉因大量包囊的寄生而变色变性使肉品废弃,给畜牧业带来巨大的经济损失,曾在新疆、青海、甘肃等地报道的绵羊住肉孢子虫感染率达90.7%~100%[3]。因此,准确鉴别住肉孢子虫不仅具有重要的学术价值和意义,而且对住肉孢子虫病的预防和控制也具有重要的意义。
传统的形态学鉴定对住肉孢子虫准确定种有一定的局限性,而利用分子遗传标记,通过研究其种群变异性对寄生虫进行分类鉴定,则能获得更加满意的结果。线粒体DNA分子量较小,结构较稳定,在细胞中含量丰富,且无组织特异性;呈现严格的母系遗传特性,在世代传递中不发生基因重组;具有较快的进化速率,约为单拷贝核基因的5~10倍,用作遗传学分析灵敏度高[4-7]。其中线粒体细胞色素C氧化酶第一亚基(cox1)基因作为线粒体基因中比较保守的基因,在寄生虫的分类鉴定和群体遗传方面,被认为是理想的遗传标记[8-9]。本研究旨在分析青海藏羊巨型住肉孢子虫线粒体cox1基因部分序列(pcox1)的遗传变异情况,并用pcox1序列构建巨型住肉孢子虫与其他住肉孢子虫的种群遗传进化树,从而明确巨型住肉孢子虫线粒体cox1基因能否成为理想的种间遗传标记。
1 材料与方法
1.1 主要试剂 血液/细胞/组织基因组DNA提取试剂盒、6×DNA loading Buffer、2×TaqPCR MasterMix、ddH2O、5×TBE、核酸染料、DL-2 000 DNA Marker,均为天根生化科技(北京)有限公司产品。
1.2 虫体样品 本试验的10个巨型住肉孢子虫样品均采自青海西宁藏羊食道内,为白色米粒状虫体,虫体大小不一,经形态学鉴定均为巨型住肉孢子虫。
1.3 样品DNA提取 将虫体用双蒸水反复吹打冲洗后置于1.5 mL灭菌离心管中,用匀浆机研磨虫体,加入200 μL缓冲液GA,震荡至彻底悬浮,加入20 μL Proteinase K,混匀后于56 ℃电浴锅消化3 h,消化好的虫体悬液按天根生化科技(北京)有限公司的DNA提取试剂盒说明书提取虫体DNA,DNA样品于-20 ℃冰箱中保存备用。
1.4 目的基因片段扩增及测序 根据参考文献[10],由华大基因科技服务有限公司合成引物,上游引物SF1:5'-ATGGCGTACAACAATCATAAAGAA-3',下游引物SR4:5'-CCACACCTGTAGTACCDCC-3'。反应体系为50 μL:模板DNA 6 μL,上、下游引物各1 μL,2×PCR Master Mix 25 μL,再加入ddH2O 17 μL,按下列条件进行PCR扩增:95 ℃预变性15 min;94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸90 s,45个循环;72 ℃延伸10 min。将PCR产物在1%琼脂糖凝胶电泳,并在紫外投射仪下观察结果,用凝胶成像系统摄像。PCR产物切胶回收后,将回收产物与T载体连接、转化、克隆后,将鉴定为阳性的菌液送至生物公司测序。
1.5 序列分析 将测序所得序列与 GenBank 中收录的住肉孢子虫cox1序列信息(见表1)进行比较分析,结合形态学鉴定定种。利用 Clustalx 1.83软件对测定序列和同源序列在默认设置下进行比对,并对比对结果进行眼观调整,将5′端和3′端剪辑对齐,导入GeneDoc软件,生成多重序列对比图,分析样品的相似性及碱基位点变异。用DNASTAR中的MegAlign程序将样品序列及GenBank中已收录的住肉孢子虫pcox1序列进行分析,比较其相似性和差异性。
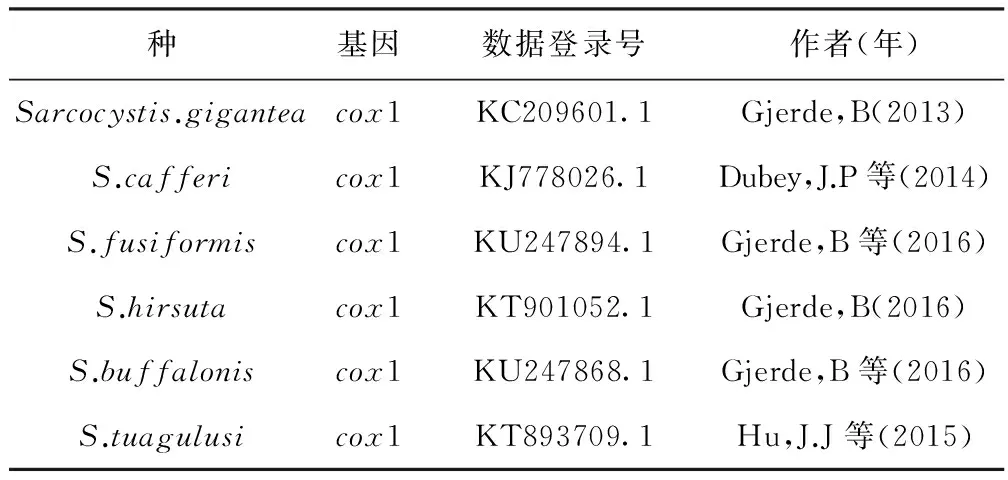
表1 不同住肉孢子虫的cox1序列信息
1.6 系统发育树的构建 选择进化距离较远的Toxoplasmagondii(龚地弓形虫)作为外群,利用生物软件MEGA5.1,以邻接法(Neighbor-Joining)对pcox1序列构建系统发育树,并采用Bootstraping法(重复1 000次)对构建的系统发育树进行评估。
2 结果
2.1 PCR扩增结果 10个样品均成功扩增出约1 000 bp的条带(图1),与预期大小相符,且无非特异性条带,表明成功扩增出目的基因。

图1 巨型住肉孢子虫pco×1扩增产物的琼脂糖电泳分析
M:DL-2 000 DNA Marker; 1-10:XN1-XN10; 11:阴性对照
2.2 测序结果及分析
2.2.1 10个样品的pcox1碱基序列长度相同,均为968 bp,经ClustalX软件转换格式后,导入GeneDoc软件,分析显示每个样品cox1序列均小于8个碱基位点变异,序列高度相似。
2.2.2 样本间序列相似性较高,为99.3%~100%,与GenBank收录的巨型住肉孢子虫基因序列(基因登录号:KC209601.1)高度相似,相似度为99.1%~99.8%。而与其他住肉孢子虫的相似性都在84.3%以下,种内差异性(0.2%~0.7%)远小于种间差异性(6.8%~20.8%)(图2)。

图2 住肉孢子虫pcox1序列差异的成对比较(对角线以上为相似率,以下为差异率)
1-10:XN1-XN10;11-16:S.gigantea、S.cafferi、S.fusiformis、S.hirsuta、S.buffalonis、S.tuagulusi
2.3pcox1的系统发育分析 种系发育显示:10个青海分离株具有高度相似性,与GenBank中已报道的巨型住肉孢子虫(S.gigantea)位于同一分枝,系统发生树中的Bootstrap值较高,并且青海分离株所属分枝与其他住肉孢子虫所属分枝相隔较远,使样本得到了很好的鉴别。
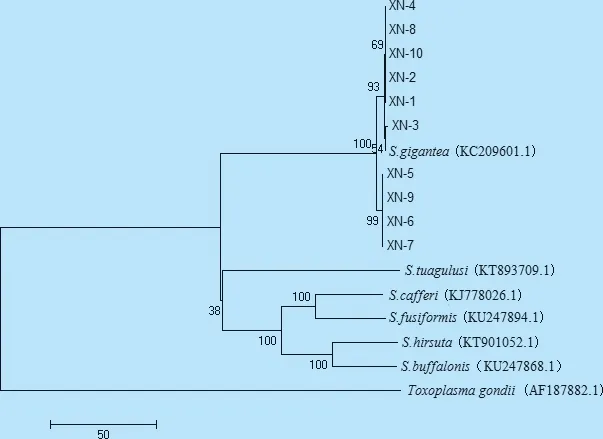
图3 以Toxoplasma gondii外群,用N-J法构建的系统发育树
3 讨论
线粒体 DNA 由于其自身在结构和功能上具有诸多优势,已经成为当前研究生物物种遗传多样性和系统发育的重要的分子标记之一。其中cox1基因是线粒体DNA中进化速率较快且相对保守的基因,本试验选用线粒体cox1基因作为分子标记,对形态学鉴定为巨型住肉孢子虫的虫体进行了分子生物学的分析,这样较大程度地保证了结果的准确性和客观性。选用引物SF1和SR4扩增线粒体cox1基因片段,是因为这一片段已被证明具有种属特异性[10]。试验结果显示,各青海分离株相应序列具有很高的相似性,碱基位点变异均小于8,与GenBank中已公布的巨型住肉孢子虫有很高的相似性,相似性在99.1%~99.8%之间。而与其他住肉孢子虫相应序列的相似性都在84.3%以下,青海分离株cox1基因序列种内差异为0.2%~0.7%,种间差异为6.8%~20.8%,由于pcox1序列种内相对保守,种间差异较大,故能为种间的遗传变异提供遗传标记,用于巨型住肉孢子虫的鉴定,这一结论可从国外学者Gjerde, B对住肉孢子虫cox1基因序列分析得出的结论获得有力的支持[10]。
采用N-J法构建的进化树显示出10个青海分离株位于同一大分枝,与巨型住肉孢子虫同在一个分支,而与其他住肉孢子虫所属分枝相隔较远,且系统发生树中的Bootstrap值较高,再次很好地证明了研究结果中所确定的分类地位。综合上述结果,确定所采青海藏羊住肉孢子虫为巨型住肉孢子虫(S.gigantea)。
试验结果表明,巨型住肉孢子虫线粒体cox1基因是比较保守的基因,适宜于种间及种下的分类鉴别和系统发育分析,可作为理想的种间分子遗传标记用于巨型住肉孢子虫的鉴定。试验结果验证了线粒体cox1基因在进化生物学研究中的可行性和可靠性,即通过线粒体cox1基因序列测定及分析可对易与该虫种混淆的虫体做出正确的分类与鉴别。同时试验结果也为住肉孢子虫的分子分类、群体遗传和系统发生规律方面提供了一定的科学依据。
[1] 汪明,孔繁瑶,肖兵南.家畜住肉孢子虫病研究进展[J].中国兽医杂志,1999,25(7):38-40.
[2] 汪明.兽医寄生虫学[M].三版.北京:中国农业出版社,2011:70-71.
[3] 韩秀敏.青海省住肉孢子虫病的流行及防治现状[J].青海畜牧兽医杂志,2008,38(4):31.
[4] 刘业发.线粒体基因揭示狼的高原适应性[D].山东:曲阜师范大学,2015.
[5] 李建华,王继文.动物线粒体DNA在进化遗传学研究中的应用[J].生物学通报,2005,40(2):5-7.
[6] WM Brown M J,George A C,Wilson.Rapid evolution of animal mitochondrial DNA[J].Proceedings of the National Academy of sciences,1979,76(4):1 967-1 971.
[7] Olivo P D,Walle MJVD,Laipis P J.Nucleotide sequence evidence for rapid genotype shifts in the bovine mitochondrial DNA D-loop[J].Nature,1983, 306(5941):400-402.
[8] RafalKolenda,Maciej Ugorski,Michal Bednarski.Molecular characterization of Sarcocystis species from olish roe deer based on ssu rRNA and cox1sequence analysis[J].Parasitology Research,2014,113(8):3 029-3 039.
[9] Gjerde B,Hilali M,Mawgood S A.Molecular characterisation of three regions of the nuclear ribosomal DNA unit and the mitochondrial cox1 gene of Sarcocystis fusiformis from water buffaloes (Bubalus bubalis) in Egypt[J].Parasitol Res,2015,114(9):3 401-3 413.
[10] Gjerde B.Phylogenetic relationships amongSarcocystisspecies in cervids, cattle and sheepinferred from themitochondrial cytochrome c oxidase subunit I gene[J].International Journal for Parasitology,2013,43:579-591
Cloningandphylogeneticanalysisofmitochondrialcox1geneofSarcocystisgiganteaisolatesfromQinghaiProvince
JU Jin-ling1, ZHANG Hong-bo2, CHEN Gang1, KANG Ming1
(1.Department of Veterinary Medicine, Qinghai University, Xining 810016, China;2.Academy of Animal Husbandry and Veterinary Science in Qinghai province, Xining 810016, China)
The objectives of this study were to examine the sequence variation in the partial mitochondrial cytochrome coxidase subunit I gene (pcox1) amongSarcocystisgiganteain Qinghai Province, and to study the phylogenetic relationship amongSarcocystisof the other family. Thepcox1 was amplified by PCR, then aligned thepcox1 sequence, analyzed the sequence homology, and constructed the phylogenetic tree. The results showed that the length ofpcox1 sequence as 968bp,and the similarity withS.giganteaavailable in GenBank was 99.1% ~ 99.8%. The phylogenetic tree revealed that the Qinghai isolates and theS.giganteaavailable in GenBank were clustered in the same branch. There were no signification variation inpcox1 sequence withinS.gigantea(0.2% ~0.7%), while inter-species differences was obvious(6.8%~20.8%). It is concluded thatpcox1 sequence can be used as genetic marker for population genetic studies.
Sarcocystisgigantea; Tibetan sheep ; mitochondrial DNA ;cox1 gene ; phylogenetic relationship
KANG Ming
S852.72+3
A
0529-6005(2017)10-0006-04
2016-11-10
青海省农牧厅科技专项(NMSY-2015-01);国家自然科学基金(31660698)
巨金玲(1992-),女,硕士,从事高原畜禽生理学研究,E-mail:545962186@qq.com
康明,E-mail:qhukang@163.com

