高粱属牧草分子指纹图谱构建及遗传聚类分析
陈 超,张宇君,赵丽丽*,王普昶,申 记,唐华江
(1.贵州大学 动物科学学院草业科学系,贵州 贵阳 550025;2 贵州省草业研究所,贵州 贵阳 550006;3 贵州省印江土家族苗族自治县农牧科技局饲草饲料工作站,贵州 印江 555200)
高粱属牧草分子指纹图谱构建及遗传聚类分析
陈 超1,张宇君1,赵丽丽1*,王普昶2,申 记3,唐华江1
(1.贵州大学 动物科学学院草业科学系,贵州 贵阳 550025;2 贵州省草业研究所,贵州 贵阳 550006;3 贵州省印江土家族苗族自治县农牧科技局饲草饲料工作站,贵州 印江 555200)
【目的】为建立快速有效的高粱属牧草品种鉴定分析方法。【方法】采用基于DNA混池的ISSR分子标记技术,遵循多态性高、稳定性强的原则,构建高粱(Sorghumbicolor)、甜高粱(Sorghumdochna)、苏丹草(SorghumSudanense)、高丹草(Sorghumbicolor×S.sudanense)4个高粱属牧草共12个品种的数字指纹,并进行鉴定分析和聚类分析。【结果】利用DNA混池技术从110个ISSR引物中筛选出13个多态性高、重复性好的扩增引物,计算出12个高粱属牧草品种的遗传相似系数在0.6867~0.9036。当Gs为0.786 时,12个品种可以聚为4类,第一类群包括6个高丹草品种和2个高粱品种;第二类群为2个甜高粱品种;第三类群为苏丹草品种超级丹;第四类群为苏丹草品种布鲁赛。根据引物扩增位点的特异性,挑选出3条核心引物(引物S37结合S35和S50),构建出能区分12个高粱属牧草品种的ISSR数字指纹。【结论】利用ISSR分子标记技术能有效鉴定高粱属牧草品种的纯度、真伪,揭示品种间遗传差异。
高粱属牧草;ISSR;数字指纹;聚类分析
【研究意义】高粱属(SorghumMoench)隶属于禾本科须芒草族(Andropogoneae,Poaceae),一年生或多年生C4植物[1-4],其种类繁多,大多数可以作为粮食、饲料和工业原料等,具有录取较高的经济价值[5-6]。其中饲用高粱(S.bicolor)、甜高粱(S.dochna)、苏丹草(S.Sudanense)和高丹草(S.bicolor×S.sudanense)具有较强的分蘖和再生能力,草质柔软,消化率高,营养价值好,在畜牧业生产中作为饲料得到广泛应用[7]。贵州自提出发展生态畜牧业以来,对优良高粱属牧草品种需求量日益提高[8]。相关科研人员开展了高粱属牧草品种引种适应性研究,指出高粱属牧草不同种甚至不同品种间由于种性差异,在生长特性和适应性方面存在较大差异,而种子外部形态差异较小,难以识别[5]。在市场上存在以次充好,用劣质品种冒充优势品种欺骗消费者的状况,常给农民带来较大经济损失[9]。因此,建立准确快捷的品种鉴定方法对规范种子市场迫切且必要。【前人研究进展】韩天文等利用酯酶同工酶电泳图谱鉴别了我国审定登记的5个苏丹草品种[10]。方雪恩以高粱属植物的种子为试验材料,基于醇溶蛋白A-PAGE建立了鉴定5种高粱属植物方法[11]。【本研究切入点】传统的种子鉴定主要采用种子形态学鉴定、化学鉴定、幼苗特征鉴定等,存在鉴定周期长,易受环境条件影响,结果不够准确等问题,不利于种子及时销售[12-13]。ISSR(Inter Simple Sequence Repeat)技术可灵敏地揭示2个亲缘关系十分相近个体之间的遗传变异,已经在作物、观赏植物、牧草种间和种下的分类中取得较好结果[14-18]。【拟解决的关键问题】以引种的12个不同高粱属牧草品种为研究对象,应用基于DNA混池的ISSR分子标记技术构建高粱属牧草分子指纹图谱,旨在建立一种快速、准确、经济的品种鉴定方法,为高粱属牧草优良品种保护提供依据。并分析供试高粱属牧草品种的遗传背景,分析不同品种之间的亲缘关系,为进一步培育适应地区生产利用的高粱属牧草新品种提供理论依据。

表1 12个高粱属牧草品种的编号及名称
1 材料与方法
1.1 供试材料
引种的12个高粱属牧草品种(表1)由百绿国际草业有限公司、北京正道生态科技有限公司等提供,种植于贵州大学农场实验地。以种植当年各品种的幼嫩叶片为试验材料。
1.2 试验方法
1.2.1 DNA提取及DNA混池构建 参照罗永聪等[19]的取样方法。每个品种随机选择20个单株,各单株收集等量嫩叶装入自封袋中,冷藏带回实验室。采用天根植物基因组提取试剂盒提取高粱属牧草DNA,用1.2 %琼脂糖凝胶电泳检测DNA提取结果。使用微量分光光度计测量DNA样品浓度3次,取其平均值,用ddH2O将所有DNA样品浓度调至100 ng/μl,-20 ℃保存备用。
1.2.2 ISSR引物的筛选 所用引物均委托上海生物工程技术有限公司合成。采用DNA混池与ISSR引物进行扩增反应,通过3次反复检测,从110个ISSR引物中筛选出13个重复性好、条带多且清晰度高、多态性良好的引物(表2)。
1.2.3 PCR扩增体系 PCR反应体系为20 μl:DNA模版1 μl,2×TaqMaster Mix 10 μl,引物1 μl,ddH2O 8 μl。
1.2.4 反应程序 设置49、50、51、52、53和54 ℃共6个温度梯度,摸索13个ISSR引物最适退火温度。PCR扩增程序为:94 ℃预变性5 min;94 ℃变性40 s,退火50 s,72 ℃延伸1 min,设置35个循环;72 ℃终延伸10 min;用1.2 %琼脂糖凝胶电泳检测扩增后的PCR产物,凝胶成像系统拍照保存。
1.3 数据处理
以1和0分别代表某个等位基因位点扩增DNA条带的有无,并将ISSR图谱转换为由1和0组成的字符串,构建DNA数字指纹。在PopGene3.2软件中假定哈迪-温伯格平衡条件下计算平均遗传相似系数,用NTSYS2.0软件进行聚类分析[20]。
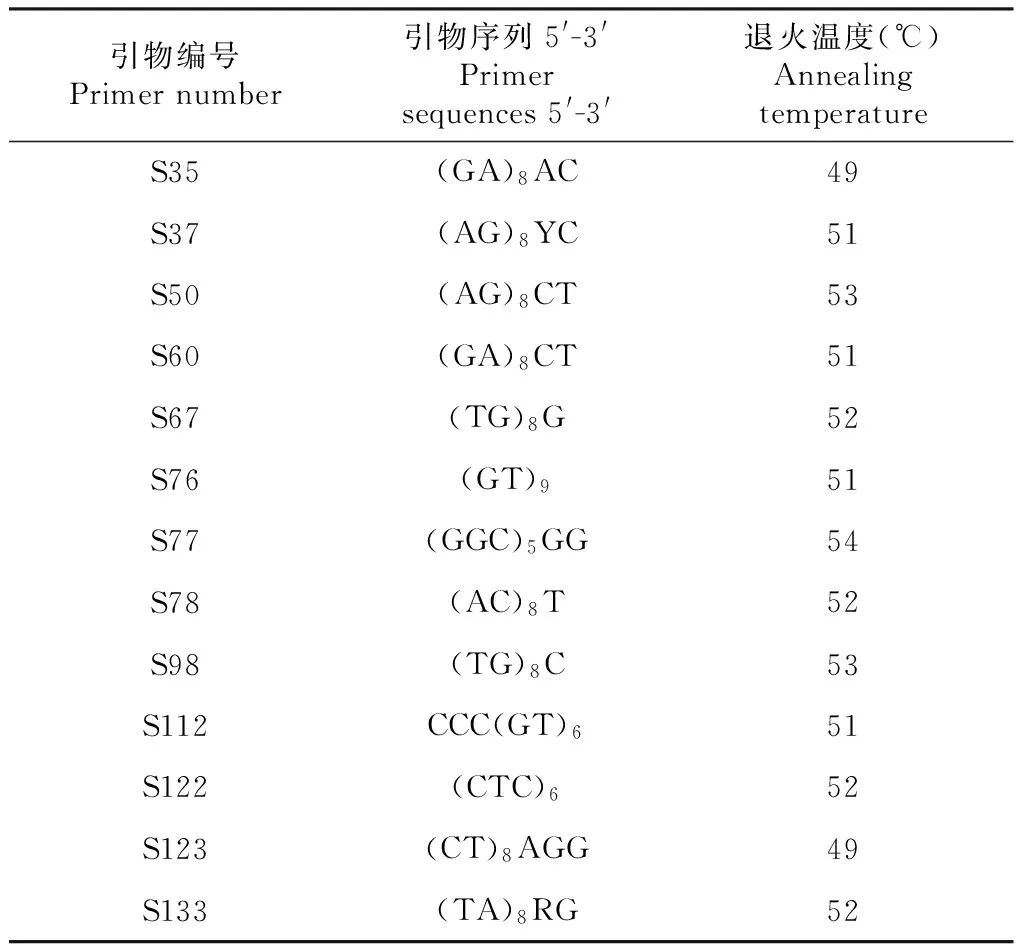
表2 13个ISSR引物序列和退火温度
2 结果与分析
2.1 PCR体系摸索
2.1.1 基因组DNA提取 从将提取的DNA用1.2 %琼脂糖凝胶电泳检测结果(图1)看出,DNA条带集中,整齐明亮无拖尾,可用于下一步试验。
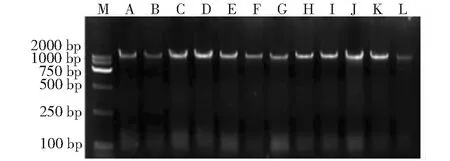
M为DL 2000 marker,A~L分别为12个高粱属牧草品种DNA提取结果M, Dl2000 marker; A-L, DNA extracted from 12 varieties respectively图1 基因组DNA的提取检测结果Fig.1 Detection results of genome DNA extraction
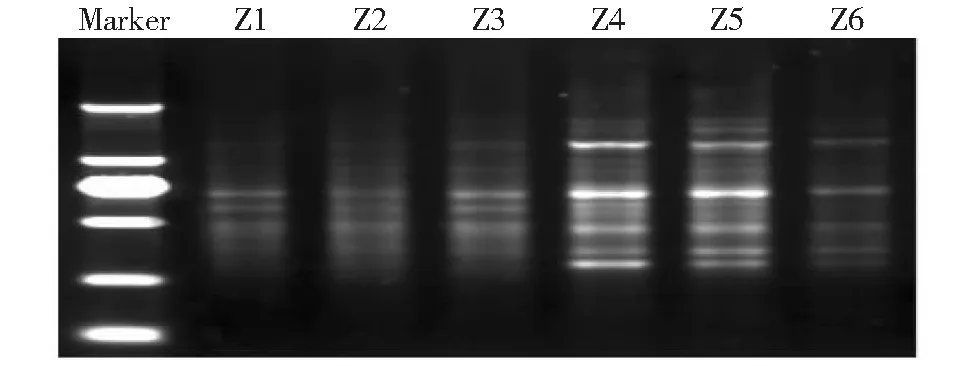
Z1~Z6依次代表49~54 ℃的退火温度Z1-Z6 represents annealing temperature from 49 ℃to 54 ℃ successively图2 引物S122最适退火温度摸索结果Fig.2 Optimum annealing temperature of S122 primer
2.1.2 最适退火温度的优化 13个ISSR引物的最适退火温度详见表2。以引物S122退火温度摸索结果(图2)为例,Z4(52 ℃)与Z5(53 ℃)条带明亮清晰,Z6(54 ℃)扩增条带不清晰,Z1(49 ℃)、Z2(50 ℃)和Z3(51 ℃)处理条带出现离散分布,确定引物S122的最适退火温度为52 ℃。
2.2 高粱属牧草ISSR数字指纹构建
利用13个ISSR引物扩增获得高粱属牧草品种的多态性图谱,其中未发现能鉴别12个品种的单个ISSR引物。由图3可知,扩增带型主要分为三类,其中高丹草C(F8421)和J(斯威特),甜高粱D(青贮大师)与高粱K(26837)扩增出的带型相同,属于第一类带型;高丹草B(甜蜜蜜)、E(F8423)和G(超级糠王),苏丹草A(布鲁赛),以及甜高粱H(大力士)扩增的带型相同,属于第二类带型;苏丹草F(超级丹)、高丹草I(22050)和高粱L(金鹰)扩增出的带型相同,属于第三类带型。结合引物S35的扩增结果,将甜高粱D(青贮大师)从第一类带型中区分出来,将高丹草B(甜蜜蜜)和E(F8423)分别从第二类带型中区分出来,将高丹草I(22050)从第三类带型中区分出来。进一步结合引物S50的扩增结果,将高丹草C(F8421)和高粱K(26837)分别从第一类带型中区分出来,将苏丹草A(布鲁赛)、高丹草G(超级糠王)和甜高粱H(大力士)分别从第三类带型中区分出来。因此,在引物S37的基础上结合S35和S50引物(图4~5),将ISSR图谱转换为由1和0组成的字符串后,形成了每个品种各自独立的数字指纹,构建出能区别12个高粱属牧草品种的ISSR数字指纹(表3)。
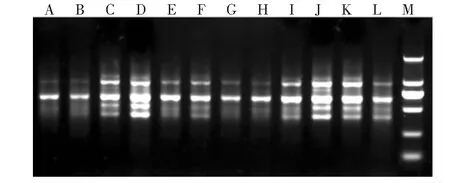
图3 引物S37在12个高粱属牧草品种中扩增结果Fig.3 Amplification of primer S37 in 12 forage varieties
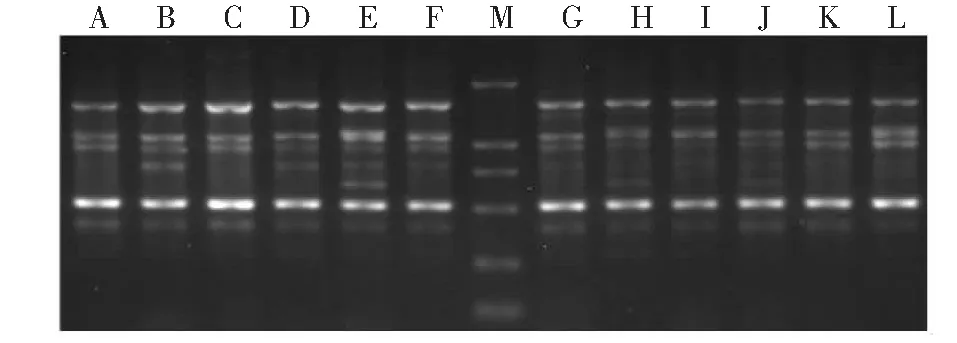
图4 引物S35在12个高粱属牧草品种中扩增结果Fig.4 Amplification of primer S35 in 12 forage varieties
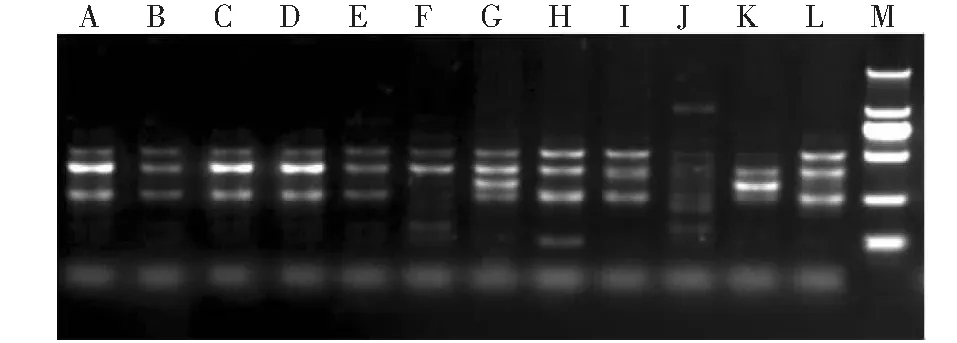
图5 引物S50在12个高粱属牧草品种中扩增结果Fig.5 Amplification of primer S50 in 12 forage varieties
2.3 高粱属牧草品种遗传结构
利用13个引物构建的ISSR数据指纹计算出供试材料的遗传相似系数,按UPGMA法进行聚类分析,建立聚类分枝树状图(图6)。12个高粱属牧草品种的遗传相似系数在0.6867~0.9036。当Gs为0.786时,可以把12个高粱属牧草品种分为4类,第一类群包括6个高丹草品种和2个高粱品种;第二类群为2个甜高粱品种;第三类群为苏丹草品种超级丹;第四类群为苏丹草品种布鲁赛。
3 讨 论
3.1 高粱属牧草品种ISSR指纹图谱构建的可行性
ISSR技术具有较稳定的试验重复性,已广泛应用于野生植物天然居群的结构分析、种质资源的评价、栽培植物品种的鉴定等,特别是在品种鉴定方面有着无可替代的简便性与实用性[21-22]。陈志丹等[23]通过ISSR技术,从11条引物中筛选出5条核心引物建立了安溪铁观音茶树分子指纹图谱,依据图谱差异可以鉴定性状不同的铁观音茶树。尹明华等[24]应用ISSR技术鉴定出上饶早梨3个主栽品种的亲缘关系及其不同继代次数试管苗的遗传变异,并利用挑选出的5条核心引物,构建了上饶早梨3个主栽品种的DNA分子指纹图谱,可鉴定上饶早梨主栽品种的归属地。Joshi等[25]利用ISSR分子标记检测了饲料高粱品种的特异性,鉴定了品种真实性,提出ISSR可以辅助饲用高粱新品种的保护。本研究借鉴前人研究经验,以20个基因型(个体)的样本量为基础,采用混合取样策略(bulking or pooling strategies),在PCR体系摸索确定的基础上,从初步筛选到的13个引物中确定出3个核心引物S37、S35和S50,形成了每个品种各自独立的数字指纹,构建出能区别12个高粱属牧草品种的ISSR数字指纹。试验条件中,采用混合取样策略可以获得供试各品种大多数个体均具有的条带,避免某些个体稀有条带出现。加之PCR体系的摸索和确定,保证了研究中12个高粱属牧草品种鉴定数字指纹的有效性。有研究指出,数字指纹只有在固定的材料范围内有效,鉴别能力相对有限[26-27]。本研究所采用的高粱属牧草品种数量有限,所获得的3对核心引物的有效性还需要在更多的品种上加以验证。
表3利用3个ISSR引物构建的12个高粱属牧草品种的数字指纹
Table 3 Digital fingerprint of 12 forage varieties established by 3 ISSR primers
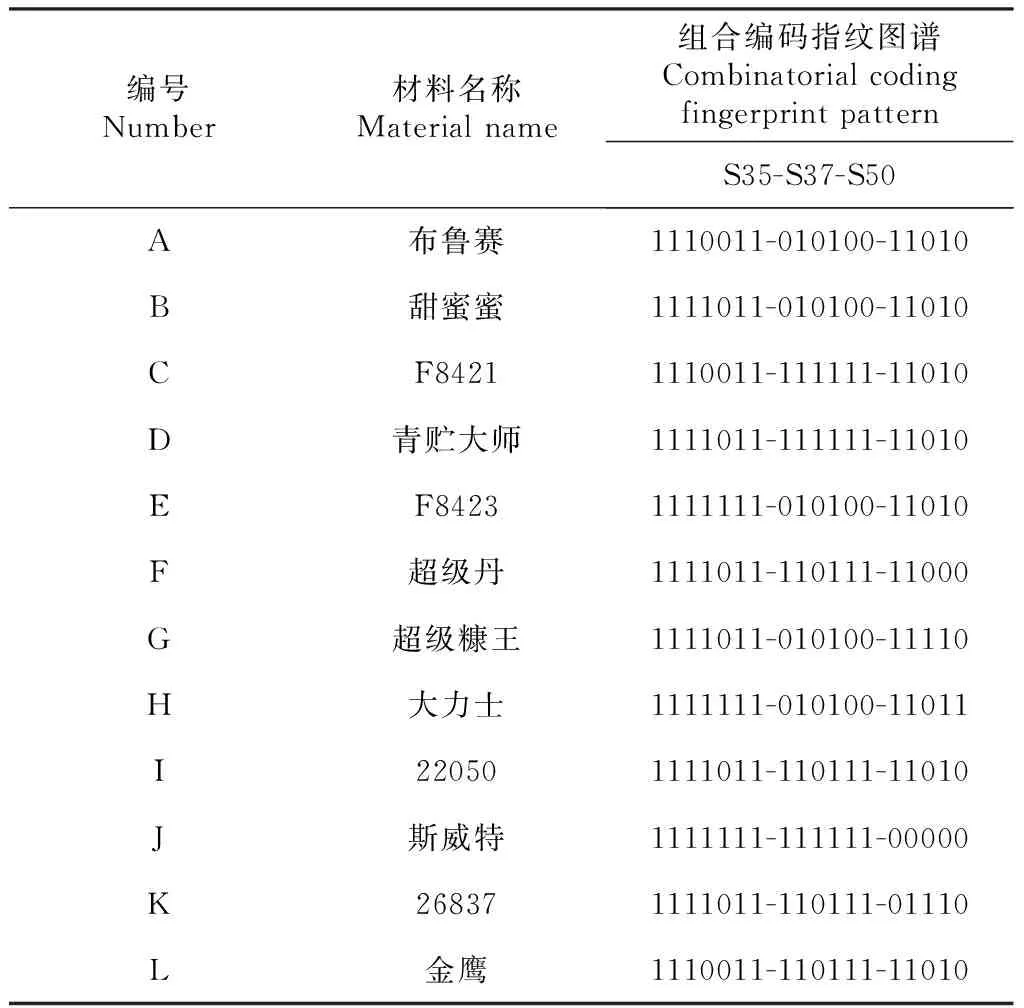
编号Number材料名称Materialname组合编码指纹图谱CombinatorialcodingfingerprintpatternS35-S37-S50A布鲁赛1110011-010100-11010B甜蜜蜜1111011-010100-11010CF84211110011-111111-11010D青贮大师1111011-111111-11010EF84231111111-010100-11010F超级丹1111011-110111-11000G超级糠王1111011-010100-11110H大力士1111111-010100-11011I220501111011-110111-11010J斯威特1111111-111111-00000K268371111011-110111-01110L金鹰1110011-110111-11010
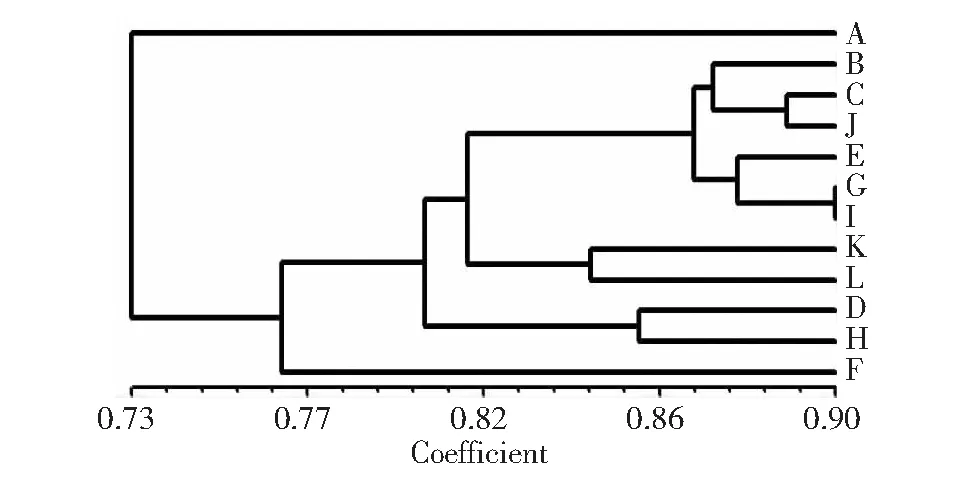
图6 高粱属牧草基于ISSR分析的聚类分析图Fig.6 Clustering dendrogram of 12 forage varieties based on ISSR analysis
3.2 供试高粱属牧草品种的遗传聚类
相关学者已经从生物学特性、分子水平等开展了高粱属牧草遗传及聚类分析,目前仍存在较多争议。杨小翠等[28]通过54个生物学性状对48个苏丹草和高粱品种进行聚类分析,李杰勤等[29]利用RAPD技术研究32个栽培高粱品种、10个苏丹草品种及2个高粱近缘种遗传差异,结果均发现使用生物学性状或RAPD分子标记进行聚类均不能将高粱和苏丹草区分开,这与Zhan Q W[30]利用SSR分子标记技术获得的研究结果吻合,研究者建议将苏丹草划为双色高粱种苏丹草亚种。王芳等[5]利用15对SSR引物对129份高粱和苏丹草的聚类分析中指出,高粱和苏丹草均各自聚为一组,SSR引物可较好地将高粱与苏丹草区分开。本研究利用ISSR技术鉴定出12个高粱属牧草品种的遗传相似系数在0.6867~0.9036,12个品种可以分为4类,第一类群包括全部的高丹草品种和高粱品种;第二类群全部为甜高粱品种;苏丹草2个品种分别被归为第三、四类。该聚类结果与目前划分的亲缘关系相符,更支持王芳等的研究结果,不同于杨小翠、李杰勤和Zhan Q W的研究结果。高丹草是以高粱为母本、苏丹草为父本育成的材料。本研究中高丹草和高粱被归为第一类群,说明高丹草的亲缘关系更接近高粱,这亦与王芳等[5]的结果相似。关于高粱和苏丹草的分类问题,需要大量的品种作为支撑,本研究所选品种有限,聚类结果仅能代表参试品种的亲缘关系,二者的分类地位需要进一步扩大参试品种进行试验证实。
4 结 论
本研究利用ISSR技术构建了12个高粱属牧草品种的DNA数字指纹图谱,能快速鉴定供试品种的真实性。聚类分析发现,12个品种的遗传聚类结果基本反映了品种间的亲缘关系。总之,利用该技术能为高粱属牧草品种的纯度、真伪鉴定和亲本选配提供参考。
[1]Liu Q, Peterson P M, Ge X J. Phylogenetic Signals in the Realized Climate Niches of Chinese Grasses (Poaceae)[J]. Plant Ecology, 2012, 212(10):1733-1746.
[2]刘 欢,曾飞燕,刘 青.高粱属植物的地理分布[J].热带亚热带植物学报,2014,22(1):1-11.
[3]Snowden J D. The Cultivated Races of Sorghum[M].London: Adlard&Son Ltd.,1936:13-15.
[4]Waston L, Dallwitz M J. The Grass Genera of the world[M].Oxfordshire: C.B.A.International, 1992:15-16.
[5]王 芳,高 秋,王 杰,等.利用SSR标记分析高粱属种质资源的遗传多样性[J].草业学报,2016, 25(5):125-133.
[6]Harlan J R, De Wet J M J. A Simplified classification of cultivated sorghum[J].Crop Science, 1972,12(2):172-176.
[7]柯 梅,朱 昊,梁维维,等.苏丹草农艺性状与产量、品质间的灰色关联度分析[J].草业科学,2016, 33(5):945-955.
[8]唐成斌,陈瑞祥,罗天琼.贵州喀斯特地区优良牧草引种及其生产性能[J].贵州农业科学,2006, 34(4):70-74.
[9]詹秋文,李杰勤,汪保华,等.42份高梁与苏丹草及其2个杂交种DNA指纹图谱的构建[J].草业学报, 2008, 17(6):85-92.
[10]韩天文,张建全.5个苏丹草品种酯酶同工酶比较研究[J].草业科学,2009,26(10):97-102.
[11]方雪恩,陈 沁,印丽萍,等.高粱属A-PAGE图谱的建立及在种子鉴定中的应用[J].西北植物学报,2007, 27(12):2399-2403.
[12]朱旭东,上官凌飞,孙 欣,等.DNA标记在植物品种鉴定上的应用现状[J].中国农学通报,2014, 30(30):234-240.
[13]詹秋文.高粱与苏丹草的遗传及其杂种优势利用的研究[D].南京:南京农业大学,2007.
[14]吴则东,江 伟,马龙彪.分子标记技术在农作物品种鉴定上的研究进展及未来展望[J].中国农学通报, 2015,31(33):172-176.
[15]Mcgregor C E, Lambert C A, Greyling M M, et al. A comparative assessment of DNA fingerprinting techniques (RAPD,ISSR,AFLP and SSR) in tetraploid potato (SolanumtuberosumL.) germplasm[J]. Euphytica,2000,113(2):135-144.
[16]Liu Q,Cheng D,Yang L, et al. Construction of digital fingerprinting and cluster analysis using ISSR markers for sugar beet cultivars (lines)[J].Transactions of the Chinese Society of Agricultural Engineering,2012,28:280-284.
[17]殷丽琴,彭云强,付绍红, 等.基于ISSR标记的彩色马铃薯遗传多样性分析及指纹图谱构建[J].西南农业学报,2016,29(1):20-25.
[18]李永清,叶 炜,江金兰,等.铁皮石斛种质亲缘关系的ISSR分析[J].西南农业学报,2015, 28(4):1530-1534.
[19]罗永聪,马 啸,张新全.利用SSR技术构建多花黑麦草品种指纹图谱[J].农业生物技术学报,2013, 21(7):799-810.
[20]Rohlf F J. NTSYS-pc,Numerical Taxonomy and Multivariate Analysis System, Version 2.10e[M]. New York: Applied Biostatistics Inc, 2000.
[21]魏青永,郭水良,曹 同, 等.基于ISSR数据探讨卷叶凤尾藓(FissidensdubiusP. Beauv.)遗传多样性[J].植物科学学报,2016,34(2):238-245.
[22]Desai P,Gajera B,Mankad M, et al.Comparative Assessment of Genetic Diversity among Indian Bamboo Genotypes Using RAPD and ISSR Markers[J].Molecular Biology Reports,2015, 42(8):1265-1273.
[23]陈志丹,孙威江,项丽慧,等.应用ISSR指纹图谱鉴定铁观音茶树[J].分子植物育种,2015, 13(12):2803-2810.
[24]尹明华,肖 芳,糜 俊, 等.应用ISSR指纹图谱鉴定上饶早梨3个主栽品种及其不同继代次数试管苗[J]. 基因组学与应用生物学,2016,35(10):2842-2850.
[25]Joshi D, Shrotria P, Singh R, et al. Assessment of RAPD and ISSR marker systems for establishing distinctiveness of forage Sorghum (SorghumbicolorL. Moench) varieties as additional descriptors for plant variety protection[J].Indian Journal of Genetics and Plant Breeding, 2011,71(1):25-36.
[26]胡晓辉,毛瑞喜,苗华荣, 等.山东省46个花生品种SSR指纹图谱构建与遗传多样性分析[J].核农学报, 2016, 30(10):1925-1933.
[27]Medraoui L, Ater M, Benlhabib O, et al. Evaluation of geneticvariability of sorghum [Sorghumbicolor(L.) Moench] in north-western Morocco by ISSR and RAPD markers[J].Comptes Rendus Biologies,2007,330(11):789-797.
[28]杨小翠,詹秋文,刘言龙,等.苏丹草与高粱的判别分析和聚类分析[J].中国草地学报,2016, 38(1):41-46.
[29]李杰勤,王丽华,詹秋文,等.应用RAPD标记对高粱属两物种之间遗传差异的研究[J].草业学报,2007, 16(5):140-144.
[30]Zhan Q W, Zhang T Z, Wang B H, et al. Diversity Comparison and Phylogenetic Relationships ofS.BicolorandS.Sudanenseas Revealed by SSR Markers[J].Plant Science,2008,174(1):9-16.
ConstructionofMolecularFingerprintPatternandGeneticClusteringAnalysisinSorghumForage
CHEN Chao1, ZHANG Yu-jun1, ZHAO Li-li1*, WANG Pu-chang2, SHEN Ji3, TANG Hua-jiang1
(1.Department of Grass Science, College of Animal Science,Guizhou University,Guizhou Guiyang 550025, China; 2.Guizhou Pratacultural Institute, Guiyang Guizhou 550006,China; 3.Station of Forage Grass and Fodder, Yinjiang Bureau of Agriculture and Animal Husbandry Science and Technology, Guizhou Yinjiang 555200, China)
【Objective】 This study aimed to establish the rapid and effective analysis method for identification ofSorghumforage varieties. 【Method】The digital fingerprint, identification and clustering of 12 forage varieties ofSorghumbicolor,Sorghumdochna,SorghumSudanenseandSorghumbicolor×S.sudanenseare respectively analyzed by the ISSR molecular marker technique based on the mixed DNA pool 【Result】13 amplification products with high polymorphism and good repeatability are screened from 110 ISSR primers by ISSR. The genetic similarity coefficient of 12 forage varieties is 0.6867-0.9036. When Gs is 0.786, 12 forage varieties can be divided into four groups. There are sixS.bicolor×S.sudanensevarieties and twoS.bicolorvarieties in Group I, twoS.dochnavarieties in Group II, oneS.sudanensevariety in Group III and oneS.sudanensevariety in Group IV. Three core primers (S37, S35 and S50) are selected to differentiate ISSR digital fingerprint of 12Sorghumforage varieties.【Conclusion】The ISSR molecular marker technique can effectively identify the purity and true or false ofSorghumforage varieties and reveal genetic difference amongSorghumforage varieties.
Sorghumforage; ISSR; Digital fingerprint; Clustering analysis
1001-4829(2017)10-2191-05
10.16213/j.cnki.scjas.2017.10.006
2017-04-12
国家科技支撑计划(2014BAD23B03);国家自然科学基金(31560664);贵州省科技计划项目[黔科合NY字(2014)3048];黔科合支撑[(2016)2516]
陈 超(1974-),男,贵州惠水人,博士,副教授,主要从事草学、畜牧学研究,*为通讯作者,E-mail:zhaolili_0508@163.com。
S431.14
A
(责任编辑 聂克艳)
——庆祝湖南农业大学草业科学系建系20 周年

