利用野栽分离群体定位水稻粒型相关QTL
梁云涛,潘英华,2,徐志健
(1.广西壮族自治区农业科学院水稻研究所,广西 南宁 530007;2.广西水稻遗传育种重点实验室,广西 南宁 530007)
利用野栽分离群体定位水稻粒型相关QTL
梁云涛1,潘英华1,2,徐志健1
(1.广西壮族自治区农业科学院水稻研究所,广西 南宁 530007;2.广西水稻遗传育种重点实验室,广西 南宁 530007)
【目的】挖掘水稻粒型相关QTL位点可为水稻的粒型遗传机制研究和优质化分子育种提供理论基础。【方法】以广西普通野生稻高代自交系材料ZY03为父本,栽培稻品种日本晴为母本,通过常规杂交获得包含160个单株的F2分离群体,并开展粒长、粒宽及粒长宽比等粒型性状的调查。利用分布于水稻12条染色体上的184个SSR标记对F2群体单株进行分子检测。应用MAPMAKER EXP 3.0软件进行数据分析,构建分子标记连锁图。应用QTLmapping3.0软件,采用复合区间作图法(composite interval mapping,CIM),以LOD=2.5为阈值检测控制粒长、粒宽和粒长宽比等性状的QTL。【结果】在F2群体中,目标性状呈现连续变异,有明显的双向超亲分离现象。共检测到与粒型相关的QTL 3个,其中1个粒长QTL位于第5染色体RM405~RM548区间内,被命名为qGL5.1,表型贡献率为10.68 %,加性效应为0.02;在第1染色体RM5501~RM486区间内检测到1个控制粒宽的QTL,被命名为qGW1.1,表型贡献率为10.56 %,加性效应为0.34;在第5号染色体RM405~RM548区间检测到1个控制粒长宽比的QTL,被命名为qLWR5.1,表型贡献率为14.77 %,加性效应为0.12。上述所有QTL的增效等位基因均来自于亲本ZY03。其中,粒长QTLqGL5.1与粒长宽比QTLqLWR5.1位于同一标记区间内。【结论】从野栽分离群体挖掘到3个野生稻的粒型QTL位点,定位结果可用于下一步主效QTL的精细定位和分子标记辅助选择育种。
粒长;粒宽,粒长宽比;QTL
【研究意义】水稻是世界上最重要的粮食作物之一,也是我国的第一大粮食作物。近年来,随着我国人民生活水平的不断提高,消费者对稻米品质的要求也越来越高,优质稻米的市场需求量逐年增加。因此,选育既高产又优质的新品种已成为当前水稻育种的趋势。粒型是水稻重要的品质性状,同时也是产量性状的重要组成部分。粒型主要由谷粒的粒长、粒宽、粒厚等3个性状构成。它们可通过协同作用调节千粒重进而影响稻谷产量。因此,改良水稻粒型是改善稻米品质和提高水稻产量的有效途径。大多数粒型性状属于数量性状,受到多基因的控制,其遗传易受环境和遗传背景等多种因素的影响。通过全面解析粒型相关性状的遗传机制和挖掘功能基因/QTL位点,可为水稻高产、优质化育种提供理论依据和优异基因资源,对推动水稻育种新突破具有重要意义。【前人研究进展】近20年来,随着分子生物学技术和现代遗传理论的进步,极大地推动了水稻分子遗传学的发展,从而可以在分子水平上深入解析不同性状的遗传机制,并发掘出大量控制水稻粒型性状的优异基因和QTL位点。到目前为止,各国科学家已经利用RFLP、RAPD、SSR等分子标记定位了许多控制粒型的QTL或基因[1],分别在水稻12条染色体上共检测到控制粒型性状的QTL252个,其中包括粒长QTL77个,粒宽QTL109个,以及粒长宽比QTL66个(http://www.gramene.org)。另外,还精细定位了GW3.1、qGL3.2、GS7、qGL7 -2和GW9.1等控制粒型的主效QTL,以及克隆了GW2[2-3]、GS2[4]、GS3[5]、GS5[6]、GW5[7-8]、GL7[9]和GL3.1[10]等7个粒型基因。这些重要的遗传位点的发掘和利用为水稻优质化育种提供了宝贵的基因材料,有效提升了稻米品质。然而,目前主要是以栽培稻为研究对象,粒型基因来源过于单一。野生稻长期生长在自然环境中,未受到人为选择干扰,保留了大量栽培稻已丧失的优质基因资源,是重要的水稻基因宝库。长期以来,绝大部分被挖掘到的基因/QTL均来自于栽培稻。如何更充分地发掘和利用野生稻基因已成为当前一个热点研究方向。【本研究切入点】野生稻基因的发掘和利用可为水稻育种提供优良的基因资源,能进一步拓宽水稻亲本的遗传基础,加快实现水稻育种的新突破。【拟解决的关键问题】本研究将ZY03和日本晴作为亲本配组杂交,构建F2分离群体,然后利用分子标记技术开展水稻粒型的QTL检测分析,旨在挖掘出控制粒型的野生稻优异基因QTL位点,并从基因组水平上揭示其遗传机制,为下一步的粒型主效QTL精细定位及稻米外观品质分子育种奠定基础。
1 材料与方法
1.1 试验材料
将1份广西普通野生稻连续进行10个世代的严格套袋自交,从而获得遗传稳定的野生稻自交系材料,其中编号为ZY03的自交系材料谷粒细长、田间综合表现优秀,是优良的水稻育种中间材料。试验以栽培稻品种日本晴作为母本,野生稻自交系ZY03作为父本,通过配组杂交获得F1代,然后进行繁殖加代,获得160个F2代单株,构成作图群体。所有试验均在广西壮族自治区农业科学院水稻研究所试验基地完成。
1.2 水稻粒型性状评价
于2015年夏季在广西农科院水稻研究所试验田种植亲本日本晴和ZY03及160个F2代单株。株行距为20 cm×23 cm,常规水肥管理。成熟后分别采收亲本和F2群体单株主穗,自然晒干,然后从主穗挑选所有饱满谷粒进行考种,取其平均值为表型值。稻谷粒型(粒长、粒宽和长宽比)的测定参照Tan等[11]的方法进行。
1.3 DNA提取及分子标记检测
在水稻分蘖期对F2群体中的单株进行挂牌取样,每个单株取2片叶子装入封口朔料袋,放置在-20 ℃冰箱里备用。DNA提取方法在参考Murray等[12]方法的基础上略有改动。
参照McCouch等[13]公布的SSR标记信息,从中挑选出均匀分布在水稻12条染色体的600对SSR标记,由北京华大生物技术服务有限公司合成标记引物。从F2群体中分别挑选出粒型性状极大和极小的各10个单株,提取其基因组DNA,分别混合建立高低池。用高低池对600对SSR标记进行多态性分析,从中筛选出184个具有多态的分子标记。然后,利用184个多态标记对F2分离群体进行分子分析,检测目标性状QTL。
分子标记PCR反应体系为10 μl体系,包含模板1 μl(50 ng·μl-1),引物1 μl(30 ng·μl-1),2×TaqMasterMix(含染料)4 μl,ddH2O 4 μl。扩增程序为95 ℃预变性5 min,95 ℃变性30 s,55 ℃退火30 s,72 ℃下延伸30 s,共35个循环,然后72 ℃下终延伸10 min,待温度降到10 ℃后取出,放置4 ℃冰箱内备用。扩增结果采用6 %聚丙烯酰胺凝胶电泳及银染法检测。PCR扩增程序、凝胶电泳和银染均参照Panaud等[14]方法。
1.4 QTL检测分析
利用分布于水稻12条染色体上的184个SSR标记对F2群体进行分子检测,应用 MAPMAKER EXP 3.0软件分析实验数据,构建分子标记连锁图谱;然后应用QTL mapping3.0软件进行QTL分析,结合表型值,采用复合区间作图法(Composite interval mapping,CIM)、以LOD=2.5为阈值检测 QTL。并计算每个QTL的表型贡献率。QTL命名原则遵循McCouch等[15]方法。采用 Excel软件对群体的性状表型值进行统计分析。
1.5 QTL连锁标记表型效应分析
将QTL连锁标记的日本晴等位基因记为A基因型,ZY03等位基因记为B基因型。从F2群体中挑选出标记基因型为AA和BB的单株,应用SPSS12.0软件分别统计AA基因型和BB基因型单株群体的目标性状表型平均值,并分析两群体表型平均值的差异程度。
2 结果与分析
2.1 亲本与F2群体的粒型表现
2亲本的粒长、粒宽、粒长宽比等性状表现差异较大。亲本日本晴与ZY03的粒长分别为0.66 cm和0.95 cm(图1、表1),亲本之间粒长差异达到极显著水平(P≤0.01)。F2群体中的粒长呈现正态分布,粒长分布范围为0.69~0.98 cm,变异系数为6.3 %,群体出现连续变异,出现明显的双向超亲分离,且存在个别超亲粒长现象。亲本日本晴与ZY03的粒宽分别为0.27和0.22cm(图1,表1),亲本之间粒宽差异达到显著水平(P≤0.05)。F2群体中的粒宽呈现正态分布,粒宽分布范围为0.28 ~0.38 cm,变异系数为5.2 %,群体出现连续变异,出现明显的双向超亲分离,且存在超亲粒宽现象。亲本日本晴与ZY03的粒长宽比分别为2.5和4.1(图1~4,表1),亲本长宽比之间差异达到极显著水平(P≤0.01)。F2群体中的长宽比呈现正态分布,长宽比分布范围为2.0~3.1,变异系数为8.9 %,群体出现连续变异,出现明显的双向超亲分离,且存在个别超亲长宽比现象。统计分析显示,粒型性状在群体上均呈正态连续分布,可以推测粒长、粒宽和粒长宽比等3种性状均属多基因控制的数量性状(图1~4)。

图1 日本晴与ZY03的粒型差别Fig.1 Difference of grain shape between Nipponbare and ZY03

图2 F2群体中粒长的频度分布Fig.2 The frequency distribution of grain length of F2 population
2.2 粒型性状之间的相关性
相关性分析表明,粒长与粒宽之间相关系数为-0.13,两者间的相关性不显著;而粒长与粒长宽比之间相关系数为0.8,两者具有高度正相关;粒宽与粒长宽比之间相关系数为-0.7,两者具有显著负相关(表2)。结果显示,粒宽与粒长宽比之间,以及粒长和粒长宽比之间均存在显著的相关性;而粒长和粒宽相关性不显著。由此可以推测,控制粒宽和粒长分别与控制粒长宽比的遗传调控机制之间相互紧密关联,而控制粒宽与粒长的调控机制则相对独立。

表1 亲本及其F2群体粒型相关性状表现
注:**为极显著差异。
Noe:**indicate the significance at the probability levels of 0.01.

图3 F2群体中粒宽的频度分布Fig.3 The frequency distribution of grain width of F2 population
2.3 粒型QTL分析
试验共检测到与粒型相关的QTL 3个。其中1个粒长QTL位于5号染色体RM405~RM548区间内,LOD值为2.53,单个QTL对表型的贡献率为10.68 %,加性效应为0.02,被命名为qGL5.1;1个粒宽QTL位于1号染色体RM5501~RM486区间内,LOD值为2.54,单个QTL对表型的贡献率为10.56 %,加性效应为0.34,被命名为qGW1.1;1个控制粒长宽比的QTL位于5号染色体RM405~RM548区间内,LOD值为3.0,单个QTL对表型的贡献率为14.77 %,加性效应为0.12,命名为qLWR5.1(表3、图5)。其中,粒长QTLqGL5.1与粒长宽比QTLqLWR5.1位于同一个区间内。上述QTL的增效等位基因均来源于亲本Y03,表明来自野生稻的等位基因将会引起粒长、粒宽和粒长宽比等性状值的增加。
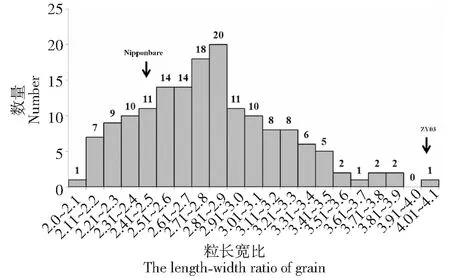
图4 F2群体中粒长宽比的频度分布Fig.4 The frequency distribution of length-width ratio of grain of F2 population
2.4 qGL5.1连锁标记表型效应分析
与qGL5.1紧密连锁的分子标记有RM405和RM548标记。RM405标记AA基因型的粒长平均值为0.80 cm,BB基因型的粒长平均值为0.86 cm,BB基因型的平均粒长比AA基因型的粒长长7.50 %,未达到显著差异;RM548标记AA基因型的粒长平均值为0.80 cm,BB基因型的粒长平均值为0.86 cm,BB基因型的平均粒长比AA基因型长7.50 %,达到显著差异(表4)。
2.5 qLWR5.1连锁标记表型效应分析
与qLWR5.1紧密连锁的分子标记是RM405和RM548标记。RM405标记的AA基因型的粒长宽比平均值为2.36,BB基因型的粒长宽比平均值为2.61,BB基因型比AA基因型长了10.60 %,差异达到极显著。RM548标记AA基因型的粒长宽比平均值为2.37,BB基因型的粒长宽比平均值为2.62,两者相差10.54 %,差异达到极显著(表4)。

表2 粒型性状之间的相关性
注:*为显著差异。
Note:*indicate the significance of correlation at the probability levels of 0.05.
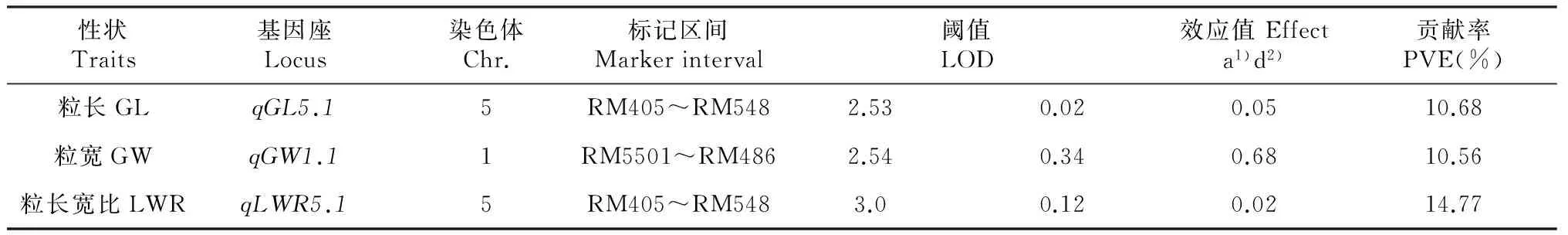
表3 粒型性状的QTL及其遗传效应
注: 1)加性效应;2)显性效应。
Note:1) Additive gene effect;2) Dominance gene effect.

表4 单标记效应分析
注:A:日本晴基因型,B:ZY03基因型,*为显著差异,**为极显著差异。
Note:A:Genotype of Japonica, B:Genotype of ZY03,*and** indicate the significance of at the probability levels of 0.05 and 0.01, respectively.
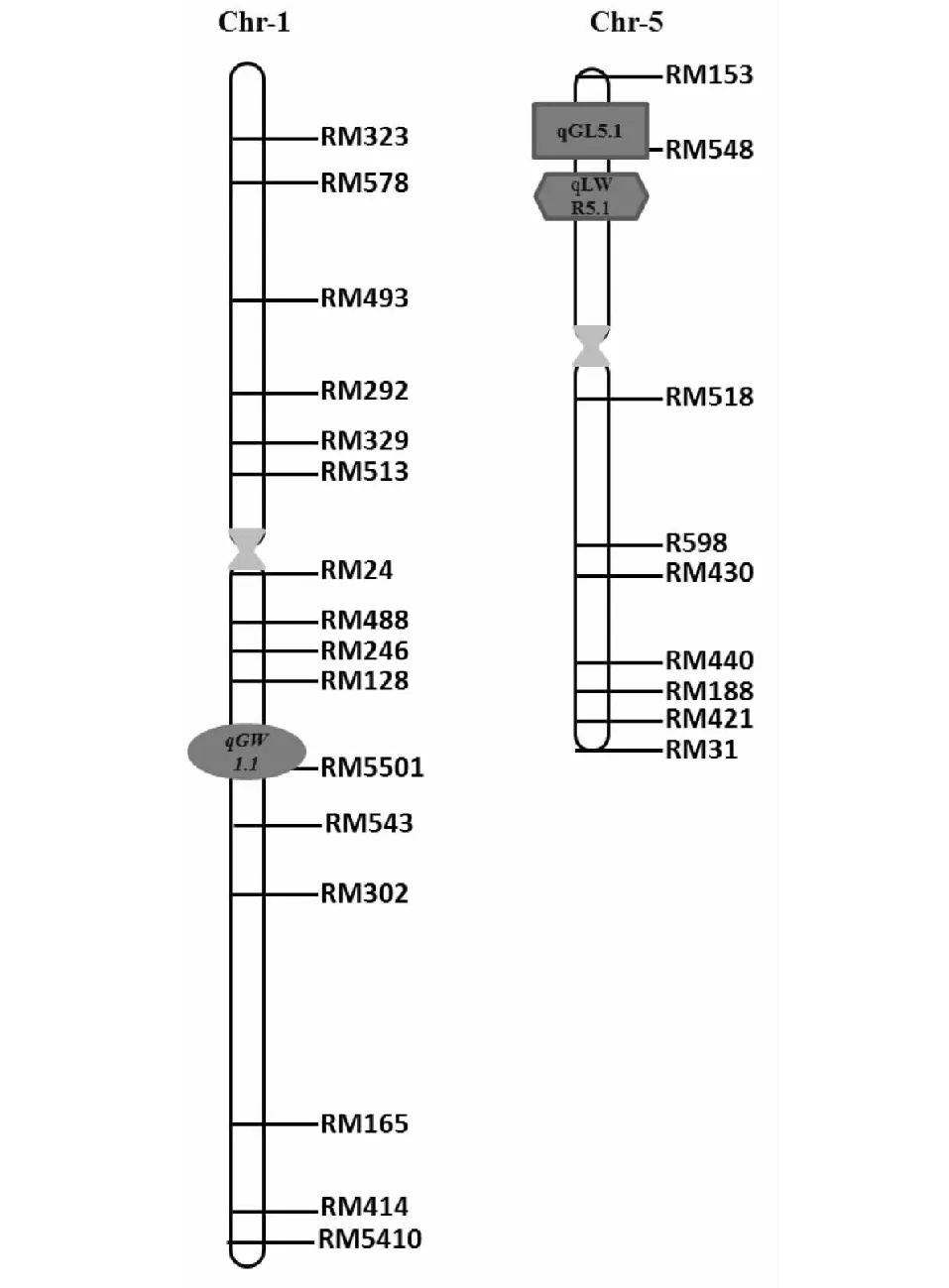
图5 粒型QTL在染色体上的分布Fig.5 The distribution of QTLs of grain shape trait on chromosomes
3 讨 论
水稻粒型性状是水稻重要的外观品质,包括粒长、粒宽、粒长宽比、粒厚等性状,这些粒型性状通过影响粒重对水稻产量有很大影响[16],并且粒型与稻米的外观、加工品质,蒸煮后的口感都有着十分密切的关系[17]。
由于稻米粒型对于水稻产量和品质的重要性,各国科学家已经做了大量工作,深入研究影响粒型性状的各种遗传因素,挖掘控制粒型的基因和QTL用于育种改良。例如,张亚东等[18]利用特大粒粳稻TD70和籼稻Kasalath构建了作图群体,定位了控制粒长的QTL5个,控制粒宽的QTL6个。汪欲鹏等[19]以大粒型水稻材料lg1与9311杂交衍生的F2遗传分离群体为对象,在3种情况下定位到5个粒长QTL和9个粒宽QTL。赵芳明等[20]利用单片段代换系(SSSL)和双片段代换系,共检测到9个水稻粒型性状QTL。邹德堂等[21]利用两个粳稻品种构建了F2∶3和BC2F2群体检测到了与粒长QTL15个,与粒宽QTL8个,籽粒长宽比QTL11个。Hu等[22]构建重组自交系,定位了7 个影响粒长和 6个影响粒宽的 QTL。Zeng等[23]利用热带粳稻Lemont和籼稻扬稻4号构建了作图群体,定位了4个控制谷粒长的QTL。Sun等[24]利用珍汕97和SLG组配了重组自交系群体,定位了2个控制粒型的QTL。Zhao等[25]用籼粳交后代的染色体片段渗入系构建了F3群体,定位了3个粒型相关主效QTL。随后,在初步定位的基础上,可通过构建重组自交系等更大的分离群体进一步精细定位粒型基因或主效QTL。Shao 等[9]利用爪哇稻D50与籼稻HB277配制了重组自交系,将控制谷粒长的QTLqGL7-2定位在7号染色体278 kb区间内。Zhang等[4]将控制大粒的基因GS2定位在2号染色体上标记GL2-35-1和GL2-12之间33.2 kb区间内。Fan等[5]则利用川7/明恢63构建回交群体进一步将粒型基因GS3精细定位在7.9 kb 区间范围内。另外,经过多年努力,至今已成功克隆了多个控制粒型的基因和主效QTL。并通过对遗传结构和功能的系统分析,人们可以更清晰和全面地了解它们的遗传规律和调控机制,为水稻优质化育种提供重要的理论依据。研究发现,已克隆的粒型基因GW2包含8个外显子,编码一个环型E3泛素连接酶,通过将其底物锚定到蛋白酶体进行降解,从而负调节细胞的分裂,显著地增加粒宽和千粒重,从而增加单株产量,该等位基因同时也能增加每株穗数、延长生育期,并显著地降低每穗粒数和主穗长度,表明GW2具有明显的一因多效[2-3]。粒型基因GW5编码一个核定位蛋白,GW5功能的缺失将不能将泛素转移到靶蛋白上,因而使得本应降解的底物不能被特异识别,进而激活颖花外壳细胞的分裂,增加颖花外壳的宽度,最终导致谷壳的宽度、粒重以及产量的增加[7-8]。GS5是同时影响粒宽和粒重的主效QTL,编码一个丝氨酸缩肽酶,启动子区域调控GS5的表达量,从而控制粒宽[6]。GW8基因通过调控细胞周期基因CDKA1、CYCD3 和E2F2的表达来影响颖壳细胞数目,最终正调控粒宽和粒重[26]。本研究分别在第1、5染色体上检测到的3个粒长、粒宽和粒长宽比QTL,其中,qGL5.1、qLWR5.1的定位区间被发现与GS5重叠,但由于定位的区间比较大,尚不能明确qGL5.1、qLWR5.1是否与GS5等位。仍需要开展进一步的工作,构建更大的分离群体,通过加大检测标记的密度,对其精细定位后才能确定与已知基因/QTL位点的关系。
本研究利用广西野生稻资源,通过采取分子标记技术,挖掘到3个野生稻的粒型QTL位点。基因载体ZY03是野生稻通过多代自交和选择而获得野生稻自交系材料,具有遗传稳定、综合农艺性状优秀等特点,是优良的育种中间材料。QTL的检测分析从分子水平上更准确的阐明了ZY03的粒型遗传机制,为下一步稻米品质的育种改良提供重要的理论依据,缩短了育种周期,加快了野生稻优异基因资源利用的步伐。
在本研究中,还通过对QTL连锁标记的表型效应分析,期望能获得可以应用于分子育种的分子标记。结果发现,在F2分离群体中,与qGL5.1连锁的分子标记RM548,以及与qLWR5.1连锁的标记RM405和RM548,它们ZY03等位基因型(AA)植株的谷粒长和谷粒长宽比平均值明显高于日本晴(BB),说明上述标记与目标性状紧密连锁,可以在今后水稻分子育种实践中作为指示标记改良水稻粒型性状。
4 结 论
本研究利用野栽杂交的F2分离群体,采用分子标记技术,共挖掘到来源于野生稻的粒型QTL 3个,1个粒长QTL位于5号染色体RM405~RM548区间内,被命名为qGL5.1;1个粒宽QTL位于1号染色体RM5501~RM486区间内,被命名为qGW1.1;1个控制粒长宽比的QTL位于5号染色体RM405~RM548区间内,被命名为qLWR5.1。所有QTL的增效等位基因均来源于亲本野生稻自交系ZY03。其中,粒长QTLqGL5.1与粒长宽比QTLqLWR5.1位于同一个染色体区间。本研究的定位结果可为下一步主效QTL的精细定位和分子标记辅助选择育种奠定了基础。
[1]高志强,占小登,梁永书,等.水稻粒形性状的遗传及相关基因定位与克隆研究进展[J].遗传,2011,33(4):314-321.
[2]Song X J, Huang W, Shi M,et al.A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J]. Nature Genetics, 2007, 39(5): 623-630.
[3]Yan S, Zou G H, Li S J, et al. Seed size is determined by the combinations of the genes controlling different seed characteristics in rice[J]. Theoretical and Applied Genetics, 2011, 123(7): 1173-1181.
[4]Zhang W H, Sun P Y, He Q, et al. Fine mapping ofGS2, a dominant gene for big grain rice[J]. The Crop Journal, 2013, 1(2): 160-165.
[5]Fan C H, Xing Y Z, Mao H L, et al.GS3, a major QTL for grain length and weight and minor QTL for grain width and thickness in rice, encodes a putative transmembrane protein[J]. Theoretical and Applied Genetics, 2006, 112(6): 1164-1171.
[6]Wang E, Xu X,Zhang L, et al. Duplication and independent selection of cell-wall invertase genesGIF1 andOsCIN1 during rice evolution and domestication[J]. BMC Evolutionary Biology, 2010, 10(1): 108.
[7]Liu J F, Chen J, Zheng X M, et al.GW5 acts in the brassinosteroid signalling pathway to regulate grain width and weight in rice[J]. Nature Plants, 2017, 3:17043.
[8]Weng J F ,Gu S H, Wan X Y, et al. Isolation and initial characterization ofGW5, a major QTL associated with rice grain width and weight[J]. Cell Research, 2008, 18(12): 1199-1209.
[9]Shao G N, Tang S Q, Luo J, et al. Mapping ofqGL7-2, a grain length QTL on chromosome 7 of rice[J]. Journal of Genetics and Genomics, 2010, 37(8): 523-531.
[10]Qi P, Lin Y S, Song X J, et al. The novel quantitative trait locusGL3.1 controls rice grain size and yield by regulating Cyclin-T1;3[J]. Cell Research, 2012, 22(12): 1666-1680.
[11]Tan Y F, Xing Y Z,Li J X, et al. Genetic bases of appearance quality of rice grains in Shanyou 63,an elite rice hybrid[J].Theoretical and Applied Genetics,2000, 101(6): 823-829.
[12]Murray M G, Thompson W F. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research,1980, 8(19): 4321-4326.
[13]McCouch S R, Kochert G, Yu Z H, et al. Molecular mapping of rice chromosomes[J]. Theoretical and Applied Genetics, 1988, 76(6): 815-829.
[14]Panaud O, Chen X, McCouch S R. Development of microsatellite markers and characterization of simple sequence length polymorphism (SSLP) in rice (OryzasativaL.)[J]. Molecular and General Genetics,1996, 252(5): 597-607.
[15]McCouch S R, Cho Y G, Yano M, et al. Report on QTL nomenclature[J]. Rice Genetics Newsletter,1997, 14: 11-13.
[16]Qiu X J, Gong R, Tan Y B, et al. Mapping and characterization of the major quantitative trait locusqSS7 associated with increased length and decreased width of rice seeds[J].Theoretical and Applied Genetics, 2012, 125(8):1717-1726.
[17]徐正进,陈温福,马殿荣,等.稻谷粒形与稻米主要品质性状的关系[J].作物学报,2004,30(9):894-900.
[18]张亚东,张颖慧,董少玲,等.特大粒水稻材料粒型性状的QTL检测[J].中国水稻科学,2013,27(2): 122-128.
[19]汪欲鹏,王根发,武志峰,等.大粒型水稻材料粒型性状的 QTL 定位[J].核农学报,2016,30(12):2295-2303.
[20]赵芳明,张桂权,曾瑞珍,等.基于单片段代换系的水稻粒型 QTL 加性及上位性效应分析[J].作物学报,2011, 37(3): 469-476.
[21]邹德堂,刘忠良,赵宏伟,等.利用F2∶3和BC2F2群体定位水稻粒型和粒重QTL[J]. 东北农业大学学报,2014,45(9): 9-17.
[22]Hu W, Wen M, Han Z M, et al. Scanning QTLs for grain shape using a whole genome SNP array in rice[J].J Plant Physiology and Biochemistry,2011(3): 104.
[23]Zeng Y X, Ji Z J, Wen Z H, et al.Combination of Eight Alleles at Four Quantitative Trait Loci Determines Grain Length in Rice[J].Plos One, 11(3): e0150832.
[24]Sun L, Ma D P, Yu H H,et al. Identification of quantitative trait loci for grain size and the contributions of major grain-size QTLs to grain weight in rice[J].Molecular Breeding,2013,31(2):451-461.
[25]Zhao F M, Tan Y, Zheng L Y, et al.Identification of rice chromosome segment substitution line Z322-1-10 and mapping QTLs for agronomic traits from the F3 population[J]. Cereal Research Communications,2016,44(3):1-11.
[26]Wang S K, Wu K, Yuan Q B,et al. Control of grain size,shape and quality byOsSPL16 in rice[J].Nature Genetics,2012,44(8):950-954.
QTLsMappingofGrainShapeofRicebyUsingOffspringDerivedfromCrossofOryzarufipogonGriff.andCultivatedRice
LIANG Yun-tao1,PAN Ying-hua1,2, XU Zhi-jian1
(1. Rice Research Institute of Guangxi Academy of Agricultural Sciences, Guangxi Nanning 530007,China;2. Guangxi Key Laboratory of Rice Genetics and Breeding, Guangxi Nanning 530007,China)
【Objective】Grain shape is an important trait affecting grain quality of rice and one of key factors determining the thousand seed weight and production of rice. Mining QTL loci associating with grain shape could provide theoretic basis for research on genetic mechanism of grain shape and molecular breeding for improving grain quality of rice.【Method】We constructed F2population with 160 plants derived from a cross of an inbred line ofOryzarufipogonGriff. ZY03 and Nipponbare. Grain length, grain width and length-width ratio of grain of F2plants were surveyed. 184 polymorphic SSR makers distributing on 12 chromosomes were used for mapping QTLs of grain length, grain width and length-width ratio of grain. QTL analysis was carried out by using both MAPMAKER EXP 3.0 and QTLmapping3.0. The QTLs was determined as value of LOD was 2.5.【Result】Phenotypic value of grain shape trait of F2plants showed continuous variation and strong transgressive segregation. Three QTLs associating with grain length, grain width and length-width ratio of grain were detected. These QTLs were distributed on the chromosome 1,5. Two QTLs, namelyqGL5.1 andqLWR5.1, were distributed on the chromosome 5.One QTL, namelyqGW1.1, was located on the chromosome 1.qGL5.1 controlling grain length was mapped in the marker interval RM405-RM548 on the chromosome 5, which explained 10.68 % of the total phenotypic variation.qLWR5.1 controlling length-width ratio of grain was located in the marker interval RM405-RM548 on the chromosome 5, which explained 14.77 % of the total phenotypic variation.qGW1.1 controlling length-width ratio of grain was mapped in the marker intervalRM5501-RM486 on chromosome 1,which explained 10.56 % of the total phenotypic variation.qGL5.1 andqLWR5.1 were mapped in the same marker interval on chromosome 5.Alleles of all QTLs were derived from ZY03.【Conclusion】By applying molecular marker, three wild rice QTLs were detected in F2population derived from cross of wild rice and cultivated rice. Molecular genetic mechanism controlling grain shape of rice was illuminated in this work,which would be helpful for fine mapping of major QTLs and molecular breeding in the furture.
Grain length; Grain width;Length-width ratio of grain; QTL
1001-4829(2017)10-2161-07
10.16213/j.cnki.scjas.2017.10.001
2017-07-03
广西壮族自治区主席科技资金项目“野生稻优异基因的挖掘和利用研究”(1517-03);国家科技资源共享服务平台项目“国家野生稻种质资源平台(南宁)”(NICGR2017-039);广西农业科学院基本科研业务专项(优势学科团队项目)“野生稻资源保护与利用研究”(2015YT14);广西农业科学院基本科研业务专项(广西药用野生稻基因MKK3的功能验证(桂农科2017YZ08),广西野生稻等保护点目标物种居群多样性分析”(2015JZ12403),野生稻抗逆基因挖掘[2015JZ24])
梁云涛(1975-),壮族,男,广西崇左人,硕士,副研究员,主要从事野生稻种质资源研究,E-mail:liangyt@sina.com。
S511
A
(责任编辑 陈 格)

