不同饲料饲养黄牛瘤胃厌氧真菌的多样性对比研究
李亚丹,杨 辉,卢向阳,田 云*
(1.湖南农业大学 生物科学技术学院,湖南 长沙 410128;2.长沙环境保护职业技术学院环境科学系,湖南 长沙 410004)
不同饲料饲养黄牛瘤胃厌氧真菌的多样性对比研究
李亚丹1,2,杨 辉1,卢向阳1,田 云1*
(1.湖南农业大学 生物科学技术学院,湖南 长沙 410128;2.长沙环境保护职业技术学院环境科学系,湖南 长沙 410004)
【目的】探究不同饲料饲养黄牛瘤胃厌氧真菌多样性的差异,了解芒草单一饲料饲养时瘤胃中特殊的真菌种类。【方法】通过构建芒草单一喂养、混合饲料饲养的黄牛瘤胃微生物18S rRNA基因文库,来对比分析单一饲喂芒草对瘤胃厌氧真菌多样性的影响。【结果】发现持续喂养18个月后,混合饲料饲养牛瘤胃真菌包含的种类较分散,无明显的优势类别,而芒草单一喂养牛瘤胃真菌包含的种类相对集中,主要集中在Orpinomycessp.,Piromycessp.,Neocallimastixfrontalis,Microdochiumsp.几种。【结论】使用芒草单一饲养确实改变了黄牛瘤胃中真菌的组成,并且其中优势真菌类群很可能与芒草的降解有密切关系,为今后开发瘤胃微生物资源在生物能源方面的利用奠定基础。
黄牛;瘤胃;芒草;18S rRNA;真菌
【研究意义】反刍动物瘤胃微生物长期处于恒定温度、恒定pH值、低氧气浓度的特殊环境,所以瘤胃微生物的纯分离培养受到极大的限制。【前人研究进展】20世纪70年代末,研究人员首次发现瘤胃内存在一个重要的微生物类群,厌氧真菌[1]。最近,仍有诸多新的微生物种陆续分离出来,如Mitsuokellajalaludinii[2],Eubacteriumpyruvativorans[3]和Cellulosilyticumruminicola[4]。瘤胃中厌氧真菌游动孢子数为103~105/mL,对于瘤胃微生物类别的认识在不断地丰富。瘤胃真菌的生物量约占瘤胃微生物总量的8 %[5]。截止2000年,瘤胃中已有15种厌氧真菌分离成功。根据其孢子、菌丝的形态分类,可将瘤胃真菌划分为多中心类型和单中心类型。其中,多中心类型真菌主要包括Orpinomyces和Anaeromyces两个属,单中心类型真菌主要包括Neocallimastix,Promyces,Caeomyces3个属,具体详见表1。
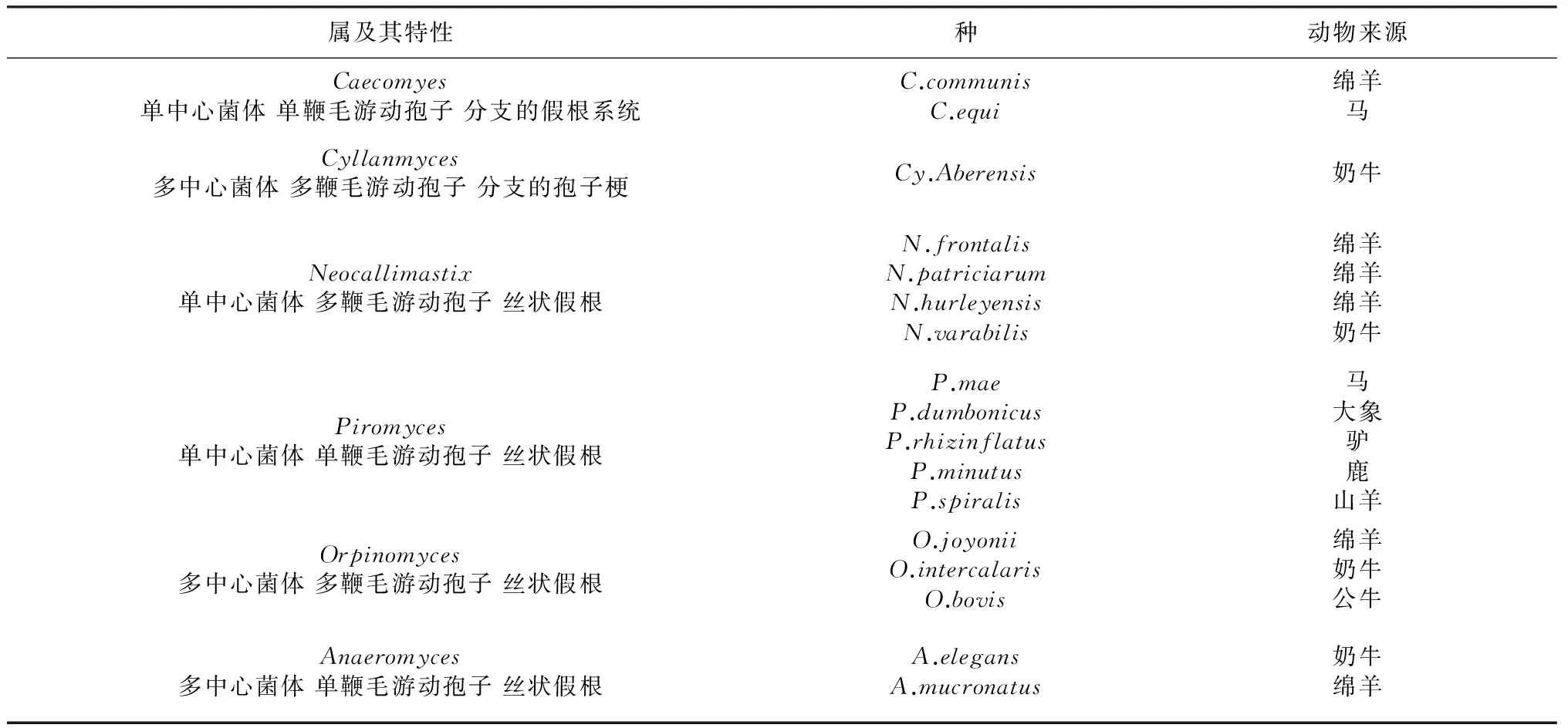
表1 瘤胃主要真菌种类[5-6]
瘤胃真菌不仅能分泌产生纤维素酶,真菌的穿透生长还使植物组织纤维内部张力减小,使其变得疏松而易于降解。有研究证实,在无瘤胃细菌存在条件下,瘤胃真菌能完成粗纤维饲料中的干物质62 %的降解[6],瘤胃真菌对于瘤胃内的纤维质成分的高效降解能力可见一斑。【本研究切入点】因此,通过高纤维含量饲料对黄牛进行驯养,研究黄牛瘤胃真菌多样性的改变,对今后研究瘤胃对食物的消化吸收,以及利用牛瘤胃真菌资源降解纤维质材料的工作存在重要的意义。【拟解决的关键问题】拟通过构建牛瘤胃真菌18S rRNA基因克隆文库的方法,分析饲喂单一芒草和普通饲料对于牛瘤胃真菌多样性的影响。
1 材料与方法
1.1 实验动物饲养及样品采集
实验动物为湘西黄牛。在长沙县江背镇乌川湖村分组饲养(3头/组),试验组 (M)仅以新鲜芒草为饲料,对照组 (C)以混合饲料酒糟、豆渣、干草为饲料,饲养时间为18个月。两种饲料的纤维素含量采用VELP型纤维素测定仪测定(表2)。
牛屠宰后带无菌手套分组取牛瘤胃内容物进行混合,再分为50 g/份,在液氮中速冻,带回实验室,置于-80 ℃备用。
1.2 高分子量瘤胃微生物总DNA的提取
以牛瘤胃内容物为实验对象,通过除杂等步骤,得到瘤胃微生物,参照文献[7]方法,提取总DNA,分别获得试验组总DNA和对照组总DNA。
1.3 PCR反应体系及条件
以真菌18S rRNA基因扩增引物[8]:(EF4:5′-GGAAGGGRTGTATTTATTAG-3′,fung5:5′-CTAAAA GTCCTGGTTCCCC-3′)和总DNA为模板,通过PCR反应扩增真菌18S rRNA基因。
1.4 18S rRNA基因克隆文库的构建
每个模板同时进行3个平行PCR反应,将3次PCR产物混合以减少随机性偏差。PCR产物用DNA纯化试剂盒纯化后的产物与pMD18-T载体按如下体系混匀,于16 ℃连接1 h。热击转化后,加入500 μl新鲜LB培养基,恢复培养1 h后在含Amp (100 μg/mL)的固体LB培养基上进行涂布。试验组18S rRNA基因文库标记为FM,对照组18S rRNA基因标记为FC。液体培养将得到的菌落的对应菌液进行备份保藏。
1.5 核糖体DNA扩增片段限制性内切酶分析
选用四碱基限制性内切酶MspⅠ和AfaⅠ先后2次对文库中菌落的PCR产物进行酶切分析。酶切产物经3 %琼脂糖电泳检测,根据酶切谱带初步确定克隆的分类单位 (OUT)。将经过两次酶切分析的具有不同酶切谱带的克隆初步确定为不同的分类单位,测序由北京六合华大基因科技股份有限公司完成。

表2 饲料成分比例
1.6 系统进化树的构建及各OTU比例分析
使测序序列进行拼接,NCBI在线Blast n比对,获得各序列在GeneBank中相似性最高的序列,下载用于与本实验结果序列共同构建系统进化树。用Neighbor-Jioning法构建进化树,Bootstrap检验进化树,自展数为1000。归纳整理序列信息,统计各运筹分类单元 (operational taxonomic units,OTU)及其所包含的克隆数,计算各个分类单元在文库中所占比例。
2 结果与分析
根据测序结果进行结果中各序列间的比对,将完全一致序列归为一个OUT,再与Genebank数据库中已知真菌18S rRNA基因进行比对分析瘤胃真菌多样性。
由表1可知,该文库中19条序列与数据库已知序列的序列相似性<97 %,约占总数的29.23 %,说明以芒草作为单一饲料的牛瘤胃真菌种类的新颖性强。其中FM-010和FM-017各包含5条序列,分别与Orpinomycessp.的18S rRNA基因序列相似性达到97 %,Orpinomycessp.很可能是试验组牛瘤胃中的优势真菌种类。进行序列的进化关系分析时,发现其中一支包括FM-012、FM-011、FM-010、FM-006、FM-009、FM-004、FM-013、FM-007、FM-016、FM-022、FM-017、FM-023,所包含33个克隆占文库容量的50.77 % (图1)。
而在对照组牛瘤胃真菌18S rRNA基因文库中的41个克隆,绝大部分的序列与数据库已知序列的相似性≥97 %,只有4条序列 (FC-009)与已知序列相似性<97 %,Gibellulopsisnigrescens(FC-010)为该库中的优势种,包含6条序列,占14.63 %(表2)。通过构建系统进化树 (图2)发现,同样该库中存在一支与已知序列亲缘关系较远的分支,包含12个OUT,分别为FC-001、FC-006、FC-008、FC-009和FC-002的分支与其他真菌的亲缘关系比较远,约占总数的34.14 %。

图1 芒草驯养牛瘤胃真菌18S rRNA基因序列系统发育树Fig.1 Phylogenetic tree of fungual 18S rRNA gene sequences from Miscanthus sinensis (+) library
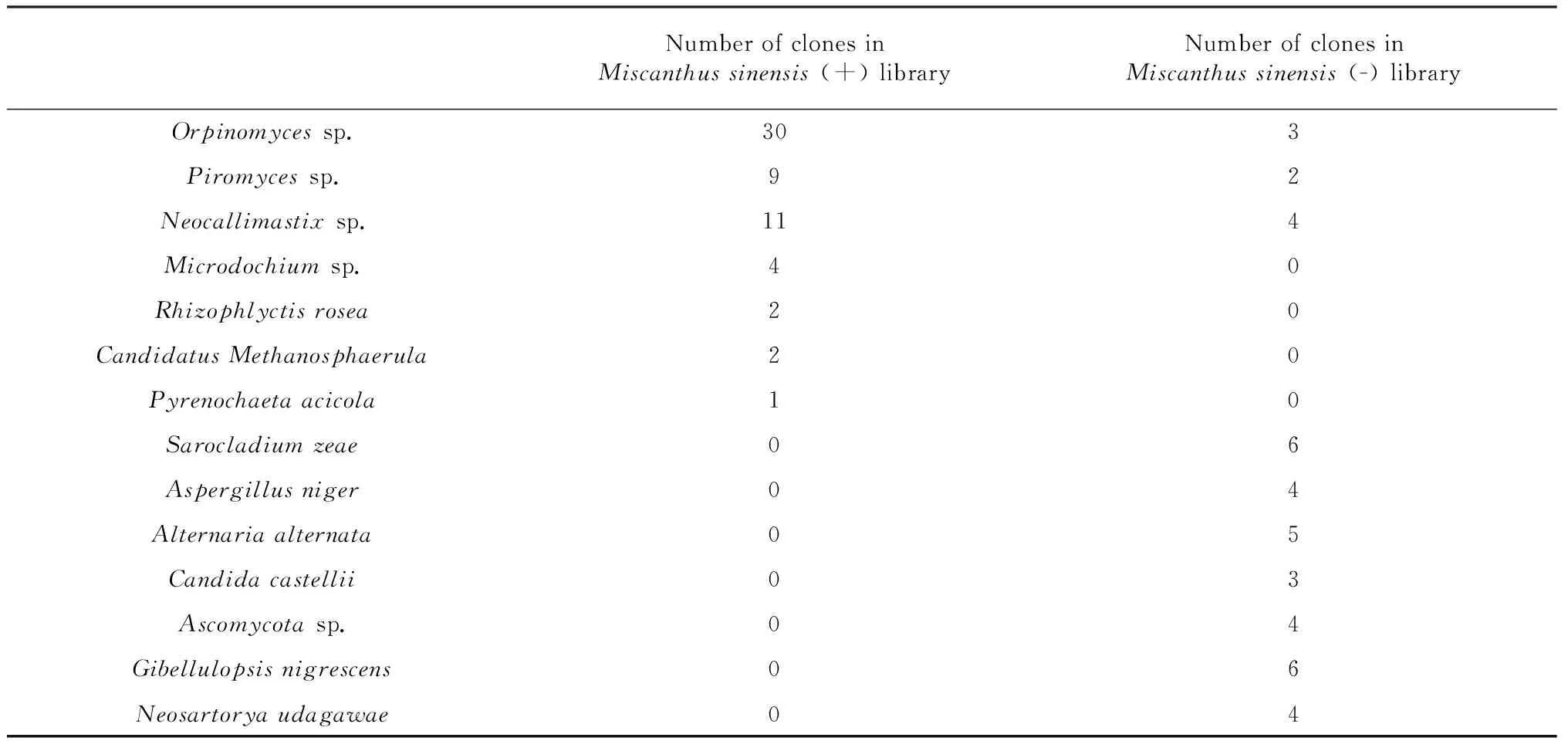
NumberofclonesinMiscanthussinensis(+)libraryNumberofclonesinMiscanthussinensis(-)libraryOrpinomycessp.303Piromycessp.92Neocallimastixsp.114Microdochiumsp.40Rhizophlyctisrosea20CandidatusMethanosphaerula20Pyrenochaetaacicola10Sarocladiumzeae06Aspergillusniger04Alternariaalternata05Candidacastellii03Ascomycotasp.04Gibellulopsisnigrescens06Neosartoryaudagawae04
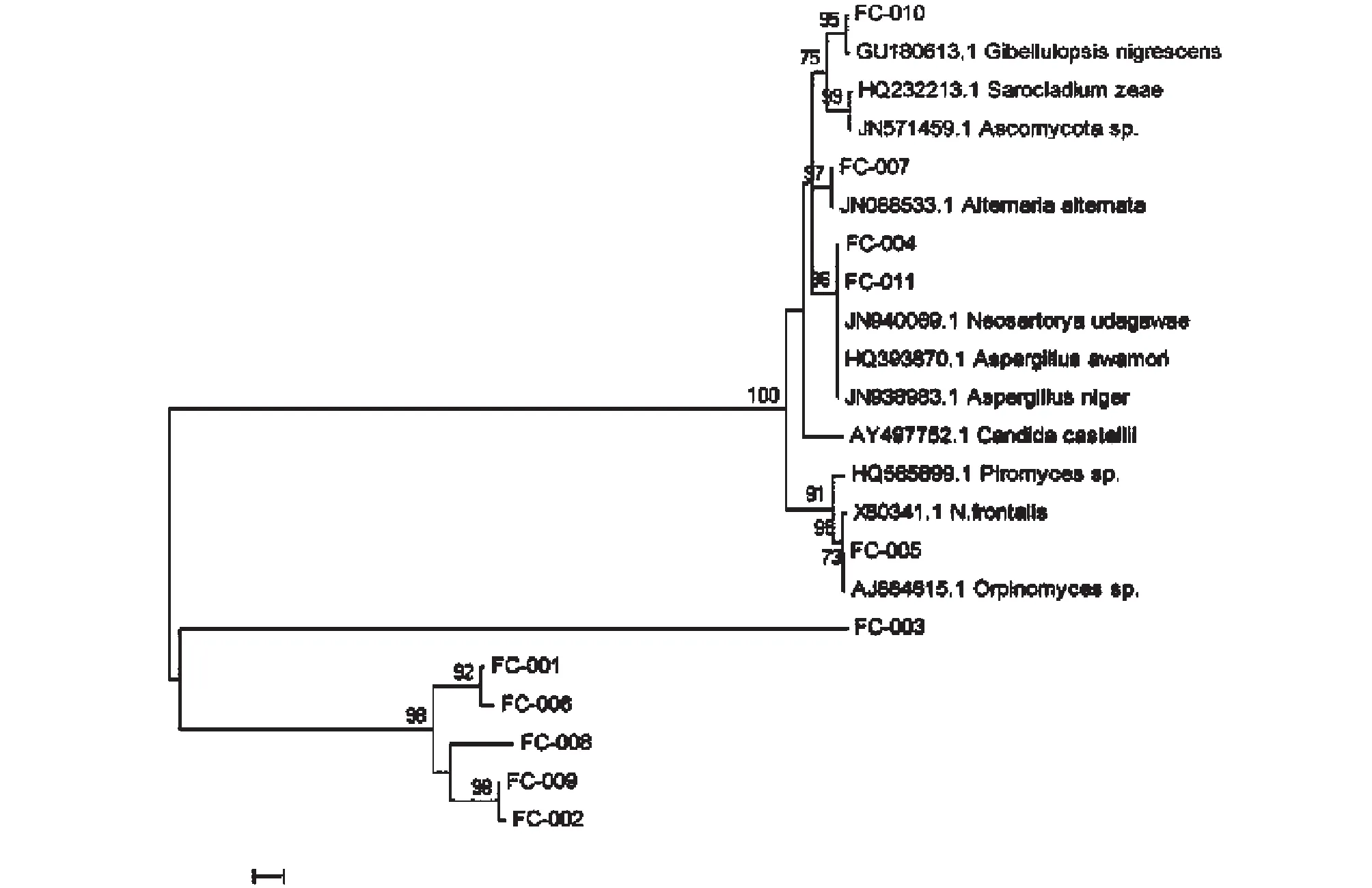
图2 混合饲料饲养牛瘤胃真菌18S rRNA基因序列系统发育树Fig.2 Phylogenetic tree of fungual 18S rRNA gene sequences from Miscanthus sinensis (-) library
比较试验组和对照组18S rRNA基因文库序列信息发现,混合饲料饲养的瘤胃真菌比对信息所包含的种类比较分散,如Orpinomycessp.,Sarocladiumzeae,Aspergillusniger,Piromycessp.,Alternariaalternata,Candidacastellii,Ascomycotasp.,Gibellulopsisnigrescens,Neosartoryaudagawae,而芒草单一喂养的牛瘤胃真菌包含的种类则相对集中,主要集中在Orpinomycessp.,Piromycessp.,Neocallimastixfrontalis,Microdochiumsp.几种 (表3)。
3 讨 论
纤维素、半纤维素以及木质素经多种化学键复合而形成天然木质纤维素,这种复杂的高分子化合物很难被任何单一的水解酶降解、破坏,因此需要多种酶协同作用完成其降解利用[9]。反刍动物之所以能够大量采食木质纤维含量高的植物茎杆类饲料,正是依靠瘤胃内的各种细菌、真菌及瘤胃原生动物所分泌的多种纤维素、木质素、半纤维素降解的酶的统一作用。其中厌氧真菌会优先附着于植物材料表面,通过真菌假根对植物细胞壁的降解作用而进入植物维管组织内,这种物理性降解,能够削弱木质化组织的抗性,穿透植物表皮角质层屏障,使得内部的成分暴露,更容易接触瘤胃微生物产生的降解酶[10]。真菌在植物材料内部扩张,使植物纤维变得疏松,更加利于其它微生物与植物材料的接触。在降解植物细胞壁的同时,厌氧真菌还分泌多种纤维素酶,据Lee等研究证实,厌氧真菌所产生的纤维素酶活力比目前工业上常用的纤维素酶活力高出很多倍[11]。
由于受到纯分离培养条件的限制,对于瘤胃内厌氧真菌多样性的认识一直不够全面。沈赞明等从水牛瘤胃中分离得到Piromycessp.,Neocallimastixsp.,Orpinomycessp.,分离到的Neocallimastixsp.和Orpinomycessp.具有CMCase活力[12]。用不同饲料喂养牛,由于饲料中植物种类不同,其木质纤维的组成成分也不相同,因此降解过程所依赖的真菌种类和比例也有相应变化[13]。另一项对日粮组成影响瘤胃厌氧真菌的研究表明,当日粮组成纤维含量较高时,厌氧真菌的数量增加,反之厌氧真菌数量则低[1,14]。
4 结 论
本文通过与数据库已知序列的比对,得到芒草驯养牛瘤胃真菌也集中在Orpinomycessp.,Piromycessp.,Neocallimastixfrontalis几种,并与已知瘤胃内纤维降解菌种类部分一致。而在混合饲料饲养的牛瘤胃内真菌种类较多,不集中。这也从一定程度上说明了长期单一饲喂芒草对于瘤胃内的厌氧真菌种类有较大影响,且芒草驯养很可能使瘤胃真菌种类显现出更利于降解纤维质材料的方向。Orpinomycessp.,Piromycessp.,Neocallimastixsp.等属与芒草降解的具体关系需要进一步深入研究加以确定。对瘤胃厌氧真菌认识的进一步丰富,有助于更好掌握瘤胃微生态总体变化,以及更加深入地利用其分泌的多种纤维质降解酶所形成的酶系统,并很可能为芒草这一高纤维质含量的非粮食类植物在生物燃料方向的开发利用扩宽了降解酶资源。
[1]Bauchop T. The rumen anaerobic Fungi:colonizers of plant fibre[J]. Ann Rech Vet, 1979, 10:246-248.
[2]Lan GQ, Abdullah N.Mitsuokellajalaludiniisp. nov., from the rumens of cattle in Malaysia[J]. International Journal of Systematic and Evolutionary Microbiology, 2002, 52:713-718.
[3]Wallace R J, McEwan N R, Miyagawa E, et al.Eubacteriumpyruvativoranssp. nov., a novel non-saccharolytic anaerobe from the rumen that ferments pyruvate and amino acids, forms caproate and utilizes acetate and propionate[J]. International Journal of Systematic and Evolutionary Microbiology, 2003, 53:965-970.
[4]Shichun C, Xiuzhu D.Cellulosilyticumruminicolagen. nov., sp. nov., isolated from yak rumen contents, and reclassification ofClostridiumlentocellumasCellulosilyticumlentocellumcomb[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60:845-849.
[5]AkinDE,WindhamWR.Influenceofdietonrumenfungi.Intheroleofprotozoaandfungiinruminantdigestion(OECD/UNEInter-nationalseminal) (EdsJVNolan,LengRA,DemeyerDI)[M].Pe-nambulbooksArmidale,NewSouthWales,Australia,1989:75-82.
[6]Wilson C A. Studies on the cellulase of the rumen anaerobic fungus Neoeallimastrix orfntalis,with special reference to the capacity of the enzyme to degrade[J]. Enzyme and Microbial Teehnology, 1994, 14:258-264.
[7]李亚丹. 芒草驯养湘西黄牛(Bostaurus)瘤胃微生物多样性及β-葡萄糖苷酶新基因的克隆与表达研究[D]. 长沙: 湖南农业大学博士论文, 2012.
[8]Jorgensen H B, Johansson T, Canback B, et al. Selective foraging of fungi by collembolans in soil[J]. Biology Letter, 2005(1):243-246.
[9]朱永涛, 刘巍峰, 王禄山, 等. 不依赖微生物培养的纤维素降解酶及基因资源的挖掘[J]. 生物工程学报, 2009, 25: 1838-1843.
[10]张 辉. 瘤胃内真菌降解粗纤维的研究进展[J]. 黄牛杂志, 1997, 23: 49-51.
[11]Lee S S, Shin K J, Kim W Y, et al. The rumen ecosystem: As a fountain source of novel enzyme [J]. Asian Aust J Anim Sci, 1999, 12(6): 988-1001.
[12]沈赞明, 韩正康. 水牛瘤胃厌氧真菌的分离和鉴定[J]. 南京农业大学学报, 1993, S1.
[13]Dipti W P, Scott E D. Rumen Bacterial Diversity Dynamics Associated with Changing from Bermudagrass Hay to Grazed Winter Wheat Diets[J]. Microbiology Ecology, 2010, 59(3):511-522.
[14]Orpin C G, Joblin K N. The rumen anaerobic fungi[M]. In:Hobson P N, Stewart C S(Eds), Rumen Microbial Eeosystem. London, UK: Blaekie Aeademic & Professional, 1997:140-195.
EvaluationofFungalDiversityinRumenofYellowCattle(Bostaurus)FedMiscanthussinensisorCommonMixedFeedstuff
LI Ya-dan1,2,YANG Hui1,LU Xiang-yang1,TIAN Yun1*
(1.College of Bioscience and Biotechnology, Hunan Agricultural University, Hunan Changsha 410128, China;2.College of Environmental Science, Changsha Environmental Protection College, Hunan Changsha 410004, China)
【Objective】This study aimed to detect whetherMiscanthussinensiscould increase the special population of rumen fibrolitic fungi. 【Methods】The experiment was conducted by constructing two 18S rRNA gene libraries using ruminal samples from yellow cattle fed with either common mixed feedstuff (group C) orM.Sinensis(group M), and the diversity of ruminal fungi in the rumens of cattle of both groups was identified. 【Results】Based on the comparative analysis of these two groups, the fungal composition in group C/M was found to be: there wereOrpinomycessp.,Sarocladiumzeae,Aspergillusniger,Piromycessp.,Alternariaalternata,Candidacastellii,Ascomycotasp.,Gibellulopsisnigrescens,Neosartoryaudagawaein rumen of mixed forage feeding cattle, in contrast, the species in rumen ofMiscanthussinensisfeeding cattle was concentrated relatively:Orpinomycessp.,Piromycessp.,Neocallimastixfrontalis,Microdochiumsp..【Conclusion】Feeding cattle withM.sinensiswill change the fungal composition in the rumen; the increased fungi may be responsible for digestingM.sinensis, which will benefit us in further screening of potentially valuable bio-enzymes.
Cattle;Rumen;Miscanthussinensis;18S rRNA;Fungi
1001-4829(2017)10-2371-05
10.16213/j.cnki.scjas.2017.10.035
2016-12-05
湖南省战略性新兴产业关键共性技术导向类项目(2015GK1017);湖南省教育厅科学研究项目(15K058, 16C0018)
李亚丹(1984-),女,河北石家庄人,博士研究生,工程师,环境微生物技术,E-mial: 469480533@qq.com;*为通讯作者:田 云,男,湖南沅江人,博士研究生,教授,微生物学,E-mial: tianyun@hunau.edu.cn。
S182
A
(责任编辑 李 洁)

