非小细胞肺癌中叉头框蛋白Q1和β连环蛋白的表达及临床意义
胡余敏,朱君飞
(1.临海市第一人民医院,浙江 临海 317000;2.台州市中心医院,浙江 台州 318000)
非小细胞肺癌中叉头框蛋白Q1和β连环蛋白的表达及临床意义
胡余敏1,朱君飞2
(1.临海市第一人民医院,浙江 临海 317000;2.台州市中心医院,浙江 台州 318000)
目的探讨非小细胞肺癌(NSCLC)组织中叉头框蛋白Q1(FOXQ1)和β连环蛋白(β-catenin)的表达及其临床意义。方法采用免疫组化En Vision法检测60例NSCLC和对应癌旁组织中FOXQ1和β-catenin的表达情况,并结合NSCLC患者临床病理资料进行分析。结果NSCLC组织中FOXQ1和β-catenin阳性表达率分别为63.33%(38/60)和 71.67%(43/60),均明显高于对应癌旁正常组织的 13.33%(9/60)和 0.00%(0/60),差异均有统计学意义 (P<0.05);FOXQ1和β-catenin蛋白表达均与临床分期及淋巴结转移有关 (P<0.05);NSCLC组织中FOXQ1和 βcatenin之间的表达呈显著正相关(r=0.662,P<0.01)。结论NSCLC组织中FOXQ1和β-catenin表达升高,其异常表达与NSCLC的转移和临床分期密切相关,FOXQ1有望成为NSCLC新的治疗靶点。
非小细胞肺癌;叉头框蛋白Q1;β连环蛋白
非小细胞肺癌(non-smallcelllungcancer,NSCLC)约占肺癌的80%~85%[1]。尽管分子机制的研究和综合治疗取得了进展,但5年生存率仍然较低[2]。因此,有必要寻找可用于NSCLC临床诊断和治疗的新分子靶点。叉头框蛋白Q1(Forkhead box Q1,FOXQ1)是FOX家族的新成员,属一种肿瘤癌基因,参与多种信号通路,调节细胞增殖、血管生成和凋亡等,促进肿瘤的发生发展[3-4]。β-连环蛋白(βcatenin)是连环蛋白家族成员之一,参与Wnt信号通路,通过与Wnt靶基因作用,诱导其转录,在肿瘤细胞的增殖、侵袭和转移过程中起着重要作用[4-5]。本研究采用免疫组化En Vision法检测FOXQ1和β-catenin在NSCLC和癌旁组织中的表达特点及其与NSCLC临床病理因素的关系,进一步分析两者的相关性,初步探讨FOXQ1和β-catenin表达与NSCLC发生、发展的关系,报道如下。
1 资料与方法
1.1 一般资料 收集2008年1月~2015年12月临海市第一人民医院20例和台州市中心医院40例手术切除的NSCLC标本,具有完整临床资料,均经病理明确诊断,术前未接受放疗和化疗。60例NSCLC中,男 35例,女 25例,年龄 30~80岁,中位年龄48岁,<60岁20例,≥60岁40例。参照2004年WHO肺肿瘤组织学分类标准[6]:鳞癌24例,腺癌36例。高中分化42例,低分化18例。依据2010年国际抗癌联盟(UICC)肺癌TNM分期标准[7]:Ⅰ期19例,Ⅱ期20例,Ⅲ期21例。有淋巴结转移者27例,无淋巴结转移者33例。同时另取同期距离肿块边缘5cm外的癌旁正常组织60例作为对照。
1.2 方法 (1)主要试剂:兔抗人FOXQ1单克隆抗体(ab99536)购自英国Abcam公司,鼠抗人βcatenin单克隆抗体(M-0442)购自美国Santa Cruz公司,即用型羊抗兔和兔抗鼠免疫组化En VisionTM检测试剂盒和DAB显色剂均购自北京中杉金桥生物科技有限公司。(2)检测方法:所有标本均采用10%的中性甲醛固定,常规石蜡包埋、切片、染色。取石蜡组织常规连续切片,厚约4μm,采用免疫组化En Vision二步法,实验步骤按照试剂盒说明书操作。用已知阳性片作阳性对照,以PBS液代替一抗作为阴性对照。
1.3 结果判定 免疫组化染色阳性信号为出现黄色至棕黄色颗粒。FOXQ1阳性表达位于细胞浆。FOXQ1细胞浆染色阳性细胞数≥10%为阳性,<10%为阴性。β-catenin≥90%细胞膜连续染色为正常表达,<90%细胞膜着色或无表达或胞质或胞核染色为异常表达,β-catenin胞质或胞核染色阳性细胞数≥10%为阳性,<10%为阴性。观察5个以上高倍视野(×400),每个视野计数≥200个细胞。
1.4 统计学处理 采用SPSS17.0软件对计数资料进行χ2检验,FOXQ1和β-catenin表达的相关性采用Spearman等级相关分析。

图1 FOXQ1在NSCLC组织中的阳性表达(En Vision×400)

表1 NSCLC和癌旁正常组织中FOXQ1和β-catenin的表达(n,%)
2 结果
2.1 NSCLC和癌旁正常组织中FOXQ1和βcatenin表达的比较 FOXQ1细胞浆出现黄色至棕黄色颗粒 (图1),β-catenin在NSCLC组织中主要于细胞质或细胞核内呈现黄色至棕黄色颗粒 (图2)。NSCLC组织中FOXQ1和β-catenin阳性表达率均明显高于对应癌旁组织,差异有统计学意义(均 P<0.05)。 详见表 1。

图2 β-catenin在NSCLC组织中的阳性表达(En Vision×400)
2.2 FOXQ1和β-catenin表达与NSCLC临床病理因素的关系 随着NSCLC临床分期进展及发生淋巴结癌转移,FOXQ1和β-catenin蛋白阳性表达率进一步升高,差异有统计学意义(P<0.05),而在不同年龄、性别、组织学类型及分化程度间的表达差异无统计学意义 (P>0.05)。详见表2。
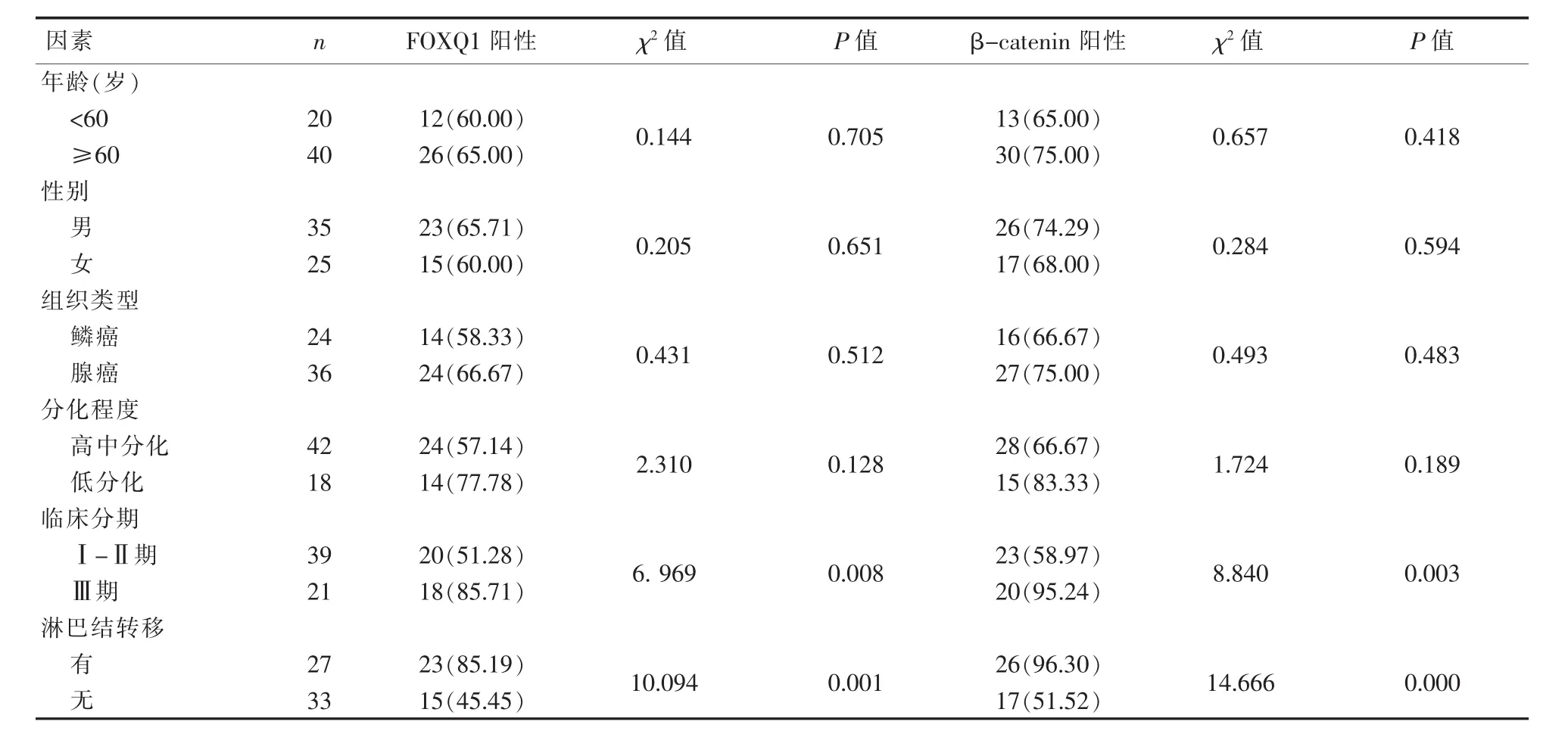
表2 FOXQ1和β-catenin表达与NSCLC临床病理因素的关系(n,%)
2.3 NSCLC组织中FOXQ1和β-catenin表达的相关性 60例NSCLC组织中FOXQ1和β-catenin表达共阳性者36例,共阴性者15例,NSCIC组织中FOXQ1 和 β-catenin 表达呈正相关(r=0.662,P<0.01)。
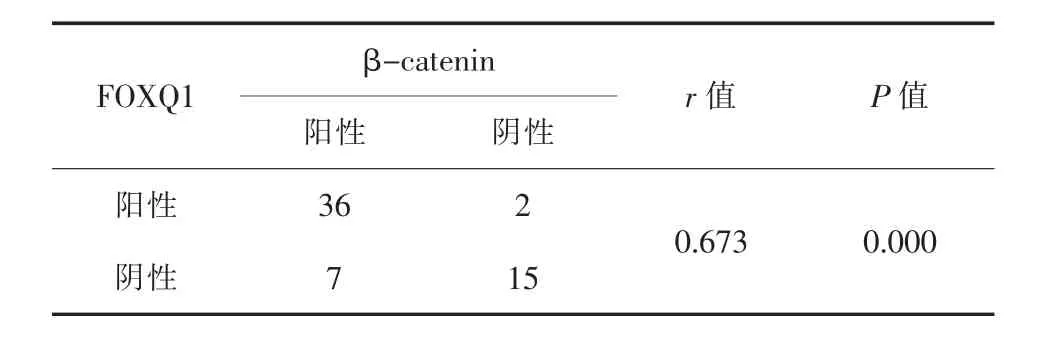
表3 NSCLC组织中FOXQ1和β-catenin表达的相关性
3 讨论
FOXQ1是FOX超家族的成员之一,广泛存在于人体的多种组织和器官,参与胚胎干细胞和脊椎动物的发育过程。FOXQ1定位于6p25.3,仅包含1个外显子,编码403个氨基酸残基的蛋白质。FOXQ1通过靶基因启动子区域的GC盒等核心元件,调节基因转录,参与细胞周期、细胞信号传导等多种生物学功能[8]。FOXQ1与肿瘤关系的研究近年来备受关注。研究证实,FOXQ1在大肠癌[9]、胃癌[10]等多种恶性肿瘤中呈高表达,与肿瘤的分化程度、TNM分期和淋巴结转移密切相关,并且FOXQ1高表达者预后差。Pei等[11]报道,与癌旁组织比较,FOXQ1在食道癌组织中呈高表达。FOXQ1过度表达促进食道鳞癌肿瘤细胞的增殖,而沉默FOXQ1表达能预防食管癌细胞增殖。FOXQ1可以作为转录抑制因子结合到CDH1启动子并阻止其转录。因此,FOXQ1可作为癌基因,通过负调节CDH1促进食管癌细胞的增殖和转移。本研究显示,NSCLC组织中FOXQ1表达率明显高于癌旁正常组织 (P<0.05),与文献报道[10-11]一致。通过分析FOXQ1表达与NSCLC临床病理因素的关系,结果发现FOXQ1蛋白表达在NSCLC患者不同临床分期和淋巴结是否转移间的差异有统计学意义(P<0.05),而在患者不同年龄、性别、组织学类型及分化程度上差异无统计学意义(P>0.05)。与一些文献报道“FOXQ1表达与肿瘤的分化程度有关”不一致,可能与不同部位来源的肿瘤有关[9-10]。表明FOXQ1蛋白表达越高,肿瘤淋巴结相对更容易发生转移,且临床分期更高,预后较差。
β-catenin是一种连接蛋白,主要表达于细胞膜,参与细胞间的连接。由于游离的β-catenin在细胞质中经磷酸泛素化,进而被蛋白酶体水解,因此始终保持在较低水平[12]。而在病理情况下,Wnt信号通路被激活时,起着向细胞核传递信号的作用。β-catenin在细胞膜中的正常表达下降,而在细胞浆和细胞核中异常增多,参与肿瘤细胞增殖、侵袭和转移过程,分化程度越低的肿瘤组织中βcatenin的异常表达率越高[13]。本研究中β-catenin在NSCLC组织中的异常表达率显著高于癌旁组织,差异有统计学意义(P<0.05)。β-catenin异常表达在NSCLC患者不同临床分期和淋巴结是否转移间的差异有统计学意义(P<0.05),而与患者年龄、性别、组织类型及分化程度无关。提示β-catenin在胞质或胞核的异常表达与NSCLC的发生、淋巴结转移及临床分期密切相关。
Spearman等级相关分析结果显示,NSCLC组织中FOXQ1和β-catenin表达呈正相关,是一对协同作用的因素,提示FOXQ1和β-catenin在NSCIC的发生、发展过程起正向调控作用。ChIP和荧光素报告实验显示[14],TCF-4高度保守的结合区域(TCAAAG)可结合FOXQ1基因启动子区域,直接导致β-catenin启动FOXQ1基因的转录。故βcatenin通过调控FOXQ1在肿瘤的发生、进展中发挥作用,这也提示FOXQ1有望成为NSCLC新的治疗靶点。
[1] Siegel RL,Miller KD,Jemal A.Cancer statistics, 2015.CA Cancer J Clin,2015,65(1):5
[2] Tang Q,Wu J,Zheng F,et al.Emodin Increases Expression of Insulin-Like Growth Factor Binding Protein 1 through Activation of MEK/ERK/AMPKα and Interaction of PPARγ and Sp1 in Lung Cancer.Cell Physiol Biochem,2017,41(1):339
[3] Peng X, Luo Z, Kang Q, et al.FOXQ1 mediates the crosstalk between TGF-β and Wnt signaling pathways in the progression of colorectal cancer.Cancer Biol Ther, 2015,16(7):1099[4] Fan DM, Feng XS,Qi PW,et al.Forkhead factor FOXQ1 promotes TGF-betal expression and induces epithelialmesenchymal transition.Mol Cell Biochem, 2014,397(1-2):179
[5] Wang JS, Ji AF, Wan HJ, et al.Gene silencing of βcatenin by RNAi inhibits proliferation of human esophageal cancer cells by inducing G0/G1 cell cycle arrest.Asian Pac J Cancer Prev,2012,13(6):2527
[6] Travis WD, Brambilla E,Muller-Hermelink HK,et al.The WHO classification of tumours:pathology and genetics of tumours of the lung, pleura, thymus and heart,chapter 3 tumours of the thymus.Lyon:IARC Press,2004:9
[7] Giroux DJ,Rami-Porta R,Chansky K, et al.International association for the study of lung cancer international staging committee.The IASLC lung cancer staging project:data elements ofr the prospective project.J Thorac Oncol,2009,4(5):679
[8] 吴红雁,范方田,刘兆国,等.转录因子叉头框Q1与肿瘤相关性的研究进展.肿瘤,2014,34(7):662
[9] Weng W,Okugawa Y,Toden S, et al.FOXM1 and FOXQ1 are Promising Prognostic Biomarkers and Novel Targets of Tumor-Suppressive miR-342 in Human Colorectal Cancer.Clin Cancer Res,2016,22(19):4947
[10]Zhang J, Liu Y, Zhang J, et al.FOXQ1 promotes gastric cancer metastasis through upregulation of Snail.Oncol Rep,2016,35(6):3607
[11]Pei Y,Wang P,Liu H,et al.FOXQ1 promotes esophageal cancer proliferation and metastasis by negatively modulating CDH1.Biomed Pharmacother,2015,74:89
[12]Isaeva AV, Zima AP, Shabalova IP, et a1.β-Catenin:structure,function and role in malignant transformation of epithelial Cells.Vestn Ross Akad Med Nauk,2015,23(4):475
[13]Tang X, Fan Z, Wang Y, et al.Expressidn of klotho and βcatenin in esophageal squamous cellcarcinoma,and their clinicopathological and prognostic significance.Dis Esophagus,2016,29(3):207
[14]Christensen J, Bentz S, Sengstag T, et al.FOXQ1, a novel target of the Wnt pathway and a new marker for activation of Wnt signaling in solid tumors.PLoS One, 2013,8 (3):e60051

