FGFR3基因单核苷酸多态与绝经前乳腺癌易感性的关联研究
李 伟 姜永冬 庞 达
FGFR3基因单核苷酸多态与绝经前乳腺癌易感性的关联研究
李 伟 姜永冬 庞 达
目的探讨FGFR3基因单核苷酸多态(SNPs)与女性绝经前乳腺癌的风险关系。方法采用多重单碱基延伸SNP分型技术(Snapshot)检测FGFR3基因的rs2234909和rs3135848的SNP基因型在绝经前乳腺癌患者和绝经前正常女性人群中的频率,并分析不同SNP基因型与绝经前乳腺癌发病的风险关系。结果FGFR3基因rs2234909和rs3135848的SNP基因型的频率在乳腺癌与对照组间无统计学差异(P>0.05)。Logistic回归分析结果显示,对于rs2234909位点,相比较于TT基因型,TC和TC+CC基因型和乳腺癌的发病风险无显著相关性(OR=1.035,95%CI:0.680~1.575,P=0.874;OR=0.985,95%CI:0.638~1.521,P=0.945);对于rs3135848位点,相比较于TT基因型,TC、CC和TC+CC基因型与乳腺癌的发病风险无关(OR=1.177,95%CI:0.846~1.636,P=0.333;OR=0.948,95%CI:0.287~3.137,P=0.931;OR=1.162,95%CI:0.548~1.112,P=0.360)。rs2234909位点突变的乳腺癌患者与未突变者相比,组织学分级(显性模型:P=0.032;共显性模型:P=0.024)以及Ki67指数(显性模型:P=0.056;共显性模型:P=0.044)显著增高;rs3135848位点突变及两位点均突变与乳腺癌患者临床病理特征无显著相关性(P>0.05)。结论FGFR3基因的rs2234909和rs3135848两位点基因多态性与乳腺癌易感性无明显相关性;而rs2234909位点突变在绝经前乳腺癌患者中与组织学分级和Ki67指数呈正相关,可能提示预后不良。
乳腺癌;FGFR3基因;单核苷酸多态;遗传易感性
乳腺癌是女性最常见的恶性肿瘤之一,其发病率在世界范围内逐年增加并呈现年轻化趋势[1]。与欧美国家发病状况相反,相比于绝经后乳腺癌患者,我国绝经前乳腺癌患者明显占有更大的比例,这可能是中国人群乳腺癌的特点[2]。目前针对乳腺癌的发生发展有大量研究报道,但其具体原因尚不明确。大量的研究结果显示遗传因素和环境因素的相互作用在乳腺癌的发病过程中发挥着重要的促进作用。BRCA1和BRCA2等高外显率易感基因在乳腺癌的发生、发展过程中发挥了重要的作用[3]。此外,一些低外显率的基因也同样扮演着不可或缺的角色。成纤维生长因子受体3(Fibroblast growth factor receptor 3,FGFR3)是一种受体型酪氨酸激酶,其在调节细胞增殖、分化、血管形成以及某些恶性肿瘤的致瘤过程中发挥重要作用[4-6]。然而,有关FGFR3的基因多态性与乳腺癌易感性的研究国内外鲜有报道。因此,本研究选取FGFR3基因的rs2234909和rs3135848这两个位点的单核苷酸多态作为研究对象,探索其与乳腺癌发病风险的关系。同时,因月经状态也影响着乳腺癌的发生[7-8],所以本研究选取所有病例标本皆为绝经前女性患者。运用病例对照研究针对FGFR3基因单核苷酸多态在绝经前乳腺癌及正常女性人群的频率进行分析,进而揭示FGFR3基因的rs2234909和rs3135848位点单核苷酸多态性与绝经前乳腺癌的发病风险关系,为乳腺癌发病机制研究提供更深层的证据。
1 对象与方法
1.1 研究对象
本研究选取2008年11月—2011年4月在哈尔滨医科大学附属肿瘤医院就诊的绝经前原发乳腺癌415例病例,所有患者均由两名独立病理科医生经组织病理学确诊。入组病例均未进行化疗、放疗以及内分泌治疗,无其他肿瘤患病史和肿瘤遗传史,平均年龄(43.53±7.01)岁。同时,在哈尔滨医科大学附属第二医院体检中心随机选取457例健康普查绝经前女性作为对照组,对照组中所有样本体检合格,无乳腺疾病患病史,无恶性肿瘤及肿瘤遗传史,平均年龄(43.14±6.16)岁。所有研究对象均获取了知情同意书,本研究经哈尔滨医科大学伦理委员会审批通过。
1.2 方法
1.2.1 基因组DNA提取 所有入组的乳腺癌患者及正常女性人群抽取2 mL外周静脉血,应用DNA提取试剂盒(AxyPrep血基因组DNA小量制备试剂盒,AP-MN-BL-GDNA)抽提基因组DNA,测定浓度及纯度后,置于-20℃保存备用。
1.2.2 PCR及基因型的测序鉴定 应用多重单碱基延伸SNP分型技术(Snapshot)对FGFR3基因rs2234909和rs3135848的SNP位点进行检测分型。应用PCR扩增包含上述SNP位点的核酸片段,设计紧邻SNP位点的延伸引物用于多重单碱基延伸(表1)。(1)PCR反应:PCR反应体系(总量20 μL):1×HotStarTaq buffer,dNTP 0.3 mmol/L,Mg2+3.0 mmol/L,上下游引物各0.1 μmol/L,HotStarTaq polymerase 1 U和样本DNA 1 μL。运用Touch-down PCR对目的片段进行扩增,反应条件:95℃预变性15 min→94℃变性20 s,65℃退火40 s,72℃延伸90 s(共11个循环,每个循环退火温度降低0.5℃)→94℃变性20 s,59℃退火30 s,72℃延伸90 s(共24个循环)→72℃延伸2 min→4℃。应用SAP/Exo I针对上述PCR产物进行纯化,简要步骤如下:10 μL PCR产物中加入SAP酶、Exonuclease I酶各1 U,37℃温浴1 h后,75℃灭活15 min,产物置于-20℃保存;(2)Snapshot多重单碱基延伸反应:应用Snapshot Multiplex试剂盒针对上述纯化后的PCR产物进行延伸反应并鉴定基因型。反应体系(总量10 μL):Snapshot Multiplex Kit 5 μL,纯化后多重PCR产物2 μL,延伸引物混合物(正反引物浓度均为0.8 mmol/L)1 μL,超纯水(PCR级别)2 μL。反应条件:96℃预变性1 min→96℃变性10 s,50℃退火5 s,60℃延伸30 s(共28个循环)→4℃。应用酶法对延伸产物进行纯化,体系如下:延伸产物10 μL,加入SAP酶1 U,37℃温浴1 h后75℃灭活15 min;(3)实验结果分析:取上述纯化后的延伸产物0.5 μL与内标Liz120 0.5 μL、甲酰胺9 μL混匀,95℃变性5 min后,应用ABI 3130XL基因测序仪进行毛细管电泳,实验结果应用GeneMapper 4.0软件进行分析。
1.3 统计分析
应用SPSS 18.0进行统计分析,基因型在病例组与对照组之间分布差异以卡方检验统计分析,各基因型和乳腺癌风险的关系采用Logistic回归分析,P<0.05为差异有统计学意义。
2 结果
2.1 FGFR3基因rs2234909和rs3135848多态性位点基因型的频率
在415例乳腺癌病例组中,rs2234909位点的TT、CT和CC基因型频率分别为89.6%、9.9%和0.5%;在457例对照组中,rs2234909位点的TT、CT和CC基因型频率分别为89.5%、10.5%和0,其分布符合Hardy-Weinberg平衡定律(P=0.23)。上述3种基因型频率分布在乳腺癌与正常女性人群中无统计学差异(P>0.05)(表2)。

表1 SNP位点PCR引物序列
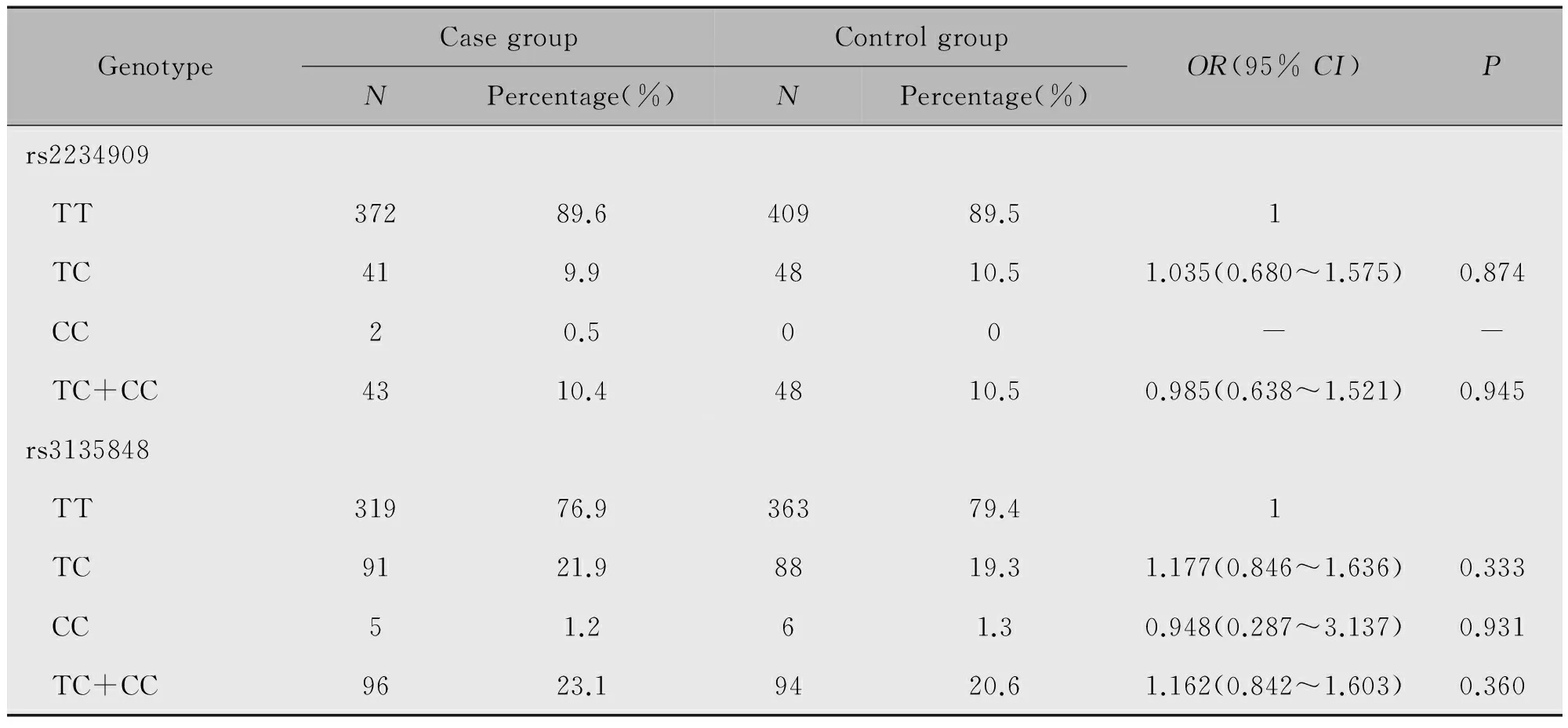
表2 FGFR3基因型频率在病例组和对照组的分布
在415例乳腺癌病例组中,rs3135848位点的TT、TC和CC基因型频率分别为76.9%、21.9%和1.2%;在457例正常对照组中,rs3135848位点的TT、TC和CC基因型频率分别为79.4%、19.3%和1.3%,其分布同样符合Hardy-Weinberg平衡定律(P=0.79)。rs3135848位点的上述3种基因型频率分布在乳腺癌与正常女性人群中无统计学差异(P>0.05)(表2)。
2.2 FGFR3基因rs2234909和rs3135848多态性位点基因型与乳腺癌发病风险
在共显性模型下,与野生纯合型TT相比,rs2234909位点的CT杂合型与乳腺癌的患病风险无显著相关性(OR=1.035,95%CI:0.680~1.575,P=0.874);与TT基因型相比,CT+TT基因型与乳腺癌的患病风险无显著相关性(OR=0.985,95%CI:0.638~1.521,P=0.945),在共显性模型下,与野生纯合型TT相比,rs3135848位点CT杂合型和突变纯合型CC均与乳腺癌的患病风险无显著相关性(OR=1.177,95%CI:0.846~1.636,P=0.333;OR=0.948,95%CI:0.287~3.137,P=0.931);与TT基因型相比,CT+TT基因型与乳腺癌的患病风险无显著相关性(OR=1.162,95%CI:0.842~1.603,P=0.360)(表2)。
2.3 FGFR3基因rs2234909和rs3135848多态性位点基因型与乳腺癌临床病理因素相关性
在415例乳腺癌患者中,与未突变者相比,FGFR3基因SNP位点rs2234909突变的乳腺癌患者组织学分级更高(显性模型:P=0.032;共显性模型:P=0.024),Ki67指数更高(显性模型:P=0.056;共显性模型:P=0.044),均具有统计学差异(表3);FGFR3基因SNP位点rs3135848位点突变及两位点均突变与乳腺癌患者临床病理因素无关(P>0.05)(表4,表5)。

表3 FGFR3基因rs2234909位点与乳腺癌临床病理因素的相关性
Note:P1 represents comparison of genotype TT、TC and CC;P2 represents comparison of genotype TT and TC+CC.
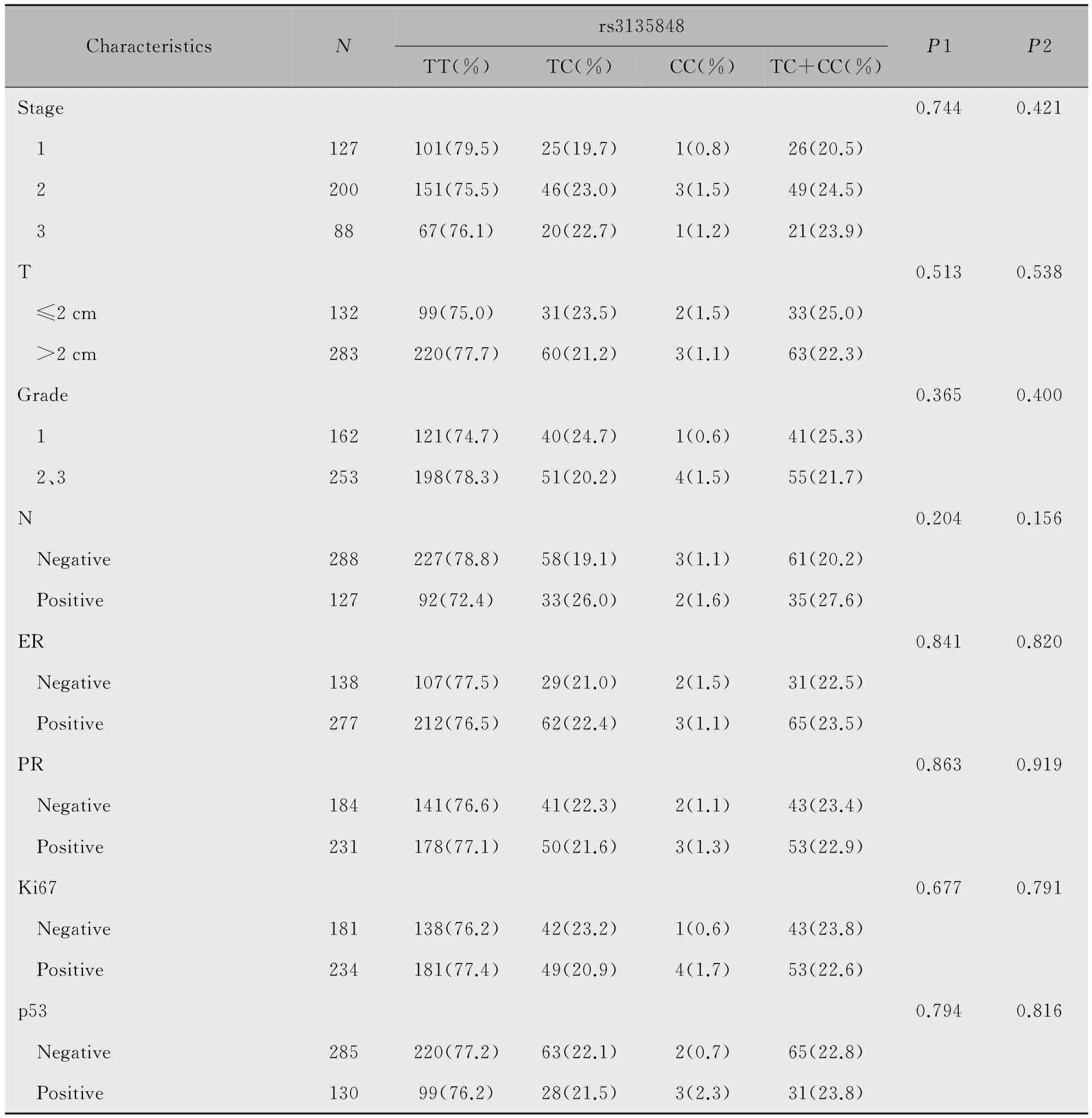
表4 FGFR3基因rs3135848位点与乳腺癌临床病理特征的关系
Note:P1 represents comparison of genotype TT、TC and CC.P2 represents comparison of genotype TT and TC+CC.

表5 FGFR3基因rs2234909和rs3135848位点共同突变与乳腺癌临床病理特征的关系
3 讨论
FGFR3基因位于人染色体4p16.3,碱基长度约16.5 kb,含有19个外显子和18个内含子[9]。FGFR3通常以两种形式存在:FGFR3Ⅲc和FGFR3Ⅲb[10]。以往的研究主要集中在FGFR3基因突变与骨发育不良相关的人类遗传性疾病的相关性,例如软骨发育不全或致死性骨发育不全等先天性异常可能与FGFR3基因的某些位点的突变存在相关性[11-15]。近期有研究证明,人类某些肿瘤的发生发展同FGFR3的突变同样存在相关性:在多发性骨髓瘤中发现FGFR3基因点突变[16-17],在尿路上皮肿瘤、膀胱癌、口腔癌和大肠癌等中发现FGFR3基因的体细胞激活突变[18-21]。
Zammit等[22]通过对80例乳腺癌样本和32例非恶性组织样本应用免疫组织化学染色检测FGFR3的表达情况,结果显示,在恶性及非恶性上皮细胞中FGFR3的表达水平无显著差异;然而,他们却发现在人乳腺癌细胞内发生以细胞核染色为主的FGFR3定位的改变。对于造成FGFR3在恶性细胞细胞核中聚积的可能原因,Johnston等[23]研究者认为可能存在外显子缺失的FGFR3基因型,导致信号多肽及跨膜区的缺失。Penault-Llorca等[24]通过对103例经手术治疗的原发性乳腺癌样本进行RT-qPCR及Northern blot分析,在乳腺癌样本中未检测到FGFR3的表达上调。本研究抛开FGFR3的表达差异,选择FGFR3的两个位点进行基因多态性检测,期许能够从基因多态性角度解释FGFR3与乳腺癌易感性的相关性。同时,本研究选取了绝经前乳腺癌患者及正常女性人群作为研究对象,从而排除月经状态对乳腺癌易感性的影响。本研究结果显示,FGFR3基因rs2234909和rs3135848位点最小等位基因频率分别为0.054和0.122,与女性绝经前乳腺癌的发病风险无显著相关性。与此结果类似,在2014年Agarwal等[25]通过对53 835例乳腺癌患者及50 156例正常女性(包括46 450例乳腺癌欧洲女性及45 600例正常的欧洲女性)进行FGFR3的SNPs分析,结果显示欧洲女性中FGFR3的rs2234909和rs3135848位点与乳腺癌风险同样无显著相关性。此外,本研究针对FGFR上述位点的分布频率与乳腺癌临床病理因素的相关性进行分析,FGFR3基因SNP位点rs2234909突变与绝经前乳腺癌的组织学分级和Ki67指数呈现显著正相关,这一结果为FGFR3基因SNP位点在乳腺癌中占据的角色提供了初步证据,但尚需深层次机制的研究以进一步揭示其具体作用与功能。
综上所述,本研究针对FGFR3基因的遗传多态性与女性绝经前乳腺癌易感性进行关联研究,结果提示rs2234909和rs3135848两个位点未增加其发病风险,但rs2234909突变与绝经前乳腺癌的组织学分级和Ki67指数显著相关。然而,SNP因人群种族的不同可能存在不同结果和意义。因此,本研究结果尚需扩大样本量以及扩展至不同种族人群进行进一步研究以获取更多证据。
1 Siegel RL,Miller KD,Jemal A.Cancer statistics,2017[J].CA Cancer J Clin,2017,67(1):7-30.
2 Fan L,Strasser-Weippl K,Li JJ,et al.,Breast cancer in China[J].Lancet Oncol,2014,15(7):279-289.
3 Kwong A,Shin VY,Ho JC,et al.Comprehensive spectrum of BRCA1 and BRCA2 deleterious mutations in breast cancer in Asian countries[J].J Med Genet,2016,53(1):15-23.
4 Helsten T,Schwaederle M,Kurzrock R.Fibroblast growth factor receptor signaling in hereditary and neoplastic disease:biologic and clinical implications[J].Cancer Metastasis Rev,2015,34(3):479-496.
5 Katoh M.FGFR inhibitors:Effects on cancer cells,tumor microenvironment and whole-body homeostasis(Review)[J].Int J Mol Med,2016,38(1):3-15.
6 Parker BC,Engels M,Annala M,et al.Emergence of FGFR family gene fusions as therapeutic targets in a wide spectrum of solid tumours[J].J Pathol,2014,232(1):4-15.
7 Phipps AI,Ichikawa L,Bowles EJ,et al.Defining menopausal status in epidemiologic studies:A comparison of multiple approaches and their effects on breast cancer rates[J].Maturitas,2010,67(1):60-66.
8 Wang J,Yang DL,Chen ZZ,et al.Associations of body mass index with cancer incidence among populations,genders,and menopausal status:A systematic review and meta-analysis[J].Cancer Epidemiol,2016,42:1-8.
9 Lafitte M,Moranvillier I,Garcia S,et al.FGFR3 has tumor suppressor properties in cells with epithelial phenotype[J].Mol Cancer,2013,12:83.
10 Chellaiah AT,McEwen DG,Werner S,et al.Fibroblast growth factor receptor(FGFR)3.Alternative splicing in immunoglobulin-like domain III creates a receptor highly specific for acidic FGF/FGF-1[J].J Biol Chem,1994,269(15):11620-11627.
11 Pannier S,Couloigner V,Messaddeq N,et al.Activating Fgfr3 Y367C mutation causes hearing loss and inner ear defect in a mouse model of chondrodysplasia[J].Biochim Biophys Acta,2009,1792(2):140-147.
12 Pannier S,Mugniery E,Jonquoy A,et al.Delayed bone age due to a dual effect of FGFR3 mutation in Achondroplasia[J].Bone,2010,47(5):905-915.
13 Kanazawa TY,Bonadia LC,Cavalcanti DP.Frequency of the allelic variant c.1150T > C in exon 10 of the fibroblast growth factor receptor 3(FGFR3)gene is not increased in patients with pathogenic mutations and related chondrodysplasia phenotypes[J].Genet Mol Biol,2014,37(4):622-624.
14 Lindy AS,Basehore MJ,Munisha M,et al.Identification of a novel insertion mutation in FGFR3 that causes thanatophoric dysplasia type 1[J].Am J Med Genet A,2016,170(6):1573-1579.
15 Del Piccolo N,Placone J,Hristova K.Effect of thanatophoric dysplasia type I mutations on FGFR3 dimerization[J].Biophys J,2015,108(2):272-278.
16 Onwuazor ON,Wen XY,Wang DY,et al.Mutation,SNP,and isoform analysis of fibroblast growth factor receptor 3(FGFR3)in 150 newly diagnosed multiple myeloma patients[J].Blood,2003,102(2):772-773.
17 Saez B,Martin-Subero JI,Lahortiga I,et al.Simultaneous translocations of FGFR3/MMSET and CCND1 into two different IGH alleles in multiple myeloma:lack of concurrent activation of both proto-oncogenes[J].Cancer Genet Cytogenet,2007,175(1):65-68.
18 Christensen E,Birkenkamp-Demtroder K,Nordentoft I,et al.Liquid biopsy analysis of FGFR3 and PIK3CA hotspot mutations for disease surveillance in bladder cancer[J].Eur Urol,2017,71(6):961-969.
19 Chou A,Dekker N,Jordan RC.Identification of novel fibroblast growth factor receptor 3 gene mutations in actinic cheilitis and squamous cell carcinoma of the lip[J].Oral Surg Oral Med Oral Pathol Oral Radiol Endod,2009,107(4):535-541.
20 Zhang Y,Hiraishi Y,Wang H,et al.Constitutive activating mutation of the FGFR3b in oral squamous cell carcinomas[J].Int J Cancer,2005,117(1):166-168.
21 Kogita A,Yoshioka Y,Sakai K,et al.Inter-and intra-tumor profiling of multi-regional colon cancer and metastasis[J].Biochem Biophys Res Commun,2015,458(1):52-56.
22 Zammit C,Barnard R,Gomm J,et al.Altered intracellular localization of fibroblast growth factor receptor 3 in human breast cancer[J].J Pathol,2001,194(1):27-34.
23 Johnston CL,Cox HC,Gomm JJ,et al.Fibroblast growth factor receptors(FGFRs)localize in different cellular compartments.A splice variant of FGFR-3 localizes to the nucleus[J].J Biol Chem,1995,270(51):30643-30650.
24 Penault-Llorca F,Bertucci F,Adelaide J,et al.Expression of FGF and FGF receptor genes in human breast cancer[J].Int J Cancer,1995,61(2):170-176.
25 Agarwal D,Pineda S,Michailidou K,et al.FGF receptor genes and breast cancer susceptibility:results from the Breast Cancer Association Consortium[J].Br J Cancer,2014,110(4):1088-1100.
AssociationbetweensinglenucleotidepolymorphismsinFGFR3geneandriskofbreastcancer
LIWei,JIANGYongdong,PANGDa
Department of Breast Surgery,Harbin Medical University Cancer Hospital,Harbin 150081,China
ObjectiveThe aim of this study was to investigate the association between single nucleotide polymorphisms(SNPs)in FGFR3 gene and the risk of breast cancer.MethodsThe frequency of SNP genotypes rs2234909 and rs3135848 of FGFR3 gene in premenopausal breast cancer patients and premenopausal normal females were detected by multiple clonal extension SNP typing technique.The SNP genotypes were compared with different SNP genotypes and the risk of premenopausal breast cancer.ResultsThere was no difference in the genotype frequencies of SNP rs2234909 and rs3135848 between breast cancer and control groups(P>0.05).Logistic regression analysis showed that there was no correlation between TC and TC +CC genotype and risk of breast cancer(OR=1.035,95%CI:0.680~1.575,P=0.874;OR=0.985,95%CI:0.638~1.521,P=0.945).For the rs3135848 locus,the genotypes of TC,CC and TC+CC were not associated with the risk of breast cancer(OR=1.177,95%CI:0.846-1.636,P=0.333;OR=0.948,95%CI:0.287-3.137,P=0.931;OR=1.162,95%CI:0.548~1.112,P=0.360).Histological grade was significantly higher in breast cancer with rs2234909 mutation than that of the non-mutation group(dominant model:P=0.032,co-dominant model:P=0.024).The Ki67 index of FGFR3 gene locus rs2234909 mutation was higher than that of the non-mutation(dominant model:P=0.056;co-dominant model:P=0.044).There was no difference between rs3135848 mutation and both site mutation with clinicopathological features of breast cancer patients(P>0.05).ConclusionThe SNP genotypes of rs2234909 and rs3135848 of FGFR3 gene were not associated with susceptibility to breast cancer in premenopausal women in North of China.Rs2234909 mutation was positively correlated with histological grade and Ki67 index in premenopausal breast cancer patients.
Breast cancer;FGFR3 gene;Single nucleotide polymorphism;Genetic susceptibility
哈尔滨医科大学附属肿瘤医院乳腺外科(哈尔滨 150081)
李伟,男,(1991-),硕士研究生,从事乳腺癌基础与临床的研究。
庞达,E-mail:pangdasir1@sina.com
R737.9
A
10.11904/j.issn.1002-3070.2017.05.006
(收稿:2017-03-13)

