液滴数字聚合酶链式反应芯片及其在致病菌检测中的应用
邓雪蕾,张苑怡,袁浩钧,刘松生,陈莹莹,郜晚蕾,周洪波,贾春平,赵建龙,卞晓军,3,4*
(1.上海海洋大学 食品学院,上海 201306;2.传感技术国家重点实验室,中国科学院上海微系统与信息技术研究所,上海 200050;3.农业部水产品贮藏保鲜质量安全风险评估实验室(上海),上海 201306;4.上海海洋大学 食品学院 食品热加工工程技术研究中心,上海 201306)
液滴数字聚合酶链式反应芯片及其在致病菌检测中的应用
邓雪蕾1,张苑怡1,袁浩钧2,刘松生2,陈莹莹1,郜晚蕾2,周洪波2,贾春平2,赵建龙2,卞晓军1,3,4*
(1.上海海洋大学 食品学院,上海 201306;2.传感技术国家重点实验室,中国科学院上海微系统与信息技术研究所,上海 200050;3.农业部水产品贮藏保鲜质量安全风险评估实验室(上海),上海 201306;4.上海海洋大学 食品学院 食品热加工工程技术研究中心,上海 201306)
设计与制作了一种基于聚二甲基硅氧烷-玻璃(PDMS-Glass)的多功能集成式液滴数字聚合酶链式反应(ddPCR)芯片,该芯片由产生液滴的PDMS模块和收集液滴的玻璃腔体模块组成。PDMS模块采用双通道的T形结构设计,液滴产生速度快且通量高,在30 min内可生成2×106个直径约为20 μm的微液滴。玻璃腔体模块中存储的液滴在整个实验过程中无需转移,可直接在原位PCR仪上进行扩增,每个液滴均是一个微反应器,经过多次热循环后,液滴仍能保持良好的稳定性。选用副溶血性弧菌(VP)作为食源性致病菌的研究模型,考察了ddPCR芯片对其基因组DNA的绝对定量能力,结果表明,该ddPCR芯片对VP基因组DNA绝对定量的线性范围宽,可跨越5个数量级(101~106copies/μL),定量结果与DNA理论参考浓度间有很好的相关性。
数字PCR;液滴微流控芯片;致病菌;副溶血性弧菌
近年来,食物中毒或死亡事件在全球频发,严重威胁着人们的健康。据世界卫生组织估计,全世界每年的食源性疾病患者,约70%由致病微生物引起[1]。我国国家卫计委关于2012至2015年各年度食物中毒事件情况的通报显示:每年由致病微生物引起的食物中毒人数均最多,占全年食物中毒人数的50%以上[2-5]。在众多的食源性致病微生物中,副溶血性弧菌(VP)是全世界范围内引起人类细菌性食源性疾病的首要病原体,在许多海产品和淡水产品中均可检出,调查显示VP在我国海产品和淡水产品中的污染率分别高达53.04%和38.78%[6-7]。VP食物中毒可导致伤口感染、败血症以及腹泻、腹痛、恶心、呕吐、发热等急性肠胃炎症状,重症患者还会出现脱水、休克昏迷甚至死亡[8]。鉴于VP污染的高发性及其危害的严重性,开发灵敏、快速的检测方法对于预防和控制食源性疾病的爆发具有重要的研究意义。
数字聚合酶链式反应(dPCR)是继实时荧光定量PCR(qPCR)之后新兴发展起来的第三代PCR技术,其原理是将样品稀释成几万甚至是几百万份,然后分配到独立的反应单元,每个反应单元不包含或包含1个到多个拷贝的目标DNA分子,各自进行独立的PCR扩增,扩增结束后包含有目标DNA分子的反应单元发出荧光,而不含目标DNA分子的反应单元则无荧光信号,最终根据阳性液滴占总液滴的比例以及泊松分布计算出起始目标DNA的浓度,dPCR可对样本中的目标DNA分子进行绝对定量[9-11]。在dPCR中,反应单元的数量直接决定了对目标DNA分子的检测范围,例如当反应单元的数量增加1个数量级,则可检测的范围也相应地增加约1个数量级。当样本中的目标DNA分子浓度较低或不同目标DNA分子间的浓度差异较小时,增加反应单元数量还有助于提高定量的精度,这类似于像素点数量与数字图像分辨率间的关系[12]。与qPCR相比,dPCR定量不需要依赖参考基因建立的标准曲线,且不易受PCR扩增效率波动的影响[9,13]。
随着液滴微流控技术的高速发展,Bio-rad和Raindance公司分别推出了QX200TM和RainDrop PlusTM液滴数字PCR(ddPCR)系统,在拷贝数变异分析、基因表达、miRNA定量等方面取得了应用进展[14-16]。然而,这两种ddPCR系统的价格比较昂贵,在操作过程中均需转移液滴,步骤较多且易污染样品,因此,发展一种集成化的简易式、经济型的ddPCR芯片十分重要。本课题组在前期研究中曾设计了一种基于矿物油饱和PDMS的ddPCR芯片,并成功应用于致病菌DNA和肺癌相关的microRNA的定量分析[17-18],该芯片在整个操作过程中无需转移液滴且成本较低。不足之处在于:矿物油外渗增加了芯片制作与进样难度;液滴数量偏低(约2×104个),限制了ddPCR的检测浓度范围。为了改进上述不足,本文设计了一种在芯片制备过程中无需掺入矿物油的PDMS-Glass芯片,该芯片由产生液滴的PDMS芯片和收集液滴的玻璃腔体紧密连接而成,制作简单,经济实用。PDMS芯片采用双通道的T型结构设计使得液滴产生速度快且通量高(约2×106个),采用玻璃腔体使整个操作过程无需转移液滴,可在玻璃腔体中进行原位PCR扩增,且扩增后可在玻璃芯片上直接监测荧光信号。选用VP作为致病菌模型,考察了PDMS-Glass芯片对VP基因组DNA 的定量分析性能。
1 实验部分
1.1 试剂与仪器
副溶血性弧菌(ATCC 33847,美国菌种保藏中心);胰蛋白胨大豆肉汤培养基(TSB);胰蛋白胨大豆琼脂培养基(TSA);硫代硫酸盐柠檬酸盐胆盐蔗糖琼脂培养基(TCBS);PDMS单体和固化剂(SYLGARD@184 BASE and CURING AGENT,Dowcorning);光敏胶(SU-8,Microchem);Plus笔形双面胶(日本);矿物油(mineral oil,Sigma);Triton X-100(Sigma Aldrich);ABIL EM 90(Evonik);Tween-20(生工生物工程股份有限公司)。
原位PCR仪(Eppendorf Mastercycler nexus flat);实时荧光定量PCR仪(ABI 7500 fast,Thermofisher公司);IX51倒置荧光显微镜(Olympus公司)。
1.2 实验方法
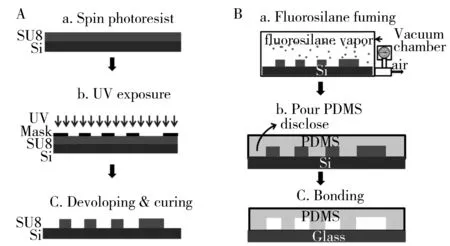
图1 液滴产生模块PDMS芯片的制作流程示意图Fig.1 The schematic of fabrication process for PDMS chip where droplets were producedA:preparation of the patterned silicon mold by SU-8 soft lithography process(SU-8光刻工艺制备硅基模具);B:fabrication of patterned PDMS chip by cast molding(模塑法制备PDMS芯片)
1.2.1PDMS-Glass芯片的设计与制作PDMS模块的结构采用autocad软件设计得到。其制作分两个步骤进行:首先采用软光刻技术制作图形化的硅基模具,之后采用模塑法制作图形化的PDMS芯片。硅基模具制作的流程如图1A所示:(a)将 SU-8负性光刻胶旋涂在洁净的硅片基底上,厚度约为30 μm,经前烘(65 ℃,5 min,95 ℃,30 min)后缓慢降至室温;(b)将打印有芯片结构的掩模(mask)放置于涂有光刻胶的硅基底上,经光刻机紫外曝光后进行后烘(65 ℃,5 min,95 ℃,12 min),缓慢降至室温;(c)显影固化后即可得到图形化的硅片模具。PDMS芯片的制作流程如图1B所示:(a)将制备好的硅基模具与10 μL全氟辛基三氯硅烷置于真空箱中保持3~6 h,(b)PDMS单体及固化剂按质量比10∶1 混匀,经真空脱气后倾注在硅基模具上,置85 ℃恒温箱中加热固化,冷却后将PDMS从硅片模具上剥离,在管道的样品入口和油相入口处分别打孔;(c)将PDMS图形面与载玻片通过等离子体键合即可得到液滴生成模块的芯片结构。对于玻璃腔体模块的制作,则在PDMS模块出口处,用厚度约30 μm的plus双面胶将一块2 cm×2 cm的盖玻片与载玻片贴合以形成封闭的玻璃腔体模块,体积约为12 μL。芯片使用前于105 ℃下加热4 h以增大其内表面的疏水性。
1.2.2VP基因组DNA的提取与浓度测定使用细菌基因组DNA提取试剂盒(TIANamp Bacteria DNA Kit,Tiagen)提取VP基因组DNA,提取步骤按操作说明执行。应用Quant-iTTMPicoGreen®dsDNA Reagents and Kits(Invitrogen)对所提取的基因组DNA的浓度进行初步定量,用无菌水将基因组DNA的浓度稀释至107copies/μL,并于-20 ℃冻存,每次实验前新鲜稀释。
1.2.3引物及探针使用Primer Premier 5.0软件设计引物和探针。VP的引物和TaqMan-MGB探针基于tlh基因序列设计得到。详细信息见表1。
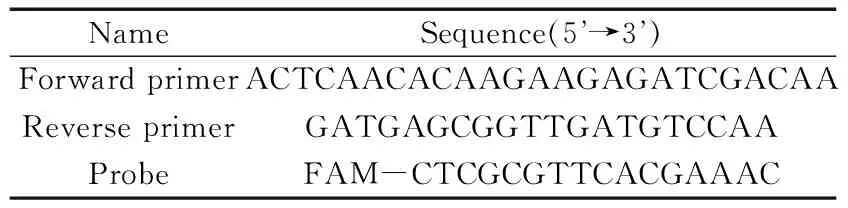
表1 副溶血性弧菌的tlh基因ddPCR的引物和探针序列Table 1 The primer and probe sequence for ddPCR based on tlh gene of VP
1.2.4ddPCR反应体系及扩增程序样品(水相)组成:1×Premix Ex Taq(Takara),0.2 μmol/L上/下游引物,0.3 μmol/L探针,0.3%(体积分数) Tween-20,1.0 μL不同浓度的细菌基因组DNA,无菌水。油相组成:含3% ABIL EM90和0.1% Triton X-100的矿物油。PCR扩增程序:95 ℃预变性30 s,再经35个循环的变性(95 ℃,5 s)和退火延伸(58 ℃,30 s)。每个反应平行做3次并设阴性对照(使用无菌蒸馏水代替目标DNA)。
1.2.5ddPCR进样将PDMS-Glass芯片固定于显微镜载物台,用多聚物管连接微量进样泵与芯片油相和样品进样口。在油相进样口和样品(水相)进样口处分别加入油相和PCR反应液,调节进样压力,控制液滴的尺寸和产生的速度。待液滴充满玻璃反应腔体后,使用掺有矿物油的PDMS(PDMS单体∶固化剂∶矿物油质量比为10∶1∶2)对玻璃腔体的进样口和出样口进行封装。
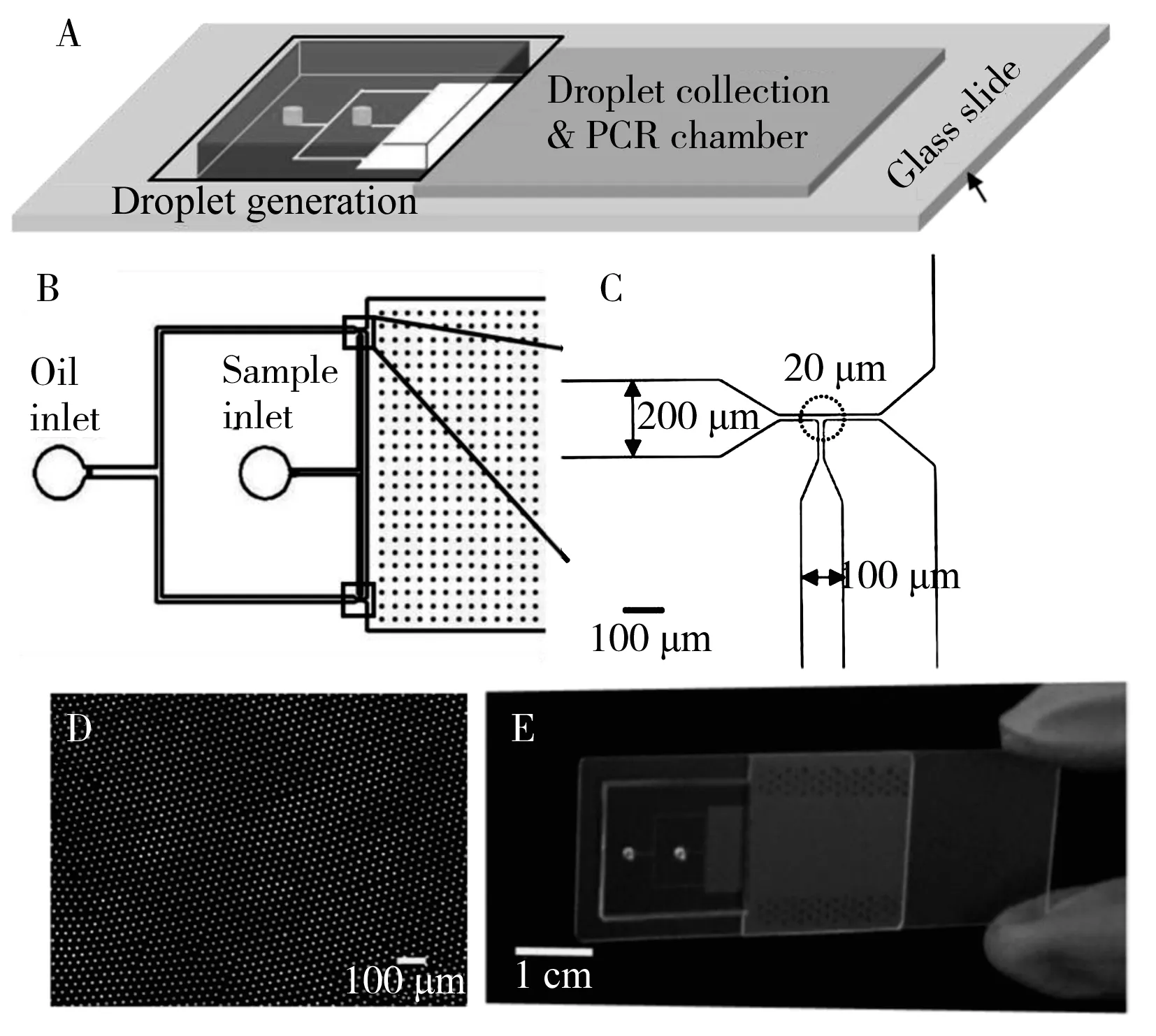
图2 PDMS-玻璃芯片结构Fig.2 The structure of the PDMS-Glass chip A:the schematic of the PDMS-Glass chip,which was composed of droplet generation module and droplet collection & PCR chamber module;B:autocad design for the droplet generation module;C:image of the enlarged T-shaped structure in Fig.1B;D:bright-field image of droplets generated by the PDMS-Glass chip:E:picture of the PDMS-Glass chip
2 结果与讨论
2.1 PDMS-Glass芯片的设计与制作
图2A是PDMS-玻璃芯片的结构示意图,该芯片由两大模块组成:左侧是用于生成液滴的PDMS模块;右侧是用于收集液滴和PCR扩增的玻璃腔体模块,两大模块紧密相连。图2B是PDMS模块的结构设计图,主要包括一个油相进样口、一个样品(水相)进样口以及双通道T型接口。图2C是T型接口的放大图,样品进样通道和油相进样通道的宽度分别为200 μm和100 μm,T型交汇处的通道宽度为20 μm,样品流经T型接口处受到油相的剪切力而被切割成一个个微液滴。在正向驱动力和毛细作用下,微液滴顺利进入收集液滴的玻璃腔体模块。图2D是储存在玻璃腔体模块中液滴的明场照片,可以看出液滴尺寸较为均匀,直径约为20 μm,体积约为4.19 pL。储存在玻璃腔体中的液滴可直接在芯片上进行原位扩增,扩增结束后用荧光显微镜直接监测玻璃腔体中液滴的荧光信号。
PDMS-Glass芯片集液滴的产生与收集、原位PCR扩增与在芯片上进行荧光信号监测等功能于一体,整个实验过程无需转移液滴,避免了多次实验操作引起的液滴破碎和融合,简化了实验过程,提高了分析效率。液滴产生模块中,双通道T型结构设计使得生成的液滴速度快、通量高,可在30 min内生成约2×106个尺寸均一的液滴,比商业化的QX200 ddPCR系统的液滴数量高2个数量级。高通量的液滴有助于提高ddPCR的检测范围,较快的液滴产生速度对于样品中Taq酶活性的保持至关重要,是ddPCR成功扩增的关键,也是实现快速检测的关键环节。相比于本课题组前期设计的矿物油饱和的PDMS芯片,采用玻璃腔体收集液滴的结构设计可以避免亲油的PDMS对油包水液滴油相的吸收,保证了ddPCR扩增的顺利进行,同时也有利于降低芯片的制作成本。
PDMS-Glass芯片制作也分两个模块分别进行,其中关键环节是PDMS模块中硅基模具的制备,即如何将图2B所示的结构转移到硅片上。图1是PDMS模块的制作流程示意图。硅基模具的制作(图1A)采用SU-8胶光刻工艺,SU-8光刻胶具有良好的力学性能和抗化学腐蚀性,经济耐用。首先制作图形化的硅基模具:先在硅片上旋涂一层SU-8光刻胶,通过紫外曝光将掩膜(mask)上的图形转移至光刻胶上,再经过显影固化得到图形化的硅基模具,一次制作可反复使用。采用模塑法制备得到图形化的PDMS,借助等离子体将图形化的PDMS与玻璃基底键合,即得到产生液滴的PDMS芯片(图1B)。玻璃腔体模块制作相对简单,通过双面胶将盖玻片与玻璃基底贴合即成。由两个模块组成的PDMS-Glass芯片实物图如图2E所示。
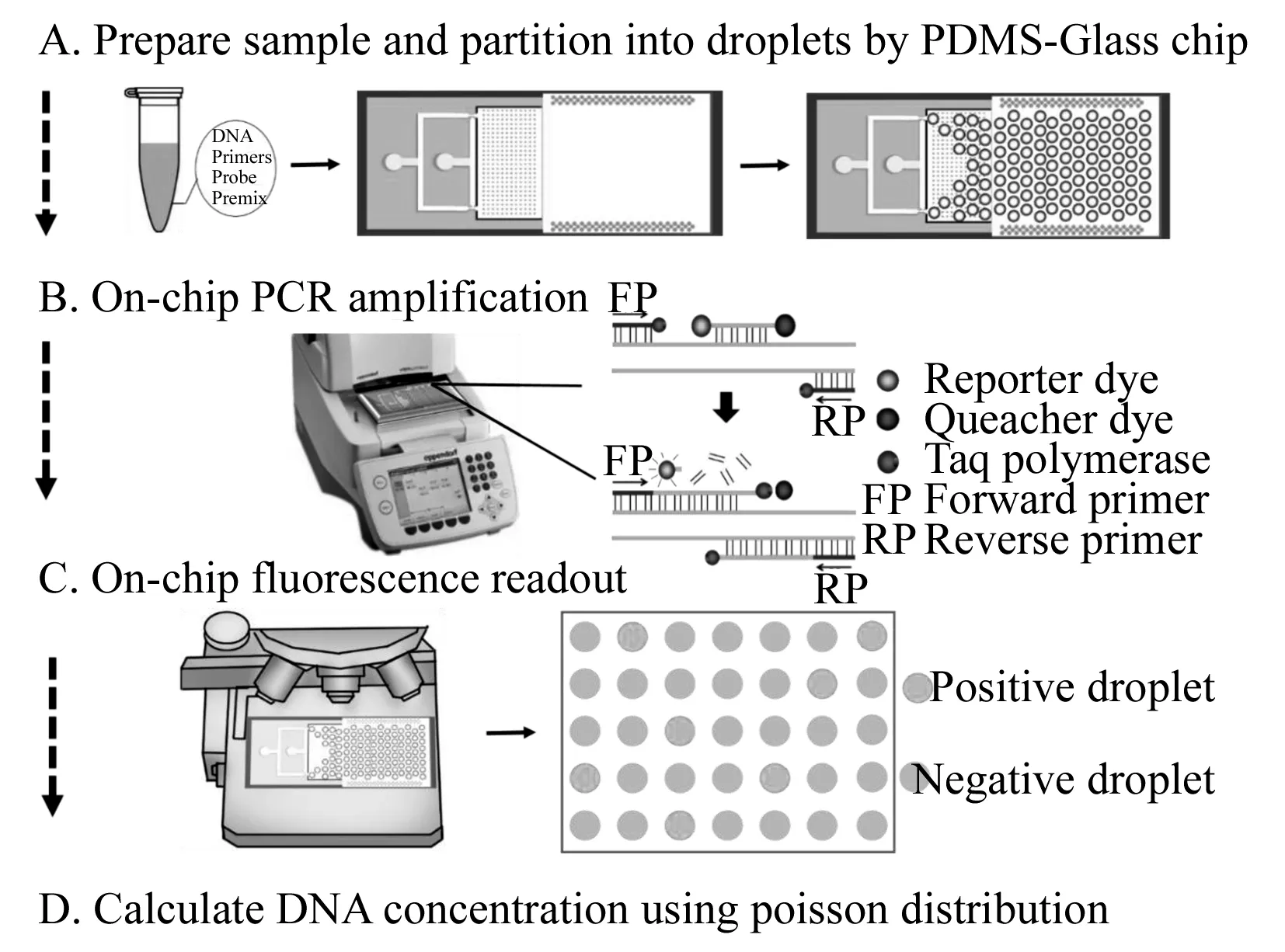
图3 基于PDMS-Glass芯片的ddPCR工作流程Fig.3 Schematic showing the PDMS-Glass chip-based ddPCR workflowA.prepare PCR mixture and partition into droplets by PDMS-Glass chip,B.on-chip insitu PCR amplification and the reaction principle in droplet,C.on-chip monitoring the fluorescence of droplets using fluorescence microscope,D.calculate the target DNA concentration in the sample using the Poisson distribution principle(A.制备样品,并通过PDMS-Glass芯片将样品平均分配到多个液滴反应单元中;B.在芯片上进行原位PCR扩增及其原理图;C.在芯片上原位读取荧光信号;D.根据泊松分布原理计算样本中目标DNA的浓度)
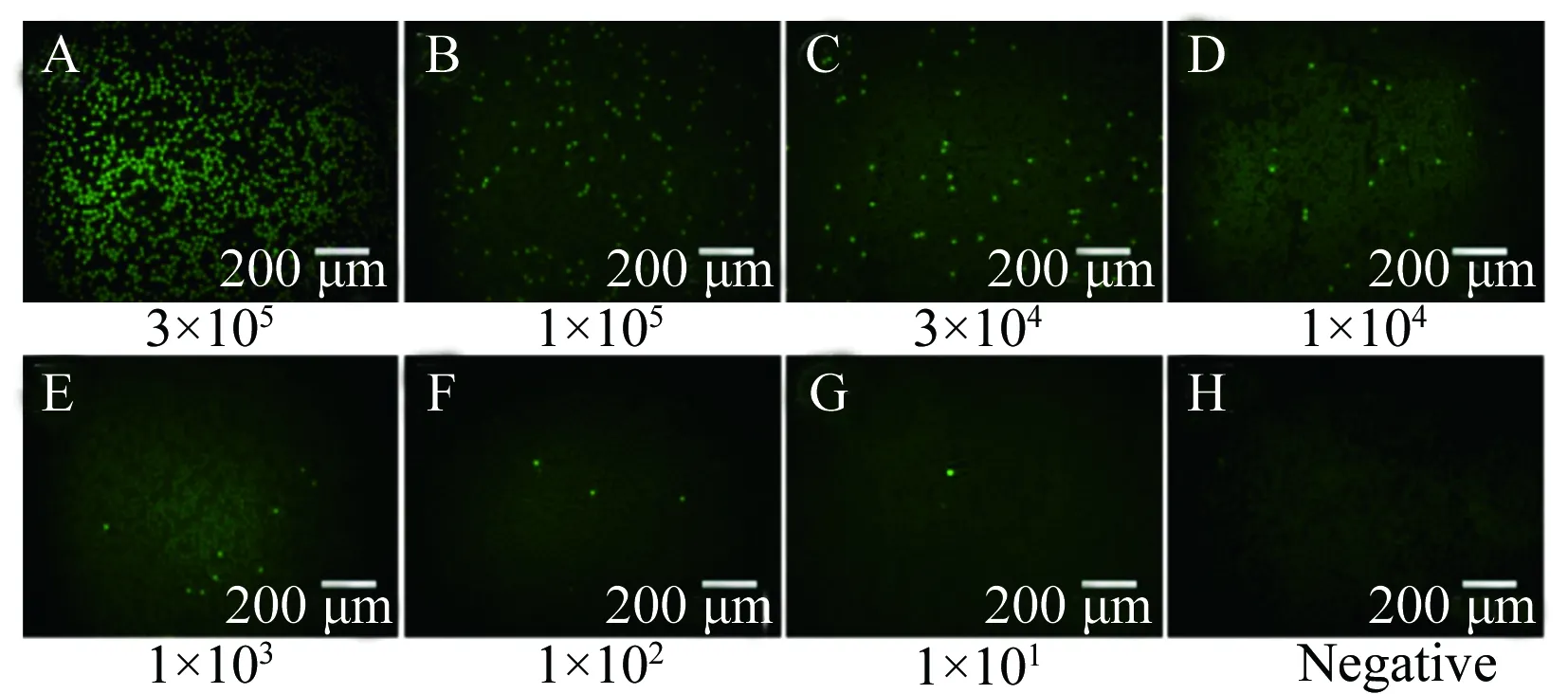
图4 基于PDMS-Glass芯片的ddPCR扩增后一系列不同浓度VP基因组DNA(3×105~1×101 copies/μL)的荧光显微镜照片Fig.4 Fluorescent images after droplet digital PCR amplifica-tion using the PDMS-Glass chip with a series of different concentrations of VP DNA from 3×105 to 1×101 copies/μL H:no DNA template as negative control;the fluorescent images were captured from FAM channel of the microscope with the excitation and emission wavelength of (485±10) nm and (524±12) nm,respectively
2.2 基于PDMS-Glass芯片的ddPCR工作流程
基于PDMS-Glass芯片的ddPCR工作流程主要包括4个步骤(图3)。第一步,制备样品并通过PDMS-Glass芯片将样品平均分配在多个液滴反应单元中(图3A)。第二步,在PDMS-Glass芯片上进行原位PCR扩增反应(图3B)。芯片中的每个液滴都是一个微反应器,液滴内包含扩增反应所需的引物、探针、目标DNA分子(有或者无)以及Taq酶等,存在目标DNA分子的液滴为阳性液滴,没有目标DNA分子的则为阴性液滴,当液滴通量足够高时,每个液滴中含有0个或1个拷贝的目标DNA分子。液滴内扩增反应的原理如图3B右图所示,液滴微反应器中包含3条短片段序列,分别为上、下游引物以及TaqMan探针,探针序列的一端标记FAM荧光报告染料分子,另一端标记猝灭染料分子,对于阳性液滴,DNA首先变性,在Taq酶的作用下,引物退火延伸,Taq酶具有核酸外切酶的活性,可以“吃掉”结合到模板分子上的DNA片段,从而使FAM报告染料分子与猝灭染料分子分离,发出绿色荧光;而对于阴性液滴,FAM报告染料分子与猝灭染料分子间发生能量共振转移,扩增结束后液滴无荧光信号。第三步,扩增结束后,将芯片置于荧光显微镜下观察储存液滴的玻璃腔体区域液滴的荧光信号,并拍摄荧光照片,阳性液滴发出绿色荧光,阴性液滴则无荧光(图3C)。第四步,使用自编的液滴自动计数软件统计出阳性液滴占总液滴的比值,并根据泊松分布原理计算出起始样本中目标DNA的浓度,当样品中目标DNA的浓度较高时,液滴中可能会分配有大于1个拷贝的模板分子,通过泊松分布可对此进行校准,以实现对目标DNA分子的绝对定量。
2.3 基于PDMS-Glass芯片的ddPCR对VP的定量分析
ddPCR对目标DNA定量的动态范围是评价该方法分析性能的重要指标。选用VP作为研究对象,以VP基因组DNA为目标基因,考察了基于PDMS-Glass芯片的ddPCR方法对DNA的定量分析性能。图4是一系列不同浓度的VP基因组DNA经ddPCR扩增后的荧光照片,随着VP基因组DNA浓度的降低,发出绿色荧光的阳性液滴的数量随之减少(图4A~G),而对于没有模板存在时的阴性对照(图4H),则全部液滴均无荧光信号。模板DNA分子在液滴中的分配遵循泊松分布(Poisson distribution)原理[19],即每个液滴中包含k个DNA模板的概率为:
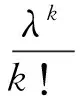
式中,λ为液滴中的平均模板数,只有当λ<0.3时,才可确保大部分微单元含有数量不多于1的DNA模板。PDMS-Glass芯片可产生2×106个液滴,则理论上当样本中的模板数<6×105copies时,才可确保单个液滴中模板数≤1。在具体实验过程中(10 μL进样体积),较低浓度(≤1 000 copies/μL)时,采用直接拍照计数阳性液滴数;对于高浓度样本(>1 000 copies/μL),则采用泊松公式[10,20],根据阳性液滴数量占总液滴数量的比值对样本浓度进行校正:
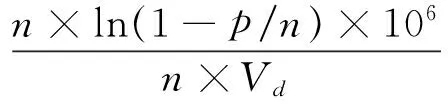
其中,C为样品中模板DNA 的浓度(copies/μL),n是液滴总数,p是阳性液滴数,Vd是液滴的平均体积(pL)。
为了评估基于PDMS-Glass芯片的ddPCR方法对VP基因组DNA定量的准确度,将ddPCR对一系列不同浓度VP基因组DNA的定量结果与其实际浓度进行了比较。结果表明,在较宽的浓度范围内(101~105copies/μL),ddPCR方法测得的结果与参考值间有很好的相关度,线性的斜率接近于1,r2=0.999,这说明基于PDMS-Glass芯片的ddPCR方法对DNA的定量具有良好的准确性。对于浓度较低(10~1 000 copies/μL)的目标基因,ddPCR测量结果与参考值间仍存在较好的相关度,相关系数(r2)为0.996。不同于qPCR的相对定量,ddPCR可进行绝对定量,无需依赖参考基因建立的标准曲线,且不敏感于扩增效率的变化。
3 结 论
本文设计与制作了一种基于PDMS-Glass的ddPCR芯片,该芯片集液滴的产生与收集、原位PCR扩增及监测荧光信号的功能于一体,整个过程无需转移液滴,操作简便省时。芯片可在半小时内产生两百万个液滴,液滴的通量比商业化QX200 ddPCR系统高两个数量级。高通量的液滴经过多次热循环之后仍能保持良好的稳定性,成功实现了对副溶血性弧菌基因组DNA的绝对定量,定量的线性范围可跨越5个数量级,检测结果与理论参考浓度间具有良好的相关性。该方法在疾病诊断中的低拷贝数变异分析、食品安全中的转基因检测、环境中的致病微生物检测等领域具有一定的应用前景。为将基于PDMS-Glass芯片的ddPCR方法应用于现场快速检测,下一步将着力改进PDMS-Glass芯片的进样方法,减少其对外部驱动力的依赖。
[1] Li J Y,Zhao Z,Jia X C,Li J,Zhang Y,Zhao L H.FoodRes.Dev.(栗建永,赵琢,贾晓川,李晶,张园,赵黎华.食品研究与开发),2013,34(18):110-115.
[2] Ministry of Health.Bulletin of the National Food Poisoning Incident in 2012(卫生部.关于2012年全国食物中毒事件情况的通报)[2013-02-26].http://www.moh.gov.cn/mohwsyjbgs/s7860/201303/b70872682e614e4189d0631ae5527625.shtml.
[3] National Health and Family Planning Commission of the People’s Republic of China.Bulletin of the National Food Poisoning Incident in 2013(国家卫生计生委.关于2013年全国食物中毒事件情况的通报).[2014-02-14].http://www.moh.gov.cn/yjb/s3585/201402/f54f16a4156a460790caa3e991c0abd5.shtml.
[4] National Health and Family Planning Commission of the People’s Republic of China.Bulletin of the National Food Poisoning Incident in 2014(国家卫生计生委.关于2014年全国食物中毒事件情况的通报).[2015-02-11].http://www.moh.gov.cn/yjb/s3585/201502/91fa4b047e984d3a89c16194722ee9f2.shtml.
[5] National Health and Family Planning Commission of the People’s Republic of China.Bulletin of the National Food Poisoning Incident in 2015(国家卫生计生委.关于2015年全国食物中毒事件情况的通报).[2016-02-19].http://www.nhfpc.gov.cn/yjb/s7859/201604/8d34e4c442c54d33909319954c43311c.shtml.
[6] Tan D M,Wang H,Lü S L,Li X G,Zeng X Y,Su A R.J.ChinaHealthSur.(谭冬梅,王红,吕素玲,李秀桂,曾献莹,苏爱荣.中国卫生检验杂志),2017,27(3):344-346.
[7] Lan Y,Xu J,Bian X J,Zhao Y,Pan Y J,Zhang W J.J.Instrum.Anal.(蓝英,徐娟,卞晓军,赵勇,潘迎捷,张炜佳.分析测试学报),2016,35(6):635-640.
[8] Xie X Q,Liu Z.FoodSci.(谢雪钦,刘舟.食品科学),2017,38(13):299-305.
[9] Hindson C M,Chevillet J R,Briggs H A,Gallichotte E N,Ruf I K,Hindson B J,Vessella R L,Tewari M.Nat.Methods,2013,10(10):1003-1005.
[10] Ottesen E A,Hong J W,Quake S R,Leadbetter J R.Science,2006,314(5804):1464-1467.
[11] Vogelstein B,Kinzler K W.Proc.Natl.Acad.Sci.,1999,96(16):9236-9241.
[12] Hindson B J,Ness K D,Masquelier D A,Belgrader P,Heredia N J,Makarewicz A J,Bright I J,Lucero M Y,Hiddessen A L,Legler T C,Kitano T K,Hodel M R,Petersen J F,Wyatt P W,Steenblock E R,Shah P H,Bousse L J,Troup C B,Mellen J C,Wittmann D K,Erndt N G,Cauley T H,Koehler R T,So A P,Dube S,Rose K A,Montesclaros L,Wang S,Stumbo D P,Hodges S P,Romine S,Milanovich F P,White H E,Regan J F,Karlin-Neumann G A,Hindson C M,Saxonov S,Colston B W.Anal.Chem.,2011,83(22):8604-8610.
[13] Hayden R T,Gu Z,Ingersoll J,Abdul-Ali D,Shi L,Pounds S,Caliendo A M.J.Clin.Microbiol.,2013,51(2):540-546.
[14] Miotke L,Lau B T,Rumma R T,Ji H P.Anal.Chem.,2015,86(5):2618-2624.
[15] Zhu Z,Yang C J.Acc.Chem.Res.,2017,50(1):22-31.
[16] Heredia N J,Belgrader P,Wang S,Koehler R,Regan J,Cosman A M,Saxonov S,Hindson B,Tanner S C,Brown A S,Karlin-Neumann G.Methods,2013,59(1):S20-S23.
[17] Bian X J,Jing F X,Li G,Fan X J,Jia C P,Zhou H B,Jin Q H,Zhao J L.Biosens.Bioelectron.,2015,74:770-777.
[18] Wang P,Jing F X,Li G,Wu Z H,Cheng Z L,Zhang J S,Zhang H L,Jia C P,Jin Q H,Mao H J,Zhao J L.Biosens.Bioelectron.,2015,74:836-842.
[19] Mazutis L,Araghi A F,Miller O J,Baret J C,Frenz L,Janoshazi A,Taly V,Miller B J,Hutchison J B,Link D,Griffiths A D,Ryckelynck M.Anal.Chem.,2009,81(12):4813-4821.
[20] Lo Y M D,Lun F M F,Chan K C A,Tsui N B Y,Chong K C,Lau T K,Leung T Y,Zee B C Y,Cantor C R,Chiu R W K.Proc.Natl.Acad.Sci.,2007,104(32):13116-13121.
Fabrication of a Droplet Digital PCR Chip and Its Application in Pathogenic Bacteria Detection
DENG Xue-lei1,ZHANG Yuan-yi1,YUAN Hao-jun2,LIU Song-sheng2,CHEN Ying-ying1,GAO Wan-lei2,ZHOU Hong-bo2,JIA Chun-ping2,ZHAO Jian-long2,BIAN Xiao-jun1,3,4*
(1.College of Food Science and Technology,Shanghai Ocean University,Shanghai 201306,China;2.State Key Laboratory of Transducer Technology,Shanghai Institute of Microsystem and Information Technology,Chinese Academy of Science,Shanghai 200050,China;3.Laboratory of Quality and Safety Risk Assessment for Aquatic Product on Storage and Preservation(Shanghai),Ministry of Agriculture,Shanghai 201306,China;4.Engineering Research Center of Food Thermal-processing Technology,College of Food Science and Technology,Shanghai Ocean University,Shanghai 201306,China)
A polydimethylsiloxane-glass(PDMS-Glass) based droplet digital polymerase chain reaction(ddPCR) chip was designed and fabricated,which was integrated with multiple functions.The PDMS-Glass chip was composed of two modules,of which one was a PDMS module for droplet generation,and the other was a multi-functional glass chamber for droplet collection,insituddPCR reaction and on-chip fluorescence readout.The PDMS module has a T-shaped structure with two channels,where the aqueous phase with sample was cut into water-in-oil droplets by the oil phase.The droplets generated by the PDMS-Glass chip have the advantages of high-throughput and high-frequency.About two million uniform droplets with an average diameter of 20 μm could be produced within 30 minutes.The glass chamber was employed for droplets collection.No transfer of the droplets were required during the whole experiment.The droplets filled in the glass chamber were directly injected into theinsituPCR instrument for amplification reaction,where each droplet was a micro-reactor.The glass chamber provided the ddPCR with a good environment for ddPCR amplification,where the droplets kept stable after many times of thermal cycling.As one of the common pathogenic bacteria,VibrioParahemolyticus(VP) was selected to investigate the property of the PDMS-Glass based ddPCR chip on the aspect of absolute quantificaiton.The results showed that the ddPCR chip has a wide linear range of five-order-magnitude towards the genomic DNA ofVPfrom 101to 106copies/μL.Meanwhile,the detected results by ddPCR approach have a good relativity with the theoretical expected DNA concentration.
digital PCR;droplet microfluidic chip;pathogenic bacteria;VibrioParahemolyticus
10.3969/j.issn.1004-4957.2017.10.004
O657;Q523
A
1004-4957(2017)10-1191-06
2017-06-21;
2017-07-19
国家自然科学基金(21405167,81472751,61271162);上海晨光计划(15CG54);国家科技重大专项(2017ZX10302201-005-006)
*
卞晓军,博士,讲师,研究方向:微流控生物分析化学技术,Tel:021-61900753,E-mail:xjbian@shou.edu.cn

