基于改进卷积神经网络的多种植物叶片病害识别
孙 俊,谭文军,毛罕平,武小红,陈 勇,汪 龙
基于改进卷积神经网络的多种植物叶片病害识别
孙 俊1,谭文军1,毛罕平2,武小红1,陈 勇1,汪 龙1
(1. 江苏大学电气信息工程学院,镇江 212013;2. 江苏大学江苏省现代农业装备与技术重点实验室,镇江 212013)
针对训练收敛时间长,模型参数庞大的问题,该文将传统的卷积神经网络模型进行改进,提出一种批归一化与全局池化相结合的卷积神经网络识别模型。通过对卷积层的输入数据进行批归一化处理,以便加速网络收敛。进一步缩减特征图数目,并采用全局池化的方法减少特征数。通过设置不同尺寸的初始层卷积核和全局池化层类型,以及设置不同初始化类型和激活函数,得到8种改进模型,用于训练识别14种不同植物共26类病害并选出最优模型。改进后最优模型收敛时间小于传统卷积神经网络模型,仅经过3次训练迭代,就能达到90%以上的识别准确率;参数内存需求仅为2.6 MB,平均测试识别准确率达到99.56%,查全率和查准率的加权平均分数为99.41%。改进模型受叶片的空间位置的变换影响较小,能识别多种植物叶片的不同病害。该模型具有较高的识别准确率及较强的鲁棒性,该研究可为植物叶片病害的识别提供参考。
病害;植物;图像处理;识别;卷积神经网络;批归一化;全局池化;深度学习
0 引 言
植物的病害问题与人们的日常生活密切相关,虽然使用化学农药能够控制植物病害,但是由于病害种类繁多,仅靠人工肉眼观察及经验判断容易发生误诊,植物病害得不到及时诊治[1-2][1]。治理植物病害最关键的是快速且精确诊断病害类型,防止农药的错误使用。
随着计算机技术的不断发展,关于植物叶片病害的智能识别研究也取得了良好的进展。谭峰等[3]通过计算叶片色度值,建立多层BP神经网络模型,实现大豆叶片的病害识别。田有文等[4]通过提取葡萄病叶的颜色与纹理特征,利用支持向量机(support vector machine,SVM)识别的方法取得了比神经网络识别更好的效果。王献锋等[5]提取叶片病斑颜色、形状、纹理等特征,结合环境信息,利用判别分析法,识别黄瓜病斑类别。Zhang等[6]也是将斑点分割之后再提取病斑的颜色、形状和纹理特征,然后通过K最近邻(K-nearest neighbor,KNN)分类算法对5种玉米叶片进行识别。以上文献均是通过提取植物特定图像特征结合传统分类方法对病害进行识别。虽然取得了较好的识别效果,但是由于特定特征并非能够完全或者较好地表征植物病害信息,且病害叶片不一定出现病斑,可能出现粉状物,这使得分割工作更加困难,从而对识别效果产生不利影响。且上述方法所选试验样本数目有限,或所选叶片仅来自一种植物,这些方法仅局限于对同一种植物的叶片病害识别。
近些年兴起的卷积神经网络[7](convolutional neural network,CNN)能够不依赖特定特征,在图像识别领域(如手写字体识别[8]、人脸识别[9-10]以及物体检测[11-12]等方面)已经得到广泛应用。在广义识别上,AlexNet[13]、GoogLeNet[14]和ResNet[15]等卷积神经网络模型都取得较好的效果。越来越多的学者将这些模型用于狭义的图像识别中,龚丁禧等[16-17]采用卷积神经网络对植物叶片分类进行了相关研究。Sladojevic等[18]、Brahimi等[19]与Amara等[20]将卷积神经网络用于植物叶片病害识别,分别在模型CaffeNet和AlexNet上使用微调方法进行改进,取得了较好的识别效果。以上文献证明卷积神经网络识别植物叶片病害是可行的,但这些模型参数量大,训练时间长,且模型不易使用。
针对训练收敛时间长,模型参数庞大的问题。本文提出一种批归一化与全局池化相结合的新型模型识别方法,对含有14种不同品种植物共26类病害的叶片图片集进行训练与测试,并与传统卷积神经网络AlexNet模型识别方法以及传统叶片病害识别方法相比较分析。以期为植物叶片病害的识别提供参考。
1 试验数据
PlantVillage工程(www.plantvillage.org)为解决植物病害诊断的问题,面向所有用户开放数据库,数据库中包含多类植物的患病与健康叶片图像数据。本文采用PlantVillage工程所收集的21 917张叶片图像作为试验数据,其中包含14种植物共26类病害叶片以及部分植物的健康叶片,如图1所示。

注:标号1-4依次为苹果疮痂病、黑腐病、锈病和健康叶;5为蓝莓健康叶;6-7依次为白粉病、樱桃健康叶;8-11依次为玉米灰斑病、锈病、枯叶病和健康叶;12-15依次为葡萄黑腐病、黑痘病、叶枯病和健康叶;16为橘子黄龙病叶;17-18依次为桃子细菌性斑点病和健康叶;19-20依次为辣椒细菌性斑点病和健康叶;21-23依次为土豆早疫病、晚疫病和健康叶;24为覆盆子健康叶;25为黄豆健康叶;26为南瓜白粉病叶;27-28依次为草莓叶焦病和健康叶;29-38依次为番茄细菌性斑点病、早疫病、晚疫病、叶霉病、斑枯病、健康、轮斑病、黄曲病、花叶病和二斑叶螨病叶。

2 叶片病害识别模型结构
卷积神经网络包含多层卷积和池化层,用于逐层提取图片深层特征[13]。AlexNet卷积神经网络作为一个经典网络,其包含输入层、卷积层、池化层、全连接层和Softmax分类器,在图像识别任务上取得了重大突破。但由于网络结构使用全连接层,需计算大量权值参数,内存占用大且收敛慢。再者由于采用批量训练的方法,每训练一个批次数据更新一次网络参数,不同批次图片数据分布不同,如果数据分布差异非常大,则需要重新大幅调整参数以适应当前批次数据。且由于层与层互相连接,随着网络层次增多,前面层的参数的微小改变会导致后面层参数发生巨大变化,增大了计算量和收敛时间[21]。
为解决模型收敛时间长和参数内存需求大的问题,本文在AlexNet的基础上做了以下2个方面的改进:1)采用批归一化方法加速网络收敛;2)采用加入全局池化层和缩减特征图数目的方法减少模型参数。
2.1 批归一化算法



式中和分别为批次均值和方差,接着将数据归一化


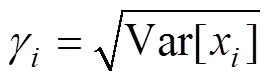


2.2 全局池化
传统的卷积神经网络模型通常设置全连接层级联(concatenate)在池化层后,但如果设置神经元数目较多,将使全连接层参数占据模型总参数的较大比重,且训练时间长。Hinton等[22]提出在全连接层设置Dropout,随机的抑制一定数目的神经元,使被抑制神经元暂时不参与网络的前向传播,但保留其权值,这能防止过拟合[23],同时提高模型的泛化能力[24]。全局池化[25]相比传统的全连接层,具有增强特征图与类别的关系、防止过拟合、对图像的空间变换保持近似不变性等优点,使用全局池化的方法可避免Dropout参数寻优。

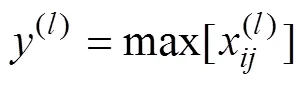
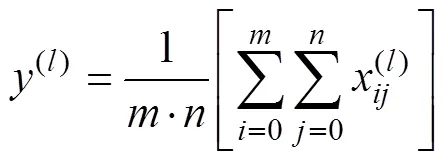
2.3 批归一化与全局池化结合的卷积神经网络模型
图2为批归一化与全局池化相结合的卷积神经网络模型,该模型对输入卷积层的数据进行了批归一化,且将全连接层替换为全局池化层,同时调节了原始AlexNet模型部分参数。改进模型的卷积层与池化层共9层,为画图方便,图中部分层次(激活层)未展示出来,但改进之处图中都有呈现。模型包含5层卷积层(Conv 1~Conv 5)和4层池化层(pooling1~global pooling 4),分类器采用Softmax分类器。考虑到不同参数设置会对模型性能产生相应的影响,本文主要比较Conv1层卷积核尺寸与初始化类型、全局池化类型以及各层激活函数类型对病害识别平均准确率(average accuracy,AA)的影响,进而对模型优化。利用平均准确率和查全率与查准率加权平均分数(1)指标来评价模型效果。

注:Conv1、Conv2、…、Conv5的卷积核数目分别为96、128、192、192、128;Conv 2的卷积核大小为5´5 dpi, Conv3、…、Conv5 卷积核大小均为3´3 dpi。
感受野(receptive field)负责直接由原始输入图像提取最低层特征,不同尺寸卷积核提取低层特征能力不同[26]。将Conv1层卷积核尺寸从11´11 dpi、9´9 dpi和7´7 dpi中选择,为了保证全局池化后的特征图大小为1´1 dpi,需调整全局池化层池化尺寸,Conv1层使用7´7 dpi时全局池化层也使用7´7 dpi大小的池化尺寸。
AlexNet中采用修正线性单元激活函数[27](rectified linear units,Relu),使梯度在反向传播时能够很好地传到前面的网络层,能防止梯度弥散的问题,同时加速网络训练。He等[28]提出PRelu激活函数,将Relu函数的负轴斜率从0改为可变参数a,在模型训练中也能取得很好效果。本文改进模型采用Relu和PRelu 2种激活函数分别训练,式(9)与式(10)分别为Relu激活函数和PRelu激活函数的表达式。


权重的初始化会对网络性能产生影响,本文选择其中最常用的高斯和Xavier方法初始卷积层Conv1,观察不同初始化方法对模型识别准确率的影响。模型的全局池化层分别使用全局平均池化和全局最大池化类型,比较不同池化类型对模型性能的影响。
3 模型训练
3.1 试验平台
试验软件环境为Ubuntu 16.04 LTS 64位系统,采用Caffe[29]深度学习开源框架,选用Python作为编程语言。计算机内存为16 GB,搭载Intel® Core™ i7-6700KCPU @ 4.00 GHz x8处理器,并采用英伟达GTX980Ti显卡加速图像处理。
3.2 试验参数设置
采用批量训练的方法将训练集与测试集分为多个批次(batch),每个批次训练64张图片,即train batch设置为64。当训练完所有训练集图片后对测试集进行测试,每次测试图片为50张,即test batch设置为50。遍历一次训练集中的所有图片作为一次迭代(epoch),共迭代100次。采用随机梯度下降优化算法(stochastic gradient descent,SGD)优化模型,设置初始学习率为0.01,为防止过拟合,将正则化系数设为0.005,学习率分阶段逐次减小为原来的0.1倍。对于原始图片集,每训练8 000个批次,改变一次学习率,对于扩充后图片集,每训练20 000个批次改变一次学习率。
4 结果与分析
4.1 初始卷积核尺寸对准确率的影响
模型的识别平均准确率AA计算如下


模型Conv1层卷积层中使用卷积核尺寸不同使感受野不同,对图像特征的提取能力也不同。模型1、2都是使用11´11 dpi的卷积核初始化模型。由表1可见,使用卷积核尺寸为9´9 dpi的模型3和4比使用大尺寸卷积核的模型1、2及使用小尺寸卷积核的模型5、6好,但在2个数据集上不同模型之间的最高与最低准确率相差仅为0.24个百分点和0.35个百分点。总体来说,使用不同尺寸卷积核对模型的识别准确率产生的影响较小。为进一步观察模型的特征提取效果,将模型2、4、6的第一层卷积核进行可视化,如图3所示。由图3可见,模型2、4的Conv1层卷积核可视化图像包含的粗糙区域较多(粗糙区域表示的卷积核主要提取边缘和纹理特征),对细粒度特征响应充分,提取的纹理特征信息更丰富。不同植物的叶片病害图片在颜色上和轮廓上差异较大,所以仅需根据颜色和轮廓特征就能区分开,而区分相同植物不同病害的叶片图片需要进一步提取出叶片的纹理信息。大尺寸卷积核易提取叶片整体信息(如颜色和轮廓特征),但对纹理特征的响应没有小尺寸卷积核充分。为获取模型识别性能,将Conv1层卷积核尺寸确定为9´9 dpi较为合适。

表1 模型参数设置与测试准确率

a. 模型2a. Model 2b. 模型4b. Model 4c. 模型6c. Model 6
图3 改进模型卷积核可视化
Fig.3 Visualizing the convolutional kernel of improved model
4.2 全局池化类型对准确率的影响
池化通常使用最大池化和平均池化2种类型,中间层使用最大池化能够提取出最利于区分不同叶片病害的特征,丢弃冗余特征。表1中模型1、3、5与模型2、4、6分别采用全局最大池化和全局平均池化类型,结果表明使用全局平均池化比使用全局最大池化效果更优。这说明使用全局最大池化容易造成提取的深层特征信息流失,从而降低识别准确率,而全局平局池化则对整张特征图所有值求平均,充分利用了每张特征图所有信息,利于提取关键特征。
4.3 初始化类型与激活函数对准确率的影响
4.4 改进模型收敛效果分析以及参数内存需求比较
训练AlexNet模型需要花费大量时间,且训练后的模型参数内存需求(模型本身所占计算机存储空间的大小)较大。为了验证改进后的网络模型收敛效果,将改进后的最优模型4与原始的AlexNet模型在2个图片集上进行准确率和收敛迭代次数的比较。图4是模型4与原始AlexNet模型迭代次数与测试准确率关系图。

图4 模型测试准确率与迭代次数关系
2个模型训练迭代100次后,其测试集识别总体准确率都能达到97%以上,模型4与AlexNet模型在扩充后的数据集上最高测试准确率分别为99.56%和99.37%。从图4中可以清楚地看到,模型4的收敛时间较AlexNet模型短,由于初始学习率较高,模型4在3次迭代训练后,测试准确率快速提升至90%以上,而AlexNet仅能达到77%左右。且在未调节学习率至0.001之前,AlexNet的测试准确率不稳定,上下波动大,而加入批归一化层的模型4测试准确率虽然也有些波动,但幅度不大,这说明使用批归一化方法的模型能适应大学习率,使网络收敛更快。本文为便于改进模型和AlexNet模型进行对比,将学习率下降步长都统一设置为20 000,从图4可以看出改进模型在迭代10次后准确率已经趋于稳定,调小学习率为0.001之后,测试准确率迅速提升且很快稳定,这说明改进之后的模型在迭代次数上可做进一步的优化。
AlexNet模型在卷积层和池化层后级联全连接层将所有特征图拉伸为4 096维的特征向量,通过Softmax分类器对叶片病害分类,训练之后模型参数内存需求为217 MB。改进模型的参数内存需求大小以及前向传播和反向传播速率(进行一次前向/反向传播所需要的时间)如表2所示,由表2可见,初始层卷积核尺寸越小,模型参数内存需求也越小,同时训练时间也将缩短。改进后的最优模型参数内存需求仅占2.6 MB,缩小至AlexNet模型的0.01倍,测试一张图片所需时间为20.79 ms,而AlexNet模型测试一张图片所需时间为25.51 ms。

表2 模型参数内存需求与训练速度
4.5 改进模型鲁棒性分析
为了更好地评价一个模型是否具有较强鲁棒性,选取测试准确率最高的模型4与原始AlexNet模型以及颜色、纹理特征+SVM[30]进行对比见表3,将每类样本分别进行测试,计算每个类别查准率(precision(i))、查全率(recall(i))以及查全率与查准率加权平均分数(1(i)),最后取平均值(1)作为模型评价标准。

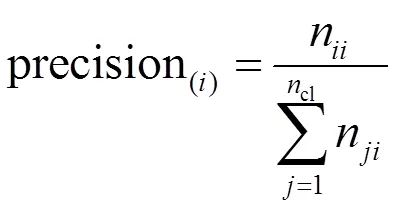

式中n为类别预测为第类的样本数。
颜色和纹理特征是叶片识别常用的特征,它们能够反映出叶片的全局信息,将提取的颜色和纹理特征融合之后用支持向量机分类是叶片病害识别的常见方法。在样本量比较少的时候,支持向量机容易抓住数据和特征之间的非线性关系,识别效果往往较好,由于本文试验采用的图片集中叶片类别和数目较多,且同一植物不同的病害叶片颜色相似度大,导致支持向量机分类方法时间和空间复杂度增大、识别准确率下降。从表3可见,基于卷积神经网络模型的叶片病害识别方法性能较好,并对图像的旋转、位移能保持近似不变性,所以AlexNet模型和改进模型在扩充后图片集上的性能都有所提升。改进模型由于采用了全局池化的方法,增强了特征图与类别的关系、对输入的空间变换的不变性,故性能比原始的AlexNet模型好,在扩充后的图片集上,模型4的查全率与查准率加权平均分数能达到99.41%。

表3 不同识别方法的性能对比
5 结 论
本文提出将卷积神经网络用于大量不同种类的植物叶片病害识别,在传统的AlexNet模型上改进,采用批归一化与全局池化相结合的卷积神经网络模型识别多种叶片病害。将改进模型与原始AlexNet模型进行对比,改进模型在训练时间和参数内存需求上都具有较大优越性,仅迭代训练3次就能达到90%以上准确率,改进的模型精简了模型参数,模型参数内存需求从217 MB缩小到2.6 MB,同时也提高模型泛化能力,性能较原始AlexNet模型有所提升,改进后最优模型在扩充后图片集上的查全率和查准率的加权平均分数为99.41%,测试准确率为99.56%。将改进模型与传统方法进行对比,改进模型对图像空间位置变化的适应性较好,具有较好鲁棒性,能够识别多种植物叶片的不同病害,而不局限于同一植物的不同病害。
本文提出模型对复杂的植物病害叶片识别效果较好,避免了特定特征选取,且训练后的模型易于使用,可为后续的植物叶片病害智能识别装置的研制提供理论依据。
[1] 李凯,袁鹤.植物病害生物防治概述[J].山西农业科学,2012,40(7):807-810. Li Kai,Yuan He.Review on biological control of plant diseases[J]. Journal of Shanxi Agricultural Siences, 2012, 40(7): 807-810. (in Chinese with English abstract)
[2] 陈志谊.植物病害生防芽孢杆菌研究进展[J].江苏农业学报,2012,28(5):999-1006. Chen Zhiyi.Research progress in biocontrol of bacillus spp. Against plant diseases[J]. Jiangsu Journal of Agricultural Sciences, 2012, 28(5): 999-1006. (in Chinese with English abstract)
[3] 谭峰,马晓丹.基于叶片的植物病虫害识别方法[J].农机化研究,2009,31(6):41-43. Tan Feng, Ma Xiaodan.The method of recognition of damage by disease and insect based on laminae[J]. Journal of Agricultural Mechanization Research, 2009, 31(6): 41-43. (in Chinese with English abstract)
[4] 田有文,李天来,李成华,等. 基于支持向量机的葡萄病害图像识别方法[J]. 农业工程学报,2007,23(6):175-180. Tian Youwen, Li Tianlai, Li Chenghua, et al. Method for recognition of grape disease based on support vector machine[J]. Transactions of The Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2007, 23(6): 175-180. (in Chinese with English abstract)
[5] 王献锋,张善文,王震,等. 基于叶片图像和环境信息的黄瓜病害识别方法[J]. 农业工程学报,2014,30(14):148-153. Wang Xianfeng, Zhang Shanwen, Wang Zhen, et al. Recognition of cucumber diseases based on leaf image and environmental information[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2014, 30(14): 148-153. (in Chinese with English abstract)
[6] Zhang S W, Shang Y J, Wang L.Plant disease recognition based on plant leaf image[J]. Journal of Animal & Plant Sciences, 2015, 25(3): 42-45.
[7] Zeiler M D, Fergus R. Visualizing and understanding convolutional networks[C]//European Conference on Computer Vision. Springer, Cham, 2014:818-833.
[8] LeCun Y, Bottou L, Bengio Y, et al. Gradient-based learning applied to document recognition[J]. Proceedings of the IEEE, 1998, 86(11): 2278-2324.
[9] Wen Y, Zhang K, Li Z, et al.A discriminative feature learning approach for deep face recognition[C]//European Conference on Computer Vision, Springer International Publishing, 2016: 499-515.
[10] Sun Y, Wang X, Tang X. Deep learning face representation from predicting 10, 000 classes[C]//Computer Vision and Pattern Recognition. IEEE, 2014: 1891-1898.
[11] Girshick R, Donahue J, Darrell T, et al. Rich feature hierarchies for accurate object detection and semantic segmentation[C]//IEEE Conference on Computer Vision and Pattern Recognition. IEEE Computer Society, 2013: 580-587.
[12] Ren S, He K, Girshick R, et al. Faster R-CNN: Towards real-time object detection with region proposal networks[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 2017, 39(6):1137-1149.
[13] Krizhevsky A, Sutskever I, Hinton G E. Imagenet classification with deep convolutional neural networks[C]// Advances in Neural Information Processing Systems, 2012: 1097-1105.
[14] Szegedy C, Liu W, Jia Y, et al. Going deeper with convolutions[C]//IEEE Computer Society. IEEE Conference on Computer Vision and Pattern Recognition, 2014: 1-9.
[15] He K, Zhang X, Ren S, et al. Deep residual learning for image recognition[C]//Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition, 2016: 770-778.
[16] 龚丁禧. 基于卷积神经网络的植物叶片分类[J]. 计算机与现代化,2014(4):12-15. Gong Dingxi. Plant leaf classification based on CNN[J]. Computer and Modernization, 2014(4): 12-15. (in Chinese with English abstract)
[17] 张帅, 淮永建. 基于分层卷积深度学习系统的植物叶片识别研究[J]. 北京林业大学学报,2016,38(9):108-115. Zhang Shuai, Huai Yongjian.Leaf image recognition based on layered convolutions neural network deep learning[J]. Journal of Beijing Forestry University, 2016, 38(9): 108-115. (in Chinese with English abstract)
[18] Sladojevic S, Arsenovic M, Anderla A, et al. Deep neural networks based recognition of plant diseases by leaf image classification[J]. Computational Intelligence and Neuroscience,2016(6): 1-11.
[19] Brahimi M, Boukhalfa K, Moussaoui A. Deep learning for tomato diseases: Classification and symptoms visualization[J]. Applied Artificial Intelligence, 2017: 1-17.
[20] Amara J, Bouaziz B, Algergawy A, et al. A deep learning- based approach for banana leaf diseases classification[C]// Datenbanksysteme Für Business, Technologie Und Web, 2017: 79-88.
[21] Ioffe S, Szegedy C. Batch normalization: Accelerating deep network training by reducing internal covariate shift[C]// International Conference on Machine Learning, 2015: 448-456.
[22] Hinton G E, Srivastava N, Krizhevsky A, et al. Improving neural networks by preventing co-adaptation of feature detectors[J]. Computer Science, 2012, 3(4): 212-223.
[23] Wan L, Zeiler M, Zhang S, et al. Regularization of neural networks using dropconnect[C]//Proceedings of the 30th International Conference on Machine Learning (ICML-13), 2013:1058-1066.
[24] Wang X Z, Xing H J, Li Y, et al. A study on relationship between generalization abilities and fuzziness of base classifiers in ensemble learning[J]. IEEE Transactions on Fuzzy Systems, 2015, 23(5): 1638-1654.
[25] Lin M, Chen Q, Yan S. Network in network[C]//Computer Vision and Pattern Recognition. ICLR, 2014.
[26] 杨国国,鲍一丹,刘子毅. 基于图像显著性分析与卷积神经网络的茶园害虫定位与识别[J]. 农业工程学报,2017,33(6): 156-162. Yang Guoguo, Bao Yidan, Liu Ziyi. Localization and recognition of pests in tea plantation based on image saliency analysis and convolutional neural network[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2017, 33(6): 156-162. (in Chinese with English abstract)
[27] Glorot X, Bordes A, Bengio Y. Deep sparse rectifier neural networks[C]//Proceedings of the Fourteenth International Conference on Artificial Intelligence and Statistics, 2011: 315-323.
[28] He K, Zhang X, Ren S, et al. Delving deep into rectifiers: Surpassing human-level performance on imagenet classification[C]// IEEE International Conference on Computer Vision. IEEE Computer Society, 2015: 1026-1034.
[29] Jia Y, Shelhamer E, Donahue J, et al. Caffe: Convolutional architecture for fast feature embedding[J]. Eprint Arxiv, 2014: 675-678.
[30] 杨倩,高晓阳,武季玲,等. 基于颜色和纹理特征的大麦主要病害识别研究[J]. 中国农业大学学报,2013,18(5):129-135. Yang Qian, Gao Xiaoyang, Wu Jiling, et al.Identification of barley diseases based on texture color feature[J].Journal of China Agricultural University,2013, 18(5): 129-135. (in Chinese with English abstract)
孙 俊,谭文军,毛罕平,武小红,陈 勇,汪 龙.基于改进卷积神经网络的多种植物叶片病害识别[J]. 农业工程学报,2017,33(19):209-215. doi:10.11975/j.issn.1002-6819.2017.19.027 http://www.tcsae.org
Sun Jun, Tan Wenjun, Mao Hanping, Wu Xiaohong, Chen Yong, Wang Long. Recognition of multiple plant leaf diseases based on improved convolutional neural network[J]. Transactions of the Chinese Society of Agricultural Engineering (Transactions of the CSAE), 2017, 33(19): 209-215. (in Chinese with English abstract) doi:10.11975/j.issn.1002-6819.2017.19.027 http://www.tcsae.org
Recognition of multiple plant leaf diseases based on improved convolutional neural network
Sun Jun1, Tan Wenjun1, Mao Hanping2, Wu Xiaohong1, Chen Yong1, Wang Long1
(1.212013,; 2.212013,)
Plant leaf diseases are a serious problem in agricultural production. To solve this problem and prevent diseases deterioration, accurate identification of diseases types is of great significance. In this paper, we proposed a recognition model of plant leaf diseases based on convolutional neural network (CNN), which combines the batch normalization and global pooling methods. The parameters of the traditional CNN model are large and have difficulty to converge. The proposed model was modified in the traditional structure of the CNN, which could optimize the training time and achieve the higher accuracy, and also reduce the size of model. In order to speed up the training convergence, we used the batch normalization layers. We put the input of every convolutional layer in batch, calculated the mean and variance of the batch, and then normalized this batch. We reduced some feature maps of some layers and removed the last full connect layer, with the global pooling layer instead. The proposed model has 5 convolutional layers and 4 pooling layers. In the last pooling layer pool5, the same kernel size of convolutional layer Conv5 was used to take advantage of the information of Conv5’s feature map comprehensively. For the image preprocessing, we had zoomed, flipped and rotated the original pictures of dataset randomly to get the augmented dataset, and used the 80% of pictures as the train dataset and the rest as the test dataset. These pictures were quantized to 256×256 dpi for CNN training, and the original dataset and augmented dataset were used to train models. To look for the best size of the first layer kernel, in the first convolutional layer, different kernel sizes i.e. 11×11, 9×9 and 7×7 dpi were used respectively. Furthermore, we chose the type of global pooling layer, like max pooling and average pooling. Then we designed 8 models with different Conv1 kernel sizes or global pooling types. To further improve the efficiency of this model, besides using the Gaussian initialization, we used the other common type of convolutional initialization such as Xavier initialization, and also used the PRelu activation function for each convolution layer. So the optimal model could be selected to recognize the 26 kinds of leaf diseases which involved 14 kinds of plants, and then we analyzed the model’s convergence rate, memory usage and robustness. After the experiment, we compared the test accuracy between the traditional model and the proposed model based on original dataset and augmented dataset. The proposed model could accelerate the training convergence, and the test accuracy could achieve about 90% while the traditional model was only about 77% after 3 training epochs. Different kernel sizes of Conv1 had little impact on the accuracy but small kernel was proved to be more beneficial to the recognition of plant diseases, which could get more texture features than the big kernel size filter, and average pooling also made better results than max pooling. We got the best performance model which used the 9×9 dpi kernel size and global average pooling layer. To show the proposed model’s performance, we tested the accuracy on each class, and the mean accuracy of augmented test dataset was 99.56%, and the weighted average score of recall and precision rate achieved 99.41%. The proposed model had the size of only 2.6 MB. In addition, compared with the traditional methods, the change of the spatial position of the pictures had little effect on the performance of the improved model, and the proposed model could identify different diseases of various plant leaves. The results show that the model has higher recognition accuracy and stronger robustness, and can be used for the identification of plant leaf diseases.
diseases; plants; image processing; recognition; convolutional neural network; batch normalization; global pooling; deep learning
10.11975/j.issn.1002-6819.2017.19.027
S126
A
1002-6819(2017)-19-0209-07
2017-05-17
2017-09-13
国家自然科学基金资助项目(No.31471413);江苏高校优势学科建设工程资助项目PAPD(苏政办发2011 6号);江苏省六大人才高峰资助项目(ZBZZ-019);江苏大学大学生科研立项资助项目(Y15A039);江苏大学大学生实践创新训练项目(No.46)
孙 俊,江苏泰兴人,教授,博士,博士生导师,研究方向为计算机技术在农业工程中的应用。Email:sun2000jun@ujs.edu.cn
中国农业工程学会会员:孙俊(E041200652S)

