朱砂叶螨β-COP和Sro基因鉴定及其沉默致死效果
敖已倩云,申光茂,王梦瑶,刘家路,潘宇,何林
(1西南大学植物保护学院/农药毒理与科学应用实验室,重庆 400716;2西南大学农业科学研究院,重庆 400716;3西南大学园艺园林学院,重庆 400716)
朱砂叶螨β-COP和Sro基因鉴定及其沉默致死效果
敖已倩云1,2,申光茂1,2,王梦瑶1,2,刘家路1,2,潘宇3,何林1,2
(1西南大学植物保护学院/农药毒理与科学应用实验室,重庆 400716;2西南大学农业科学研究院,重庆 400716;3西南大学园艺园林学院,重庆 400716)
【目的】明确朱砂叶螨(Tetranychus cinnabarinus)β-COP和Sro两条基因分子生物学信息,并基于优化的RNAi体系评价它们的致死效应,为筛选适用于RNAi防控的靶基因打下基础。【方法】首先克隆目的基因的全长,并通过序列的同源比对、保守区域及蛋白结构预测以及系统进化树的构建明确其分子生物学信息;其次通过减少dsRNA降解因素、并适时补充dsRNA等方法改进朱砂叶螨的RNAi体系,从而延长干扰时间。利用定量PCR技术检测特定时间点的沉默效率,评价优化RNAi体系后的沉默效果;最后在沉默目的基因β-COP和Sro后,检测朱砂叶螨在各特定时间点的死亡率,评价目的基因在RNAi下的致死效果,并观察相应的致死表型。【结果】β-COP开放阅读框长度为2 688 bp,编码895 aa,属于“WD40 superfamily”及“Coatomer_ WDAD superfamily”,包含了“WD 40”和“Coatomar_WDAD”的保守区域。Sro的开放阅读框长度为1 119 bp,编码372 aa,具有典型的短链脱氢酶所特有的两个特征,即与NADP+biding 结合位点基序“TGxxxGx”和“YxxxK”及其上游的天冬氨酸(Asn)、丝氨酸(Ser)活性位点。优化后的RNAi体系可在96 h内能稳定保持50%左右的沉默效率,使用该方法干扰朱砂叶螨β-COP和Sro两条致死基因48 h后试验组与对照组的死亡率均有显著性差异,用β-COP的dsRNA片段干扰雌成螨108 h后,死亡率达57.4%;用Sro的dsRNA片段干扰若螨96 h后,死亡率达28.8%。死亡个体均具有明显的表型,β-COP试验组死亡表型为4对足蜷缩在体侧;Sro试验组则在静伏期死亡或在蜕皮过程中无法蜕皮而死亡。【结论】优化的朱砂叶螨RNAi技术有稳定持续的沉默效果。β-COP和Sro与RNAi技术相结合,验证了这两条目的基因的沉默可对朱砂叶螨产生一定的致死效应,表明此类基因与技术加成的模式极具开发和利用的潜力,为今后基因功能验证和筛选以及开发以 RNAi技术为基础的朱砂叶螨防控方法提供了依据。
朱砂叶螨;RNA干扰技术;致死效应;综合绿色防控
Abstract:【Objective】 The objective of this study is to clarify the molecular information of Sro and β-COP genes of Tetranychus cinnabarinus, using the optimized RNAi system to study the lethal effects of these two genes on the T. cinnabarinus,and to provide a basic information to identify target genes, which are useful in the application of RNAi control. 【Method】 Firstly,full sequences of the two genes were identified and the bioinformatics were analyzed by homologous alignment of the sequence,prediction of the conserved region, protein structure and construction of the phylogenetic tree. Secondly, the RNAi system was improved by reducing the factors that will degrade the dsRNA, timely replenishment of dsRNA and other methods, and the gene silencing effect of the new RNAi system was evaluated by quantitative PCR technique. Finally, based on the optimized RNAi system,the expressions of β-COP and Sro genes were silenced to evaluate the lethal effects, and the lethal phenotypes were observed.【Result】The length of open reading frame of β-COP is 2 688 bp, encoding a 895 aa protein, which belonging to “WD40 superfamily” and “Coatomer_WDAD superfamily”, containing the conserved regions of “WD 40” and “Coatomar_WDAD”. The length of open reading frame of Sro is 1 119 bp, encoding a 372 aa protein. It has two characteristics of typical short chain dehydrogenase: one is binding sequence of NADP+biding binding sequence “TGxxxGx”, the other is “YxxxK” Motif and its upstream aspartic acid (Asn), serine (Ser) active sites. The optimized RNAi could maintain the silencing efficiency of about 50%after 96 h. Based on this system, the expressions of β-COP and Sro were silenced and the death rate showed significant differences between experimental group and control group after 48 h. β-COP dsRNA caused 57.4% mortality rates at 108 h post-treatment, and Sro dsRNA caused 28.8% death rates at 96 h post-treatment. In addition, the phenotype of dead individuals in the β-COP group showed that the four pairs of feet were curled up on the body side and died, and in the Sro group, the mites dead during the molting process. 【Conclusion】The optimized RNAi technique has stable and continuous silencing effect on the T. cinnabarinus. The combination of β-COP and Sro genes functional verification with RNAi technique showed that the silencing of the two target genes can produce a certain lethal effect on the mite. It also shows that the combination of gene and technology has great potential for development and utilization, which can lay a basis for the future functional verification, screening gene and developing the RNAi technology to control the T. cinnabarinus.
Key words:Tetranychus cinnabarinus; RNA interference technology; lethal effect; comprehensive prevention and green control
0 引言
【研究意义】朱砂叶螨(Tetranychus cinnabarinus)是一种在温带地区广泛分布的杂食性农林害螨,在中国各地均有发生,可危害140科1 100种作物或杂草,以成若螨在叶背吸取汁液危害,严重时导致全叶干枯脱落,缩短结果期,影响产量。多年来,施用化学农药是防治该害螨的主要方式,但由于螨体小、世代多、繁殖速度快、发育历期短的特点,极易产生抗药性[1]。RNAi技术作为一种基因沉默的有效工具,近年来成为一种有效的有害生物防治新方法而受到广泛关注[2-4]。因此,筛选适用于RNAi技术的朱砂叶螨致死基因,既可增进对基因功能的了解,对该害螨新型防治方法的开发也具有重要意义。【前人研究进展】RNAi技术是验证基因功能的重要技术之一,如何将 dsRNA成功导入目标生物体内是沉默目的基因的关键步骤,目前主要使用的方法有饲喂法、注射法和利用载体媒介将 dsRNA导入等[5-11]。2006年,ARAUJO 等[12-13]通过饲喂法沉默了长红烈蝽(Rhodnius prolixus)NP2和苹果透翅蛾(Epiphyas postvittana)EposCXE1,结果发现通过饲喂dsRNA降低了靶标基因转录水平,表明通过饲喂可以达到基因沉默的效果。由于该方法操作简单方便,对虫体造成的伤害小,因此其不仅适用于一些难于采用注射RNAi研究的小型害虫,而且还是一种快速筛选靶基因的途径,为害虫、害螨的可持续治理提供重要的依据与保障。植食性叶螨的RNAi一般采用饲喂法,由于体外合成的dsRNA易降解,因此SHI等[14]基于RNAi的朱砂叶螨基因功能验证试验基本限定在48 h内。但要验证目的基因的沉默致死效应需要观察更长的时间,因此亟需对朱砂叶螨的饲喂法进行改良。同时,要将RNAi技术应用于害虫、害螨的防控中,需要选择合适的靶基因,目前常用的候选基因功能大都与害虫、害螨的生长发育、生殖行为和繁殖能力密切相关,干扰后能使害虫、害螨出现畸形、交配能力下降、繁殖力减弱,甚至死亡等现象,这些基因有的是调控能量代谢方面的酶(质子代谢的V-ATPase,毒物质代谢的细胞色素P450酶等)[14-16],也有与生长发育相关的激素(蜕皮激素、保幼激素等)[17-20],还有针对神经调控通路的受体和酶[7]。在此基础上通过与其他技术相结合,使RNAi技术用于防控害虫、害螨的潜能大大提升,例如TIAN等[21]用大肠杆菌表达dsRNA方法来饲喂甜菜夜蛾(Spodoptera exigua),发现连续取食dsRNA后几丁质合成酶基因SeCHSA的表达受到了抑制,且昆虫的蜕皮发育也受到了阻碍;PITINO等[22]通过对桃蚜(Acyrthosiphon pisum)饲喂表达肠道基因Rack1和唾腺基因MpC002 dsRNA的转基因烟草和拟南芥,发现目标基因的表达被抑制达60%以上,而且目标基因被抑制的桃蚜所产的后代数量更少;THAIRU等[9]将siRNA与纳米颗粒结合形成复合物,沉默了3种蚜虫(Acyrthosiphon pisum、Aphis glycines和Schizaphis graminum)的胡萝卜素脱氢酶(tor)基因,可影响蚜虫的生长发育并出现相关表型;ZHANG等[5]利用质体介导的 RNAi技术防控马铃薯甲虫(Leptinotarsa decemlineata),并取得较好的效果。在生物体中,β-COP与其他外被体蛋白相互作用共同形成一种复合物COPI。COPI介导的囊泡逆向转运在维持内质网和高尔基体结构与形态、调节神经系统、生长发育和能量代谢等方面发挥重要作用,特别是对卵巢的发育及食物消化代谢有影响。ISOE等[23]报道COPI缺陷的埃及伊蚊(Aedes aegypti)在96 h后死亡率高达80%。Sro作为蜕皮激素合成过程中的限速还原酶,在家蚕(Bombyx mori)和果蝇(Drosophila melanogaster)的报道中均表明其对生长发育中的蜕皮过程可产生重要影响,该基因缺陷后可致畸、致死[11,24-25]。【本研究切入点】虽然适用于 RNAi技术防控的靶基因筛选已在多种昆虫中进行了研究,但是关于朱砂叶螨靶基因的筛选还未见报道。因此,选取可能会对螨类的生长发育和生命活动有重要影响的两条基因,即与能量代谢、神经调节相关的β′-外被体蛋白(简称β-COP)和与生长发育相关的蜕皮激素-β-11-短链脱氢酶(简称 Sro)基因为研究对象,在明确其分子生物学信息的基础上评估它们用于RNAi时的致死效果。【拟解决的关键问题】以朱砂叶螨为研究对象,使用改良的RNAi技术,在保证目的基因沉默效率的基础上延长干扰时间,记录朱砂叶螨的死亡率及死亡表型等,筛选适用于RNAi技术的致死基因,为基因功能的研究提供新思路,并为该螨的绿色防控打下基础。
1 材料与方法
试验于 2016—2017年在西南大学植物保护学院农药毒理与科学应用实验室完成。
1.1 供试螨源
供试朱砂叶螨于 1997年采于重庆市北碚区田间豇豆苗上,饲养于室内豇豆苗上,饲养条件:温度25—27℃,相对湿度35%—55%,光周期14 h光照∶10 h黑暗,在饲养期间没有接触或使用任何药剂。
1.2 主要试剂
动物组织总RNA提取试剂盒(TIANGEN公司);反转录试剂盒(PrimeScriptTMRT reagent Kit,TaKaRa公司);Taq DNA聚合酶(TaKaRa公司);琼脂糖凝胶 DNA回收试剂盒(TIANGEN公司);载体(pGEM-T Easy Vector,TaKaRa公司);感受态细胞T1(TaKaRa公司);qPCR 试剂(GoTaq® qPCR Master Mix,Promega公司);dsRNA基因体外转录试剂盒(Thermo公司)。
1.3 主要仪器
核酸蛋白质浓度测定仪 NanoVue(GE Health care);q TOWER 2.2荧光定量梯度PCR仪(quantitative PCR instrument,Analytik Jena AG公司);立式压力蒸汽灭菌器 YXQ-LS-50G(上海博迅实业公司);CT14RD高速冷冻离心机(上海天美生化仪器设备工程有限公司);德国 Leica莱卡体现显微镜 M205A(Leica公司)。
1.4 基因克隆
1.4.1 总RNA的提取 从豇豆苗上挑取成螨,收集雌成螨200头于1.5 mL离心管中,至少重复收集3管,按照动物组织总 RNA提取试剂盒使用说明书逐步提取,完成后使用核酸蛋白质浓度测定仪NanoVue(GE Health care)检测OD260/OD280是否在1.8—2.2范围内,保证所提取的RNA样品的浓度和纯度,然后再利用1%琼脂糖凝胶电泳进一步检测其完整性。将检测合格的总 RNA保存于-80℃冰箱备用或立即进行后续试验。
1.4.2 cDNA第一链合成 采用 PrimeScriptTMRT reagent Kit(TaKaRa)反转录试剂盒合成cDNA第一链。试验全程使用RNase离心管,反应液的配置均在冰上完成并按照说明书逐步进行,合成cDNA保存于-20℃或立即进行后续试验。
1.4.3 β-COP与Sro全序列扩增 将两条致死基因的核苷酸序列在二斑叶螨(Tetranychus urticae)基因组数据库中进行比对,记录比对后匹配度最高的基因(二斑叶螨与朱砂叶螨是姐妹种,具有最为相似的基因组,因此可以作为本研究的参考基因组)。使用Primer Premier 5.0 软件分别设计引物,然后使用朱砂叶螨cDNA作为模板进行两端扩增。扩增所用的25 μL PCR 体系:2 μL cDNA、14 μL ddH2O、2.5 μL 10×PCR buffer、2.5 μL MgCl2、2 μL dNTPs、0.25 μL Taq 酶以及上下游引物各1 μL。扩增条件:95℃预变性3 min,95℃变性30 s,60℃复性30 s,72℃延伸1 min,共35个循环,最后72℃延伸10 min。用1%琼脂糖凝胶电泳检测并回收目的条带,胶回收按试剂盒说明书操作,回收产物连接到pGEM-T Easy Vector上,再转化至感受态大肠杆菌中,通过蓝白斑筛选,经PCR鉴定的阳性克隆送往华大基因公司测序。
1.4.4 信息学分析 将测序正确的核苷酸序列,使用NCBI数据库BLAST程序进行同源序列比对以及保守区域的检测(http://blast.ncbi.nlm.nih.gov/Blast.cgi),并使用作图软件IBS 1.0.1进行绘制[26]。选取NCBI数据库氨基酸比对结果,下载模式物种黑腹果蝇等及与目的基因相似性最高的数条氨基酸序列,采用邻接法(neighbor-joining,NJ)在MEGA7.0.2软件中完成系统发育树的构建[27],各分支重复检验次数均为1 000。
1.5 RNAi
1.5.1 前期准备 从豇豆苗上挑取成螨,使用叶碟法饲养。产卵24 h后挑除叶碟上的成螨,保证试验用螨为同一历期,后逐日观察,收集3日龄雌成螨,用于β-COP RNAi试验;收集5日龄若螨,用于Sro RNAi试验,开始干扰试验前高温灭菌所需要的培养皿及焦碳酸二乙酯(以下简称DEPC)处理的枪头、PCR管、水等。
使用表1所列出的目的基因特异性片段引物和绿色荧光蛋白GFP(ACY56286)基因片段引物,按1.4.3方法克隆扩增特异性靶标基因片段,获得胶回收产物,严格按照试剂盒说明书合成及纯化dsRNA,合成纯化过程均在冰上进行并使用DEPC处理后无酶枪头及离心管,具体步骤:(1)使用基因体外转录试剂盒(Themo公司),冰上融化所需试剂;(2)质粒模板 1 μg,10×Reaction Buffer 2 μL,ATP/CTP/GTP/UTP 各 2 μL,T7 Enzyme Mix 2 μL,Neclease-free water补至 20 μL,37℃孵育 2 h;(3)加 2 μL DNaseI,37℃反应15 min;2 μL 0.5 mol·L-1EDTA,65℃反应 10 min;(4)加 115 μL DEPC-treated water,15 μL 3 mol·L-1Sodium Acetate Solution(pH 5.2),混匀后依次加150 μL苯酚氯仿混合液(苯酚∶氯仿=1∶1,现配现用)和300 μL氯仿,混匀,12 000 r/min、4℃离心10 min;(5)取上清至新管,加入350 μL无水乙醇,-20℃放置2 h;(6)12 000 r/min、4℃离心 10 min。去上清,加 500 μL 70%乙醇(预冷),7 500 r/min、4℃离心5 min;(7)弃废液,晾干沉淀,加入100 μL DEPC-treated water,检测 dsRNA浓度;(8)DEPC-treated water稀释至200 ng·μL-1,分装存于-80℃备用。
以饲喂dsGFP作为阴性对照,饲喂DEPC处理的水作为空白对照。
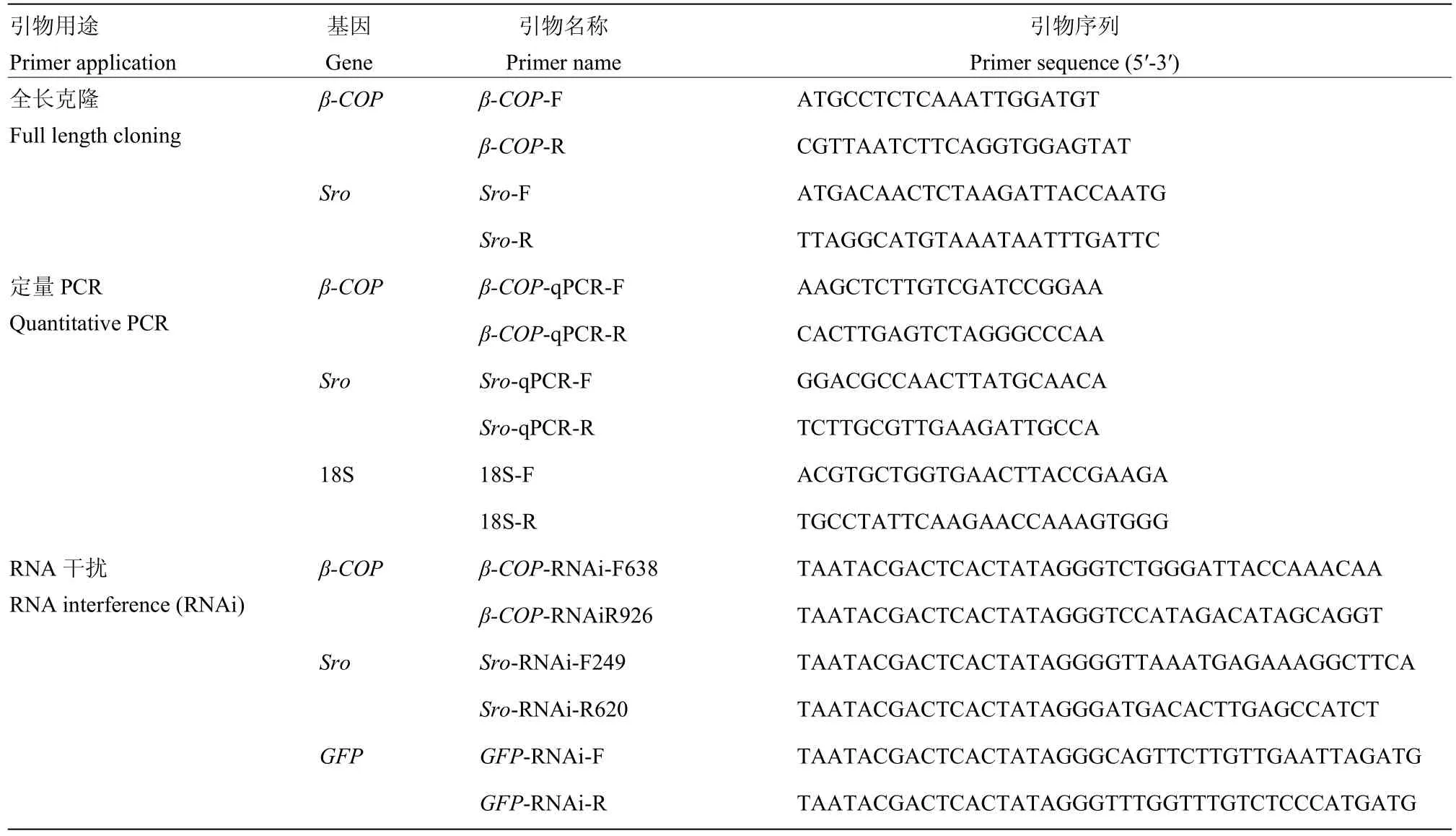
表1 引物信息Table 1 Primers used in this study
1.5.2 RNAi具体操作 参照SHI等[14]干扰方法并进行改进(图 1)。首先需要提前将朱砂叶螨的雌成螨饥饿处理24 h(由于Sro干扰试验使用的是若螨,所以没有进行饥饿处理),试验时用新鲜的嫩豇豆叶剪裁为合适大小的正方形(约2 cm×2 cm),60℃烘干1 min;在小培养皿中加入 100 μL 200 ng·μL-1的dsRNA,将叶片正面朝上放置2 h,充分吸收;后在小培养皿中放置滤纸,并将叶片背面朝上,晾干后(约2 h),在小培养皿中加入2 mL无酶水(每隔36 h向培养皿中添加 100 μL 200 ng·μL-1的 dsRNA),每片叶子上最多可挑约180头成螨。根据试验需要,用于每时段定量检测沉默效率的每片叶子上挑150—180头,各个时段收集雌成螨/若螨约200头于1.5 mL离心管中,至少重复收集3管,按照1.4.1中方法提取总RNA并检测,并使用反转录试剂盒(PrimeScriptTMRT reagent Kit,TaKaRa公司),转录后可保存于-20℃冰箱备用。而用于统计死亡率的试验,每片叶子上挑40—50头螨,试验至少重复3次,每12或24 h后开盖计数(跳水的螨将其挑至滤纸,等待一段时间后观察存活数量)。
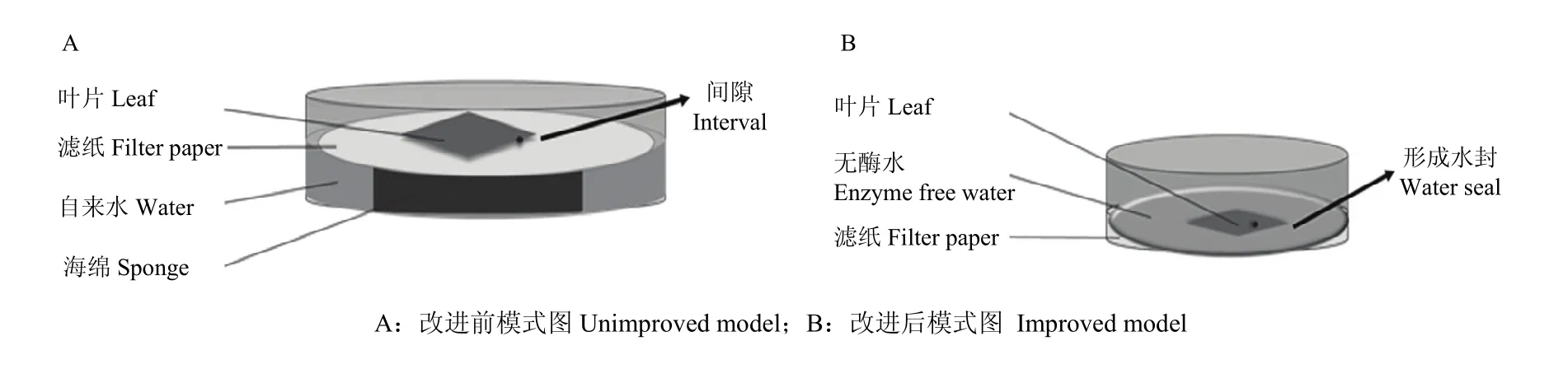
图1 RNA干扰方法对比图Fig. 1 Comparison of RNAi methods
1.6 沉默效率检测
使用实时荧光定量PCR技术检测饲喂dsRNA后目的基因沉默效率。设计并筛选基因特异的qPCR引物(表 1),反应体系为 20 μL:荧光染料 GoTaq® qPCR Master Mix 10 μL,Nuclease-free Water 7 μL,上下游引物各1 μL,模板cDNA 1 μL。反应条件:95℃预变性2 min;95℃变性15 s,60℃退火30 s,共40个循环;并在每个循环中插入熔解曲线,并选取 18S作为朱砂叶螨各时段饲喂dsRNA后qPCR检测的内参基因[28]。
1.7 数据处理及图片整合
通过饲喂目的基因的片段dsRNA后,对朱砂叶螨Sro和β-COP进行干扰,对特定时间目的基因的qPCR 数据采用 2-ΔΔCT方法进行计算[29],使用 IBM SPSS 22.0软件对目的基因相对表达量及死亡率进行显著性分析(ANOVA,Duncan’s tests,P<0.05),数据表示为平均值±标准误,各试验均独立重复 3次。死亡表型拍摄使用德国Leica M205A显微镜,在同一时间,使用相同倍数进行拍摄,图片使用Adobe Photoshop CS6软件编辑,固定选区大小(240×300像素),并调整选区,将选区中轴线尽量与虫体中线对齐截取并复制选区,最后将截取的图片排列整齐。
2 结果
2.1 基因克隆与全长分析
β-COP的开放阅读框长度为2 688 bp,编码895 aa,GenBank登录号为MF045039。通过NCBI比对发现该基因属于“WD40 superfamily”及“Coatomer_WDAD superfamily”,并且包含了“WD 40”和“Coatomar_WDAD”的保守区域。在预测的蛋白三维结构图中可清晰地看到“WD 40”的重复结构(图2-A、2-B)。
Sro的开放阅读框长度为1 119 bp,编码372 aa,GenBank登录号为MF045040。通过NCBI比对发现该基因具有典型的短链脱氢酶所特有的两个特征:一是位于氨基酸70—76位的NADP+biding结合位点基序“TGxxxGx”,二是位于229—233位的“YxxxK”基序与上游的180位天冬氨酸(Asn)、216位丝氨酸(Ser)等活性位点可形成典型的催化四分体(图2-C、2-D)。
2.2 系统发育分析
将β-COP氨基酸序列提交NCBI数据库对比后,收集参考主要模式物种以及其他物种的相关序列,使用邻接法构建系统发育树,各分支重复检验次数均为1 000。结果表明,朱砂叶螨β-COP与二斑叶螨β-COP的同源性最高(99%),然后与疥螨(Sarcoptes scabiei)聚为一支(图3),从外被体蛋白-β的聚类情况来看,各个纲或目的物种均单独成一支,与传统分类一致。
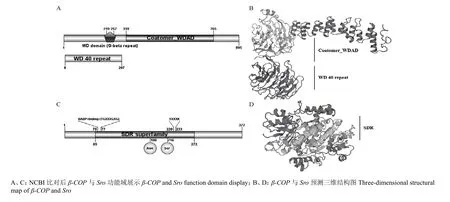
图2 β-COP与Sro生物信息学分析Fig. 2 β-COP and Sro bioinformatics analysis
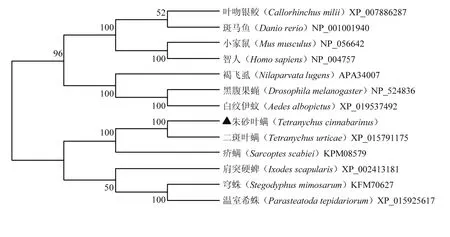
图3 朱砂叶螨β-COP系统发育分析Fig. 3 Phylogenetic analysis of β-COP of T. cinnabarinus
将Sro氨基酸序列提交NCBI数据库对比后,收集主要模式物种以及其他物种的 shroud或 17-β-羟化类固醇脱氢酶(简写为 17-β-D)的氨基酸序列,使用邻接法构建系统发育树,各分支重复检验次数均为1 000。结果与β-COP相似,即目的基因与二斑叶螨的shroud同源性最高,其次是疥螨等(图4)。
2.3 RNAi沉默效率及死亡率检测
改进的RNAi体系在干扰3日龄雌成螨108 h后依然可保持对β-COP 50%左右的沉默效率,死亡个体与正常个体相比较,有较为明显的死亡表型,均表现为 4对足蜷缩在体侧(图5-A、图 6)。试验组与对照组的死亡率在干扰36 h后差异显著,并且在108 h达到57.4%的死亡率(图7)。
而干扰3日龄雌成螨的Sro 96 h后,目的基因的沉默效率下降(表 2),且不能观察到相应表型。但干扰若螨的Sro 96 h,目的基因的沉默效率在50%左右,死亡个体与正常个体相比较,均出现蜕皮过程受阻的现象,具体表现为在静伏期死亡,或在蜕皮过程中不能蜕皮而死亡(图5-B、图8)。试验组与对照组的死亡率在干扰48 h后差异显著(图9)。
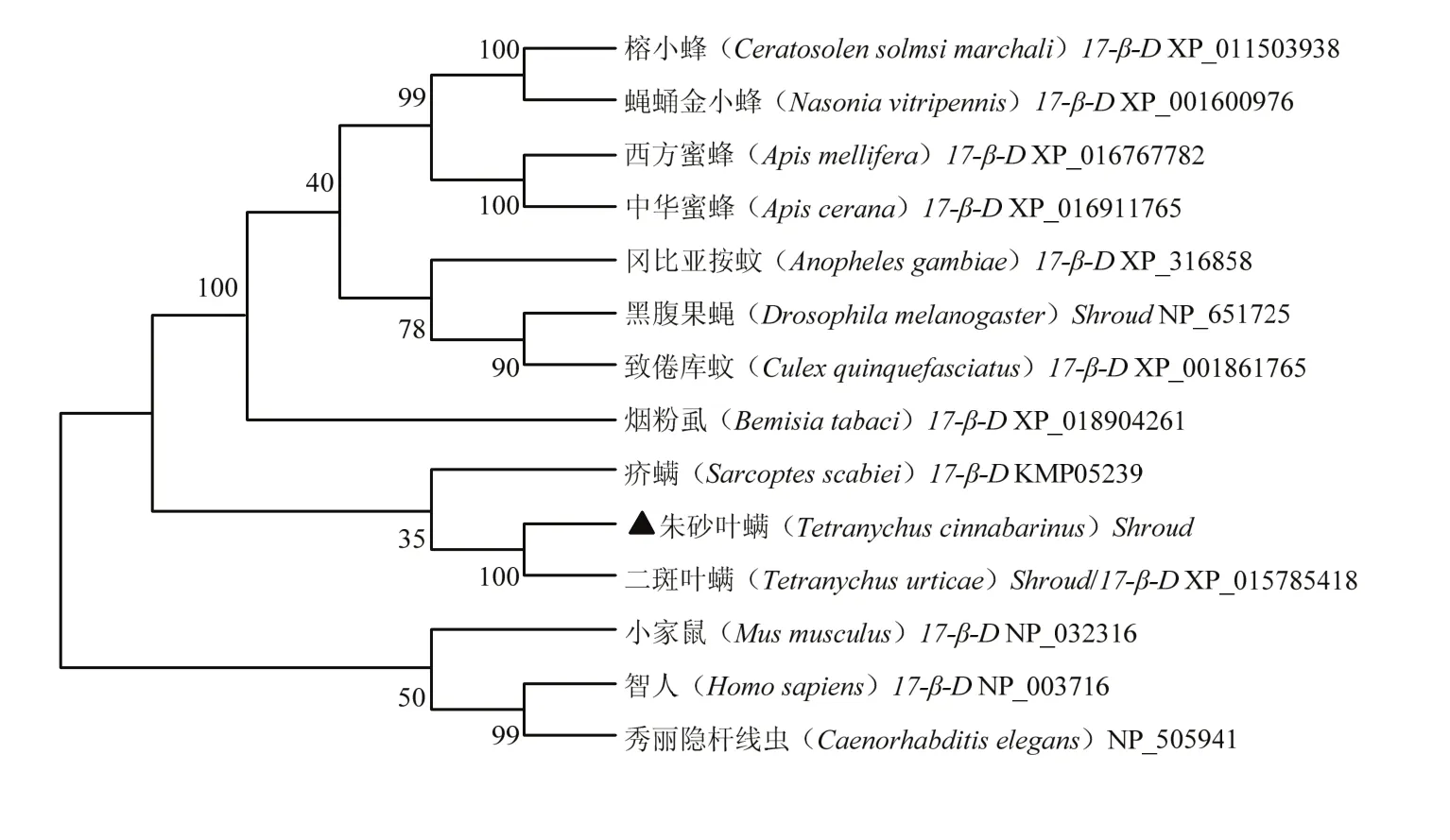
图4 朱砂叶螨Sro系统发育分析Fig. 4 Phylogenetic analysis of Sro of T. cinnabarinus

表2 干扰3日龄雌成螨Sro的qPCR检测结果Table 2 The results of qPCR of Sro RNAi with 3-day-old female adult mite
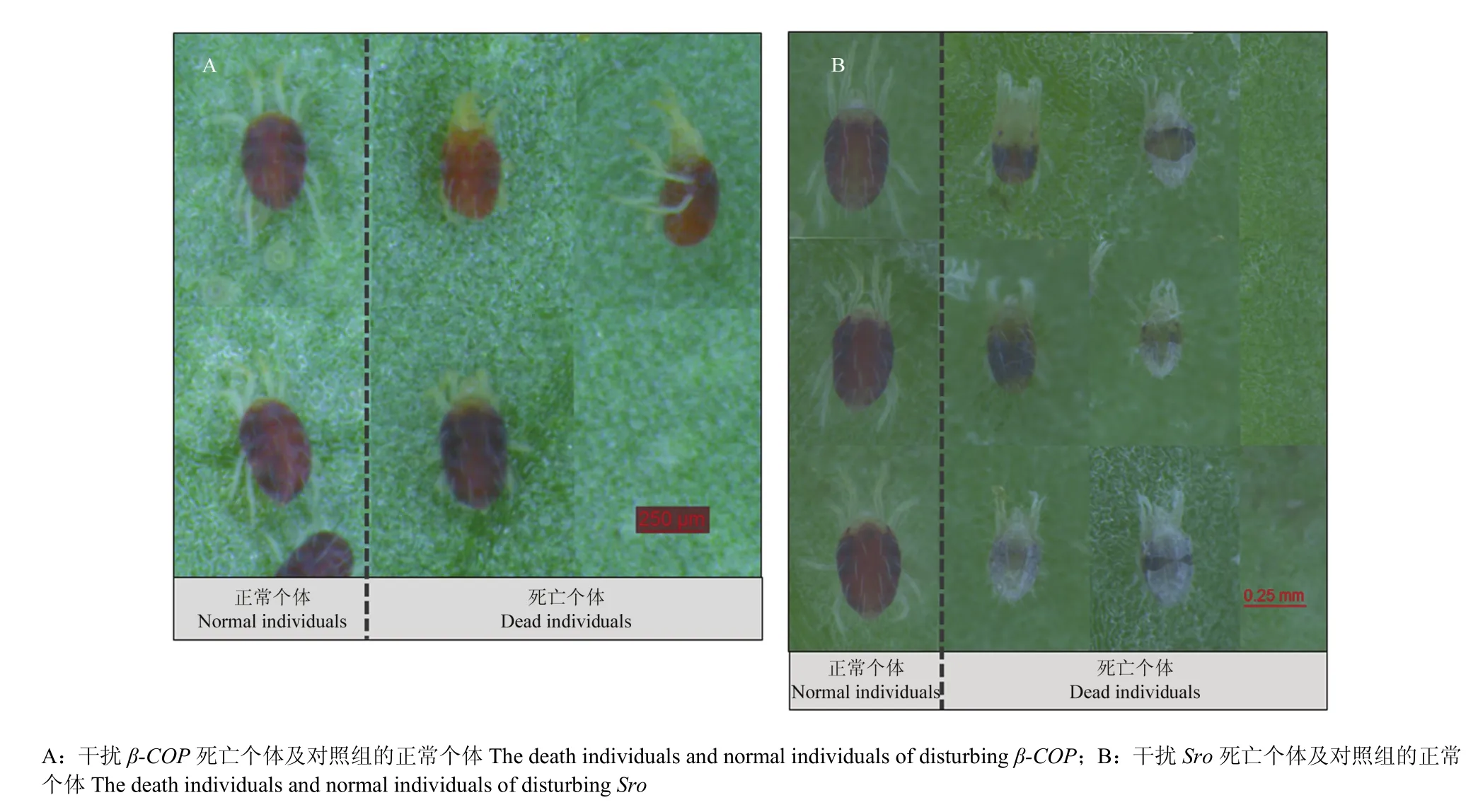
图5 显微拍摄死亡表型Fig. 5 Microscopic pictures of the death phenotype
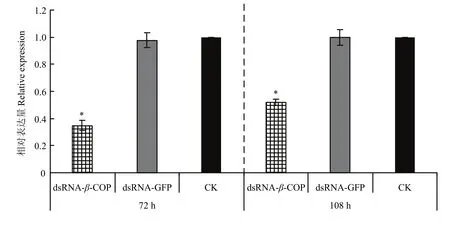
图6 各时间点β-COP相对表达量Fig. 6 Relative expression of β-COP at different time points
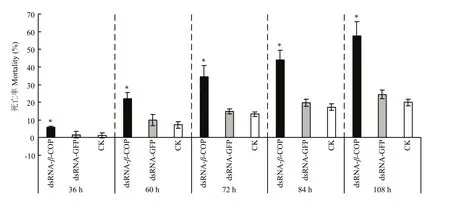
图7 β-COP RNAi后死亡率检测Fig. 7 The mortality of β-COP RNAi
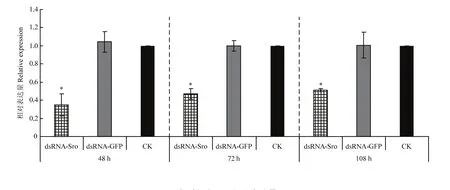
图8 各时间点Sro相对表达量Fig. 8 Relative expression of Sro at different time points
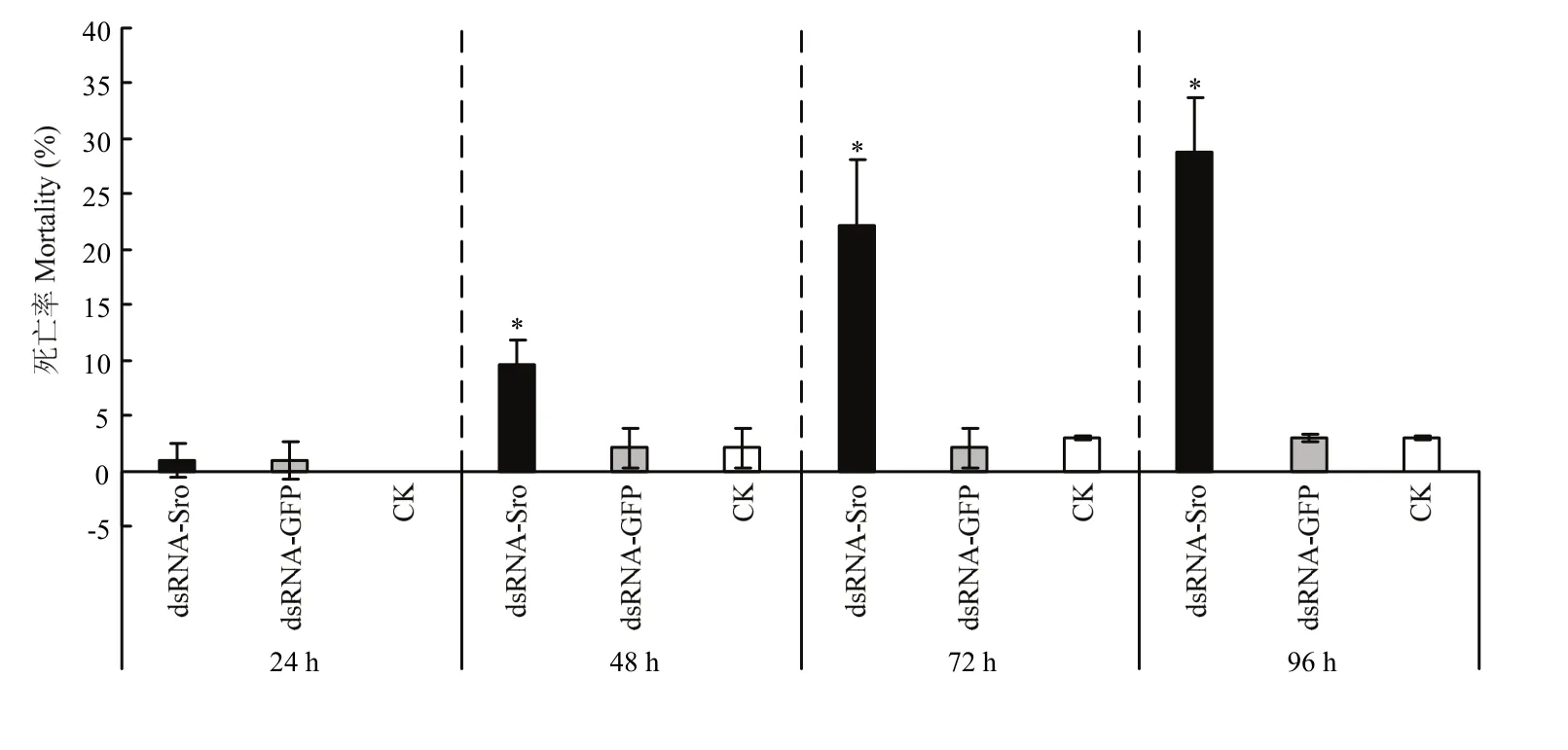
图9 Sro RNAi后死亡率检测Fig. 9 The mortality of Sro RNAi
3 讨论
改进后的RNAi技术主要有以下3个优点:(1)试验材料经DEPC处理灭菌后形成无酶环境,从而减少可能降解 dsRNA的因素;(2)利用叶片与无酶dsRNA溶液间的张力,形成液面略高于叶面的自然“水封”,减少朱砂叶螨跳水或避免其爬入背面的间隙中。朱砂叶螨在叶面背面取食危害,有研究认为朱砂叶螨在叶背取食可能是其躲避天敌的策略或本身具有避光的习性[30],改进后的方法可减少该螨跳水及爬进背面间隙等情况的发生频率,有利于死亡率等数据的统计;(3)持续添加dsRNA溶液,试验中每隔36 h,就添加约20 μg的dsRNA,保证试验时dsRNA浓度在相对适合的范围,从而延长干扰时间,保证目的基因沉默效率维持在较高水平。
RNAi技术是否取得成功,还受到以下因子的影响[31-32]:(1)最佳的dsRNA浓度,浓度不佳会影响最终的干扰效果。本试验不仅最大限度地减少dsRNA降解的各类因素,还采用持续添加dsRNA的方法来保证试验时 dsRNA的浓度,而试验结果证明改进的RNAi方法可以在保证目的基因沉默效率较稳定的情况下实现较长时间的干扰试验;(2)核苷酸序列的特异性,主要从检测目的基因沉默效率和特定表型来判断dsRNA是否具有特异性。但由于同一个靶标基因在害虫的不同龄期、不同生育阶段会有不同的干扰效果[33],这就要根据靶标基因的功能来决定试验龄期,如本试验在进行朱砂叶螨Sro干扰时,曾采用与β-COP一样的3日龄雌成螨作为供试对象,结果观察不到相关表型,其根本原因就是Sro的功能是影响蜕皮过程而对成螨影响较小。因此,选择对若螨进行干扰时,观察到了与基因功能相符的表型。
β-COP是功能复合物COPI的重要组成部分[34],研究表明在埃及伊蚊中注射能引起COPI、COPII和网格蛋白缺陷的dsRNA后,发现只有缺失COPI后,蚊卵巢的发育和中肠消化血液的进程都受到了阻碍,证明该基因在消化代谢中起十分重要的作用[23]。本研究表明,干扰该基因后死亡个体均表现为4对足蜷缩,根据该基因的功能可推测其受到干扰可能会影响朱砂叶螨对植物汁液的消化,由于能量代谢不足导致出现类似于足无力而蜷缩、击倒的效果。基于该基因表现出较好的致死率,因此笔者认为β-COP可作为RNAi防控朱砂叶螨的靶基因。蜕皮激素是一类由前胸腺合成,具有蜕皮活性的甾类化合物,蜕皮激素在昆虫体内的合成过程为先借助Rieske加氧酶Neverland(Nvd)将胆固醇或植物甾醇转变为 7-脱氢胆固醇[19,25];经“black box”过程后,利用几种P450酶完成由三脱氧蜕皮酮向 20-羟基蜕皮酮的转变。该过程环环相扣,缺一不可,而11β-羟化类固醇脱氢酶(Sro)则是“black box”中重要的限速还原酶。NIWA 等[25]研究表明,干扰该基因能影响昆虫(家蚕)的蜕皮过程以致死亡,或过表达该基因导致昆虫发育和变态出现异常,甚至死亡。朱砂叶螨的生长发育过程中涉及多次蜕皮,而对 Sro的干扰可能会影响蜕皮激素合成中的“black box”过程。本研究表明沉默Sro对朱砂叶螨若螨具有致死效应,若将Sro应用于RNAi防控朱砂叶螨,通过影响蜕皮而干扰朱砂叶螨新螨态的形成,对于减少基础虫量有重大意义。
由于朱砂叶螨极易产生抗药性,因此其抗性治理也是研究的重点。目前,低毒农药及防控新技术的研发和应用是主要的发展方向。基于RNAi原理的有害生物防治技术已被多次报道,如结合转基因技术以植物或细菌为载体、或利用纳米材料作为运载工具实现对有害生物靶基因的RNAi等[5-6,9,35]。本研究得到两条适用于RNAi技术的靶标基因,不仅可用于室内基因功能等研究,还具有大田防治应用的潜力,在后续试验中将尝试用这些候选靶标基因通过转基因技术或纳米材料载体进行实际防控效果及植物互作等研究,为绿色防控提供依据。
4 结论
沉默朱砂叶螨β-COP和Sro后具有明显的致死效应,这两条基因作为RNAi的靶标在朱砂叶螨的防控中具有应用价值。
[1]何林, 赵志模, 邓新平, 王进军, 刘怀, 刘映红. 朱砂叶螨对3种杀螨剂的抗性选育及抗性治理研究. 中国农业科学, 2003, 36(4):403-408.HE L, ZHAO Z M, DENG X P, WANG J J, LIU H, LIU Y H.Resistance breeding of Tetranychus urticae to three kinds of acaricides and its resistance treatment. Scientia Agricultura Sinica,2003, 36(4): 403-408. (in Chinese)
[2]HUNTER C P. Genetics: a touch of elegance with RNAi. Current Biology, 1999, 9(12): R440-R442.
[3]CAPLEN N J, FLEENOR J, FIRE A, MORGAN R A. dsRNA-mediated gene silencing in cultured Drosophila cells: a tissue culture model for the analysis of RNA interference. Gene, 2000, 252(1/2):95-105.
[4]FAY D S, STANLEY H M, HAN M, WOOD W B. A Caenorhabditis elegans homologue of hunchback is required for late stages of development but not early embryonic patterning. Developmental Biology, 1999, 205(2): 240-253.
[5]ZHANG J, KHAN S A, HASSE C, RUF S, HECKEL D G, BOCK R.Full crop protection from an insect pest by expression of long double-stranded RNAs in plastids. Science, 2015, 347(6225):991-994.
[6]SHEN G M, SONG C G, AO Y Q Y, XIAO Y H, ZHANG Y J, PAN Y, HE L. Transgenic cotton expressing CYP392A4 double-stranded RNA decreases the reproductive ability of Tetranychus cinnabarinus.Insect Science, 2016, 24(4): 559-568.
[7]ARAKANE Y, LOMAKIN J, BEEMAN R W, MUTHUKRISHNAN S, GEHRKE S H, KANOST M R, KRAMER K J. Molecular and functional analyses of amino acid decarboxylases involved in cuticle tanning in Tribolium castaneum. The Journal of Biological Chemistry,2009, 284(24): 16584-16594.
[8]PRABHA S, VYAS R, GUPTA N, AHMED B, CHANDRA R,NIMESH S. RNA interference technology with emphasis on delivery vehicles-prospects and limitations. Artificial Cells, Nanomedicine, and Biotechnology, 2016, 44(6): 1391-1399.
[9]THAIRU M W, SKIDMORE I H, BANSAL R, NOVÁKOVÁ E,HANSEN T E, LI-BYARLAY H, WICKLINE S A, HANSEN A K.Efficacy of RNA interference knockdown using aerosolized short interfering RNAs bound to nanoparticles in three diverse aphid species. Insect Molecular Biology, 2017, 26(3): 356-368.
[10]SINGH N, AGRAWAL A, LEUNG AKL, SHARP P A, BHATIA S N. Effect of nanoparticle conjugation on gene silencing by RNA interference. Journal of the American Chemical Society, 2010,132(24): 8241-8243.
[11]ENDO S, MIYAGI N, MATSUNAGA T, HARA A, IKARI A.Human dehydrogenase/reductase (SDR family) member 11 is a novel type of 17β-hydroxysteroid dehydrogenase. Biochemical and Biophysical Research Communications, 2016, 472(1): 231-236.
[12]ARAUJO R N, SANTOS A, PINTO F S, GONTIJO N F, LEHANE M J, PEREIRA M H. RNA interference of the salivary gland nitrophorin 2 in the triatomine bug Rhodnius prolixus (Hemiptera:Reduviidae) by dsRNA ingestion or injection. Insect Biochemistry and Molecular Biology, 2006, 36(9): 683-693.
[13]TURNER C T, DAVY M W, MACDIARMID R M, PLUMMER K M,BIRCH N P, NEWCOMB R D. RNA interference in the light brown apple moth, Epiphyas postvittana (Walker) induced by doublestranded RNA feeding. Insect Molecular Biology, 2006, 15(3):383-391.
[14]SHI L, ZHANG J, SHEN G, XU Z, WEI P, ZHANG Y, XU Q, HE L.Silencing NADPH-cytochrome P450 reductase results in reduced acaricide resistance in Tetranychus cinnabarinus (Boisduval).Scientific Reports, 2015, 5: 15581.
[15]YOON J, SHUKLA J N, GONG Z J, MOGILICHERLA K, PALLI S R. RNA interference in the Colorado potato beetle, Leptinotarsa decemlineata: Identification of key contributors. Insect Biochemistry and Molecular Biology, 2016, 78: 78-88.
[16]KWON D H, PARK J H, LEE S H. Screening of lethal genes for feeding RNAi by leaf disc-mediated systematic delivery of dsRNA inTetranychus urticae. Pesticide Biochemistry and Physiology, 2013,105(1): 69-75.
[17]程道军, 李志清, 孟勐, 彭健, 钱文良, 康丽霞, 夏庆友. 家蚕蜕皮激素合成相关的细胞色素 P450基因的鉴定分析. 中国农业科学,2014, 47(3): 594-604.CHENG D J, LI Z Q, MENG M, PENG J, QIAN W L, KANG L X,XIA Q Y. Characterization of cytochrome P450 genes involving in ecdysteroidogenesis in silkworm (Bombyx mori). Scientia Agricultura Sinica, 2014, 47(3): 594-604. (in Chinese)
[18]TAN A, PALLI S R. Ecdysone receptor isoforms play distinct roles in controlling molting and metamorphosis in the red flour beetle,Tribolium castaneum. Molecular and Cellular Endocrinology, 2008,291(1/2): 42-49.
[19]YAMAZAKI Y, KIUCHI M, TAKEUCHI H, KUBO T. Ecdysteroid biosynthesis in workers of the European honeybee Apis mellifera L.Insect Biochemistry and Molecular Biology, 2011, 41(5): 283-293.
[20]OGIHARA M H, HIKIBA J, IGA M, KATAOKA H. Negative regulation of juvenile hormone analog for ecdysteroidogenic enzymes.Journal of Insect Physiology, 2015, 80: 42-47.
[21]TIAN H, PENG H, YAO Q, CHEN H, XIE Q, TANG B, ZHANG W.Developmental control of a lepidopteran pest Spodoptera exigua by ingestion of bacteria expressing dsRNA of a non-midgut gene. PLoS ONE, 2009, 4(7): e6225.
[22]PITINO M, COLEMAN A D, MAFFEI M E, RIDOUT C J,HOGENHOUT S A. Silencing of aphid genes by dsRNA feeding from plants. PLoS ONE, 2011, 6(10): e25709.
[23]ISOE J, STOVER W, MIESFELD R B, MIESFELD R L. COPI-mediated blood meal digestion in vector mosquitoes is independent of midgut ARF-GEF and ARF-GAP regulatory activities. Insect Biochemistry and Molecular Biology, 2013, 43(8): 732-739.
[24]KWON D H, PARK J H, ASHOK P A, LEE U, LEE S H. Screening of target genes for RNAi in Tetranychus urticae and RNAi toxicity enhancement by chimeric genes. Pesticide Biochemistry and Physiology,2016, 130: 1-7.
[25]NIWA R, NAMIKI T, ITO K, SHIMADA-NIWA Y, KIUCHI M,KAWAOKA S, KAYUKAWA T, BANNO Y, FUJIMOTO Y,SHIGENOBU S, KOBAYASHI S, SHIMADA T, KATSUMA S,SHINODA T. Non-molting glossylshroud encodes a short-chain dehydrogenase/reductase that functions in the ‘Black Box’ of the ecdysteroid biosynthesis pathway. Development, 2010, 137(12):1991-1999.
[26]LIU W, XIE Y, MA J, LUO X, NIE P, ZUO Z, LAHRMANN U,ZHAO Q, ZHENG Y, ZHAO Y, XUE Y, REN J. IBS: an illustrator for the presentation and visualization of biological sequences.Bioinformatics, 2015, 31(20): 3359-3361.
[27]KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis Version 7.0 for bigger datasets.Molecular Biology and Evolution, 2016, 33(7): 1870-1874.
[28]SUN W, JIN Y, HE L, LU W C, LI M. Suitable reference gene selection for different strains and developmental stages of the carmine spider mite, Tetranychus cinnabarinus, using quantitative real-time PCR. Journal of Insect Science, 2010, 10: Article 208.
[29]PFAFFL M W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research, 2001, 29(9): e45.
[30]TAKABAYASHI J, SHIMODA T, DICKE M, ASHIHARA W,TAKAFUJI A. Induced response of tomato plants to injury by green and red strains of Tetranychus urticae. Experimental & Applied Acarology, 2000, 24(5/6): 377-383.
[31]PAIM R M, ARAUJO R N, LEHANE M J, GONTIJO N F,PEREIRA M H. Long-term effects and parental RNAi in the blood feeder Rhodnius prolixus (Hemiptera; Reduviidae). Insect Biochemistry and Molecular Biology, 2013, 43(11): 1015-1020.
[32]YANG D, LU H, ERICKSON J W. Evidence that processed small dsRNAs may mediate sequence-specific mRNA degradation during RNAi in Drosophila embryos. Current Biology, 2000, 10(19):1191-1200.
[33]LINZ D M, CLARK-HACHTEL C M, BORRÀS-CASTELLS F,TOMOYASU Y. Larval RNA interference in the red flour beetle,Tribolium castaneum. Journal of Visualized Experiments, 2014, 92:e52059.
[34]LI W, ELLIOTT R W, NOVAK E K, SWANK R T. cDNA sequence and mapping of the mouse Copb gene encoding the beta subunit of the COPI coatomer complex. Somatic Cell and Molecular Genetics, 1999,25(3): 177-183.
[35]WU K, HOY M A. Oral delivery of double-stranded RNA induces prolonged and systemic gene knockdown in Metaseiulus occidentalis only after feeding on Tetranychus urticae. Experimental & Applied Acarology, 2014, 63(2): 171-187.
(责任编辑 岳梅)
Identification of β-COP and Sro of Tetranychus cinnabarinus and Their Lethal Effects After Silencing
AO YiQianYun1,2, SHEN GuangMao1,2, WANG MengYao1,2, LIU JiaLu1,2, PAN Yu3, HE Lin1,2
(1College of Plant Protection, Southwest University/Laboratory of Pesticide Toxicology and Science, Chongqing 400716;2Academy of Agricultural Sciences, Southwest University, Chongqing 400716;3College of Horticulture and Landscape Architecture, Southwest University, Chongqing 400716)
2017-05-22;接受日期:2017-06-26
国家自然科学基金(31672085)、中央高校团队项目(XDJK2016A005)
联系方式:敖已倩云,Tel:18375639762;E-mail:hacy0538@163.com。通信作者何林,Tel:023-68251541;E-mail:helinok@vip.tom.com

