不同植物无色花青素还原酶及其基因的生物信息学分析
李 蒙,鲍 锋
(西安文理学院 生物与环境工程学院,陕西 西安 710065)
不同植物无色花青素还原酶及其基因的生物信息学分析
李 蒙,鲍 锋
(西安文理学院 生物与环境工程学院,陕西 西安 710065)
对9种不同植物的无色花青素还原酶及其基因进行生物信息学分析,探索该蛋白的结构并预测LAR的功能。通过生物信息学软件对GenBank中已经登录的不同植物LAR基因和氨基酸序列进行分析,对其组成成分、理化性质、翻译后修饰位点、功能域、二级结构、亚细胞定位、分子进化等进行预测和推断。结果表明,9种植物无色花青素还原酶基因全长在1.0~1.2 kb,编码342~391个氨基酸,不具有信号肽;9种植物LAR都具有蛋白激酶C磷酸化位点、酪蛋白激酶Ⅱ磷酸化位点和N-端肉豆蔻酰化位点,个别植物LAR具有N-端糖基化位点、酪氨酸激酶磷酸化位点和cAMP和cGMP依赖蛋白激酶磷酸化位点;二级结构以α螺旋和无规则卷曲为主;进化分析表明禾本科的玉米与其他科植物亲缘关系较远。
无色花青素还原酶;生物信息学;结构预测;功能分析
无色花青素还原酶(Leucoanthocyanidin reductase, LAR, EC 1.17.1.3)是植物黄酮类化合物合成路径中一个关键酶,在苯丙氨酸代谢通路中,其催化无色花青素转化为儿茶素[1],而儿茶素是具有重要生理活性的次生代谢产物,具有如免疫调节、抗氧化、抗癌活性等多种功效[2-4]。
在多种植物中研究发现儿茶素的积累与LAR基因的表达相关[5-7]。目前在GenBank数据库中登陆的植物LAR的基因序列有1000多条,已从葡萄[6]、苹果[8]、豆类[9]、苜蓿[10]等植物中分离出不同拷贝数的LAR基因,而LAR的活性也已经在大麦[11]、豆类[12]、茶树[13]等植物中得到鉴定。LAR在儿茶素合成途径中尤为重要[14],有研究发现植物儿茶素的积累与LAR的转录因子调控相关[5],并且受种内和种间差异的明显影响[15]。因此,探讨LAR基因的表达调控及酶的功能特性具有重要的科学意义。
随着大多数儿茶素合成路径中相关酶的基因得到分离与克隆[16],在后续基因的表达调控的研究中,将有利于进一步了解植物生长发育过程中黄酮类物质的积累过程。本文通过生物信息学方法,对不同植物LAR基因及其氨基酸序列的结构特点与特征进行了预测与分析,以期为研究LAR家族蛋白的生化特性和调控机理提供理论依据。
1 材料与方法
1.1 材料
在NCBI中查询9种不同植物的无色花青素还原酶的核酸序列及其对应的氨基酸序列:玉米(NM-001155409)、苦荞(KC404849)、苹果(DQ139837)、葡萄(NM-001281160)、茶树(EF205148)、西洋梨(DQ251190)、草莓(JX134096)、大豆(JF433916)、玫瑰(KP768080)作为研究对象。
1.2 方法
(1)不同植物LAR核苷酸与氨基酸序列分析与比对采用DNAStar、ClustalX2和genedoc软件;(2)蛋白分子量、等电点、半衰期和稳定性等重要理化性质分析采用ProtParam;(3)蛋白质亚细胞定位、分泌信号肽等采用TargetP 和SignalP 4.0,二级结构预测采用SOPMA;(4)蛋白质糖基化采用NetNGlyc在线分析,脂酰化、磷酸化、cAMP和cGMP依赖蛋白激酶磷酸化位点采用Motif Scan;(5)多重序列比对和构建系统发育树采用ClustalX 2.0和TreeView。
2 结果与分析
2.1LAR基因与氨基酸序列理化性质分析
利用DNAStar和ProtParam在线分析,结果如表1所示,9种植物LAR基因的ORF全长在1029~1176 bp之间,GC含量适中(除玉米外),其氨基酸数目在342~391,分子量36.92~43.30 kD,pI值在5.16~6.38,说明LAR呈酸性,与酸碱氨基酸比例结果一致。经ProParam分析氨基酸组成,发现氨基酸Val在所有植物LAR中含量较高,比例在7.1%~9.9%;氨基酸Ile、Ala在大多数植物LAR中含量较高;氨基酸Trp和Gln在所有植物LAR中含量较低。所有植物LAR蛋白质中负电荷氨基酸残基比例高于正电荷氨基酸残基比例,疏水氨基酸比例高于酸性、碱性和极性氨基酸比例,LAR在溶液中的不稳定指数为28.86~49.28,除苦荞外均低于阀值(<40为稳定蛋白),推测LAR为稳定蛋白。脂肪系数为89.23~96.88,亲水性平均系数为-0.258~0.036,说明LAR蛋白质总体亲水性较低。

表1 9种植物中LAR核苷酸及氨基酸序列组成和理化性质
2.2 LAR氨基酸序列比对分析
不同植物的LAR氨基酸序列经ClustalX 2.0比对后,除了玉米,其他植物的LAR氨基酸序列同源性较高,都存在RFLP、ICCN和THD结构域(图1),推断不同植物中LAR的活性差异可能与保守结构域中个别氨基酸的替换相关。
2.3 LAR翻译后修饰与功能位点分析
植物蛋白质序列分析对其生物学功能的确定具有重要意义。9种植物蛋白质翻译后修饰位点表明(表2),通过NetNGlyc在线N-糖基化位点分析,玉米、葡萄、茶树和玫瑰4种植物的LAR蛋白含有不同的糖基化位点(玉米270NNTV、葡萄227NKSL、茶树50NKTL和212NKSV、玫瑰229NLSD),说明这些蛋白质只能在真核生物中翻译才具有生物学功能。通过Motif Scan预测cAMP和cGMP依赖蛋白激酶、脂酰化等,所有植物LAR蛋白都具有蛋白激酶C磷酸化位点、酪蛋白激酶Ⅱ磷酸化位点和N端肉豆蔻酰化位点,而酪氨酸激酶磷酸化位点的修饰唯有玉米LAR蛋白含有,cAMP和cGMP依赖蛋白激酶磷酸化唯有苦荞蛋白序列(343KKQS和361KKRS)和大豆蛋白序列(355KKCS)位点修饰。
2.4 蛋白的亚细胞定位和二级结构预测与分析
TargetP在线分析表明(表3),信号肽预测分值为0.012~0.618,cutoff值为0.430,说明9种植物LAR蛋白不存在信号肽,其结果与Signal P 4.0预测的结果相符;亚细胞定位分析表明,线粒体定位预测值为0.071~0.652,定位于其他位置的预测值为0.058~0.548;经SOPMA对LAR进行二级结构预测,预测结果表明(表3),不同植物LAR二级结构α螺旋所占比例为30.39%~43.73%,β折叠所占比例为8.16%~11.11%,无规则卷曲所占比例为29.26%~38.00%,延伸链所占比例为16.91%~24.85%,说明LAR蛋白的二级结构以α螺旋和无规则卷曲为主。

黑色和灰色分别表示氨基酸的相似性;方框标明保守结构域。物种名分别为GmLAR:大豆(Glycinemax);CsLAR:茶树(Camelliasinensis);PcLAR:西洋梨(Pyruscommunis);MdLAR:苹果(MalusXdomestica);FaLAR:草莓(Fragariaxananassa);RrLAR:玫瑰(Rosarugosa);FtLAR:苦荞(Fagopyrumtataricum);VvLAR:葡萄(Vitisvinifera);ZeLAR:玉米(Zea)。
图1 多重氨基酸序列比对结果
2.5 进化分析
利用ClustalX 2.0和TreeView对不同植物的LAR进行氨基酸的进化分析,结果表明(图2),不同植物LAR在进化过程中,禾本科的玉米LAR与其他科植物LAR距离较远,亲缘性较远;蔷薇科的西洋梨、苹果、草莓和玫瑰LAR相聚为一个分支,说明亲缘性较近;而蓼科的苦荞、豆科的大豆、葡萄科的葡萄和山茶科的茶树LAR具有亲缘性。进化分析与LAR氨基酸序列比对结果相符。

图2 不同植物LAR的系统进化树
3 讨论
植物儿茶素代谢路径中LAR属于短链脱氢酶/还原酶家族和PIP家族[17],在植物中普遍存在并参与多种生长发育调控。随着不同植物转录组测序的开展,生物信息学的发展为功能基因组学的研究开辟了更加便利的手段,通过对克隆的基因进行功能预测,有助于揭示物种基因的进化,发掘蛋白质功能,为遗传育种筛选经济性状和抗性候选基因提供一定的理论依据。
本文通过一系列生物信息学方法对9种植物儿茶素类化合物合成关键酶LAR的核酸和蛋白质序列进行分析,发现9种植物LAR核酸组成中G+C的含量正常(除玉米偏高外)。基因序列G+C含量直接影响mRNA二级结构及其热力学稳定性,最终会影响植物组织中LAR的转录和翻译,比如外源基因G+C含量(40%~50%)要与毕赤酵母表达手册要求相一致,才能确保成功表达[18]。蛋白质的理化性质影响蛋白质的结构和生理功能;而蛋白质的糖基化修饰能够调控其在组织和细胞中的定位、活性与功能,是参与蛋白质功能的主要修饰手段,其位点的变异与疾病的发生可能有关[19];同样蛋白质磷酸化修饰能够调节蛋白质在细胞信号传导过程中离子通道和改变激酶的活性,改变基因的表达,在一定程度上影响细胞的再生和分化[20];蛋白质的二级或空间结构与蛋白质行使其功能密切相关;进化关系分析可以为植物的繁殖与育种提供理论依据。
植物黄酮类物质的生物活性功能不断被发掘,以及在未来医药领域开阔的市场前景[21],其黄酮类物质代谢相关酶的基因得到克隆,后续可能利用基因工程手段改良植物品种,筛选代谢产物累积相关基因,研究其表达调控模式具有重要的意义。

表2 翻译后修饰和结构特征性序列分析结果
注:“N-端糖基化位点”和“蛋白激酶C磷酸化位点”的阀值均为0.5。
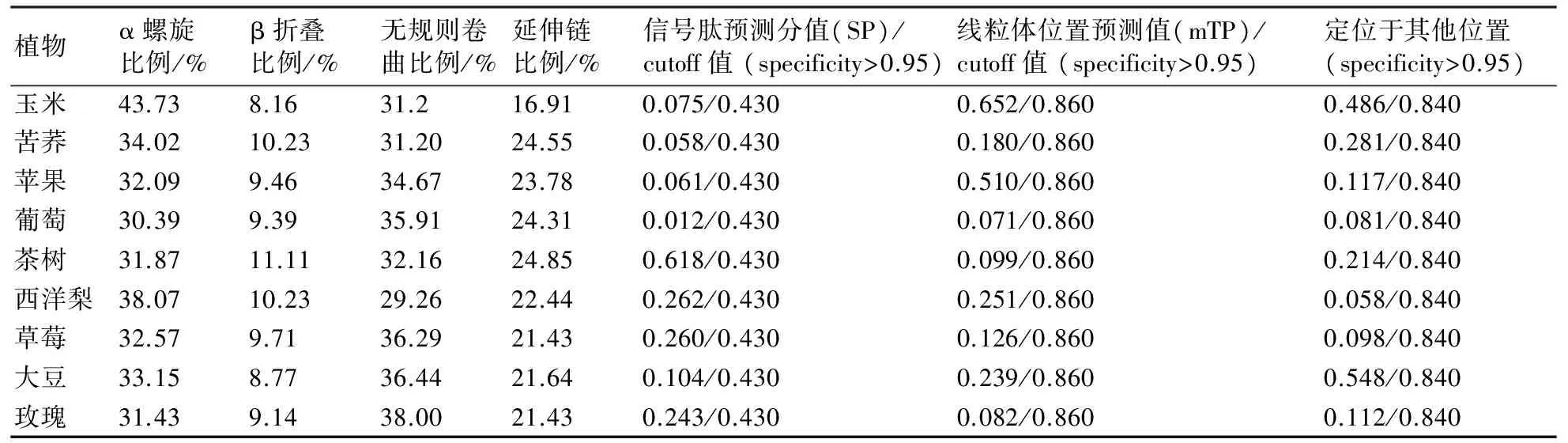
表3 亚细胞定位及二级结构预测与分析结果
[1] Devic M, Guilleminot J, Debeaujon I, et al. TheBANYULSgene encodes a DFR-like protein and is a marker of early seed coat development[J]. The Plant Journal, 1999, 19(4): 387-398.
[2] Lin Y L, Juan I M, Chen Y L, et al. Composition of polyphenols in fresh tea leaves and associations of their oxygen-radicalabsorbing capacitiy with antiproliferative actions in fibroblast cells[J]. Journal of Agricultural and Food Chemistry, 1996, 44: 1387-1394.
[3] Rao L J M, Yada H, Ono H, et al. Occurrence of antioxidant and radical scavenging proanthocyanidins from the Indian minor spice nagkesar(Mammealongifoliaplanch and triana syn) [J]. Bioorg Med Chem, 2004, 12: 31-36.
[4] Sano T, Oda E, Yamashita T, et al. Anti-thrombotic effect of proanthocyanidin, a purified ingredient of grape seed[J]. Thromb Res, 2005, 115(1/2): 115-121.
[5] Paolocci F, Robbins M P, Madeo L, et al. Ectopic expression of a basic helix-loop-helix gene transactivates parallel pathways of proanthocyanidin biosynthesis: structure, expression analysis and genetic control of leucoanthocyanidin 4-reductase and anthocyanidin reductase genes inLotuscorniculatus[J]. Plant Physiology, 2007, 143(1): 504-516.
[6] Bogs J, Downey M O, Harvey J S, et al. Proanthocyanidin synthesis and expression of genes encoding leucoanthocyanidin reductase and anthocyanidin reductase in developing grape berries and grapevine leaves[J]. Plant Physiology, 2005, 139(2): 652-663.
[7] Wang Y P,Yang Z M,Zhang B. Identification of protein related to biosynthesis of condensed tannin inOnobrychistanaiticaSpreng[J]. Acta Agrestia Sinica, 2000, 88(2): 126-131.
[8] Takos A M, Ubi B E, Robinson S P, et al. Condensed tannin biosynthesis genes are regulated separately from other flavonoid biosynthesis genes in apple fruit skin[J]. Plant Science, 2006, 170(3): 487-499.
[9] Tanner G J, Francki K T, Abrahams S, et al. Proanthocyanidin biosynthesis in plants. Purification of legume leucoanthocyanidin reductase and molecular cloning of its cDNA[J]. Journal of Biological Chemistry, 2003, 278(34): 31647-31656.
[10] Pang Y, Peel G J, Wright E, et al. Early steps in proanthocyanidin biosynthesis in the model legumeMedicagotruncatula[J]. Plant Physiology, 2007, 145(3): 601-615.
[11] Kristiansen K N. Conversion of (+)-dihydroquercetin to (+)-2,3-trans-3,4-cis-leucocy anidin and (+)-catechin with an enzyme extract from maturing grains of barley[J]. Carisberg Research Communications, 1986(51): 51-60.
[12] Joseph R, Tanner G, Larkin P. Proanthocyanidin synthesis in the forage legumeOnobrychisviciifolia. A study of chalcone synthase, dihydroflavonol 4-reductase and leucoanthocyanidin 4-reductase in developing leaves[J]. Australian Journal of Plant Physiology, 1998, 25(3): 271-278.
[13] 高可君,夏涛,高丽萍,等.茶叶二氢黄酮醇还原酶/无色花青素还原酶反应产物的快速检测方法研究[J].园艺园林科学,2008,6(24):287-290.
[14] Yuan L, Wang L, Han Z, et al. Molecular cloning and characterization of PtrLAR3,a gene encoding leucoanthocyanidin reductase fromPopulustrichocarpa, and its constitutive expression enhances fungal resistance in transgenic plants[J]. Journal of Experiment Botany, 2012, 63(7): 2513-2524.
[15] Pang Y, Wenger J P, Saathoff K, et al. A WD40 repeat protein fromMedicagotruncatulais necessary for tissue-specific anthocyanin and proanthocyanidin biosynthesis but not for trichome development[J]. Plant Physiology, 2009, 151(3):1114-1129.
[16] 夏涛,高丽萍.类黄酮及茶儿素生物合成途径及其调控研究进展[J].中国农业科学,2009,42(8):2899-2908.
[17] Maugé C, Granier T, D′Estaintot B L, et al. Crystal structure and catalytic mechanism of leucoanthocyanidin reductase fromVitisvinifera[J]. Journal of Molecular Biology, 2010, 397(4): 1079-1091.
[18] 张云博,白仁惠,王春迪,等.嗜热棉毛菌木糖苷酶Xy143基因优化及其在毕赤酵母中高效表达[J].微生物学通报,2017,44(1):9-19.
[19] Defaus S, Gupta P, Andreu D, et al. Mammalian protein glycosylation-structure versus function[J]. Analyst, 2014, 139(12): 2944-2967.
[20] Beenstock J, Mooshayef N, Engelberg D. How do protein kinases take a selfie(Autophosphorylate) [J]. Trends in Biochemical Sciences, 2016, 41(11): 938-953.
[21] Choi S U, Ryu S Y, Yoon S K, et al. Effects of flavonoids on the growth and cell cycle of cancer cells[J]. Anticancer Research, 1999, 19(6B): 5229-5233.
(责任编辑:许晶晶)
Bioinformatics Analysis of Leucoanthocyanidin Reductase and Gene in Different Plants
LI Meng, BAO Feng
(College of Biological and Environmental Engineering, Xi’an University of Arts and Sciences, Xi’an 710065, China)
The leucoanthocyanidin reductase (LAR) and its gene in nine kinds of plants were analyzed by using bioinformatic method. The nucleic acid sequences and amino acid sequences of LARs registered in GenBank were analyzed by using bioinformatic software, and their compositions, physical and chemical properties, post-translational modification sits, functional domains, secondary structure, subcellular location and molecular phylogenetic evolution were predicted and inferred. The results demonstrated that the LAR genes in nine kinds of plants was 1.0~1.2 kb in overall length, and they coded 342~391 amino acids, but had no signal peptide. All LARs had protein kinase C phosphorylation sites, casein kinase Ⅱ phosphorylation sites and N-myristoylation sites, and the LAR in several plants also had N-glycosylation sites, tyrosine kinase phosphorylation sites, and cAMP-dependent and cGMP-dependent protein kinase phosphorylation sites. The secondary structure mainly was of alpha helix and random coil. The evolutionary tree showed that the gramineous maize and the plants in other families had a distant genetic relationship.
Leucoanthocyanidin reductase; Bioinformatics; Structural prediction; Functional analysis
2017-05-14
李蒙(1988─),男,助理实验师,硕士,研究方向为生物化学与分子生物学。
Q811.4
A
1001-8581(2017)09-0005-05

