葡萄球菌CRISPR/Cas系统
叶立娜,陈力,瞿涤
复旦大学基础医学院病原生物学系,医学分子病毒学教育部/卫生部重点实验室,上海 200032
·综述·
葡萄球菌CRISPR/Cas系统
叶立娜,陈力,瞿涤
复旦大学基础医学院病原生物学系,医学分子病毒学教育部/卫生部重点实验室,上海 200032
成簇的规律间隔的短回文重复序列及其相关蛋白〔clustered regularly interspaced short palindromic repeat(CRISPR)/ CRISPR-associated protein, CRISPR/Cas〕是原核生物在进化过程中形成的获得性免疫系统,能抵抗噬菌体、质粒及可移动遗传因子等外源性DNA或RNA的入侵。目前,在多种葡萄球菌基因组中均发现CRISPR序列存在,其间隔序列通常与葡萄球菌的噬菌体或接合性质粒具有同源性,可能对葡萄球菌的毒力、耐药性传递和生物膜形成等生理学特性有影响。本文在简单介绍细菌CRISPR/Cas系统的基础上,对葡萄球菌CRISPR/Cas系统的构成、防御机制等进行综述。
成簇的规律间隔的短回文重复序列及其相关蛋白;葡萄球菌;外源性核酸;噬菌体
近年来发现,成簇的规律间隔的短回文重复序列及其相关蛋白〔clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein,CRISPR/Cas〕是细菌的一种免疫系统,主要防御外源性核酸如噬菌体或质粒等的入侵。CRISPR/Cas系统的作用与限制修饰(restriction-modification,R-M )及DNA磷硫酰化修饰系统相似,均以识别外源性DNA为基础[1];其中CRISPR/Cas系统通过对入侵基因的免疫记忆识别外源性DNA或RNA。CRISPR/Cas系统的记忆产生是通过外源性核酸的短核苷酸序列作为间隔序列插入CRISPR位点,转录后识别再次入侵的外源性DNA或RNA[2],因此CRISPR/Cas系统是一种获得性免疫系统。金黄色葡萄球菌是易产生耐药性的重要致病性球菌,而表皮葡萄球菌是引起医院内感染及生物膜相关疾病的重要机会致病菌,易被噬菌体侵袭或外源基因转入。随着表皮葡萄球菌RP62A基因组中CRISPR序列的发现(2005年)[3],多个课题组对葡萄球菌CRISPR/Cas系统的特性开展了研究。本文重点介绍葡萄球菌CRISPR/Cas系统。
1 细菌CRISPR/Cas系统
CRISPR/Cas9作为简单、快速、高效的基因编辑工具,已成功应用于人类细胞、斑马鱼、小鼠及细菌的基因组精确修饰,其在基因工程领域尤其是动物模型的构建中具有广阔应用前景,并有望为血液病、肿瘤及其他遗传疾病提供新的治疗手段。
在已解析的细菌基因组中,84%的古细菌和47%的真细菌具有CRISPR/Cas系统,多位于染色体上,仅少部分存在于质粒(如硫化叶菌)(CRISPRdb,http://crispr.i2bc.paris-saclay.fr/crispr)[4-5]。CRISPR/Cas系统由前导序列(leader)、CRISPR序列和多个cas基因组成。CRISPR 是一类结构特殊的重复序列,由21~48 bp的同向重复序列和间隔序列组成。间隔序列的大小与重复序列相似。重复序列通常呈回文序列,起始于GTTT,终止于GAAAC[6],转录后可形成茎环结构。间隔序列大多与入侵质粒或噬菌体的基因组序列具有同源性。在CRISPR位点上游有一段20~534 bp富含AT的前导区,而CRISPR序列转录的启动子就位于前导区内。另外,新间隔序列的插入位点往往位于前导区与第1个重复序列之间,提示前导序列可能与间隔序列的插入有关,也有研究证实了前导区的DNA片段在外源基因获取中的作用[7]。CRISPR/Cas系统通常包含4~20个不同的cas基因[8],位于CRISPR位点的上游或下游,其编码的Cas蛋白在外源性核酸入侵防御过程中起重要作用。Cas1、Cas2蛋白在新间隔序列插入时发挥作用,而其他Cas蛋白的基序相差很大,在各防御阶段行使不同功能。
1.1 细菌CRISPR/Cas系统的分型
在噬菌体及其他外源遗传因子的选择压力下,细菌CRISPR/Cas系统不断进化演变,导致菌种内和菌种之间CRISPR/Cas系统的数量和功能呈现多样性[9]。根据CRISPR位点结构、cas基因库和免疫作用机制的不同,CRISPR/Cas系统主要分为Ⅰ~Ⅴ 5个型别,并进一步划分为16个亚型(表1)[10]。Ⅰ、Ⅱ、Ⅲ型均有其特有的cas基因,分别为cas3、cas9和cas10。葡萄球菌CRISPR/Cas系统归类于Ⅲ型[10]。Ⅳ型和Ⅴ型是新发现的型别,其功能与作用机制尚不清楚。
1.2 细菌CRISPR/Cas系统的作用机制
CRISPR/Cas系统对外源性DNA发挥防御作用分3个阶段:适应、表达和干扰阶段,整个过程由Cas蛋白和CRISPR RNA(crRNA)共同参与(图1)。适应阶段(外源性核酸获取阶段):在Cas1和Cas2蛋白的参与下,外源性核酸的原型间隔序列(proto-spacer)插入细菌的CRISPR位点中,形成一个新的带有免疫记忆和遗传性的间隔序列,从而获得对同种外源性核酸再次入侵的抵抗力[2]。表达阶段(crRNA合成阶段):先由CRISPR序列转录产生一段长的crRNA前体(precursor crRNA,pre-crRNA),通过进一步剪切,处理成仅包含一个间隔序列的成熟crRNA,其两端带有部分重复序列[11-12]。在大多数Ⅰ型和Ⅲ型CRISPR/Cas系统中,crRNA合成依赖Cas6蛋白家族参与,在重复序列的特定位点对pre-crRNA进行识别和剪切[11];而Ⅱ型系统是利用反式激活crRNA(trans-activating crRNA,tracrRNA)及宿主编码的核糖核酸酶Ⅲ(ribonuclease Ⅲ,RNase Ⅲ)来对pre-crRNA进行处理[12]。干扰阶段(攻击阶段):crRNA作为引导序列,在Ⅱ型系统中与tracrRNA和具有多个功能域的Cas9结合[13],在Ⅰ、Ⅲ型系统中与多个Cas蛋白结合形成核糖核蛋白效应复合物[14]。crRNA通过碱基配对识别再次入侵的靶核酸,介导Cas蛋白对其进行切割和降解。虽然不同型别CRISPR系统的核糖核蛋白效应复合物的组成成分和核酸剪切机制不同,但Ⅰ和Ⅱ型效应复合体可抵御外源性DNA入侵[13,15],Ⅲ型对DNA和RNA均有防御作用[16-17]。研究显示,Ⅲ型CRISPR/Cas系统不仅具有抗病毒的免疫功能,还能抵抗基因的水平转移,如抵抗接合性质粒的转移[16]和外源性DNA的自然转化[18]。然而,致病菌的毒力和耐药性的水平转移及噬菌体的入侵对CRISPR/Cas系统也存在免疫逃逸,前者是基于质粒及辅助遗传因子的选择作用,涉及宿主自身CRISPR 位点或间隔序列的修饰或删除[9], 而后者与噬菌体靶基因的变异有关[19]。
表1 细菌CRISPR/Cas系统的分型
Tab.1 The classification of bacterial CRISPR/Cas systems

TypesSignatureproteinsSubtypesSignatureproteinsRepresentativestrainsⅠCas3ⅡCas9ⅢCas10ⅣCsf1ⅤCpf1Ⅰ-ACas8a,Csa5ArchaeoglobusfulgidusⅠ-BCas8bClostridiumkluyveriDSM555Ⅰ-CCas8cBacillushaloduransC-125Ⅰ-DCas10dCyanothecesp.PCC8802Ⅰ-ECse1,Cse2EscherichiacoliK12Ⅰ-FCsy1,Csy2,Csy3,Cas6fYersiniapseudotuberculosisYPIIIⅠ-UGSU0054GeobactersulfurreducensⅡ-ACsn2StreptococcusthermophilusCNRZ1066Ⅱ-BCas4Legionellapneumophilastr.ParisⅡ-C—*Neisserialactamica020-06Ⅲ-ACsm2StaphylococcusepidermidisRP62AⅢ-BCmr5PyrococcusfuriosusDSM3638Ⅲ-CMTH326Methanothermobacterthermautotrophicusstr.DeltaHⅢ-DCsx10Roseiflexussp.RS-1——AcidithiobacillusferrooxidansATCC23270——Francisellacf.novicidaFx1
*Only three genes are present in the Ⅱ-C operon:cas1,cas2,cas9.
目前,表皮葡萄球菌等凝固酶阴性葡萄球菌(coagulase-negative staphylococci,CoNS)引起的医院内感染(包括细菌生物膜相关疾病)日趋增多[20]。由于基因操作技术困难,对CoNS致病性相关基因及其功能的研究受到限制,推测其与细菌存在CRISPR/Cas等防御外源性DNA入侵的系统有关。葡萄球菌可通过基因水平转移获取毒力因子,噬菌体在其中发挥重要作用[21-22]。抗生素的广泛使用导致多重耐药菌株出现,与细菌获取携带耐药基因的接合性质粒密切相关[3]。CRISPR/Cas系统可抵抗此类外源性基因的水平转移,分析致病性葡萄球菌CRISPR/Cas系统的间隔序列与噬菌体和质粒的同源性,预测其携带的毒力和耐药基因,可为细菌致病性研究及抗菌药物的选择提供依据。
2 葡萄球菌CRISPR/Cas系统
Gill等于2005年对表皮葡萄球菌RP62A基因组分析时,首次发现其存在CRISPR序列[3]。Marraffini等于2008年发现表皮葡萄球菌RP62A的CRISPR/Cas系统限制外源性质粒的水平转移[16]。CRISPRdb[4](http://crispr.i2bc.paris-saclay.fr/crispr/)最新数据显示,在已解析的55株葡萄球菌菌株中,37株菌株共存在51个CRISPR序列,其中32株是金黄色葡萄球菌,1株是表皮葡萄球菌(RP62A,ATCC 35984),4株是其他种类的葡萄球菌。表皮葡萄球菌ATCC 12228株基因组中仅发现了CRISPR的可疑结构(questionable structure)。Yang等对其中32株金黄色葡萄球菌的45个CRISPR序列进行分析,仅有两株菌(08BA02176和MSHR1132)具有cas基因[22]。鉴于表皮葡萄球菌RP62A的CRISPR/Cas系统中间隔序列与所有已测序的葡萄球菌(包括医院和社区获得性的耐甲氧西林和耐万古霉素金黄色葡萄球菌)的接合性质粒中nickase(nes)基因具有同源性[3,23-25],其在抵抗耐药性传播中具有重要意义。本文主要以表皮葡萄球菌RP62A为例,介绍葡萄球菌CRISPR/Cas系统。
根据葡萄球菌CRISPR/Cas系统所含的csm基因(Cas subtypeM.tuberculosisgene), 可将其归类于Ⅲ-A型[10]。Ⅲ型CRISPR/Cas系统分为Ⅲ-A、Ⅲ-B、Ⅲ-C、Ⅲ-D 4个亚型,均具有cas10基因,其编码的蛋白Cas10与辅助基因(csm或cmr)编码的蛋白构成Cas10-辅助蛋白复合体[26]。辅助基因中,csm为Ⅲ-A、Ⅲ-D型CRISPR/Cas系统所特有,csm2为其标志基因;而cmr为Ⅲ-B、Ⅲ-C型所特有,cmr5为其标志基因[10]。Ⅲ-A型的Cas10-Csm复合物主要攻击外源性DNA,但在少数细菌中可作用于RNA(如嗜热链球菌[27]),且两者剪切外源性核酸的过程也不相同,由Cas10-Csm复合物中不同的活性区所催化[28]。ⅢB型的Cas10-Cmr复合物主要攻击外源性RNA[10]。

The adaptation, expression and interference mechanisms of staphylococcal CRISPR/Cas systems. The leader (gray boxes) provides locus for integration of the proto-spacer and contains transcriptional promoter for CRISPR sequence. The repeats (yellow diamonds) and spacers (colored rectangles) are transcribed as a long precursor crRNA, and are processed as several short mature crRNAs by specific Cas-Csm proteins (colored pies). The spacers within the mature crRNAs guide Cas10-Csm effector complexes to recognize exogenous nucleic acids by matching the nucleotides, which is followed by cleavages and degradations of the targets.
图1 葡萄球菌CRISPR/Cas系统的防御机制
Fig.1 Defensive mechanisms of staphylococcal CRISPR/Cas systems
2.1 葡萄球菌CRISPR/Cas系统的组成
利用CRISPRfinder[4](http://crispr.i2bc.paris-saclay.fr/Server/)和美国国立生物技术信息 中心(National Center for Biotechnology Information,NCBI)基本局部比对搜索工具(Basic Local Alignment Search Tool,BLAST)(http://blast.ncbi.nlm.nih.gov/)分析金黄色葡萄球菌MSHR1132和08BA0217的CRISPR/Cas系统,发现其不同于传统的CRISPR/Cas系统,含有两个CRISPR序列(图2A、2B)。在利用BLAST对金黄色葡萄球菌08BA0217的重复序列进行分析时,发现了与其含有相同重复序列的金黄色葡萄球菌JS395,而在对JS395基因组进行分析时发现其具有完整的CRISPR/Cas系统(图2C),与Cao等的描述结果一致[29]。
分析表皮葡萄球菌RP62A的CRISPR/Cas系统,结果与Marraffini等的描述结果相同[16]。CRISPR序列中含3个间隔序列(spa1、spa2和spa3)(图2D):spa1与目前已测序的葡萄球菌质粒具有不同程度的同源性,其中同源性为100%的质粒有20种(表2);spa2与表皮葡萄球菌的噬菌体具有不同程度的同源性,其中与葡萄球菌噬菌体PH15[21]具有100%同源性;而spa3在NCBI数据库中未找到同源性基因。
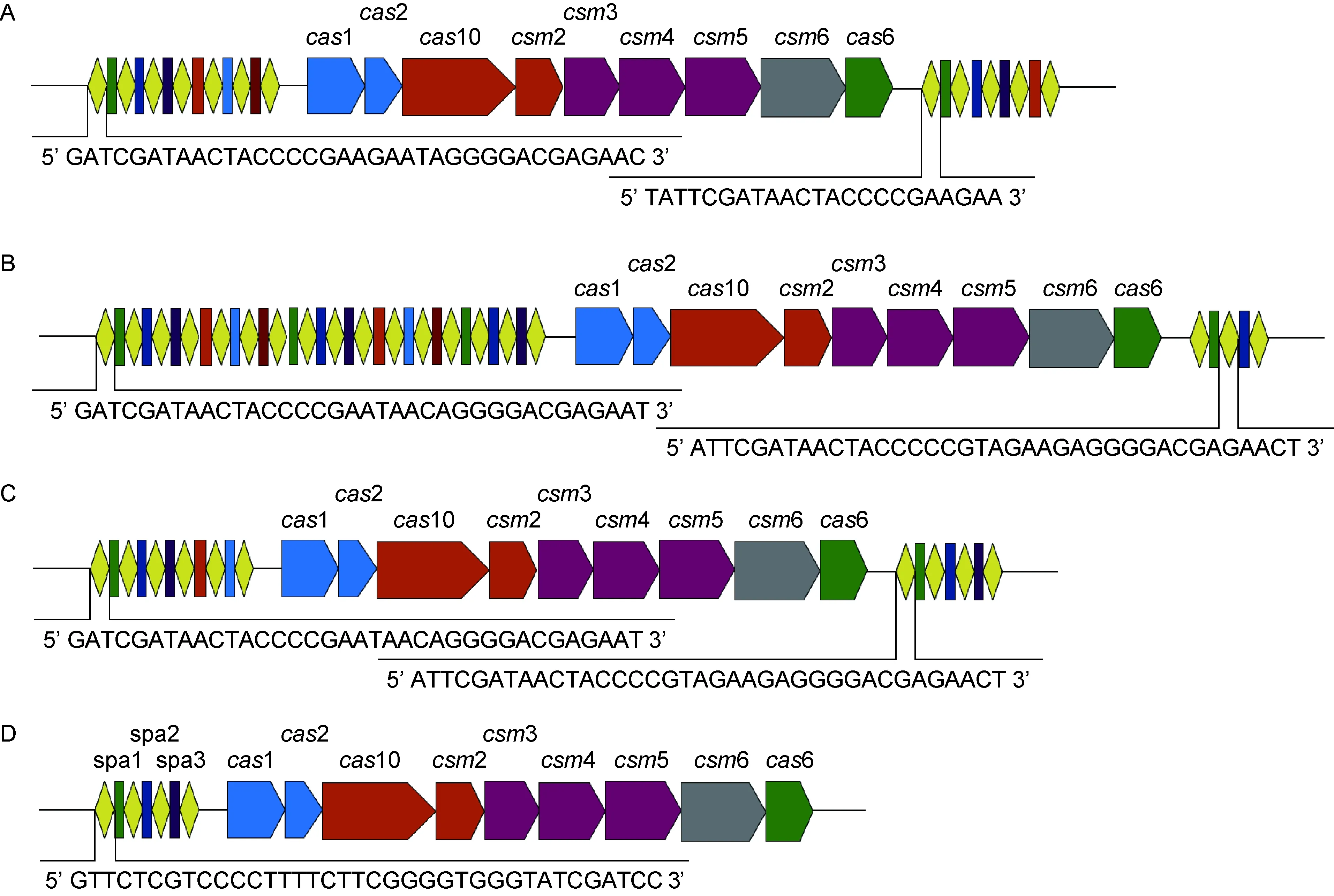
The CRISPR loci ofStaphylococcusaureussubsp.aureusMSHR1132 (A),Staphylococcusaureus08BA0217 (B),StaphylococcusepidermidisRP62A (D) were found in CRISPRs database (http://crispr.i2bc.paris-saclay.fr/crispr/), while that ofStaphylococcusaureussubsp.aureusJS395 (C) was identified by BLAST against the repeat sequence ofStaphylococcusaureus08BA0217. The arrangements of repeats (yellow diamonds), spacers (colored rectangles), CRISPR-associated genes andcassubtypeM.tuberculosis(csm) genes (colored pentagons) of the four strains are shown according to their genomic graphics in NCBI (http://www.ncbi.nlm.nih.gov/). The repeat sequence of each CRISPR locus is also presented.
图2 葡萄球菌CRISPR/Cas系统的结构
Fig.2 Structures of staphylococcal CRISPR/Cas systems
葡萄球菌CRISPR/Cas系统包含9个cas-csm基因(图2),其在金黄色葡萄球菌MSHR1132、08BA0217和JS395中位于两个CRISPR序列之间,而在表皮葡萄球菌RP62A中位于CRISPR位点下游。其中cas1和cas2在噬菌体或质粒入侵时对其间隔序列的获取发挥作用[30],其他7种基因作用于防御过程。Cas10(又称Csm1)是Ⅲ型CRISPR/Cas系统的特征性蛋白,也是Ⅲ型效应复合物中的最大亚单位[26]。Csm3、Csm4、Csm5和Cas6均属于RAMP超级家族[8],该家族的蛋白在Ⅰ型和Ⅲ型CRISPR/Cas系统中分布广泛。Cas10、 Csm2、Csm3、Csm4和Csm5参与crRNA成熟和Cas10-Csm核糖核蛋白复合物的形成[26],其中Cas10、Csm3和 Csm4在聚集蛋白形成复合物中起重要作用[31]。
2.2 葡萄球菌CRISPR/Cas系统对外源性核酸的防御机制
葡萄球菌CRISPR/Cas系统的防御过程与其他CRISPR/Cas系统一样,分3个阶段,且在适应阶段均以Cas1和Cas2发挥外源性核酸的获取作用。然而,在表达和干扰阶段,葡萄球菌对外源性核酸的防御作用机制较为复杂,区别于Ⅰ型和Ⅱ型CRISPR/Cas系统,详见表3。
在crRNA表达阶段,CRISPR序列转录成 pre-crRNA后的处理过程对靶基因的识别和剪切至关重要,因其作用位点在离成熟crRNA末端特定距离处[17],且多余的侧翼片段可能增加与靶基因同源的机会而阻碍免疫识别作用。在表皮葡萄球菌RP62A CRISPR/Cas系统的crRNA表达过程中,pre-crRNA处理过程分为初步处理和成熟两个阶段(图1)。在pre-crRNA初步处理阶段,Cas6将pre-crRNA剪切成71个核苷酸(nucleotide,nt)的crRNA中间体[26],包含一个完整的34~44 nt间隔序列,其5′端是来自重复序列3′端8 nt片段(称为crRNA tag),3′端是来自重复序列5′端的15~16 nt残余片段[32]。Cas6是RAMP家族的一种内切核糖核酸酶[8],对重复序列具有特异性。Ⅲ-A型系统的Cas6在pre-crRNA的初步处理过程中单独作用,无其他Cas蛋白参与[26,31],而大多数Ⅰ型系统中的Cas6与其直系同源物(orthologue)形成识别效应复合物共同作用[33-34]。与此不同,Ⅱ型系统pre-crRNA的初步处理由tracrRNA与pre-crRNA互补而介导[12]。在crRNA成熟阶段,Ⅲ-A型CRISPR/Cas系统的Csm2、Csm3、Csm5与crRNA中间体聚集,Csm3在crRNA中间体3′端以6 nt为单位进行度量,由Csm2、Csm3和Csm5共同作用将多余重复序列切除,形成31~67 nt的成熟crRNA[26],并与Csm4和Cas10结合,共同形成稳定的Cas10-Csm核糖核蛋白效应复合物[26,31]。Ⅱ型系统的crRNA成熟剪切位点在crRNA中间体5′端[12],而Ⅰ型系统无该成熟阶段[35-36]。
表2 表皮葡萄球菌RP62A基因组 CRISPR中spa1的100%同源性质粒
Tab.2 Plasmids homologous to spa1 in the CRISPR locus ofS.epidermidisRP62A with 100% identity

PlasmidsBacteriaStrainsReferencespT33G-1S.lugdunensisTLUG033G-4Ho,2016pK93GS.lugdunensisKLUG093G-4Ho,2016pT15G-1S.lugdunensisTLUG015G-4Ho,2016pKM01S.pseudintermediusHK547/11Calcutt,2015pMIS.aureusMIKatayama,2016pGO400S.aureusRN4220Ray,2015pRM27S.aureusRN4220Ray,2015pN315GS.aureusN315GZhang,2012pI5S5S.aureusST228Vogel,2012SAP082AS.aureusCDCPANICUMcDougal,2010SAP080AS.aureusCDCTN147McDougal,2010SAP079AS.aureusCDCGA672McDougal,2010SAP069AS.aureusPM79Summers,2009SAP068AS.aureusPM64Summers,2009SAP014AS.aureusCDC58Summers,2009SAP015BS.aureusCDC61Summers,2009pUSA03S.aureusUSA300_FPR3757Diep,2006pLW043S.aureus—*Weigel,2003pGO1S.aureus4220Climo,1996pSK41S.aureus—*Firth,1993
*The strain is not mentioned in the nucleotide database of NCBI.
表3 葡萄球菌Ⅲ-A型CRISPR/Cas系统与Ⅰ、Ⅱ型系统防御机制的比较
Tab.3 Comparison of defensive mechanisms among Ⅲ-A and Ⅰ/Ⅱ CRISPR/Cas systems

CRISPR/Cas 系统型别crRNA表达阶段crRNA初步处理crRNA的成熟靶核酸干扰阶段靶向选择剪切机制Ⅲ-A型系统Cas6与同源物形成复合物共同作用Csm2、Csm3、Csm5参与,剪切crRNA3'端要求crRNAtag与靶核酸的5'端侧翼序列不具有同源性需外源性DNA转录,Cas10-Csm剪切DNA和mRNAⅠ型系统Cas6单独作用无此过程识别前间区序列邻近基序(protospacer-adjacentmotif,PAM)Cas3、Cas9直接剪切外源性DNAⅡ型系统tracrRNA介导RNase剪切crRNA 5'端
在靶核酸干扰阶段,葡萄球菌对外源性DNA的识别和剪切机制亦不同于Ⅰ型和Ⅱ型CRISPR/Cas系统(表3)。除crRNA与外源性DNA配对外,葡萄球菌Ⅲ-A型CRISPR/Cas系统的靶向选择还有特殊要求,以区分外源性DNA与自身CRISPR。当crRNA tag与靶核酸5′端侧翼序列不具有同源性时,靶核酸被识别为外源性核酸;而crRNA tag与自身CRISPR的部分重复序列完全配对时,自身核酸被识别,以防止自身免疫[37]。因此,当crRNA tag与外源性核酸5′端侧翼序列具有完全同源性时,可出现免疫逃逸。噬菌体和质粒对CRISPR/Cas系统的免疫逃逸机制可能成为葡萄球菌致病性基因水平转移的有利条件。此外,葡萄球菌Ⅲ-A型系统Cas10-Csm复合物剪切外源性DNA需特殊条件:在外源性DNA转录后,由Cas10-Csm复合物的不同活性区分别对DNA和转录的mRNA进行剪切[28],而Ⅰ、Ⅱ型系统则分别由Cas3、Cas9蛋白直接行使对外源性DNA的剪切功能[38-39]。
3 结语
CRISPR/Cas系统是原核生物在进化过程中形成的适应性免疫系统,除能抵抗病毒侵袭外,还能阻止质粒或其他遗传因子的水平转移,主要取决于间隔序列与外源性核酸的同源性。葡萄球菌具有Ⅲ-A型CRISPR/Cas系统,包含9个cas-csm基因,其间隔序列通常与噬菌体和质粒的基因具有同源性,表明这些外源性基因的不断入侵在葡萄球菌CRISPR/Cas系统进化过程中起重要作用。鉴于葡萄球菌CRISPR/Cas系统可抵抗毒力及耐药性相关外源性基因的水平传播,其间隔序列的生物信息学分析对选择临床用药具有重要指导意义。基于质粒转化的基因操作在很多CoNS中常常失败,推测可能与细菌存在CRISPR/Cas系统等防御外源性DNA入侵的遗传屏障有关。因此,研究葡萄球菌CRISPR/Cas系统对外源性核酸转移的免疫作用及机制,通过改造CRISPR/Cas系统用于葡萄球菌基因操作,将为葡萄球菌生理学特性和致病性相关功能基因的研究提供重要工具。
[1] Makarova KS, Wolf YI, Koonin EV. Comparative genomics of defense systems in archaea and bacteria [J]. Nucleic Acids Res, 2013, 41(8): 4360-4377.
[2] Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea [J]. Nature, 2012, 482(7385): 331-338.
[3] Gill SR, Fouts DE, Archer GL, Mongodin EF, Deboy RT, Ravel J, Paulsen IT, Kolonay JF, Brinkac L, Beanan M, Dodson RJ, Daugherty SC, Madupu R, Angiuoli SV, Durkin AS, Haft DH, Vamathevan J, Khouri H, Utterback T, Lee C, Dimitrov G, Jiang L, Qin H, Weidman J, Tran K, Kang K, Hance IR, Nelson KE, Fraser CM. Insights on evolution of virulence and resistance from the complete genome analysis of an early methicillin-resistant Staphylococcus aureus strain and a biofilm-producing methicillin-resistant Staphylococcus epidermidis strain [J]. J Bacteriol, 2005, 187(7): 2426-2438.
[4] Makarova KS, Wolf YI, Alkhnbashi OS, Costa F, Shah SA, Saunders SJ, Barrangou R, Brouns SJ, Charpentier E, Haft DH, Horvath P, Moineau S, Mojica FJ, Terns RM, Terns MP, White MF, Yakunin AF, Garrett RA, van der Oost J, Backofen R, Koonin EV. An updated evolutionary classification of CRISPR-Cas systems [J]. Nat Rev Microbiol, 2015, 13(11): 722-736.
[5] Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats [J]. BMC Bioinformatics, 2007, 8: 172. doi: 10.1186/1471-2105-8-172.
[6] Lillestøl RK, Shah SA, Brügger K, Redder P, Phan H, Christiansen J, Garrett RA. CRISPR families of the crenarchaeal genus Sulfolobus: bidirectional transcription and dynamic properties [J]. Mol Microbiol, 2009, 72(1): 259-272.
[7] Godde JS, Bickerton A. The repetitive DNA elements called CRISPRs and their associated genes: evidence of horizontal transfer among prokaryotes [J]. J Mol Evol, 2006, 62(6): 718-729.
[8] Yosef I, Goren MG, Qimron U. Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli [J]. Nucleic Acids Res, 2012, 40(12): 5569-5576.
[9] Makarova KS, Aravind L, Wolf YI, Koonin EV. Unification of Cas protein families and a simple scenario for the origin and evolution of CRISPR-Cas systems [J]. Biol Direct, 2011, 6: 38. doi: 10.1186/1745-6150-6-38.
[10] Jiang W, Maniv I, Arain F, Wang Y, Levin BR, Marraffini LA. Dealing with the evolutionary downside of CRISPR immunity: bacteria and beneficial plasmids [J]. PLoS Genet, 2013, 9(9): e1003844.
[11] Charpentier E, Richter H, van der Oost J, White MF. Biogenesis pathways of RNA guides in archaeal and bacterial CRISPR-Cas adaptive immunity [J]. FEMS Microbiol Rev, 2015, 39(3): 428- 441.
[12] Deltcheva E, Chylinski K, Sharma CM, Gonzales K, Chao Y, Pirzada ZA, Eckert MR, Vogel J, Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III [J]. Nature, 2011, 471(7340): 602-607.
[13] Gasiunas G, Barrangou R, Horvath P, Siksnys V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria [J]. Proc Natl Acad Sci USA, 2012, 109(39): E2579-E2586.
[14] Plagens A, Richter H, Charpentier E,Randau L. DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes [J]. FEMS Microbiol Rev, 2015, 39(3): 442-463.
[15] Westra ER, van Erp PB, Künne T, Wong SP, Staals RH, Seegers CL, Bollen S, Jore MM, Semenova E, Severinov K, de Vos WM, Dame RT, de Vries R, Brouns SJ, van der Oost J. CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3 [J]. Mol Cell, 2012, 46(5): 595-605.
[16] Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA [J]. Science, 2008, 322(5909): 1843-1845.
[17] Hale CR, Zhao P, Olson S, Duff MO, Graveley BR, Wells L, Terns RM, Terns MP. RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex [J]. Cell, 2009, 139(5): 945-956.
[18] Zhang Y, Heidrich N, Ampattu BJ, Gunderson CW, Seifert HS, Schoen C, Vogel J, Sontheimer EJ. Processing-independent CRISPR RNAs limit natural transformation in Neisseria meningitidis [J]. Mol Cell, 2013, 50(4): 488-503.
[19] Deveau H, Barrangou R, Garneau JE, Labonté J, Fremaux C, Boyaval P, Romero DA, Horvath P, Moineau S. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus [J]. J Bacteriol, 2008, 190(4): 1390-1400.
[20] Otto M. Staphylococcal infections: mechanisms of biofilm maturation and detachment as critical determinants of pathogenicity [J]. Annu Rev Med, 2013, 64: 175-188.
[21] Daniel A, Bonnen PE, Fischetti VA. First complete genome sequence of two Staphylococcus epidermidis bacteriophages [J]. J Bacteriol, 2007, 189(5): 2086-2100.
[22] Yang S, Liu J, Shao F, Wang P, Duan G, Yang H. Analysis of the features of 45 identified CRISPR loci in 32 Staphylococcus aureus [J]. Biochem Biophys Res Commun, 2015, 464(3): 894-900.
[23] Weigel LM, Clewell DB, Gill SR, Clark NC, McDougal LK, Flannagan SE, Kolonay JF, Shetty J, Killgore GE, Tenover FC. Genetic analysis of a high-level vancomycin-resistant isolate of Staphylococcus aureus [J]. Science, 2003, 302(5650): 1569-1571.
[24] Diep BA, Gill SR, Chang RF, Phan TH, Chen JH, Davidson MG, Lin F, Lin J, Carleton HA, Mongodin EF, Sensabaugh GF, Perdreau-Remington F. Complete genome sequence of USA300, an epidemic clone of community-acquired methicillin-resistant Staphylococcus aureus [J]. Lancet, 2006, 367(9512): 731-739.
[25] Climo MW, Sharma VK, Archer GL. Identification and characterization of the origin of conjugative transfer (oriT) and a gene (nes) encoding a single-stranded endonuclease on the staphylococcal plasmid pGO1 [J]. J Bacteriol, 1996, 178(16): 4975-4983.
[26] Hatoum-Aslan A, Samai P, Maniv I, Jiang W, Marraffini LA. A ruler protein in a complex for antiviral defense determines the length of small interfering CRISPR RNAs [J]. J Biol Chem, 2013, 288(39): 27888-27897.
[27] Tamulaitis G, Kazlauskiene M, Manakova E, Venclovas,Nwokeoji AO, Dickman MJ, Horvath P,Siksnys V. Programmable RNA shredding by the type III-A CRISPR-Cas system of Streptococcus thermophilus [J]. Mol Cell, 2014, 56(4): 506-517.
[28] Samai P, Pyenson N, Jiang W, Goldberg GW, Hatoum-Aslan A, Marraffini LA. Co-transcriptional DNA and RNA cleavage during type III CRISPR-Cas immunity [J]. Cell, 2015, 161(5): 1164-1174.
[29] Cao L, Gao CH, Zhu J, Zhao L, Wu Q, Li M, Sun B. Identification and functional study of type III-A CRISPR-Cas systems in clinical isolates of Staphylococcus aureus [J]. Int J Med Microbiol, 2016, 306(8): 686-696. doi: 10.1016/j.ijmm.2016.08.005.
[30] Datsenko KA, Pougach K, Tikhonov A, Wanner BL, Severinov K, Semenova E. Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system [J]. Nat Commun, 2012, 3: 945. doi: 10.1038/ncomms1937.
[31] Hatoum-Aslan A, Maniv I, Samai P, Marraffini LA. Genetic characterization of antiplasmid immunity through a type III-A CRISPR-Cas system [J]. J Bacteriol, 2014, 196(2): 310-317.
[32] Carte J, Wang R, Li H, Terns RM, Terns MP. Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes [J]. Genes Dev, 2008, 22(24): 3489-3496.
[33] Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, Dickman MJ, Makarova KS, Koonin EV, van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes [J]. Science, 2008, 321(5891): 960-964.
[34] Haurwitz RE, Sternberg SH, Doudna JA. Csy4 relies on an unusual catalytic dyad to position and cleave CRISPR RNA [J]. EMBO J, 2012, 31(12): 2824-2832.
[35] Gesner EM, Schellenberg MJ, Garside EL, George MM, Macmillan AM. Recognition and maturation of effector RNAs in a CRISPR interference pathway [J]. Nat Struct Mol Biol, 2011, 18(6): 688-692.
[36] Wiedenheft B, van Duijn E, Bultema JB, Waghmare SP, Zhou K, Barendregt A, Westphal W, Heck AJ, Boekema EJ, Dickman MJ, Doudna JA. RNA-guided complex from a bacterial immune system enhances target recognition through seed sequence interactions [J]. Proc Natl Acad Sci USA, 2011, 108(25): 10092-10097.
[37] Marraffini LA, Sontheimer EJ. Self versus non-self discrimination during CRISPR RNA-directed immunity [J]. Nature, 2010, 463(7280): 568-571.
[38] Sinkunas T, Gasiunas G, Fremaux C, Barrangou R, Horvath P, Siksnys V. Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system [J]. EMBO J, 2011, 30(7): 1335-1342.
[39] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science, 2012, 337(6096): 816-821.
敬请关注《微生物与感染》微信公众号
. QU Di,E-mail:dqu@shmu.edu.cn
CRISPR/Cas systems ofStaphylococcus
YE Lina, CHEN Li, QU Di
DepartmentofMedicalMicrobiologyandParasitology,KeyLaboratoryofMedicalMolecularVirologyofMinistriesofEducationandHealth,SchoolofBasicMedicalSciences,FudanUniversity,Shanghai200032,China
Clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein (CRISPR/Cas) is an acquired immune system developed in evolution of prokaryotes. It provides an immune protection to resist invasion of foreign DNAs or RNAs, such as phages, plasmids and the other mobile genetic elements. CRISPR loci have been identified in genomes ofStaphylococcusspecies. The spacers of the CRISPR arrays usually match with the genes on phages or plasmids derived from the other species or strains ofStaphylococcus, and may provide a function to interfere the transmission of exogenous elements related with bacterial virulence, drug resistance and biofilm formation. This review focuses on the unique structure and function of staphylococcal CRISPR/Cas systems.
Clustered regularly interspaced short palindromic repeat (CRISPR)/CRISPR-associated protein;Staphylococcus; Exogenous nucleic acid; Bacteriophage

国家自然科学基金 (81271791)
瞿涤
2016-08-25)

