结核分枝杆菌临床分离株链霉素耐药性的检测及耐药基因突变位点分析
杨彩虹,曹旭东,史静雪,姬祥,于璐,袁俐,吴长新,陈创夫*
(1石河子大学动物科技学院,新疆 石河子 832003;2石河子大学医学院,新疆 石河子 832002;3石河子大学生命科学学院,新疆 石河子 832003)
结核分枝杆菌临床分离株链霉素耐药性的检测及耐药基因突变位点分析
杨彩虹1,曹旭东2,史静雪1,姬祥3,于璐2,袁俐2,吴长新2,陈创夫1*
(1石河子大学动物科技学院,新疆 石河子 832003;2石河子大学医学院,新疆 石河子 832002;3石河子大学生命科学学院,新疆 石河子 832003)
为检测结核分枝杆菌链霉素(streptomycin,SM)耐药性,分析耐药基因突变,探讨DNA测序分析技术在结核分枝杆菌链霉素耐药性检测中的应用价值。采用方法:1、用传统的比例法药敏试验对49株结核分枝杆菌临床分离株进行链霉素耐药性检测。2、用DNA测序分析技术对49株结核分枝杆菌临床分离株进行链霉素耐药相关基因Rpsl和rrs的突变位点分析,筛查耐药相关突变位点。3、探讨DNA测序分析技术对结核分枝杆菌临床分离株进行链霉素耐药性检测的效率。结果:1、传统比例法药敏试验结果显示,49株实验菌株中16株为链霉素耐药株,33株为链霉素敏感株。2、Rpsl基因DNA测序。结果显示:Rpsl基因只存在一个突变位点,为第43位密码子AAG的突变,突变形式为AAG→AGG,氨基酸由赖氨酸(Lys)变为精氨酸(Arg)。rrs基因DNA测序结果发现:rrs基因共存在5个突变位点,分别为第513位碱基A突变为G、第523位碱基A缺失、第598位碱基A突变为G、第599位碱基A缺失、第612位碱基A缺失,其中第513位碱基突变只存在于链霉素耐药株中,其余4个突变位点在耐药株和敏感株中均存在。即初步认为Rpsl基因第43位密码子的突变及rrs基因第513位碱基的突变与结核分枝杆菌链霉素耐药性有关。3、分析Rpsl基因和rrs基因突变与结核分枝杆菌链霉素耐药性之间的关系,结果显示:16株链霉素耐药株中13株存在Rpsl基因第43位密码子的突变,1株存在rrs基因第513位碱基的突变。以传统的比例法药敏结果为参照,以DNA测序分析Rpsl基因或rrs基因发现Ppsl基因发生第43位密码子或rrs基因第513位碱基突变为判断菌株耐药的标准,DNA测序分析技术对结核分枝杆菌链霉素耐药性检测的敏感性为87.5%,特异性为96.97%。由此可知,初步认为Rpsl基因第43位密码子的突变及rrs基因第513位碱基的突变与结核分枝杆菌链霉素耐药性有关。用DNA测序分析结核分枝杆菌Rpsl基因和rrs基因链霉素耐药相关突变,判断结核杆菌链霉素耐药性具有较好的灵敏度和特异性,耗时短,在链霉素耐药结核病的快速诊断方面具有一定的应用价值。
结核分枝杆菌;链霉素;DNA测序;耐药性
结核病是当今全球范围内最具威胁性的感染性疾病之一,是单一病菌致死率最高的传染病。目前,我国结核病人数位居世界第二,仅次于印度,卫生部将其列为全国重点控制的重大疾病之一。大量结核分枝杆菌(Mycobacterium tuberculosis,MTB)耐药菌株的出现及不断扩散使结核病的防控更加困难,给结核病的防治带来了更大的挑战,现已成为全球关注的公共卫生和社会问题[1-2]。
因此快速准确的检测结核杆菌的耐药性,为指导早期用药、提高结核病疗效,防止耐药菌的产生和传播具有重要意义。
链霉素(streptomycin,SM)是第一个发现与核糖体相互作用的抗生素。1944年,Selman Waksman发现SM可用于治疗结核病[3]。早在1946年,就已经有关于结核分枝杆菌对SM产生耐药性的报道[4]。2000年第4次全国结核病流行病学调查[5]与2010年第5次全国结核病流行病学调查[6]比较显示,经我国结核分枝杆菌SM 耐药率为有上升的趋势,应当引起人们的高度关注。
本实验首先采用传统的比例法药敏试验对49株结核分枝杆菌临床分离株进行耐药性检测;再参考相关文献[5]及Rpsl基因和rrs基因序列,设计引物,对49株结核分枝杆菌临床分离株进行Rpsl基因和rrs基因的测序分析,以传统的比例法药敏试验检测结果为参照,评价DNA测序分析技术检测链霉素耐药性的敏感性、特异性,探讨DNA测序分析技术检测结核分枝杆菌链霉素耐药性的应用价值,为耐药结核病的快速辅助诊断提供依据。
1 材料与方法
1.1 材料
1.1.1 菌株
49株结核分枝杆菌由本实验室保存。
1.1.2 主要试剂
氨酸钠、磷酸二氢钾、硫酸镁、柠檬酸镁、丙三醇、天泽恩基因柱式分支杆菌DNAout试剂盒、高分辨率熔解曲线试剂盒(罗氏公司)、谷核酸染料及2×Taq PCR Master Mix购与生工生物科技有限公司、引物均由上海祥音生物科技有限公司合成、DNA ladder购自北京康为世纪生物科技有限公司。
1.1.3 主要仪器与设备
涡旋混合仪、恒温培养箱、实时荧光定量PCR仪(LightCycler480,瑞士罗氏公司)。Nanodrop2000分光光度计。
1.2 方法
1.2.1 比例法药敏实验
将培养至出现可见菌落的结核分枝杆菌进行药敏实验,实验操作遵循《结核分枝杆菌药物敏感性试验标准化操作程序及质量保证手册》进行,链霉素在含药培养基中的浓度为4 μg/mL[7]。
1.2.2 结核分枝杆菌DNA的提取
在改良的罗氏培养基上挑取适量生长良好的结核分枝杆菌加入磨菌瓶,磨菌瓶提前加入磨菌株高压备用,使用时再加入5-6滴生理盐水。每株菌磨3 min,静止5 min,然后分装到0.5 mL的灭菌管中,再将其放入灭活仪85℃,1 h。灭活后的菌液按照柱式分枝杆菌DNAout试剂盒说明书提取,获得的DNA溶液用分光光度计检测浓度以备用。
1.2.3 结核分枝杆菌链霉素耐药相关基因Rpsl和rrs的DNA测序分析
Rpsl(Rv0682)基因位于结核分枝杆菌标准株H37Rv基因组的第 781560-781934,全长 375 bp,Gene ID为888259,编码核糖体蛋白S12。测序引物如表1所示,测序区从第2位密码子-第103位密码子之间,引物扩增片段长度为306 bp。
rrs(Rvnr01)基因位于结核分枝杆菌标H37Rv基因组的第 1471846-1473382,Gene ID为 2700429,全长1537 bp,编码16srRNA。测序区覆盖rrs513-517和 rrs905-908及1400三个区域,从第456位碱基-第1449位碱基之间,引物扩增片段长度为994 bp(表 1)。

表1 测序引物Tab.1 The primers of DNA sequencing
对49株实验菌株进行结核分枝杆菌链霉素耐药相关基因 Rpsl和rrs测序区的PCR扩增,PCR体系为25 μL,包括9.7 μL dd H2O、25 um上下游引物各 0.4 μL、模板 2 μL、Mix 12.5 μL。PCR 反应条件为95℃预变性 5 min、94℃变性 40 s、59.5℃(59℃)退火30 s、72℃延伸40 s、重复30个循环、72℃再延伸10 min,4℃保存。
扩增产物经电泳检测有条带后送上海生工生物工程股份有限公司测序,测序结果与GeneBank上公布的结核分枝杆菌H37Rv标准株对应基因序列进行比对分析,获得每株菌在测序区对应的突变信息,将获得的突变信息与文献报道过的突变位点进行比较,分析二者之间的异同,并统计各突变位点在耐药菌株里所占比例。
最后分析耐药株耐药相关基因的突变位点及突变类型、分析用DNA直接测序法对结核分枝杆菌链霉素耐药性检测的效率。
1.2.4 统计学分析
应用SPSS17.0统计软件进行统计分析,比较传统药敏结果、DNA测序结果之间的符合率。用卡方检测分析比例法药敏结果与DNA测序结果的差异性,P﹤0.05提示两种检测方法检测结果有统计学意义。用Kappa检测分析两种结果的一致性。1≥Kappa≥0.75时为高度一致性;0.75≥Kappa≥0.4时为中度一致性;Kappa<0.4时为低度一致性。
2 结果
2.1 比例法药敏试验结果
发现49株实验菌株中有16株为链霉素耐药株,33株为链霉素敏感株。
2.2 链霉素耐药相关基因Rpsl和rrs DNA测序分析结果
2.2.1目的基因Rpsl和rrs的扩增结果
用测序引物PCR分别扩增实验菌株的基因组DNA,取5 μL扩增产物经1%琼脂糖凝胶电泳。49株结核分枝杆菌及1株H37Rv标准株全部扩增出Rpsl基因306 bp片段长度(图1)。rrs基因扩增出994 bp片段长度(图 2)。
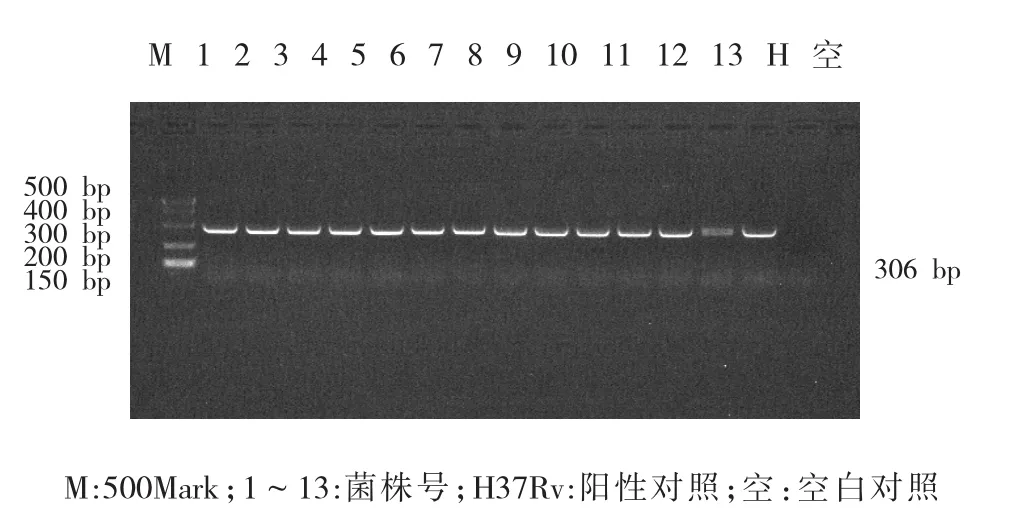
图1 不同菌株 Rpsl基因扩增结果Fig.1 PCR amplification results of Rpsl gene

图2 不同菌株 rrs基因扩增结果Fig.2 Different strains of rrs gene amplification results
2.2.2 目的基因Rpsl和rrs的测序分析
将DNA测序结果分别与Genebank上公布的结核分枝杆菌H37Rv基因Rpsl和rrs基因对应序列进行比较,49株结核分枝杆菌中链霉素耐药相关基因Rpsl存在1个突变位点,为第43位密码子 AAG突变为AGG,氨基酸由赖氨酸(Lys)突变为精氨酸(Arg)(3a)。49株结核分枝杆菌中链霉素耐药相关基因rrs存在5个突变位点(3b-f)。
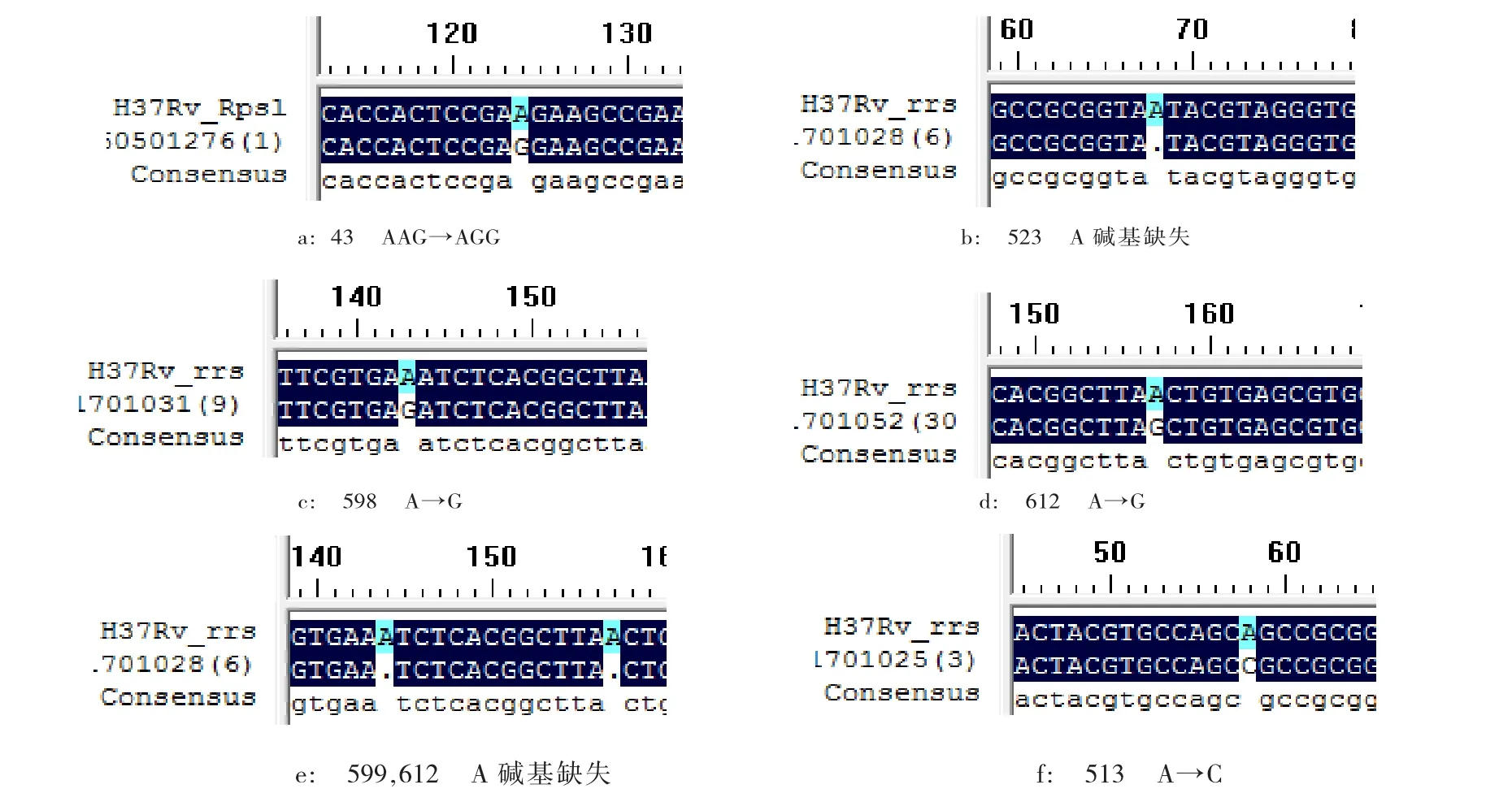
图3 DNA序列比对结果Fig.3 DNA sequence alignment results
2.2.3 各菌株在链霉素耐药相关基因Rpsl和rrs的测序区的突变位点分析
将每株菌的测序结果与GeneBank上公布的标准株H37Rv的Rpsl基因和rrs基因对应序列进行比对分析,结果见表2。

表2 不同株菌在链霉素耐药相关基因Rpsl和rrs基因的突变Tab.2 Streptomycin Resistance conferring mutation within rpsl and rrs in different Mycobacterium tuberculosis isolares
2.2.4 Rpsl基因和rrs基因各位点突变类型及密码子变化
通过对49株结核分枝杆菌Rpsl基因突变位点分析发现,16株链霉素耐药株中出现了1种突变类型,为第43位密码子AAG→ AGG,氨基酸由赖氨酸(Lys)突变为精氨酸(Arg);33株链霉素敏感株中出现了1种突变类型,为第43位密码子AAG→AGG,氨基酸由赖氨酸(Lys)突变为精氨酸(Arg)(表 3)。

表3 Rpsl基因分别在链霉素耐药菌株和敏感菌株中的突变Tab.3 Rpsl mutations in Streptomycin Resistant strains and Streptomycin susceptible strains,respectively
49株分枝杆菌临床分离株的Rpsl基因测序区分析发现,有14株存在第43位密码子的突变,其中13株为链霉素耐药株,占链霉素耐药株的81.25%。1株出现在链霉素敏感株中,在链霉素敏感株中占3.03%。综上所述,Rpsl基因第43位密码子的突变在链霉素耐药株中占较高比率,因此,初步认为该位点的突变与链霉素耐药性存在密切关系,可作为检测菌株链霉素耐药性的靶点之一(图4)。
对49株结核分枝杆菌rrs基因进行突变位点分析发现,16株链霉素耐药株中9株不存在突变位点;7株发现存在突变情况,其中1株存在513 A→C、3株存在598 A→G、1株存在523 A碱基缺失及612 A→G、1株存在 599A碱基缺失、1株存在 523及599位点A碱基缺失。
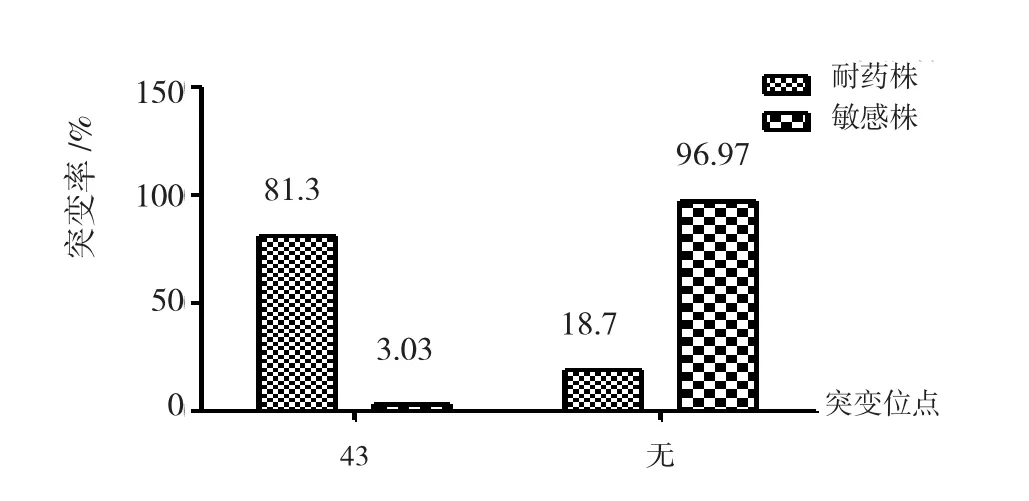
图4 Rpsl各突变位点在敏感株及耐药株中所占比例Fig.4 The mutations proportion of Rpsl in sensitive strains and resistant strains
33株链霉素敏感株中22株不存在突变位点;11株发现存在突变情况,其中4株存在598 A→G的突变、1株存在599A缺失、1株存在523A缺失及598 A→G的突变、1株存在 523,599,612 位点 A碱基缺失、1株存在598 A→G的突变及612位点A碱基缺失、3株存在523及599位点A碱基缺失(表 4)。
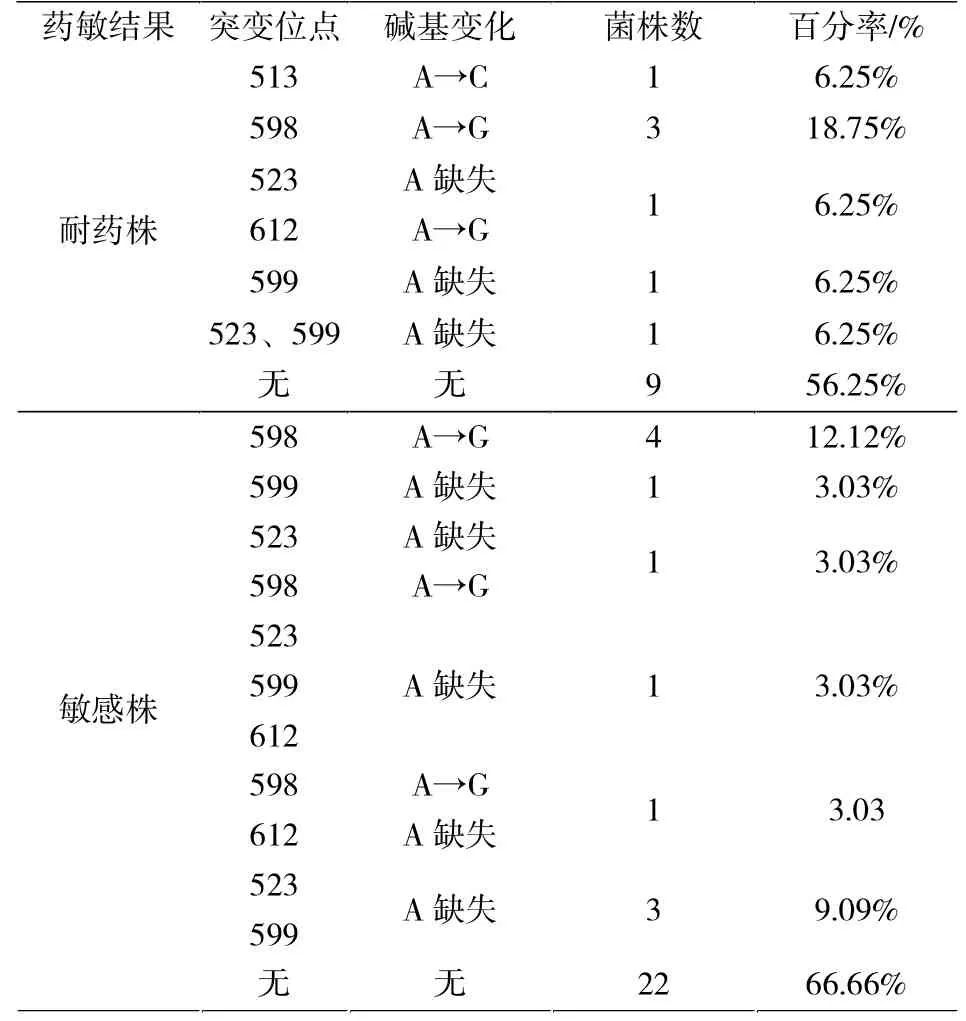
表4 rrs基因分别在链霉素耐药菌株和敏感菌株中的突变Tab.4 rrs mutations in Streptomycin Resistant strains and Streptomycin susceptible strains,respectively
rrs基因的 DNA 测序结果显示,523、598、599、612位点突变不仅在链霉素耐药株中出现,在链霉素敏感株中同样也出现,在耐药株中各突变位点所占比例依次为 12.5%、18.75%、12.5%、6.25;在敏感株中各突变位点所占比例依次为15.15%、18.18%、15.15%、6.06%。因此,初步认为 523、598、599、612位点的突变与链霉素耐药无关。而513位点的突变只在链霉素耐药株中存在,初步认为该位点的突变与链霉素耐药性存在一定关系,可作为检测菌株链霉素耐药性的靶点之一,本实验中该位点的突变占链霉素耐药株的6.25%(图5)。

图5 rrs基因各突变位点在敏感株及耐药株中所占比例Fig.5 The mutations proportion of rrs in in sensitive strains and resistant strains
2.3 评价DNA测序分析技术检测结核分枝杆菌链霉素耐药性的应用价值
2.3.1 以DNA测序检测到耐药突变为判断菌株耐药性的标准
将传统比例法药敏结果与基因突变结果进行比对分析发现,16株链霉素耐药株中13株存在Rpsl基因的第43位密码子的突变,1株存在rrs基因的第513位碱基的突变,2株不存在Rpsl基因和rrs基因中任何一个基因的突变。若以比例法药敏结果为参照,以DNA测序检测到耐药突变为判断菌株耐药性的标准,在链霉素耐药株中,耐药突变的菌株占87.5%,耐药未突变的菌株占12.5%。即通过DNA测序分析方法检测结核杆菌链霉素耐药性的敏感性为87.5%、特异性为 96.97%(表 5)。
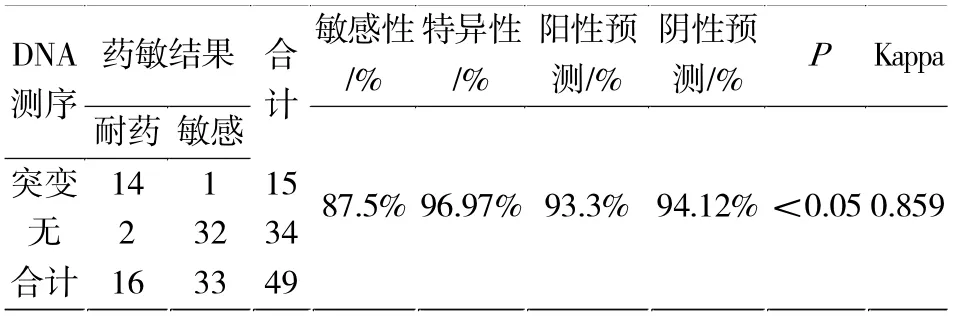
表5 传统耐药与基因测序结果比较分析Tab.5 Comparison of traditional drug resistance and gene sequencing results
3 讨论
链霉素(SM)是1945年发明的一种氨基环醇糖苷类抗生素,是第一种有效的抗结核药物[8]。链霉素的抗菌活性主要是通过与结核分枝杆菌中核糖核酸蛋白体亚单位结合,干扰细菌的翻译,进而干扰蛋白质的合成[5]。对于结核杆菌链霉素耐药性的检测,目前应用比较普遍的是传统的药物敏感性检测(DST),该方法包括实验室常用的耐药诊断金标法——罗氏培养基(L-J)比例法、快速诊断耐药结核病的 Bactec MGIT 960检测系统以及检测细菌最低抑菌浓度值(MIC)的微孔板 Alamar Blue显色法(MABA 法)。罗氏培养基(L-J)比例法是目前运用最广泛的药敏检测方法,国内外大多数研究报道均用比例法药敏结果作为金标准[9-10]。虽然该方法具有较高的准确性,但耗时长,对结核分枝杆菌耐药性的检测至少需要1个月,不能及时有效的指导临床用药,患者可能因此耽误最佳的治疗时机,在药敏结果出来前已死亡。因此快速检测结核分枝杆菌的耐药性有利于指导临床医生在病程早期正确用药,加快结核病的治愈,降低病人的费用,抑制耐多药结核分枝杆菌的传播,具有重大的现实意义。
目前普遍认为结核分枝杆菌的耐药机制包括细胞壁通透性改变、泵蛋白的调控、酶失活、靶基因的突变等因素,其中引起结核分枝杆菌耐药的主要原因之一是基因突变。经多项流行病学调查研究结果显示,结核分枝杆菌Rpsl和rrs基因突变与链霉素耐药有关[11],约 80%的耐链霉素结核杆菌临床分离株可见rpsL和 rrs基因突变[12]。其中约52%-59%的SM耐药菌株可见rpsL基因发生突变,突变位点主要集中在第43位和第 88位氨基酸 ,二者密码子均由 AAG突变为AGG,氨基酸由赖氨酸(Lys)突变为精氨酸(Arg)[13],且rpsL基因70%以上的突变发生在Rpsl43位点[14]。还有 8%-21%的SM耐药菌株rrs基因发生突变,突变主要集中在530环区和904位碱基[15]。由此可见,Rpsl基因和rrs基因的突变与结核分枝杆菌链霉素耐药性有密切关系,因此,可通过分析这两个基因的突变检测大部分结核分枝杆菌对链霉素的耐药性。基于结核分枝杆菌链霉素耐药性主要是耐药相关基因的突变引起的,许多研究者建立了以检测耐药基因突变为基础的耐药检测方法。因此,基因诊断技术近年来在结核病的快速诊断中取得了重大突破。
本实验使用的方法是DNA测序分析技术,其优点是检测结果直观可靠,排除了人为因素造成的实验结果的不可靠性,且可在一周内得到结果。不足之处是对每一分离株来说,全部检测所有位点需要多次反应,操作较为复杂[16]。
分析本实验通过DNA测序分析方法检测结核分枝杆菌链霉素耐药性的结果显示,其敏感性为87.5%、特异性为96.97%,虽然具有较好的敏感性和特异性。但仍有2株耐药株未检测到Rpsl基因或rrs基因发生突变,导致假阴性结果的出现,分析其原因,可能是因为通过DNA测序分析方法检测结核杆菌链霉素耐药性时,设计的检测范围未能覆盖所有的耐药相关基因或耐药突变位点、也可能是结核杆菌链霉素耐药性存在其他一些耐药机制,这些耐药机制并不是基因突变导致的耐药,可能与细胞膜的通透性有关,所以部分耐药菌株未能检测到基因突变。
总的来说,DNA测序分析方法与传统的药敏实验相比,在结核杆菌链霉素耐药性检测中可作为较好的快速辅助诊断,可尽快为耐药结核病的临床治疗方案提供依据。
[1] 宋育明,杨色娟.997例结核分枝杆菌的耐药性分析[J].中国微生态学杂志,2016,28(3):333-336.Song Y M,Yang S J.Analysis of drug resistance of Mycobaterium tuberculosis in 997 cases[J].Chinese Journal of Microecology,2016,28(3):333-336.
[2] 温书香,王慧勤,曹旭东,等.PMA-qPCR快速检测结核活菌方法的建立[J].石河子大学学报(自然科学版),2014,32(2):143-147.Weng S X,Wang H Q,Cao X D,et al.Establisment of rapid detection for Live/Dead Mycobacterium tuberculosis with PMA-q PCR.[J].Journal of Shihezi University(Natural Science),2014,32(02):143-147.
[3] Schatz A,Waksman S A.Effect of Streptomycin and other antibiotic substances upon Mycobacterium tuberculosis and related organisms[J].Experimental Biology& Medicine,1944,57(2):244-248.
[4] Klein M,Kimmelman L J.The role of spontaneous variants in the acquisition of streptomycin resistance by the Shigellae[J].Journal of Bacteriology,1946,52(4):471-479.
[5] 张俊仙,吴雪琼,邢婉丽,等.应用基因芯片技术检测结核分枝杆菌链霉素耐药性的研究[J].中国人兽共患病学报,2009,25(11):1046-1048.Zhang J X,Wu X Q,Xin W L,et al.Rapid detection of rpsl and rrs mutations in Mycobacterium tuberculosis by DNA chip.[J].Chinese Journal of Zoonoses,2009,25(11):1046-1048.
[6] 王黎霞,成诗明,陈明亭,等.2010年全国第五次结核病流行病学抽样调查报告 [J].中国防痨杂志,2012,34(08):485-508.Wang L X,Cheng S M,Chen M T,et al.The fifth national tuberculosis epidemiological survey in 2010.[J].Chinese Journal of Antituberculosis.,2012,34(8):485-508.
[7] 王仙,袁俐,何树梅,等.结核分枝杆菌不同药物敏感性检测方法的比较[J].石河子大学学报(自然科学版),2007,25(3):342-344.Wang X,Yuan L,He S M,et al.Comparison of drug susceptibility testing methods for M.tuberculosis.[J].Journal of Shihezi University(Natural Science),2007,25(3):342-344.
[8] 席向宇,王炳花,张瑞梅.结核分枝杆菌耐链霉素和乙胺丁醇基因的突变特征分析[J].中外医学研究,2016,14(21):135-137.Xi X Y,Wang B H,Zhang R M.Analysis on the characteristics of mutation Gene streptomycin and ethambutol Drug-resistance in Mycobacterium tuberculosis.[J].Chinese and Foreign Medical Research,2016,14(21):135-137.
[9] 彭亦平,谭彩萍,熊国亮.BACTEC MGIT 960快速培养及药敏在肺结核诊治中的应用评价 [J].江西医药,2016,51(1):21-22.Peng X Y,Tan C P,Xiong G L.Performance evalution of bactec mgit 960 system in culture and durg susceptibility test of Mycobacterium in Patients with pulmonary tuberculosis[J].Jiangxi Medicine,2016,51(1):21-22.
[10] 李奇凤,张向晖,杜燕,等.结核分枝杆菌耐链霉素与rpsL和rrS基因的相关性研究[J].石河子大学学报(自然科学版),2008,26(1):16-18.Li Q F,Zhang X H,Du Y,et al.A Study on the relevence between the rpsL and rrS Gene and streptomycin-resistant M.tuberculosis[J].Journal of Shihezi University(Natural Science),2008,26(1):16-18.
[11] 孙荣,欧维正,王燕,等.贵阳市结核分枝杆菌链霉素和乙胺丁醇耐药相关基因突变的检测与分析[J].中国人兽共患病学报,2016,32(8):760-764.Son R,Ou W Z,Wang Y,et al.Detection of mutations associated with streptomycin and ethambutol resistance in Mycobacterium tuberculosis clinical isolates[J].Chinese Journal of Zoonoses,2016,32(8):760-764.
[12] Tracevska T,Jansone I,Nodieva A,et al.Characterisation of rpsL,rrs and embB mutations associated with streptomycin and ethambutol resistance in Mycobacterium tuberculosis[J].Research in Microbiology,2004,155(10):830-834.
[13]Lipin M Y,Stepanshina V N,Shemyakin I G,et al.Association of specific mutations in katG,rpoB,rpsL and rrs genes with spoligotypes of multidrug-resistant Mycobacterium tuberculosis isolates in Russia[J].Clinical Microbiology&Infection,2007,13(6):620-626.
[14] 张向东,张婷,牛建军,等.探针熔解分析法快速检测结核分枝杆菌链霉素耐药突变[J].中国人兽共患病学报,2011,27(1):53-57.Zhang X D,Zhang T,Niu J J,et al.Probe melting analysis for rapid detection of streptomycin resistant mutations in Mycobacterium tuberculosis clinical isolates[J].Chinese Journal of Zoonoses,2011,27(1):53-57.
[15]Fukuda M,Koga H,Ohno H,et al.Relationship between genetic alteration of the rpsL gene and streptomycin susceptibility of Mycobacterium tuberculosis in Japan[J].Journal of Antimicrobial Chemotherapy,1999,43(2):281-284.
[16]Taylor T B,Patterson C,Hale Y,et al.Routine use of PCR-restriction fragment length polymorphism analysis for identification of mycobacteria growing in liquid media[J].Journal of Clinical Microbiology,1997,35(1):79-85.
Streptomycin resistance testing and analysis of resistance gene mutations of Mycobacterium tuberculosisin clinical isolates
Yang Caihong1,Cao Xudong2,Shi Jingxue1,Ji Xiang3,Yu lu2,Yuan Li2,Wu Changxin2,Chen Chuangfu1*
(1 Collage of Animal Science and Technology,Shehezi University,Shihezi,Xinjiang 832003,China;2 School of Medicine,Shihezi University,Shihezi,Xinjiang 832002,China;3 Collage of Life Science,Shehezi University,Shihezi,Xinjiang 832003,China)
To analysis the drug resistance and gene mutation inMycobacterium tuberculosis,and to investigate the clinical application value of DNA sequence analysis detecting ofMycobacterium tuberculosisisolates streptomyci-resistance.First,traditional drug susceptibility test(DST)was used to analysis streptomycin-resistance for 49 strainsMycobacterium tuberculosis.Then,analysis assay for detection of mutation in two drug resistance associated genes of 49 strainsMycobacterium tuberculosis clinical isolates (rpsl,rrs)by DNA sequencing,and screening for mutations.According to the mutations of genes,the design of primersof DNA Sequenceanalysisfordetection of streptomycin resistance.DST resultsshowed that16 strainswere Streptomycin-resistant strains,33 strains were streptomycin-sensitive strains.Sequencing results showed that there was only a mutations of rpsl,for the 43rd codon mutation of AAG,mutation forms for AAG-AGG,amino acid from lysine (Lys)into arginine (Arg).There were five site mutations of rrs gene,513 A to G,523,599,612 of lack of A,598A to G,respectively.The 513th base mutations existed only in the streptomycin resistant strains,the remaining four mutations in resistant strains and sensitive strains existed.The preliminary think the 43rd codon Rpsl gene mutation and rrs 513th of base mutations associated with streptomycin resistance Mycobacterium tuberculosis.Culture-based phenotypic drug susceptibility testing and DNA sequencing to analyze the level of rpsl gene,rrs gene mutation,the results showed 1 strain existed 513th base mutations of rrs genes,13 strains existed 43th codon mutations of rpsl gene in 16 strains of streptomycin resistant strains.With culture-based phenotypic drug susceptibility testing as the gold standard,the DNA sequencing detected rpsl gene 43th codes or rrs gene 513th base mutations for strains resistant,DNA sequencing results showed that the sensitivity and specificity was 87.5%and 96.97%.It can beconcluded that DNA sequencing analysistechnology hasgood sensitivity and specificity to detect streptomycin resistance ofMycobacterium tuberculosisclinical isolates,which is short time-consuming and in early diagnosis of streptomycin drug-resistant TB.
Mycobacterium tuberculosis;streptomycin;DNAsequencing;drug-resistance
S855.2
A
10.13880/j.cnki.65-1174/n.2017.02.007
1007-7383(2017)02-0169-07
2016-10-11
国家自然科学基金项目(31060333),新疆兵团重大科技专项(2014AA001)
杨彩虹(1990-),女,硕士研究生,专业方向为:畜禽病原分子生物学。
*通信作者:陈创夫(1962-),男,教授,博士生导师,从事分子病理学和免疫学研究,e-mail:ccf-xb@163.com。

