水稻耐盐性和耐碱性相关性状的QTL定位及环境互作分析
梁银培,孙健,索艺宁,刘化龙,王敬国,郑洪亮,孙晓雪,邹德堂
(东北农业大学农学院水稻研究所,哈尔滨 150030)
水稻耐盐性和耐碱性相关性状的QTL定位及环境互作分析
梁银培,孙健,索艺宁,刘化龙,王敬国,郑洪亮,孙晓雪,邹德堂
(东北农业大学农学院水稻研究所,哈尔滨 150030)
【目的】探索水稻在盐和碱胁迫下产量相关性状的变化规律,寻找耐盐碱主效QTL,并分析QTL加性、上位性与环境互作效应。揭示单株有效穗数、结实率、千粒重和单株穗重在盐、碱胁迫下的遗传机制,为水稻耐盐碱性分子标记辅助育种提供理论依据。【方法】以东农425和长白10号杂交得到的重组自交系为材料,构建包含120个SSR标记的遗传连锁图。以浓度6 ds·m-1的NaCl水溶液,pH9.0的Na2CO3水溶液进行全生育期处理,正常水灌溉为对照。对2014年和2015年盐、碱胁迫和自然条件下水稻的单株有效穗数、结实率、千粒重和单株穗重分别采用2种作图方法同时定位研究,即完备区间作图法进行加性QTL定位和混合线性模型的复合区间作图法进行加性、上位性QTL与环境互作联合分析。【结果】2014年和2015年碱胁迫条件下与盐胁迫条件下各性状表型值相比,耐碱相关性状降低较明显,表明水稻对碱胁迫更为敏感,碱胁迫更大程度地限制了高产和稳产。并且 2年的碱胁迫条件下各性状与盐胁迫条件下各性状均未表现出显著相关性。水稻在耐盐性和耐碱性上可能存在遗传机制上的差异。运用ICIM共检测到61个水稻耐盐碱相关性状加性效应QTL,分布在第1、2、3、4、5、6、7、8、10、11和12染色体上。运用MCIM在6个环境下进行加性及环境互作效应的联合定位分析,共检测到17个加性QTL存在环境互作效应,分布在第1、3、5、7、8、9、11和12染色体上。其中,运用ICIM同时在自然条件和盐胁迫条件下2年重复检测到qPN1-1,仅在碱胁迫下2年重复检测到qPN11-2,同时在盐胁迫和碱胁迫条件下2年重复检测到qPN3-3,在盐胁迫与自然条件比值下2年重复检测到qRPN1-1,仅在自然条件下2年重复检测到qGW7和同时在盐、碱胁迫和自然条件下2年重复检测到qPW11均被MCIM检测到。qPW11是1个新的耐盐碱QTL,其贡献率为7.94%—20.13%。运用MCIM对水稻耐盐碱相关性状在6个环境下进行上位性与环境互作效应分析,共检测到13对上位性QTL与环境发生互作效应。检测到2对有关单株有效穗数的上位性QTL与环境互作,检测到2对胁迫与自然条件比值下单株有效穗数的上位性QTL与环境互作;检测到2对有关结实率的上位性QTL与环境互作,检测到2对胁迫与自然条件比值下结实率的上位性QTL与环境互作;检测到1对有关千粒重的上位性QTL与环境互作,检测到1对胁迫与自然条件比值下千粒重的上位性QTL与环境互作;检测到3对有关单株穗重的上位性QTL与环境互作。【结论】盐胁迫和碱胁迫都能影响水稻的产量相关性状,但二者是性质有所差别的2种胁迫,碱胁迫破坏更强,降低产量更明显。
水稻;盐胁迫;碱胁迫;产量性状;QTL定位;环境互作分析
0 引言
【研究意义】水稻(Oryza sativa L.)作为中国乃至世界最为重要的粮食作物[1],稳产高产的重要性不言而喻。盐碱胁迫是影响水稻生长发育的一种严重逆境危害,在盐碱化的土地上种植水稻将使其产量减少近 30%[2]。地球环境的日益恶化,使得土地沙漠化和盐碱化日益加剧,水稻的生产和发展也面临着巨大的威胁。盐碱地的改良方案,一般是建造排灌工程,通过灌水排水洗盐,这种方法浪费水资源、经济投入大,效果不显著[3]。相比之下,种植耐盐碱的水稻来修复盐碱化土地改善板结土壤生态[4],更为直接、有效,同时具有投资小见效快和推动农业经济可持续发展等优点。因此,培育耐盐碱水稻品种意义重大。【前人研究进展】近年来,水稻耐盐碱的研究很多,WANG 等[5]利用RIL群体和PRASAD等[6]利用DH群体检测到有关发芽率的QTL,KIM等[7]定位有关苗高、叶干重、叶鲜重和叶面积的8个QTL,SABOURI等[8]定位有关苗高耐盐等级和苗干重的 6个 QTL,THOMSON等[9]利用 NILs群体定位苗期耐盐相关性状的27个QTL,LEE等[10]利用NILs群体定位苗期盐害级别的2个QTL,ZHAO等[11]利用F2分离群体检测到3个苗期耐盐主效QTL,LIN等[12]应用RIL群体定位到盐胁迫下3个苗存活天数的QTL,龚继明等[13]定位到耐盐主效基因 Std,顾兴友等[14]利用耐盐品Pokkali和Peta回交群体检测到苗期的5个QTL,KOYAMA等[15]在第1、4、6、9染色体上定位到与Na+、K+有关的11个QTL,SUN等[16]利用RIL群体在盐胁迫下检测到6个控制苗高和3个控制分蘖数的QTL,钱益亮等[17]以4个BC2F3群体定位出与水稻幼苗耐盐性有关的43个QTL,邢军等[18]在盐胁迫下检测到5个QTL,其中,位于第3染色体上qSNRC3-1贡献率最大,与苗期根部Na+浓度有关,CHAI等[19]运用回交育种策略寻找籼稻中耐盐基因,检测到产量相关性状的 47个加性主效 QTL 和 40个上位性 QTL。【本研究切入点】以上研究多集中在苗期耐盐性,对大田生育期耐盐性QTL定位的研究较少,针对指标主要是耐盐级别、幼苗存活天数和Na+/K+等相关性状,而水稻的耐盐性在不同的生育期存在差异[20],苗期的耐盐生理指标与盐胁迫下成熟期的产量没有密切联系[21]。仅依靠苗期表现评定品种的耐盐性并不合理,对水稻进行全生育期盐胁迫,考察累积的最终产量判定耐盐性更能满足实际育种需求。前人研究大多集中在水稻耐盐性上,碱胁迫下水稻的QTL定位鲜见报道。研究表明耐盐机理和耐碱机理可能存在差异[22],耐盐性强的水稻品种耐碱性不一定强。同时进行盐、碱胁迫下水稻产量相关性状的研究,探究2种胁迫下水稻产量相关性状变化规律和遗传机制有重要意义。【拟解决的关键问题】本研究利用东农425和长白10号衍生的重组自交系为材料,连续2年分别在盐和碱2种胁迫方式下处理,考察盐、碱胁迫下成熟期水稻的单株有效穗数、结实率、千粒重和单株穗重,对其进行加性QTL、上位性QTL与环境互作效应分析,旨在挖掘同时控制耐盐性和耐碱性的QTL,为水稻耐盐碱分子标记辅助育种奠定基础。
1 材料与方法
1.1 试验材料
以黑龙江省广泛种植的粳稻品种东农 425 (DN425)为母本,吉林省农业科学院水稻研究所选育的耐盐碱性强的粳稻品种长白10号(CB10)为父本配置杂交组合,通过“单粒传”获得180个株系的F6和F7群体作为试验材料。
1.2 田间试验方法
试验于2014年和2015年在东北农业大学实验基地进行。亲本和F6(2014年)、F7(2015年)RIL群体分别于4月20日和25日播种,采用旱育秧苗的方法,分别于5月25日和29日单本移栽到大田。2年的平均气温、降水量和日照时长均与往年相近。试验田土壤全氮0.23%,碱解氮216.4 mg·kg-1、全钾2.32%、速效钾 167.4 mg·kg-1、总磷 0.16%和磷 27 mg·kg-1。模拟盐碱池分为盐处理、碱处理和对照 3个小区,采用随机排列,2行区,行长 2 m,行距30 cm,穴距10 cm,3次重复。盐、碱处理小区返青后开始进行胁迫处理。盐胁迫处理方式是灌溉浓度定为6 ds·m-1的 NaCl[16]水溶液(0.36%),碱胁迫处理方式是灌溉 pH=9.0[22]的 Na2CO3水溶液,2种处理是保证钠离子浓度相近的同时胁迫强度为中等胁迫水平,对照为正常水灌溉。病虫害防治和除草管理方式都保持与大田生产一致。为了确保盐、碱处理强度稳定,浓度不波动,需每天分别在早、中、晚监测盐池内NaCl浓度和碱池内的pH,若遇降雨或高温干旱天气应及时排水,重新灌溉。
在盐、碱胁迫和对照3种条件6个环境:E1(2014年对照)、E2(2015年对照)、E3(2014年盐处理)、E4(2015年盐处理)、E5(2014年碱处理)和E6(2015年碱处理),每株系随机选取非边株的5个单株。测定亲本及F6、F7群体的4项产量相关性状:单株有效穗数(panicle number per plan,PN)、结实率(seed setting rate,SS)、千粒重(thousand grain weight,GW)和单株穗重(panicle weight per plant,PW),以3次重复的平均值作为统计单元。
1.3 产量相关性状表型数据统计分析
利用Excel 2013软件对产量相关性状的数据进行处理,运用SPSS(Version 17.0 for Windows)软件对各性状进行统计分析和相关性检验分析。
1.4 遗传连锁图谱构建与QTL定位
采用DNA提取试剂盒提取叶片DNA,试剂盒由天根生化科技(北京)有限公司提供。从水稻基因组(http//www.gramene.org)中获得1 000对SSR引物序列,引物由华大基因合成。利用两亲本间有多态性的138对引物对F7代180个单株DNA的进行PCR扩增,PCR反应和电泳方法参考邢军等[18]方案,最终确定多态性好的SSR标记130对。去除10个偏分离标记,剩余120个SSR标记用于构建连锁图谱,利用Mapchart 2.2软件绘制遗传连锁图谱。
取2014年和2015年耐盐碱性相关性状数据的平均值进行2种方案分析。利用QTL IciMapping v3.3软件的完备区间作图法(ICIM)进行加性QTL定位,设定LOD值为2.5。利用QTL Network 2.1软件把不同年份和盐、碱胁迫作为环境因子,对水稻耐盐碱性相关性状进行基于混合线性模型的复合区间作图法(MCIM)多环境联合分析加性、上位性与环境互作效应[23],选取临界阈值P=0.05,当QTL效应P≤0.05时,判断QTL存在。QTL命名参照MCCOUCH等[24]的方式。
2 结果
2.1 水稻耐盐和耐碱相关性状的表型值分析
2.1.1 水稻耐盐相关性状的表型分析 通过对水稻2014年和2015年的盐胁迫和自然条件下亲本及RIL群体单株的表型分析(表1)。2年自然条件下亲本DN425和 CB10的单株有效穗数和结实率表现并无显著差异,千粒重、单株穗重存在显著差异(P<0.05),且DN425高于CB10。盐胁迫下单株有效穗数、千粒重和单株穗重在两亲本之间表现出极显著差异,结实率在两亲本之间表现出显著差异。盐胁迫与自然条件比值下,单株有效穗数和单株穗重在两亲本之间表现出极显著差异,千粒重在两亲本之间表现出显著差异。双亲各性状间的遗传差异较大且在耐盐性表现方面存在明显不同,为QTL分析提供了较好的遗传背景。
2.1.2 水稻耐碱相关性状的表型分析 通过对水稻2014年和2015年碱胁迫和自然条件下亲本及RIL群体的表型分析(表 1)。发现碱胁迫下千粒重和单株穗重在两亲本之间表现出极显著差异,单株有效穗数和结实率在两亲本之间表现出显著差异。碱胁迫与自然条件比值下,单株有效穗数和单株穗重在两亲本之间表现出极显著差异。双亲各性状间的遗传差异较大且在耐碱性表现方面存在明显不同,为QTL分析提供了较好的遗传背景。在盐、碱胁迫下亲本及RIL群体的耐盐碱相关性状均受到了胁迫的影响,有减产的情况发生,CB10表现出较强的耐盐碱特性,稳产性优于DN425。且在碱胁迫条件下减产更为明显,单株有效穗数、结实率、千粒重和单株穗重以及碱胁迫与自然条件比值下各性状的平均值都比盐胁迫条件下低。表明水稻对碱胁迫更为敏感,碱胁迫更大程度地限制了高产和稳产。各个性状平均值都介于两亲本之间,且变异范围较大,存在明显的超亲分离现象,峰度、偏斜度的绝对值均小于或接近 1.0。对数据进行正态分布的适合性检验,表明各个性状都基本符合正态分布,符合数量性状的遗传特性(图1)。
2.2 水稻盐胁迫和碱胁迫下产量相关性状的相关分析
2.2.1 水稻盐胁迫下产量相关性状间相关分析通过对水稻2014年和2015年的盐、碱胁迫和自然条件下产量的相关性分析(表2)。2014年自然条件下,结实率与单株有效穗数呈显著正相关;千粒重与结实率呈显著正相关;单株穗重与单株有效穗数成极显著正相关,与结实率呈显著正相关,与千粒重呈极显著正相关。2015年自然条件下,结实率与单株有效穗数呈极显著正相关;千粒重与单株有效穗数呈显著正相关,与结实率呈极显著正相关;单株穗重与结实率呈极显著正相关,与千粒重呈极显著正相关。
2014年盐胁迫条件下,结实率与单株有效穗数呈显著正相关;千粒重与单株有效穗数呈显著正相关,与结实率呈极显著正相关;单株穗重与单株有效穗数呈极显著负相关;结实率和千粒重呈极显著正相关。2015年盐胁迫条件下,结实率与单株有效穗数呈显著正相关;千粒重与单株有效穗数呈显著正相关,与结实率呈极显著正相关;单株穗重与单株有效穗数呈极显著负相关,与结实率呈极显著正相关,与千粒重呈显著正相关。
2.2.2 水稻碱胁迫下产量相关性状间相关分析
2014年碱胁迫条件下,结实率与单株有效穗数呈显著负相关;千粒重与单株有效穗数呈显著负相关,与结实率呈极显著正相关;单株穗重与单株有效穗数呈极显著负相关,与千粒重呈极显著正相关,与结实率呈显著正相关。2015年碱胁迫条件下,结实率与单株有效穗数呈显著负相关;千粒重与单株有效穗数呈显著负相关,与结实率呈极显著正相关;单株穗重与单株有效穗数呈极显著负相关,与结实率呈极显著正相关,与千粒重呈显著正相关。
2014年和2015年碱胁迫条件下各性状与盐胁迫条件下各性状均未表现出显著相关性。
2.3 遗传连锁图谱构建
利用 120对 SSR引物对两亲本衍生的后代进行基因型鉴定,最终构建了遗传连锁图谱,总长2 397.28 cM,覆盖水稻12条染色体,标记间平均距离为19.97 cM。

表1 水稻大田生育期亲本及重组自交系群体在不同环境下产量相关性状的表型分析Table 1 Variation of different environments yield related traits of RIL population and parents in field growth period of rice
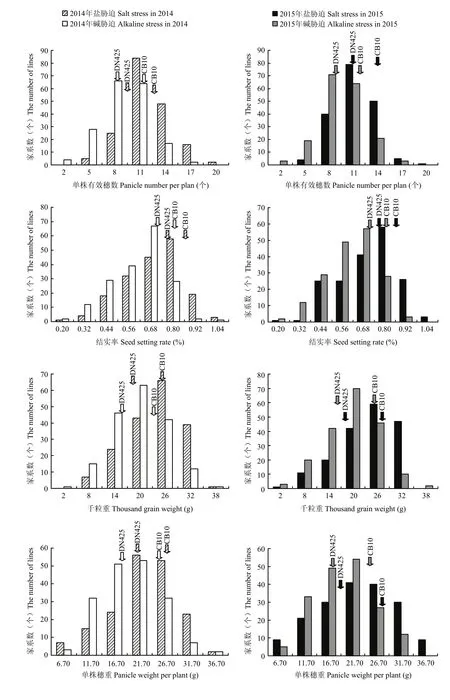
图1 盐胁迫和碱胁迫下水稻产量相关性状的分布Fig. 1 Distribution of yield-related traits under salt stress and alkaline stress in rice
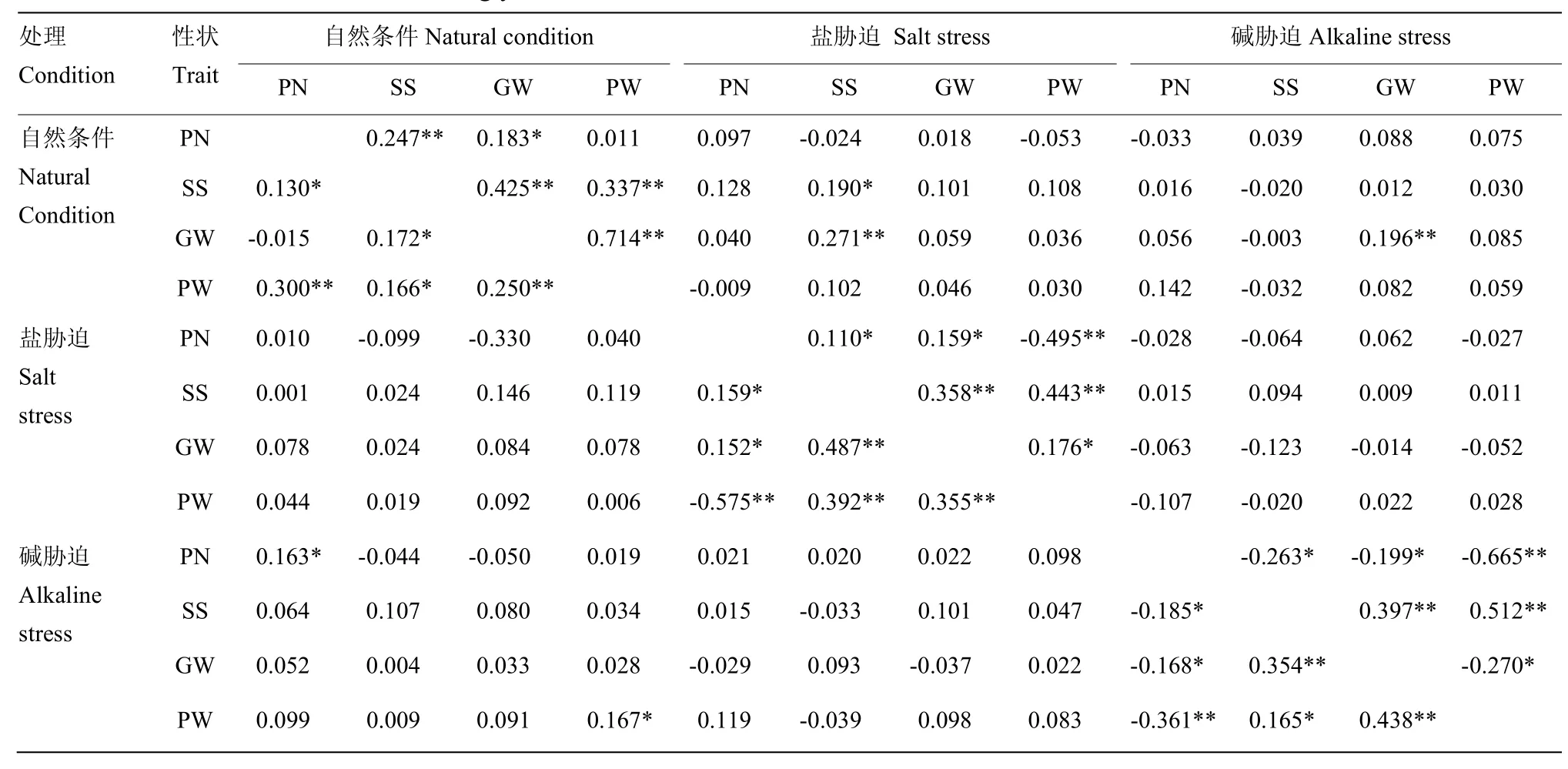
表2 产量相关性状间的相关系数Table 2 Correlation coefficients among yield related traits
2.4 水稻耐盐和耐碱相关性状的加性QTL定位分析
2.4.1 水稻耐盐相关性状的ICIM加性QTL定位分析运用ICIM对水稻的耐盐碱相关性状进行加性QTL的定位分析(表3)。共检测到61个加性效应QTL,分布在第1、2、3、4、5、6、7、8、10、11和12染色体上。
在盐胁迫下,检测到8个与单株有效穗数相关的QTL,分布在第1、3、5和11染色体上。其中,qPN1-1同时在自然条件和盐胁迫条件下2年重复检测到,增效等位基因来自父本CB10。没有检测到仅在盐胁迫条件下2年重复表达的QTL。盐胁迫与自然条件比值下,检测到5个QTL,分布在第1、2、10和12染色体上。其中,qRPN1-2在E3/E1下检测到,增效等位基因来自母本DN425;qRPN2在E6/E2下检测到,增效等位基因来自母本DN425;qRPN10在E3/E1下检测到,增效等位基因来自父本CB10;qRPN12在E3/E1下检测到,增效等位基因来自母本 DN425;qRPN1-1在2年重复检测到,增效等位基因来自父本CB10。
在盐胁迫下,检测到1个与结实率相关的QTL,分布在第 1染色体上;检测到 2个与千粒重相关的QTL,分布在第6和11染色体上。仅在盐胁迫条件下2年重复表达的QTL、同时在自然条件和盐胁迫条件下2年重复表达的QTL均没有检测到。盐胁迫与自然条件比值下,检测到结实率的2个QTL,分布在第4 和7染色体上。其中,qRSS4在E4/E2下检测到,增效等位基因来自父本CB10;qRSS7-2在E3/E1下检测到,增效等位基因来自母本DN425。盐胁迫与自然条件比值下,检测到千粒重的2个QTL,分布在第3和7染色体上。其中,qRGW3-1在E3/E1下检测到,增效等位基因来自父本CB10;qRGW7在E4/E2下检测到,增效等位基因来自父本 CB10;结实率和千粒重在盐胁迫与自然条件比值下,均没有检测到2年重复表达的QTL。
在盐胁迫下,检测到3个与单株穗重相关的QTL,分布在第1和11染色体上。仅在盐胁迫条件下2年重复表达的QTL、同时在自然条件和盐胁迫条件下2年重复表达的QTL均没有检测到。盐胁迫与自然条件比值下,检测到3个QTL,分布在第3和11染色体上。其中,qRPW3在E4/E2下检测到,增效等位基因来自母本DN425;qRPW11-1在E3/E1下检测到,增效等位基因来自父本CB10;qRPW11-2在E4/E2下检测到,增效等位基因来自父本CB10;没有检测到2年重复表达的QTL。
综上所述,李白模仿鲍照的同题新作,多有创新,情感指向个性化,用典密度变小,叙事线条变得疏朗,主题寓义宽泛,成功地超越了鲍照诗歌,如刘熙载说:“太白诗以庄、骚为大源,而于嗣宗之渊放、景之儁上、明远之驱迈、玄晖之奇秀,亦各有所取,无遗美焉。 (《艺概·诗》)”成为后代诗人学习的典范。
2.4.2 水稻耐碱相关性状的ICIM加性QTL定位分析
在碱胁迫下,检测到7个与单株有效穗数相关的QTL,分布在第1、2、3、5和11染色体上。其中,qPN11-2仅在碱胁迫下2年重复检测到,增效等位基因来自母本 DN425;qPN3-3同时在盐胁迫和碱胁迫条件下 2年重复检测到,增效等位基因来自母本DN425;没有检测到同时在自然条件和碱胁迫条件下2年重复表达的QTL。碱胁迫与自然条件比值下,检测到2个QTL,其中,qRPN1-1在E5/E1下检测到,增效等位基因来自母本DN425;qRPN2在E6/E2下检测到,增效等位基因来自母本DN425;没有检测到2年重复表达的QTL。
在碱胁迫下,检测到3个与结实率相关的QTL,分布在第1和5染色体上;检测到1个与千粒重相关的QTL,分布在第11染色体上。仅在碱胁迫条件下2年重复表达的QTL、同时在自然条件和碱胁迫条件下2年重复表达的QTL均没有检测到。碱胁迫与自然条件比值下,检测到结实率的3个QTL,其中,qRSS1-1 在E5/E1下检测到,增效等位基因来自母本DN425;qRSS12在 2年重复检测到,增效等位基因来自父本CB10。检测到千粒重的2个 QTL,其中,qRGW3-2在E5/E1下检测到,增效等位基因来自父本CB10;qRGW11在E6/E2下检测到,增效等位基因来自母本DN425;没有检测到2年重复表达的QTL。
在碱胁迫下,检测到3个与单株穗重相关的QTL,分布在第1和11染色体上。其中,qPW11同时在盐、碱和自然条件下2年重复检测到,增效等位基因来自母本DN425,且盐、碱胁迫下的贡献率高于自然条件。仅在碱胁迫条件下2年重复表达的QTL、同时在自然条件和碱胁迫条件下2年重复表达的QTL均没有检测到。碱胁迫与自然条件比值下,检测到 1个 QTL,qRPW11-1在E6/E2下检测到,增效等位基因来自父本CB10;没有检测到2年重复表达的QTL。
2.5 水稻耐盐和耐碱相关性状的MCIM加性QTL定位分析
运用MCIM对水稻耐盐碱相关性状在6个环境下进行加性及环境互作效应的联合定位分析(表 4)。共检测到17个加性QTL存在环境互作效应,分布在第1、3、5、7、8、9、11和12染色体上。
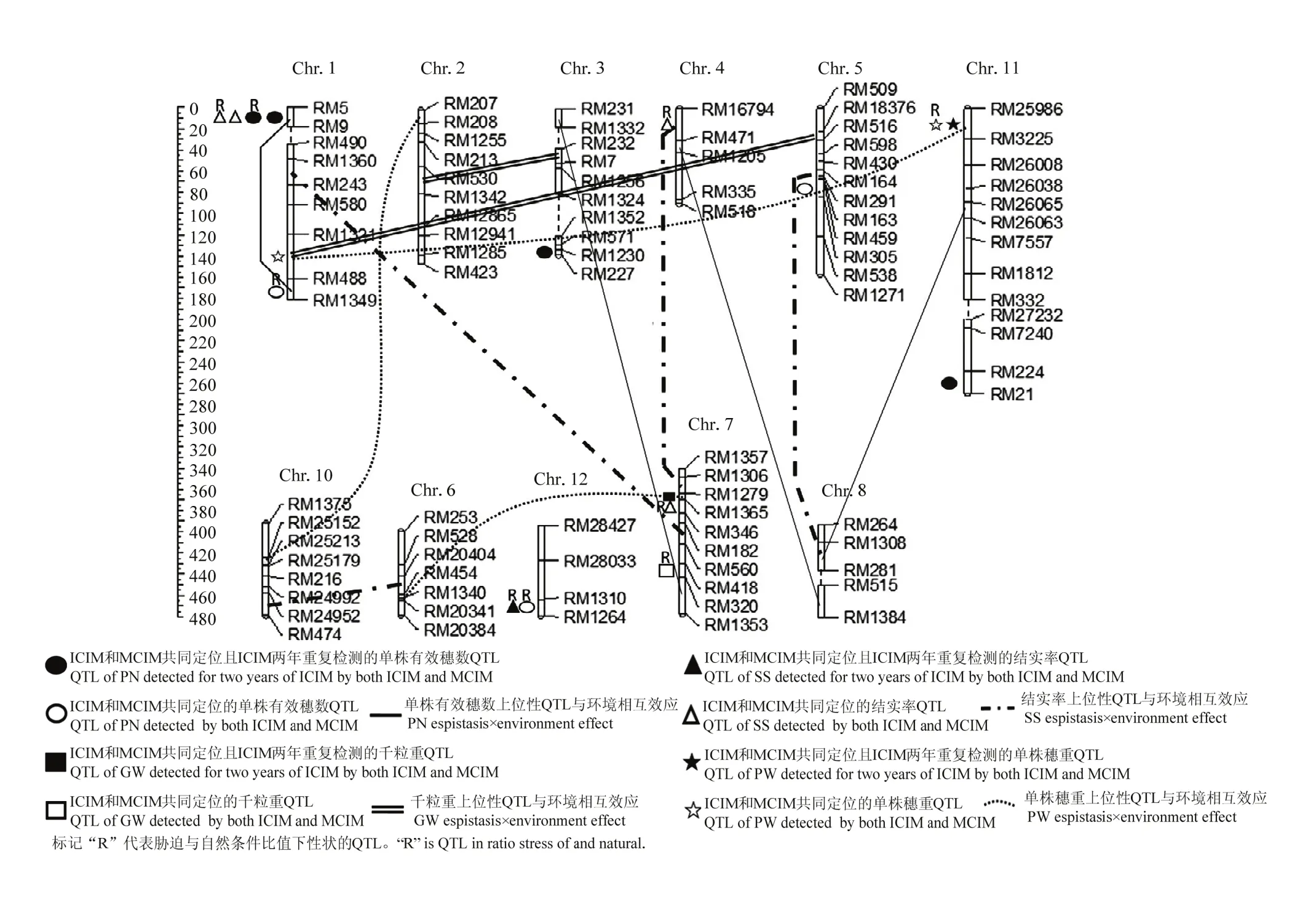
图2 水稻耐盐碱相关性状的加性QTL及上位性QTL与环境互作效应在染色体上的分布Fig. 2 Additive QTL and epistasis QTL×environment effect of salt and alkali tolerance related traits in rice on chromosomes

表3 RIL群体耐盐碱相关性状的QTL及遗传效应Table 3 QTL mapping for salt and alkali tolerant related traits of the RIL poupulation

续表3 Continued table 3
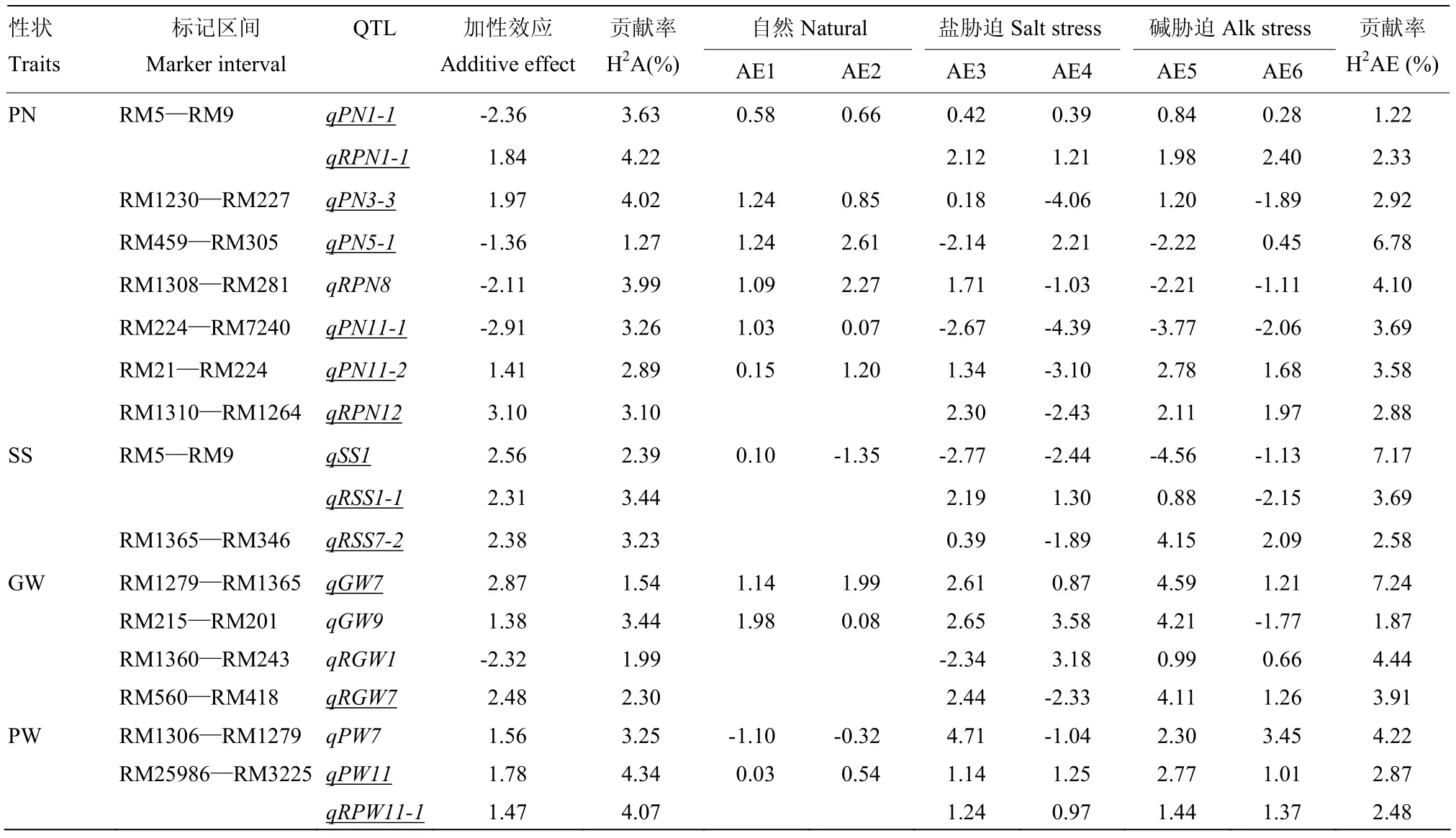
表4 RIL群体耐盐碱相关性状的加性QTL与环境互作效应Table 4 Additive QTL for environment interaction of salt and alkali tolerant related-traits in the RIL poupulation
检测到5个有关单株有效穗数的加性与环境互作QTL,分布在第1、3、5和11染色体上。其中,运用ICIM同时在自然条件和盐胁迫条件下 2年重复检测到的qPN1-1也被MCIM检测到,贡献率是3.63%,加效效应值是-2.36,环境互作贡献率是1.22%;运用ICIM仅在碱胁迫下2年重复检测到的qPN11-2也被MCIM检测到,贡献率是2.87%,加效效应值是1.41,环境互作贡献率是3.58%;运用ICIM同时在盐胁迫和碱胁迫条件下2年重复检测到的qPN3-3也被MCIM检测到,贡献率是4.02%,加效效应值是1.97,环境互作贡献率是2.92%;qPN5-1和qPN11-1被ICIM和MCIM共同检测到,但是ICIM没有2年重复检测到。胁迫与自然条件比值下,检测到3个的加性与环境互作QTL,分布在第1、8和12染色体上。其中,运用ICIM在2年重复检测到的qRPN1-1也被MCIM检测到,贡献率是4.22%,加效效应值是1.84,环境互作贡献率是2.33%。
检测到2个有关千粒重的加性与环境互作QTL,分布在第7和9染色体上。运用ICIM仅在自然条件下2年重复检测到的qGW7也被MCIM检测到。胁迫与自然条件比值下,检测到2个的加性与环境互作QTL,分布在第1和7染色体上。qRGW7被ICIM和MCIM共同检测到,但是ICIM没有2年重复检测到。
检测到 2个有关单株穗重的加性与环境互作QTL,分布在第7和11染色体上。运用ICIM同时在盐、碱胁迫和自然条件下 2年重复检测到的 qPW11也被MCIM检测到,贡献率是4.34%,加效效应值分1.78,环境互作贡献率是 2.87%。胁迫与自然条件比值下,检测到1个加性与环境互作QTL,分布在第11染色体上。qRPW11-1被ICIM和MCIM共同检测到,但是ICIM没有2年重复检测到。
2.6 水稻耐盐和耐碱相关性状的MCIM上位性QTL定位分析
运用MCIM对水稻耐盐碱相关性状在6个环境下进行上位性与环境互作效应分析(表5)。检测到13对上位性QTL与环境发生互作效应。其中,2对互作QTL影响单株有效穗数,qPN3-1/qPN7贡献率为1.55%,上位性效应值是1.24,亲本型大于重组型,qPN4/qPN8贡献率为1.75%,上位性效应值是-5.57,重组型大于亲本型,同时这2对QTL均检测到与环境之间存在互作效应,环境互作贡献率分别是 0.78%和2.14%。胁迫与自然条件比值下,检测到2对互作QTL,qRPN1-1/qRPN1-2贡献率为2.47%,上位性效应值是2.47,亲本型大于重组型;qRPN8/qRPN11贡献率为3.01%,上位性效应值是 0.98,亲本型大于重组型。同时这两对QTL均检测到与环境之间存在互作效应,环境互作贡献率分别是1.03%和1.20%。

表5 RIL群体耐盐碱相关性状的上位性QTL与环境互作效应Table 5 Epistatic QTL for environment interaction of salt and alkali tolerant related traits in the RIL poupulation
检测到2对互作QTL影响结实率,qSS4/qSS7和qSS6/qSS10贡献率分别为2.42%和3.14%,均表现为重组型大于亲本型,同时这2对互作QTL检测到与环境之间存在互作效应,环境互作贡献率分别是 1.80% 和1.24%。胁迫与自然条件比值下,检测到2对互作QTL,qRSS1-2/qRSS7-1贡献率为2.95%,上位性效应值是-1.22,重组型大于亲本型;qRSS5/qRSS8贡献率为2.34%,上位性效应值是1.35,亲本型大于重组型。同时这2对QTL均检测到与环境之间存在互作效应,环境互作贡献率分别是2.04%和2.18%。
检测到1对互作QTL影响千粒重,qGW1/qGW5贡献率为 1.99%,亲本型大于重组型,同时这对互作QTL检测到与环境之间存在互作效应,环境互作贡献率是3.22%。胁迫与自然条件比值下,检测到1对互作QTL,qRGW2/qRGW3贡献率为2.34%,上位性效应值是1.35,亲本型大于重组型,环境互作贡献率是1.12%。
检测到 3对互作 QTL影响单株穗重,是qPW6/qPW7、qPW1-3/qPW11和qPW2/qPW10,贡献率分别是5.03%、4.48%和6.03%,重组型大于亲本型,同时这3对互作QTL检测到与环境之间存在互作效应,环境贡献率分别是0.93%、1.79%和1.44%。胁迫与自然条件比值下,没有检测到的上位性与环境互作QTL。
3 讨论
3.1 盐、碱胁迫对水稻产量相关性状的影响
一直以来,高产、稳产性都是评定水稻抗性强弱的最为重要的标准。提高盐碱胁迫下的产量是水稻育种的主要目的之一。水稻最早起源于淡水沼泽环境,是一种对盐碱胁迫中度敏感的作物[25]。土壤盐碱化自然也成为了影响水稻产量的主要不利因素之一[2]。
土壤的盐碱化会影响作物的正常的新陈代谢和各项生理活动。主要是离子毒害、渗透胁迫和营养吸收不平衡等方面[16]。高盐分环境下植物生长发育受限,叶片发黄、卷曲,影响细胞分裂和伸长,作物生长缓慢,产量降低[26]。值得关注的是,2年的碱胁迫条件下各性状与盐胁迫条件下各性状均未表现出显著相关性。碱胁迫下水稻单株有效穗数、结实率、千粒重和单株穗重降低幅度均大于盐胁迫,也表明碱胁迫对水稻的伤害更严重。碱胁迫与自然条件比值下各性状降低幅度同样均大于盐胁迫与自然条件比值下各性状,表明水稻耐盐性和耐碱性存在差异。水稻在耐盐性和耐碱性上可能存在遗传机制上的差异,盐胁迫和碱胁迫可能是性质有差别的 2种胁迫。这与前人以星星草[27]、小冰麦[28]、羊草[29]、向日葵[30]、燕麦[31]、黄瓜[32]和玉米[33]为材料的研究结果一致。原因可能是由于碱胁迫下的较高 pH更严重的破坏细胞原生质膜系统和光合结构,导致有氧呼吸、光合功能下降有关[34]。此外,pH升高还可能导致Fe、Mg等叶绿素合成有关的微量元素利用率降低[35],影响光合色素合成,色素含量明显降低[36]从而导致作物更大程度的生长受限,产量下降。
3.2 ICIM和 MCIM对水稻耐盐碱性相关性状的加性QTL定位分析
大量的研究表明,水稻的耐盐碱性是多种耐盐、耐碱生理生化变化的综合呈现,是由不同染色体上的不同基因所调节的数量性状[37]。耐盐碱QTL定位研究有许多报道,并多以单个环境的数据为基础进行QTL检测,同时以盐、碱胁迫和自然条件为环境因素的QTL检测报道较少。而且从统计上来看,同时分析多个环境下的数据,能够增强QTL检测强度,精准估计QTL的位置和效应[38]。本研究还进行了2年检测,验证2年重复表达的QTL,进一步提高定位的准确性。
ICIM和MCIM的分析结果对比发现,ICIM所得到的QTL基本包括了MCIM的分析结果,ICIM定位结果更多,贡献率也高于MCIM。其原因可能是MCIM对加性、上位性以及与环境互作效应的综合分析的结果[39]。QTL的作图原理都是数学统计中的概率事件。虽然ICIM发现QTL得能力优于MCIM[40],但还不能排除其可能存在假阳性的可能。MCIM的综合分析,可能更符合实际的遗传规律[39]。但是任何单一的作图方法真实性都需要加以验证,故本研究采用双作图方法,认为共同发现的QTL可信度较高。
3.3 与前人耐盐碱性QTL分析结果的比较
本研究耐盐碱性定位结果与前人研究比较发现(表3),部分大田生育期耐盐性相关性状QTL与苗期耐盐碱性处在相同区间或相临区间,但还是有部分QTL结果处于不同的区间。水稻的苗期和大田生育期的耐盐碱性可能存在一定联系,但也保持着一定独立关系。
定位在第1染色体上的qPN1-1在多个环境下定位到而且贡献率高达8.76%—16.68%,这与邢军等[18]的定位结果一致,并且环境互作效应值较低,表示该QTL受环境影响不明显。这也符合了当QTL的自身的加性效应较强,环境互作不明显的规律。而且还发现对提高冷、热、盐和干旱胁迫忍耐力有关的已克隆基因OsGSK1[6]也在该区间,这些证据表明该区间可能存在控制水稻耐盐碱有关的基因。qGW7的定位结果与LIN等[12]结果一致,只在自然条件下检测到并没有在盐、碱环境下检测到,可能对盐碱环境敏感,影响了他们的表达。qPN3-3的定位结果与MOHAMMADI等[43]一致,与有利于长日照下粳稻早开花,调控抽穗期的已克隆基因CKI/EL1[46]定位在相同区间。与粒长粒重主效控制已克隆基因 GS3[47]定位在相邻区间。qRSS12的定位结果与LI等[48]一致,与在细胞壁的形成和植株生长过程中发挥着重要的作用的已克隆基因OsCSLD4[48]定位在在相邻区间
qPN3-3和qPW11既能在盐环境下检测到又能在碱环境下检测到,表明盐和碱胁迫之间存在共同遗传机制。而qPN1-1和qRPN1-1只在盐环境下检测到,qPN11-2和qRSS12只在碱环境下检测到,却又揭示2种胁迫可能存在独立的遗传机制,2种胁迫间的关系需要进一步研究。这些QTL能在多种环境、不同性状或者不同的遗传背景下都定位到,是遗传表现稳定且可靠的QTL,对分子标记辅助选育耐盐碱的水稻品种有应用价值。此外,本研究检测到的qRPN10、qRPN12、qGW11、qRGW3-2、qRPW3、qPW11和qRPW11-1在前人的报道中未被检测到,可能是新的 QTL位点。qPW11同时在盐、碱胁迫和自然条件下2年重复检测到,并且贡献率高达7.94%—20.13%,但在自然条件下的贡献率要低于盐、碱胁迫下检测到的贡献率,可能是因为盐碱胁迫促进了QTL表达,qPW11由双作图方法共同检测到,还与qPW1-3存在上位性互作效应,推断该QTL可能是1个同时控制耐盐和耐碱性的主效QTL。
另外相同区间定位到的不同QTL,第1染色体上同一区间RM5—RM9定位到qPN1-1、qRPN1-1、qSS1-1、qRSS1和qPW1-2,在RM243—RM580定位到的qSS1-2和qPW1-1,在第11染色体上同一区间RM25986—RM3225定位到的qGW11、qRPW11-1、qPW11,在第12染色体上同一区间RM1310—RM1264定位到qRPN12和qRSS12,可能存在一因多效性或遗传连锁。这就可以解释产量性状之间的相互关联性,产量相关的不同性状可能存在着共同的遗传基础[49]。
3.4 水稻耐盐碱相关性状 QTL上位性与环境互作效应分析
单纯的定位单个加性 QTL不能全面的反映出QTL对性状调节的真实效应。因为一个性状可能需要不同的基因共同调节[50-51],并且环境条件也能影响其表达。本试验定位结果也表明同一个性状定位的QTL分布在不同染色体上,且QTL之间存在上位性互作效应和环境互作效应,它们共同决定了QTL的主效应。环境互作效应的存在也解释了数量性状易受环境影响的现象。qPW1-3/qPW11、qRPN1-1/qRPN1-2是2对控制同一性状的加性QTL之间发生上位性效应,共同调节同一性状。这种多个基因共同调节同一性状的现象,可能对遗传的稳定性有一定作用。QTL研究中如若忽略了上位性和环境互作效应,那么,QTL的效应将无法被正确估计[52]。
本研究利用RIL群体进行2年产量相关性状定位,目的是定位出耐盐碱的产量性状 QTL,需要继续重复鉴定,准确定位QTL位点。可以考虑提高标记数目,复杂化数学模型,提高QTL定位精度,再配合常规育种手段实现分子标记辅助育种,大力促进耐盐碱性和高产2种优良性状结合。针对2年多环境2种作图方法共同发现的qPW11,可以考虑培育近等基因系群体进行精细定位,为克隆耐盐碱基因做准备。另外,值得关注的是多重逆境胁迫下的水稻遗传机理,调查发现,土壤的盐碱化一般伴随着干旱,且干旱的地区盐碱化也更为严重[53],故联合干旱和盐碱多重逆境探究同时控制耐旱性和耐盐碱性的 QTL也相当有实际意义。
4 结论
盐胁迫和碱胁迫都能影响水稻的产量相关性状,但二者是性质有所差别的2种胁迫,碱胁迫破坏更强,降低产量更明显。
[1] 周锡跃, 徐春春, 李凤博, 方福平. 世界水稻产业发展现状、趋势及对我国的启示. 农业现代化研究, 2010, 31(5): 525-528.ZHOU X Y, XU C C, LI F B, FANG F B. Status quo and trends of world’s rice industry development and its enlightenment to China. Research of Agricultural Modernization, 2010, 31(5): 525-528. (in Chinese)
[2] KUMAR K, KUMAR M, KIM S R, RYU H, CHO Y G. Insights into genomics of salt stress response in rice. Rice, 2013, 6(1): 1.
[3] 石玉海, 李彻, 张三元, 张俊国, 相桂兰. 水稻耐盐品种选育初报.吉林农业科学, 1992(4): 39-41. SHI Y H, LI Q, ZHANG S Y, ZHANG J G, XIANG G L. At the beginning of salt tolerance in rice breeding. Jilin Agricultural Sciences, 1992(4): 39-41. (in Chinese)
[4] 梁正伟, 王志春, 马红媛. 利用耐逆植物改良松嫩平原高 pH盐碱土研究进展. 吉林农业大学学报, 2008, 30(4): 517-528. LIANG Z W, WANG Z C, MA H Y. The progress in improvement of high pH saline-alkali soil in the Songnen Plain by stress tolerant plants. Journal of Jilin Agricultural University, 2008, 30(4): 517-528. (in Chinese)
[5] WANG Z, WANG J, BAO Y, WU Y, ZHANG H. Quantitative trait loci controlling rice seed germination under salt stress. Euphytica, 2011, 178(3): 297-307.
[6] PRASAD S R, BAGALI P G, HITTALMANI S, SHASHIDHAR H E. Molecular mapping of quantitative trait loci associated with seedling tolerance to salt stress in rice (Oryza sativa L.). Current Science, 2000, 78(2): 162-164.
[7] KIM D M, JU H G, KWON, OH C S, AHN S N. Mapping QTLs for salt tolerance in an introgression line population between Japonica cultivars in rice. Journal of Crop Science and Biotechnology, 2009, 12(3): 121-128.
[8] SABOURI H, REZAI A M, MOUMENI A , KAVOUSI A, KATOUZI M, SABOURI A. QTLs mapping of physiological traits related to salt tolerance in young rice seedlings. Biologia Plantarum, 2009, 53(4): 657-662.
[9] THOMSON M J, OCAMPO M, EGDANE J, RAHMAN M A, SAJISE A G, ADORADA D L, GREGORIO G B. Characterizing the Saltol quantitative trait locus for salinity tolerance in rice. Rice, 2010, 3(2/3): 148-160.
[10] LEE S Y, ANN J H, CHA Y S, YUN D W, LEE M C, KO J C, EUN M Y. Mapping QTL related to salinity tolerance of rice at the young seedling stage. Plant Breeding, 2007, 126(1): 43-46.
[11] ZHAO K, TUNG C W, EIZENGA G C, WRIGHT M H, ALI M L, PRICE A H, MCCLUNG A M. Genome-wide association mapping reveals a rich genetic architecture of complex traits in Oryza sativa. Nature Communications, 2011, 2: 467.
[12] LIN H X, ZHU M Z, YANO M, GAO J P, LIANG Z W, SU W A, CHAO D Y. QTLs for Na+and K+up-take of the shoots and roots controlling rice salt tolerance. Theoretical and Applied Genetics, 2004 108(2): 253-260.
[13] 龚继明, 何平, 钱前, 沈利爽, 朱立煌, 陈受宜. 水稻耐盐性 QTL的定位. 科学通报, 1998, 43(17): 1847-1850. GONG J M, HE P, QIAN Q, SHEN L S, ZHU L H, CHEN S Y. Preliminary detection of quantitative trait loci for salt tolerance in rice. Chinese Science Bulletin, 1998, 43(17): 1847-1850. (in Chinese)
[14] 顾兴友, 梅曼彤, 严小龙, 郑少玲, 卢永根. 水稻耐盐性数量性状位点的初步检测. 中国水稻科学, 2000, 14(2): 2-7. GU X Y, MEI M T, YAN X L, ZHENG S L, LU Y G. Preliminary detection of quantitative trait loci for salt tolerance in rice. Chinese Journal of Rice Science, 2000, 14(2): 2-7. (in Chinese)
[15] KOYAMA M L, LEVESLEY A, KOEBNER R M D, FLOWERS T J, YEO A R. Quantitative trait loci for component physiological traits determining salt tolerance in rice. Plant Physiology, 2001, 125(1): 406-422.
[16] SUN J, ZOU D T, LUAN F S, ZHAO H W, WANG J G, LIU H L, LIU Z L. Dynamic QTL analysis of the Na+content, K+content, and Na+/K+ratio in rice roots during the field growth under salt stress. Biologia Plantarum, 2014, 58(4): 689-696.
[17] 钱益亮, 王辉, 陈满元, 张力科, 陈冰嬬, 崔金腾, 刘海燕, 朱苓华, 石英尧, 高用明, 黎志康. 利用 BC2F3产量选择导入系定位水稻耐盐QTL. 分子植物育种, 2009, 7(2): 224-232. QIAN Y L, WANG H, CHEN M Y, ZHANG L K, CHEN B X, CUI J T, LIU H Y, ZHU L H, SHI Y Y, GAO Y M, LI Z K. Detection of salt-tolerant QTL using BC2F3yield selected introgression lines of rice (Oryza sativa L.). Molecular Plant Breeding, 2009, 7(2): 224-232. (in Chinese)
[18] 邢军, 常汇琳, 王敬国, 刘化龙, 孙健, 郑洪亮, 赵宏伟, 邹德堂.盐、碱胁迫条件下粳稻Na+、K+浓度的QTL分析. 中国农业科学, 2015, 48(3): 604-612. XING J, CHANG H L, WANG J G, LIU H L, SUN J, ZHENG H L, ZHAO H W, ZOU D T. QTL analysis of Na+and K+concentrations in Japonica rice under salt and alkaline stress. Scientia Agricultura Sinica, 2015, 48(3): 604-612. (in Chinese)
[19] CHAI L, ZHANG J, PAN X B, ZHANG F, ZHENG T Q, ZHAO X Q, WANG W S, ALI JAUHAR, XU J L, LI Z T. Advanced Backcross QTL Analysis for the Whole Plant Growth Duration Salt Tolerance in Rice(Oryza sativa L.). Journal of Integrative Agriculture, 2014, 13(8): 1609-1620.
[20] 程广有, 许文会, 黄永秀. 植物耐盐碱性的研究-水稻耐盐性与耐碱性相关分析. 吉林林学院学报, 1996, 12(4):214-217. CHENG G Y, XU W H, HUANG Y X. Study on the saline-alkaline tolerance in plants (1) -correlation analysis between saline tolerance and alkaline-tolerance of rice. Journal of Jilin Forestry University , 1996, 12(4): 214-217. (in Chinese)
[21] ZAIDEM M L, MENDOZA R D, TUMIMBANG E B. Genetic variability of salinity tolerance at different growth stages of rice. PBGB 2003 Annual Report. Los Banos, the Philippines: International Rice Research Institute.
[22] 程广有, 许文会, 黄永秀, 刘宪虎, 李范洙. 水稻品种耐盐碱性的研究. 延边农学院学报, 1995, 17(4): 195-201. CHENG G Y, XU W H, HUANG Y X, LIU X H, LI F Z. Study on the saline-alkaline tolerance of rice varieties II. A screening method for alkline-tolerance of rice varieties. Journal of Yanbian Agricultural College, 1995, 17(4): 195-201. (in Chinese)
[23] YANG J, ZHU J, WILLIAMS R W. Mapping the genetic architecture of complex traits in experi-mental populations. Bioinformatics, 2007, 23(12): 1527-1536.
[24] MCCOUCH S R, CHO Y G, YANO M, PAUL E, BLINSTRUB M, MORISHIMA H, KINOSHITA T. Report on QTL nomenclature. Rice Genetics Newsletter, 1997, 14(11): 11-13.
[25] QI D L, GUO G Z, LEE M C, CAO G L, ZHANG J G, ZHOU Q Y, HAN L Z. Progress of physiology and genetic research on saline-alkaline tolerance in rice. Journal of Plant Genetic Resources, 2007, 8(4): 486-493.
[26] 胡兴旺, 金杭霞, 朱丹华. 植物抗旱耐盐机理的研究进展. 中国农学通报, 2015, (24): 137-142. HU X W, JIN H X, ZHU D H. Research progress on drought resistance and salt tolerance mechanism of plants. Chinese Agricultural Science Bulletin, 2015, (24): 137-142. (in Chinese)
[27] SHI D C, YIN L J. Difference between salt (NaCl) and alkaline (Na2CO3) stresses on Pucinellia tenuiflora (Griseb. ) Scribn. et Merr. plants. Acta Botanica Sinica (Chinese Edition), 1993, 35: 144-149.
[28] 杨春武, 李长有, 尹红娟, 鞠淼, 石德成. 小冰麦(Triticum aestivum-Agropyron intermedium)对盐胁迫和碱胁迫的生理响应.作物学报, 2007, 33(8): 1255-1261. YANG C W, LI C Y, YIN H J, JU M, SHI D C. Physiological response of xiaobingmai(Triticum aestivum-Agropyron intermedium) to salt-stress and alkali-stress. Acta Agronomica Sinica, 2007, 33(8): 1255-1261. (in Chinese)
[29] SHI D, WANG D. Effects of various salt-alkaline mixed stresses on Aneurolepidium chinense (Trin.) Kitag. Plant and Soil, 2005, 271(1/2): 15-26.
[30] SHI D, SHENG Y. Effect of various salt-alkaline mixed stress conditions on sunflower seedlings and analysis of their stress factors. Environmental and Experimental Botany, 2005, 54(1): 8-21.
[31] 刘建新, 王金成, 贾海燕. 燕麦幼苗对盐胁迫和碱胁迫的生理响应差异. 水土保持学报, 2015, 29(5): 331-336. LIU J X, WANG J C, JIA H Y. Difference between physiological responses of Avena nuda seedlings to salt and alkali stresses. Journal of Soil and Water Conservation, 2015, 29(5): 331-336. (in Chinese)
[32] 张丽平, 王秀峰, 史庆华, 高青海, 刘泽洲. 黄瓜幼苗对氯化钠和碳酸氢钠胁迫的生理响应差异. 应用生态学报, 2008, 19(8): 1854-1859. ZHANG L P, WANG X F, SHI Q H, GAO Q H, LIU Z Z. Differences of physiological responses of cucumber seedlings to NaCl and NaHCO3stress. Chinese Journal of Applied Ecology, 2008, 19(8): 1854-1859. (in Chinese)
[33] 曲元刚, 赵可夫. NaCl和Na2CO3对玉米生长和生理胁迫效应的比较研究. 作物学报, 2004, 30(4): 334-341. QU Y G, ZHAO K F. Comparative studies on growth and physiological reaction of Zea mays under NaCl and Na2CO3stresses. Acta Agronomica Sinica, 2004, 30(4): 334-341. (in Chinese)
[34] 白文波, 李品芳, 李保国. NaCl和NaHCO3胁迫下马蔺生长与光合特性的反应. 土壤学报, 2008, 45(2): 328-335. BAI W B, LI P F, LI B G. Response of iris Lactea Var. chinensis to NaCl and NaHCO3stress in growth and photosynthesis. Acta Pedologica Sinica, 2008, 45(2): 328-335. (in Chinese)
[35] YANG C, CHONG J, LI C, KIM C, SHI D, WANG D. Osmotic adjustment and ion balance traits of an alkali resistant halophyte Kochia sieversiana during adaptation to salt and alkali conditions. Plant and Soil, 2007, 294(1/2): 263-276.
[36] 刘建新, 王鑫, 王瑞娟, 贾海燕. 黑麦草对 NaHCO3胁迫的光合生理响应. 草业学报, 2012, 21(3): 184-190. LIU J X, WANG X, WANG R J, JIA H Y. Photosynthetic physiological response of Lolium prenne to NaHCO3stress. Acta Prataculturae Sinica, 2012, 21(3): 184-190. (in Chinese)
[37] FLOWERS T J. Improving crop salt tolerance. Journal of Experimental Botany, 2004, 55(396): 307-319.
[38] 梁慧珍, 余永亮, 杨红旗, 许兰杰, 董薇, 牛永光, 张海洋, 刘学义, 方宣钧. 大豆异黄酮及其组分含量的遗传分析与 QTL检测.作物学报, 2015, 41(9): 1372-1383. LIANG H Z, YU Y L, YANG H Q, XU L J, DONG W, NIU Y G, ZHANG H Y, LIU X Y, FANG X J. Genetic analysis and QTL mapping of isoflavone contents and its components in soybean. Acta Agronomica Sinica, 2015, 41(9): 1372-1383. (in Chinese)
[39] 梁慧珍, 余永亮, 杨红旗, 张海洋, 董薇, 崔暐文, 杜华, 刘学义,方宣钧. 大豆小区产量及其相关性状QTL间的上位性和环境互作效应. 植物学报, 2014, 49(3): 273-281. LIANG H Z, YU Y L, YANG H Q, ZHANG H Y, DONG W, CUI W W, DU H, LIU X Y, FANG X J. Epistatic effects and quantitative trait loci (QTL) × environment (QE) interaction effects for yield per plot and botanical traits in soybean. Chinese Bulletin of Botany, 2014, 49(3): 273-281. (in Chinese)
[40] 李杰勤, 张启军, 叶少平, 赵兵, 梁永书, 彭勇, 吴发强, 王世全,李平. 四种不同QTL作图方法的比较研究. 作物学报, 2005, 31(11): 1473-1477. LI J Q, ZHANG Q J, YE S P , ZHAO B, LIANG Y S, PENG Y, WU F Q, WANG S Q, LI P. Comparative research on four mapping methods of QTLs. Acta Agronomica Sinica, 2005, 31(11): 1473-1477. (in Chinese)
[41] TAKEHISA H, SHIMODATE T, FUKUTA Y, UEDA T, YANO M, YAMAYA T, SATO T. Identification of quantitative trait loci for plant growth of rice in paddy field flooded with salt water. Field Crops Research, 2004, 89(1): 85-95.
[42] AMMAR M H M, PANDIT A, SINGH R K, SAMEENA S, CHAUHAN M S, SINGH A K, SINGH N K. Mapping of QTLs controlling Na+, K+and CI−ion concentrations in salt tolerant indica rice variety CSR27. Journal of Plant Biochemistry and Biotechnology, 2009, 18(2): 139-150.
[43] MOHAMMADI R, MENDIORO M S, DIAZ G Q. Mapping quantitative trait loci associated with yield and yield components under reproductive stage salinity stress in rice (Oryza sativa L. ). Journal of Genetics, 2013, 92(3): 433-443.
[44] LIANG J, QU Y, YANG C, MA X D, CAO G L, ZHAO Z W, HAN L Z. Identification of QTLs associated with salt or alkaline tolerance at the seedling stage in rice under salt or alkaline stress. Euphytica, 2015, 201(3): 441-452.
[45] ZHENG H, ZHAO H, LIU H, WANG J, ZOU D. QTL analysis of Na+and K+concentrations in shoots and roots under NaCl stress based on linkage and association analysis in japonica rice. Euphytica, 2015, 201(1): 109-121.
[46] KWON C T, YOO S C, KOO B H, CHO S H, PARK J W, ZHANG Z Y, LI J J, LI Z C, PAEK N C. Natural variation in early flowering1 contributes to early flowering in japonica rice under long days. Plant, Cell & Environment, 2014, 37(1): 101-112.
[47] TAKANO K N, DOI K, YOSHIMURA A. GS3 participates in stigma exsertion as well as seed length in rice. Breeding Science, 2011, 61(3): 244-250.
[48] LI M, XIONG G Y, LI R, CUI J J, TANG D, ZHANGB C, PAULY M, CHENG Z K, ZHOU Y H. Rice cellulose synthase-like D4 is essential for normalbiosynthesis and plant growth. The Plant Journal, 2009, 60(6): 1055-1069.
[49] 李婕, 王忠, 李永慈, 盖钧镒, 黄中文, 邬荣领. 异速生长的 QTL定位模型及一因多效性扩展. 南京林业大学学报(自然科学版), 2014, 38(3): 35-39. LI J, WANG Z, LI Y C, GAI J Y, HUANG Z W, WU R L. A novel QTL mapping model for allometric growth and pleiotropic extension. Journal of Nanjing Forestry University (Natural Sciences Edition) , 2014, 38(3): 35-39. (in Chinese)
[50] WALIA H, WILSON C, CONDAMINE P, LIU X, ISMAIL A M, ZENG L, CLOSE T J. Comparative transcriptional profiling of two contrasting rice genotypes under salinity stress during the vegetative growth stage. Plant Physiology, 2005, 139(2): 822-835.
[51] WALIA H, WILSON C, ZENG L, ISMAIL A M, CONDAMINE P, CLOSE T J. Genome-wide transcriptional analysis of salinity stressed japonica and indica rice genotypes during panicle initiation stage. Plant Molecular Biology, 2007, 63(5): 609-623.
[52] 高用明, 朱军, 宋佑胜, 何慈信, 石春海, 邢永忠. 水稻永久 F2群体抽穗期 QTL的上位性及其与环境互作效应的分析. 作物学报, 2004, 30(9): 849-854. GAO Y M, ZHU J, SONG Y S, HE C X, SHI C H, XING Y Z. Use of permanent F2population to analyze epistasis and their interaction effects with environments for QTLs controlling heading date in rice. Acta Agronomica Sinica, 2004, 30(9): 849-854. (in Chinese)
[53] MUNNS R, TESTER M. Mechanisms of salinity tolerance. Annual Review Plant Biology, 2008, 59: 651-681.
(责任编辑 李莉)
QTL Mapping and QTL × Environment Interaction Analysis of Salt and Alkali Tolerance-Related Traits in Rice(Oryza sativa L.)
LIANG YinPei, SUN Jian, SUO YiNing, LIU HuaLong, WANG JingGuo, ZHENG HongLiang, SUN XiaoXue, ZOU DeTang
(Rice Research Institute, College of Agriculture, Northeast Agricultural University, Harbin 150030)
【Objective】Experiments were carried out in 2014 and 2015 to study the yield related traits in rice under salt andalkaline stress in order to explore the QTL of major genes of salt and alkaline tolerance, and analyze the interaction effects between QTL and environment, thus revealing the genetic mechanism of panicle number per plant, seed setting rate, thousand grain weight and panicle weight per plant in rice under salt and alkaline stress. The results of the present study will provide a scientific basis for the rice genetic mechanism of salt and alkaline tolerance and molecular marker assisted breeding. 【Method】The recombinant inbred line (RIL) population derived from a cross between Dongnong 425 (DN425) with high yielding ability and quality as the female parent and Changbai 10 (CB10) with salt and alkaline tolerance as the male parent. A genetic linkage map was constructed with 120 SSR markers. The panicle number per plant, seed setting rate, thousand grain weight and panicle weight per plant in rice were measured under 6ds·m-1NaCl solution of salt stress, Na2CO3solution (pH = 9.0) of alkaline stress and the normal water irrigation as control conditions during the whole growing period in 2014 and 2015. The additive quantitative trait loci(QTL)analysis was conducted by using the complete interval mapping method (ICIM),the additive and epistatic QTL×environment interaction effects was analyzed by using the mixed composite interval mapping method (MCIM). 【Result】Compared with the salt stress, the alkaline tolerant traits of rice decreased significantly, and were more sensitive to the alkaline stress, the alkaline stress was more restrictive to the high-yield and stable-yield in 2014 and 2015. Under the condition of alkaline stress for two years, no significant correlation was found between the traits of salt stress. There may be genetic differences in rice under salt stress and alkaline stress. By using ICIM, a total of 61 additive QTLs for salt and alkali tolerance-related traits were detected, which were distributed on chromosomes 1, 2, 3, 4, 5, 6, 7, 8, 10, 11 and 12. By using MCIM, a total of 17 the additive QTL×environment interaction effects QTLs for salt and alkali tolerance-related traits were detected, which were distributed on chromosome 1, 3, 5, 7, 8, 9, 11 and 12. By using ICIM, qPN1-1 which was repeatedly detected under both natural and salt stress conditions for two years, qPN11-2 which was repeatedly detected only under alkaline stress conditions for two years, qPN3-3 which was repeatedly detected under both salt and alkaline stress conditions for two years, qRPN1-1 which was repeatedly detected under both natural and salt stress conditions for two years, qGW7 which was repeatedly detected only under natural conditions for two years and qPW11 which was repeatedly detected under salt, alkaline stress and natural conditions for two years, all of them were detected by MCIM. A new salt and alkali tolerance QTL qPW11, can explain 7.94%—20.13% of phenotypic variance. By using MCIM, a total of 13 epistatic QTL×environment interaction effects QTLs for salt and alkali tolerance-related traits were detected. Two pairs of epistatic QTLs which are related to panicle number per plant were detected have significant environmental interaction effects. Two pairs of epistatic QTLs which are related to panicle number per plant under ratio of stress and natural were detected have significant environmental interaction effects. Two pairs of epistatic QTLs which are related to seed setting rate were detected have significant environmental interaction effects, two pairs of epistatic QTLs which are related to seed setting rate under the ratio of stress and natural conditions were detected have significant environmental interaction effects. One pairs of epistatic QTLs which are related to thousand grain weight were detected have significant environmental interaction effects. One pairs of epistatic QTLs which are related to thousand grain weight under the ratio of stress and natural conditions were detected have significant environmental interaction effects. Three pairs of epistatic QTLs which are related to panicle weight per plant were detected have significant environmental interaction effects.【Conclusion】Both the salt stress and the alkaline stress could affect the yield - related traits in rice, but they are two kinds of stresses with different properties. Alkaline stress damage is more severe, and yield reduction is more significantly.
rice; salt stress; alkaline stress; yield; QTL mapping; environment interaction
2016-11-25;接受日期:2017-01-19
黑龙江省重大科技招标项目(GA14B102-02)
联系方式:梁银培,E-mail:747486836@qq.com。孙健,E-mail:277347680@qq.com。梁银培和孙健为同等贡献作者。通信作者邹德堂,E-mail:zoudt@163.com

