微生物来源羰基还原酶分子改造研究进展
岑佳善,弓添添,陈蔚青,王 普
(1.浙江工业大学 药学院,浙江杭州310014;2.浙江树人大学 生物与环境工程学院,浙江杭州310015)
微生物来源羰基还原酶分子改造研究进展
岑佳善1,弓添添1,陈蔚青2,王 普1
(1.浙江工业大学 药学院,浙江杭州310014;2.浙江树人大学 生物与环境工程学院,浙江杭州310015)
羰基还原酶具有高度的立体选择性和催化活性,是生物催化合成手性化合物中应用最为广泛的催化剂之一。由于天然来源的羰基还原酶表达水平低、分离纯化困难以及催化效率不理想等原因,限制了羰基还原酶的工业化应用。利用基因工程技术对羰基还原酶进行分子改造,可显著提高其催化活性。本文中,笔者就羰基还原酶体外分子改造的不同策略、研究进展及其成果进行综述,总结其分子改造的一般规律,展望其发展方向,为微生物来源羰基还原酶的体外分子改造研究提供借鉴。
羰基还原酶;催化机理;定向进化;理性设计
羰基还原酶(carbonyl reductase)因其具有高度的化学、区域和立体选择性,常用于生物催化合成光学活性手性化合物,在化学、医药和精细化学品等领域具有重要应用[1-2]。虽然越来越多的生物催化剂已被应用于工业生产,但由于自然界的环境与工业生产需求上的实际差异,现有的生物催化剂资源并不能完全满足生物化工产业的需求。因此,对生物催化剂的改造——体外分子改造技术应运而生[3],特别是近年来分子生物学和生物信息学技术的快速发展,使得在实验室模拟自然进化机制对现有的生物催化剂进行人为改造成为可能[4]。积极探索羰基还原酶的分子催化机理,并运用分子生物技术改造酶的性能,也可为今后基于分子改造创制性能优良的新型还原酶催化剂奠定理论基础[5]。
羰基还原酶在酶学分类上属于短链脱氢/还原酶类(short-chain dehydrogenases/reduetases,SDR),为非金属还原酶,约250个氨基酸残基,通常以二聚体或四聚体形式存在[6]。SDR拥有3 000多个主要酶蛋白,其中有30多个三维结构编入蛋白质数据库(PDB)。虽然不同的酶序列相似度较低(大约在15%~30%),但位于中心部位的β-夹层结构,其三维结构的α/β-折叠模式具有高度相似性[7],如人类胆绿素还原酶(PDB:1hdo)和假单胞菌的转录调控酶(PDB:1k6x)以SDR作为最优同源结构的模式(图1)。
图1 SDR的中心β-夹层结构与其最优 同源结构之间的结构关系Fig.1 Diagram displaying the structural relationship between SDR enzyme along the central (β-sheet) and related structural neighbours
1 催化机理
基于一级结构和三维结构的生物信息学分析,SDR含有保守催化序列Tyr-X-X-X-Lys和N末端NAD(P)H结合的保守序列Gly-(X)3-Gly-X-Gly。保守催化序列Tyr-(X)3-Lys中的Tyr和Lys与另外的2个氨基酸残基(Asn和Ser)形成Asn-Ser-Tyr-Lys催化四联体。在这催化四联体中,Tyr作为最保守的氨基酸残基,也是主要的催化基团,其与底物结合并发挥作用;Ser稳定底物作用;Lys与辅酶形成氢键,且降低Tyr—OH的pKa值[8]。
羰基化合物的羰基C一般呈平面结构,属于sp2杂化,具有re和si面的两个非对映面。在还原过程中,NAD(P)H的pro-S氢分别从si面和re面传递给底物的羰基,形成S-构型的醇,称为Prelog规则。而pro-R氢分别从si面和re面传递给底物的羰基,形成R-构型的醇,称为反Prelog规则[9]。
羰基还原酶在催化羰基化合物还原反应过程中需要NAD(P)H作为氢或电子传递体,在还原酶的作用下,羰基底物从R或S面进攻羰基生成相应的单一对映体醇,同时NAD(P)H被转化成NAD(P)+。酶反应一般遵循顺序机制:首先,酶和辅酶形成的“二元复合物”与底物结合形成三联体中间物;其次,NAD(P)H上的氢转移给底物生成相应的醇,同时NAD(P)H变为NAD(P)+;最后,酶释放出产物醇,与NAD(P)+脱离[10],如图2所示。
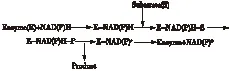
图2 还原酶顺序机制Fig.2 Order mechanism of reductase
2 羰基还原酶的分子改造策略
目前,羰基还原酶的体外分子改造策略主要分为三种:一是基于已知蛋白质结构和机理的“理性设计”;二是基于未知蛋白质信息的“非理性设计”;三是将理性设计与非理性设计相结合延伸出的“半理性设计”[11]。分子改造技术不仅为研究酶的底物特异性和催化活性位点、改善酶的热稳定性以及提高酶作用温度等提供了有效手段,更有利于深入了解酶的蛋白结构与功能之间的关系。下面从这三种分子改造策略来分析羰基还原酶体外分子改造的研究进展。
2.1 理性设计策略
理性设计策略是分子改造的重要手段之一。近年来,由于生物信息学等相关学科的进步,理性设计越来越受到人们的重视,并已取得了可喜的进展,在蛋白质工程方面的应用也愈加广泛[12]。目前,应用较为广泛的理性设计策略主要分为基于蛋白质构象的理性设计和基于同源建模的理性设计。
2.1.1 基于蛋白质构象的理性设计
基于酶的结构、功能和催化机理等分析酶的不同结合域对酶催化活性、底物特异性和稳定性以及对映体选择性等特性的改造具有指导作用[13]。Jakoblinnert等[14]通过分析近平滑假丝酵母(Candidaparapsilosis)羰基还原酶(CPCR2)的蛋白结构(PDB:1R37),发现A275位于二聚体界面,接近活性中心且内部含有亚基氢键,对其进行定点突变,得到突变株CPCR2-(A275N,L276Q),使其酶活和在两相介质中的稳定性均提高1.5倍。Zhang等[15]通过分析近平滑假丝酵母羰基还原酶(SCR)蛋白结构得到其辅酶结合区域,组合突变点S67D/H68D对羟基苯乙酮催化反应速率提高1.9倍,同时对映体选择性发生反转。由此可见,通过基于蛋白质构象的理性设计,可以有效提高羰基还原酶的稳定性和催化特性。
2.1.2 基于同源建模的理性设计
理性设计通常需要对酶分子的三维结构有清晰的认识,但对于尚未有晶体结构报道的羰基还原酶,只能寻找相似度最高的序列进行同源建模[16]。通过软件模拟进行同源建模,考察某氨基酸对目标蛋白与底物的影响,再通过突变这些氨基酸以期得到特殊优良性状的菌株。对来自于睾酮丛毛单胞菌(Comamonastestosteroni)的羰基还原酶,通过酶动力学、稳定性研究以及荧光检测得到N86、Y155和K159与NADH的结合作用有关(图3),因此Chang等[17]根据图3模拟结果对其位点进行定点突变得到N86A、Y155F和K159A突变体酶,经研究发现,其催化常数降低了37~220倍,而解离常数增加了3~75倍。Heiss等[18]研究嗜热厌氧乙醇杆菌(Thermoanaerobacterethanolicus)脱氢酶时,以布洛克热厌氧菌(Thermoanaerobiumbrockii)来源的乙二醇脱氢酶(1YKF.PDB)的蛋白结构为同源模型,经研究发现,C295为烷基键口袋,其大小决定酮基底物的取向键,经定点突变后获得突变株C295A,以此来催化反应,发现所得产物的对映选择性发生反转,而动力学常数保持一致。Luo等[19]利用同源建模与分子对接技术分析乳酸克鲁维酵母(Kluyveromyceslactis)醛酮还原酶,发现其295位和296位氨基酸靠近活性中心,通过两轮定点突变得到组合突变株KlAKR-Y295W/W296L,研究发现,其催化效率比野生型提高了11.25倍。同样,利用同源建模与分子对接技术,Chao等[20]发现白毛杨(Populustomentosa)肉桂酰辅酶还原酶(CCR)的192、155和208位氨基酸组成了底物结合区域,对其进行定点突变,得到催化效率较野生型提高4.7倍的突变酶F155Y。由此可见,在目的酶蛋白的三维结构不够明晰的情况下,通过同源建模的理性设计,亦能达到改造酶蛋白的目的。
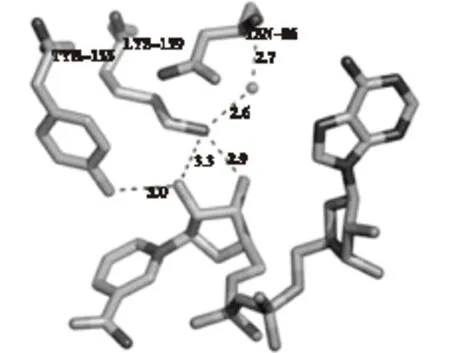
由PyMOL软件设计出的源于PDB:1fk8的3α-HSD(/CR)的二维结构,虚线为氢键[17]图3 活性位点N86、Y155和K159与NAD+的结合构型Fig.3 The N86,Y155,and K159 residues interact with NAD+ as shown
2.2 定向进化的策略
由于大部分羰基还原酶的晶体结构尚未解析出来,因此,目前的改造工程主要是依靠定向进化等非理性手段[21]。定向进化主要包括建立突变文库、选择合适的表达体系以及建立快速、高效的高通量筛选方法等[22]。
2.2.1 易错PCR
采用易错PCR不需要了解酶蛋白的任何信息,仅需通过改变传统PCR反应体系中的各因素,使碱基随机引入错误而创造序列多样性文库[23-24]。Rao等[25]利用易错PCR方法构建了含有3 000多个突变子的文库,以65 ℃为筛选温度,获得了7个热稳定性有所提高的单点突变株,再通过组合饱和突变得到了四点突变株,热稳定性提高了24.7倍。Huang等[26]同样利用定向进化和饱和突变相结合的方法,将还原酶CgKR1的溶解温度提高了12 ℃。Machielsen等[27]采用一轮易错PCR构建了1 500个突变子,30 ℃筛选酶活,得到的乙醇脱氢酶突变株对底物2,5-己二酮的催化能力是野生型菌株的10倍。虽然易错PCR技术可以达到定向进化的目的,但此方法的关键问题是要有高效的筛选策略。
2.2.2 DNA改组
DNA改组技术是利用基因重组和重排对某一个酶进行改造,同样不需要知道蛋白质的三维结构与功能之间的关系,便可达到体外进化的目的,加速了基因的进化程度[28]。金庆超等[28]利用DNA改组技术研究多种链霉菌的spyl调控基因,筛选得到71个spyl改组基因,接合子的普那霉素Ⅰ产量为出发菌株的7.5倍。Suzuki等[29]运用DNA改组技术对嗜热菌(Thermusthermophilus) HICDH进行定向进化,得到突变株LR5-1,使其对3-异丙基苹果酸(3-IPM)的催化效率提高了65倍。
2.2.3 筛选方法的建立
虽然通过定向进化可以获得性状优良的菌株。但是,定向进化技术的成功依赖两个关键因素,一是建立大容量的突变体库,二是建立简单、高效、灵敏和特异性强的筛选方法。目前,常用的突变体筛选方法主要有平板筛选法、荧光法、显色法以及表面展示技术[30]。Makino等[31]在平板中进行苯乙醛脱氢酶催化2-氯-1-(3-氯苯基)乙酮和4-氯-乙酰乙酸乙酯的还原反应,并添加5-溴-4-氯-3-吲哚基-磷酸盐(BCIP)/四唑硝基蓝(NBT)碱性磷酸酯酶显色试剂,生成的NADH与BCIP/NBT碱性磷酸酯酶显色试剂反应,在菌落上形成不溶性的深蓝色或紫蓝色物质,根据颜色深浅的变化挑取单菌落,以期得到高活力的突变株,从而降低筛选通量。平板筛选方法最为简便,但局限于提高酶活性等的突变体筛选,对于筛选改变了最适pH和反应温度等条件的突变体,可考虑采用荧光法或显色法。张航等[32]根据羰基还原酶催化可逆氧化还原反应的原理,耦合偶氮还原酶催化偶氮染料的颜色变化筛选出的突变酶CMCR对产物(S)-1-苯基-1,2-乙二醇的立体选择性达到99.9%。
定向进化技术已成为改善酶的催化性能和开发新型酶蛋白的重要策略,并被应用于改善酶的诸多特性,如热稳定性、酸碱稳定性,催化活性和效率等方面[33]。然而,羰基还原酶作为一类重要的生物催化剂,有关其分子改造的研究报道还不多,同时由于缺乏具有普适性的高通量筛选方法,在改造工程上仍存在一定的难度。表1列举了部分羰基还原酶定向进化的实例。
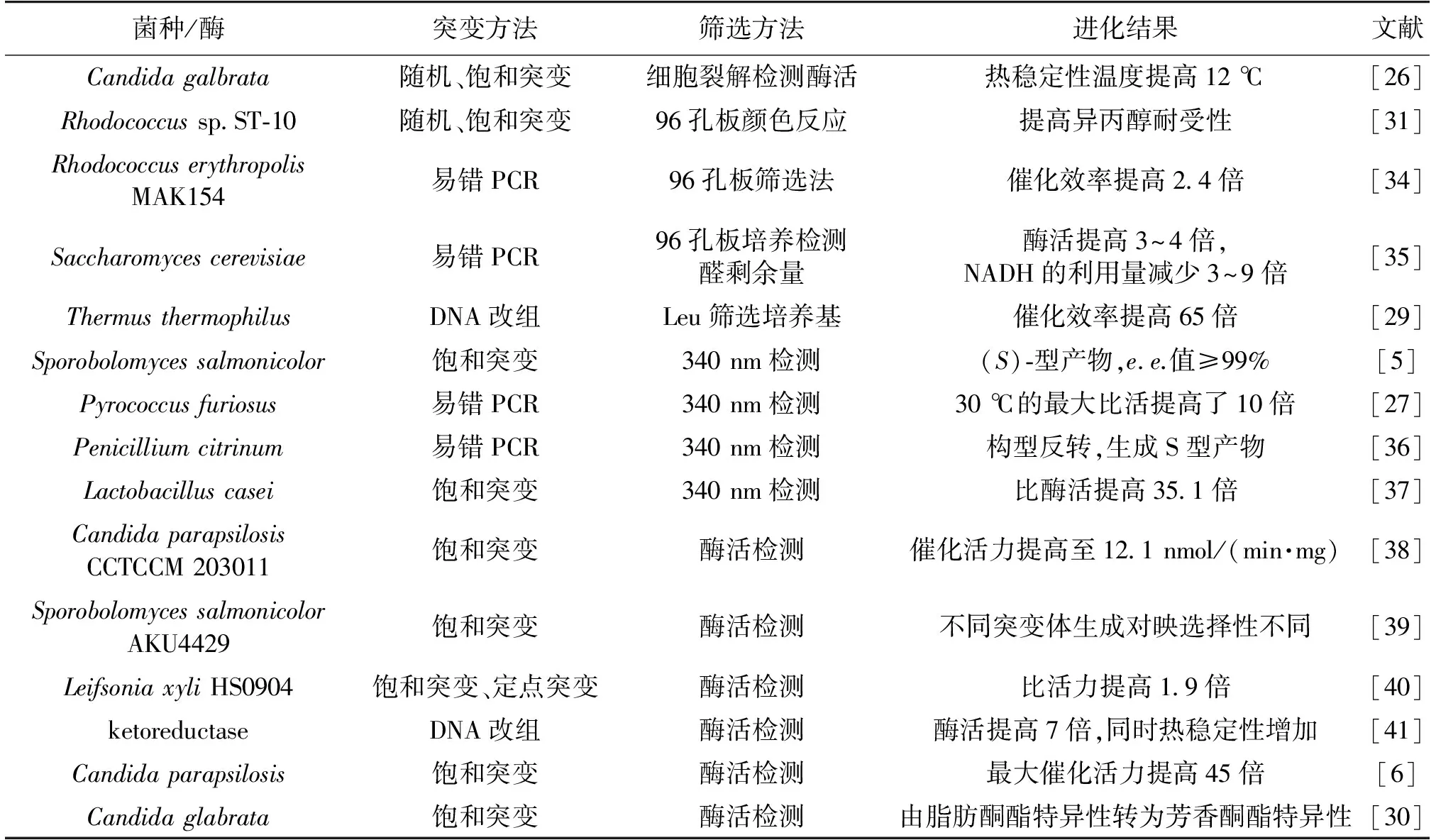
表1 羰基还原酶的部分定向进化实例
2.3 半理性设计的策略
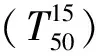
半理性设计组合方法较多,除基于蛋白质结构分析外,还有结合计算机辅助的半理性设计[43]。Zhang等[39]通过软件模拟羰基还原酶催化拆分β-氨基酮,发现L174处于底物结合位点,其大小影响底物的取向,经饱和突变得到的突变体L174Y可催化得到(R)-型产物,e.e.值为93 %;而L174W催化得到(S)-型产物,e.e.值为96 %。笔者所在课题组采用Discovery Studio 3.0软件和基于PDB数据库酶蛋白的氨基酸序列比对,以相似度最高的苜蓿中华根瘤菌(Sinorhizobiummeliloti)1021的短链脱氢酶(PDB:3TOX)和嗜热菌(Thermusthermophilus)HB8的氧化还原酶(PDB:2D1Y)的晶体结构为模板,对雷弗松氏菌(Leifsoniaxyli)HS0904羰基还原酶LXCAR进行同源建模(图4)。由三维结构分析发现,活性中心附近的Ser154和Leu194可能会影响活性位点,从而影响LXCAR的催化活力,因此对这两个位点进行饱和突变,构建饱和突变库,并从中筛选得到正突变体S154Y,其对底物3,5-双三氟甲基苯乙酮(BTAP)具有较高的催化活力,酶比活达到2.89 U/mg,是野生型的1.9倍[40]。
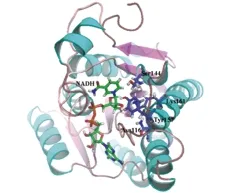
绿色标记的为辅酶NADH,蓝色球棍结构是的催化四联体(N166-S144-Y157-K161)图4 同源建模得到的LXCAR三维结构Fig.4 The homology modeled structure of LXCAR describing the NADH (green stick) and the catalytic tetrad (N116-S144-Y157-K161) (blue stick)
3 结论与展望
近年来,分子改造技术的日趋成熟,对蛋白质工程的发展产生了巨大的影响,酶基因序列、空间结构及催化方式等信息也逐渐被人们所了解。然而,许多蛋白质的结构以及结构与功能之间的关系还不是很清楚,在进行酶分子设计时具有较大的盲目性。因此,深入研究现有酶的分子结构与功能,利用分子生物学技术对具有特殊优良性状的基因进行克隆、改造,可提高酶分子设计的针对性,提高分子改造效率。
一般而言,只要突变体库足够大,通过定向进化就可以对羰基还原酶多方面的催化性能进行改造,从而产生具有优良性状的突变菌株。但是突变体库越庞大,就越需要高效简便的高通量筛选方法。构建高质量、功能丰富的小型库,可以降低突变体筛选的压力,反而有可能取得更好的进化结果。因此,建立突变库高通量筛选方法,快速从突变体库中筛选出所需的基因是该技术成功应用的关键所在。此外,微生物来源的羰基还原酶分子改造的研究大多以提高其催化效率及酶的热稳定性为主,对底物专一性的改造则相对较少。随着羰基还原酶应用领域的进一步拓展,底物分子的多样性与羰基还原酶底物专一性的矛盾将日渐突出。因此,在注重对羰基还原酶底物专一性改造的同时,也应注重筛选和研究还原酶家族中的其他种类羰基还原酶。
[1] MANO J I.Reactive carbonyl species:their production from lipid peroxides,action in environmental stress,and the detoxification mechanism[J].Plant Physiol Biochem,2012,59:90-97.
[2] ZHANG R Z,WANG L,XU Y,et al.In situ expression of (R)-carbonyl reductase rebalancing an asymmetric pathway improves stereoconversion efficiency of racemic mixture to (S)-phenyl-1,2-ethanediol inCandidaparapsilosisCCTCC M203011[J].Microb Cell Fact,2016,15:143-152.
[3] KATAOKA M,MIYAKAWA T,SHIMIZU S,et al.Enzymes useful for chiral compound synthesis:structural biology,directed evolution,and protein engineering for industrial use[J].Appl Microbiol Biotechnol,2016,100(13):5747-5757.
[4] MULLER M,KATZBERG M,BERTAU M,et al.Highly efficient and stereoselective biosynthesis of (2S,5S)-hexanediol with a dehydrogenase fromSaccharomycescerevisiae[J].Org Biomol Chem,2010,8(7):1540-1550.
[5] LI H M,YANG Y,ZHU D M,et al.Highly enantioselective mutant carbonyl reductases created via structure-based site-saturation mutagenesis[J].J Org Chem,2010,75(22):7559-7564.
[6] LODERER C,DHOKE G V,DAVARI M D,et al.Investigationof structural determinants for the substrate specificity in the zinc-dependent alcohol dehydrogenase CPCR2 fromCandidaparapsilosis[J].ChemBioChem,2015,16(10):1512-1519.
[7] OPPERMANN U,FILLING C,HULT M,et al.Short-chain dehydrogenases/reductases(SDR):the 2002 update[J].Biochemistry,2003,143(18):247-253.
[8] GANI O A,ADEKOYA O A,GIURATO L,et al.Theoretical calculations of the catalytic triad in short-chain alcohol dehydrogenases/reductases[J].Biophys J,2008,94(4):1412-1427.
[9] HUANG M M,HU H H,MA L,et al.Carbon-carbon double-bond reductases in nature[J].Drug Metab Rev,2014,46(3):362-378.
[10] LIANG P,QIN B,MU M,et al.Prelog and anti-Prelog stereoselectivity of two ketoreductases fromCandidaglabrata[J].Biotechnol Lett,2013,35:1469-1473.
[11] 冯旭东,吕波,李春.酶分子稳定性改造研究进展[J].化工学报,2016,76(1):277-284.
[12] DENARD C A,REN H Q,ZHAO H M.Improving and repurposing biocatalysts via directed evolution[J].Curr Opin Chem Biol,2015,25:55-64.
[13] GARST A D,BASSALO M C,PINES G,et al.Genome-wide mapping of mutations at single-nucleotide resolution for protein,metabolic and genome engineering[J].Nature Biothchnol,2017,35:48-55.
[14] JAKOBLINNERT A,WITTENBOER A V D,SHIVANGE A V,et al.Design of an activity and stability improved carbonyl reductase fromCandidaparapsilosis[J].J Biotechnol,2013,165:52-62.
[15] ZHANG R Z,XU Y,SUN Y,et al.Ser67Asp and His68Asp substitutions inCandidaparapsilosiscarbonyl reductase alter the coenzyme specificity and enantioselectivity of ketone reduction[J].Appl Environ Microbiol,2009,75(7):2176-2183.
[16] KRIES H,BLOMBERG R,HILVERT D,et al.De novo enzymes by computational design[J].Curr Opin Cheml Biol,2013,17(2):221-228.
[17] CHANG Y H,WANG C Z,CHIU C C,et al.Contributions of active site residues to cofactor binding and catalysis of 3α-hydroxysteroid dehydrogenase/carbonyl reductase[J].Biochim Biophys Acta,2010,1804:235-241.
[18] HEISS C,LAIVENIEKS M,ZEIKUS J G,et al.Mutation of cysteine-295 to alanine in secondary alcohol dehydrogenase fromThermoanaerobacterethanolicusaffects the enantioselectivity and substrate specificity of ketone reductions[J].Bioorg Med Chem,2001,9(7):1659-1666.
[19] LUO X,WANG Y J,SHEN W,et al.Activity improvement of aKluyveromyceslactisaldo-keto reductase KIAKR via rational design[J].J Biotechnol,2016,224:20-26.
[20] CHAO N,LI N,QI Q,et al.Characterization of the cinnamoyl-CoA reductaes (CCR) gene family inPopulustomentosareveals the enzymatic active sites and evolution of CCR[J].Planta,2017,245(1):61-75.
[21] KIPNIS Y,DELLUS-GUR E,TAWFIK D S.Trins:a method for gene modification by randomized tandem repeat insertions[J].Protein Eng Des Sel,2012,25(9):437-444.
[22] KOYAMA Y,HIDAKA M,NISHIMOTO M,et al.Directed evolution to enhance thermostability of galacto-N-biose/lacto-N-biose I phosphorylase[J].Protein Eng Des Sel,2013,26(11):755-761.
[23] COBB R E,CHAO R,ZHAO H.Directed evolution:past,present,and future[J].AIChE J,2013,59(5):1432-1440.
[24] ZHAO F J,PEI X Q,REN Z Q,et al.Rapid asymmetric reduction of ethyl 4-chloro-3-oxobutanoate using a thermostabilized mutant of ketoreductase ChKRED20[J].Appl Microbiol Biotechnol,2016,100(8):3567-3575.
[25] RAO G D,LEE J K,ZHAO H M.Directed evolution of phloroglucinol synthase PhlD with increased stability for phloroglucinol production[J].Appl Microbiol Biotechnol,2013,97(13):5861-5867.
[26] HUANG L,XU J H,YU H L.Significantly improved thermostability of a reductase CgKR1 fromCandidaglabratawith a key mutation at Asp138 for enhancing bioreduction of aromatic α-keto esters[J].J Biotechnol,2015,203:54-61.
[27] MACHIELSEN R,LEFERINK N G H,HENDRIKS A,et al.Laboratory evolution ofPyrococcusfuriosusalcohol dehydrogenase to improve the production of (2S,5S)-hexanediol at moderate temperatures[J].Extremophiles,2008,12:587-594.
[28] 金庆超,沈娜,杨郁,等.spyl的DNA改组提高普那霉素的产量[J].中国抗生素杂志,2015,40(3):178-191.
[29] SUZUKI Y,ASADA K,MIYAZAKI J,et al.Enhancement of the latent 3-isopropylmalate dehydrogenase activity of promiscuous homoisocitrate dehydrogenase by directed evolution[J].Biochem J,2010,431(3):401-410.
[30] 黄磊.光滑假丝酵母酮酯还原酶的分子改造及应用研究[D].上海:华东理工大学,2014.
[31] MAKINO Y,ITOH N.Development of an improved phenylacetaldehyde reductase mutant by an efficient selection procedure[J].Appl Microbiol Biotechnol,2014,98:4437-4443.
[32] 张航,陈曦,冯进辉,等.高通量筛选具有催化活性和立体选择性羰基还原酶[J].生物工程学报,2015,31(2):220-230.
[33] CHEN Z,ZENG A P.Protein design in systems metabolic engineering for industrial strain development[J].Biotechnol J,2013,8(5):523-533.
[34] URANO N,FUKUI S,KUMASHIRO S,et al.Directed evolution of an aminoalcohol dehydrogenase for efficient production of double chiral aminoalcohols[J].J Biosci Bioeng,2011,111(3):266-271.
[35] MOON J,LIU Z L.Engineered NADH-dependent GRE2 fromSaccharomycescerevisiaeby directed enzyme evolution enhances HMF reduction using additional cofactor NADPH[J].Enzyme Microb Technol,2012,50(2):115-120.
[36] ASAKO H,SHIMIZU M,ITOH N.Biocatalytic production of (S)-4-bromo-3-hydroxybutyrate and structurally related chemicals and their applications[J].Appl Microbiol Biotechnol,2009,84(3):397-405.
[37] 高秀珍.烯酮/烯酯还原酶和D-氨基酸脱氢酶的筛选、性质及定向进化[D].北京:中国科学院大学,2013.
[38] 宫绪敏,聂尧,徐岩,等.羰基还原酶辅酶结合域位点突变对其不对称催化性能的影响[J].催化学报,2012,33(2):330-335.
[39] ZHANG D L,CHEN X,CHI J,et al.Semi-rational engineering a carbonyl reductase for the enantioselective reduction ofβ-amino ketones[J].ACS Catal,2015,5(4):2452-2457.
[40] WANG N Q,SUN J,HUANG J.Cloning,expression,and directed evolution of carbonyl reductase fromLeifsoniaxyliHS0904 with enhanced catalytic efficiency[J].Appl Microbiol Biotechnol,2014,98:8591-8601.
[41] MA S K,GRUBER J,DAVIS C,et al.A green-by-design biocatalytic process for atorvastatin intermediate[J].Green Chem,2010,12:81-86.
[42] WIJMA H J,FLOOR R J,JANSSEN D B.Structure- and sequence-analysis inspired engineering of proteins for enhanced thermostability[J].Curr Opin Struct Biol,2013,23(4):588-594.
[43] 王晓玥,王白云,王智文,等.蛋白质定向进化的研究进展[J].生物化学与生物物理进展,2015,42(2):123-131.
[44] SUN Z T,LONSDALE R,LLIE A,et al.Catalytic asymmetric reduction of difficult-to-reduce ketones:triple-code saturation mutagenesis of an alcohol dehydrogenase[J].ACS Catal,2016,6(3):1598-1605.
(责任编辑 荀志金)
Progress in molecular modification of microbial carbonyl reductase
CEN Jiashan1,GONG Tiantian1,CHEN Weiqing2,WANG Pu1
(1. College of Pharmaceutical Science,Zhejiang University of Technology,Hangzhou 310014,China;2. College of Biological and Environmental Engineering,Zhejiang Shuren University,Hangzhou 310015,China)
Carbonyl reductase is widely used in the synthesis of chiral compounds due to its excellent stereoselectivity and catalytic activity.However,low expression level,difficult to purify intracellular enzyme,and unsatisfactory enzyme activity hammer industrial applications of native microbial carbonyl reductase.Using protein engineering techniques to modify carbonyl reductaseinvitrocould substantially improve its ability for asymmetric synthesis of chiral compounds.In this paper,we reviewed different molecular modification strategies,advances and achievements in molecular modificationinvitroof carbonyl reductase.The general rules in carbonyl reductase molecular modification process are also summarized.Future development in this field is also discussed.This review can provide useful reference for the study on microbial carbonyl reductase molecular modificationinvitro.
carbonyl reductase; catalysis mechanism; directed evolution; rational design
10.3969/j.issn.1672-3678.2017.03.007
2017-02-27
国家自然科学基金(21676250);浙江省自然科学基金(LY16B060010)
岑佳善(1991—),女,浙江慈溪人,研究方向:生物催化与生物转化;王 普(联系人),教授,博士生导师,E-mail:wangpu@zjut.edu.cn
Q78
A
1672-3678(2017)03-0036-06
——紫 苏

